| |
| Med Sci (Paris). 2010 May; 26(5): 497–503. Published online 2010 May 15. doi: 10.1051/medsci/2010265497.Évolution de l’empreinte parentale chez les mammifères Quelle ménagerie ! Charlotte Proudhon* and Déborah Bourc’his* UMR 3215/Inserm U934, Unité de génétique et biologie du développement, Institut Curie, 26 rue d’Ulm, 75248 Paris Cedex 05, France |
La majorité des espèces animales peuvent se reproduire sans partenaire sexuel, par activation d’un œuf non fécondé ou parthénogenèse. Les abeilles et les salamandres alternent naturellement entre reproduction sexuée et parthénogenèse au cours de leur vie reproductive. La parthénogenèse peut aussi être déclenchée par une disponibilité limitée en partenaires sexuels : plusieurs cas de naissances parthénogénétiques ont été documentés chez des femelles requins et dragons de Komodo1 maintenues en captivité prolongée [
1]. Chez les mammifères en revanche, la reproduction monoparentale est impossible. Des expériences de transfert nucléaire chez la souris ont démontré dans les années 1980 que le développement des mammifères requiert obligatoirement un génome maternel et un génome paternel [
2,
3]. Nous avons célébré en 2009 les 25 ans de cette preuve formelle de la spécificité reproductive des mammifères liée à l’existence de l’empreinte parentale. Les mammifères ne sont cependant pas tous égaux face à l’empreinte parentale. Des contraintes physiologiques et génomiques ont ainsi contribué à l’émergence et à l’évolution de l’empreinte au cours de la spéciation des différents groupes de mammifères. |
Définition biologique, génétique et épigénétique de l’empreinte parentale La restriction à un mode de reproduction sexué chez les mammifères résulte d’une non-équivalence fonctionnelle des génomes parentaux, dictée par l’empreinte parentale ou empreinte génomique. L’empreinte parentale est un phénomène épigénétique qui affecte une centaine de gènes connus chez la souris et l’homme, leur imposant une expression monoallélique et dépendante de l’origine parentale. Le déterminisme parental résulte d’un marquage par méthylation de l’ADN, différentiellement apposé sur les allèles parentaux au moment de leur passage par la gamétogenèse. La méthylation gamétique est spécifiquement ciblée sur des régions régulatrices appelées ICR (imprinting control regions ou centres d’empreinte) qui contrôlent en cis l’expression d’un ou plusieurs gènes. Les ICR maternelles sont méthylées spécifiquement dans l’ovocyte, tandis que les paternelles le sont dans le spermatozoïde. Ces marques d’empreinte ont la particularité d’être maintenues après la fécondation et interprétées par la répression d’un des deux allèles parentaux. Une question essentielle dans le champ de la biologie de l’empreinte est de comprendre l’identité unique des ICR. En particulier, quels sont les facteurs trans et cis responsables de l’établissement de l’empreinte et du maintien de la mémoire de l’origine parentale ? La mise en place des marques de méthylation sur les centres d’empreinte est régulée lors de la gamétogenèse par l’ADN méthyltransférase de novo Dnmt3A et son cofacteur Dnmt3L [
4,
5] (Figure 1). Les marques d’empreinte sont conservées au cours de la vie par l’action de Dnmt1, l’ADN méthyltransférase de maintenance, qui reproduit les profils de méthylation au moment de la réplication [
6]. Au cours du développement pré-implantatoire (de la fécondation à l’implantation dans l’utérus), les gènes soumis à empreinte doivent résister à des remaniements drastiques des profils de méthylation [
7]. Les facteurs PGC7/Stella et Zfp57 (KRAB zinc finger protein) jouent un rôle de protection contre la déméthylation qui suit la fécondation [
8,
9]. Au moment de l’implantation, les allèles non méthylés des gènes soumis à empreinte doivent cette fois résister à la phase de reméthylation que subit l’embryon mais les facteurs responsables ne sont pas connus. Des caractéristiques nucléotidiques et épigénétiques particulières sont impliquées dans le recrutement des facteurs évoqués ci-dessus. Les gènes soumis à empreinte sont notamment préférentiellement associés à des régions riches en dinucléotides CpG, en éléments rétrotransposables de type LINE (long interspersed elements) et en motifs de liaison pour CTCF (CCCTC-binding factor)2 [
10]. Cependant, ces caractéristiques n’étant ni obligatoires ni exclusives, la méthylation parentale des ICR est plus vraisemblablement corrélée à un état chromatinien particulier. Il a ainsi été récemment montré que la mise en place de la méthylation sur les ICR maternelles dans l’ovocyte requiert le retrait des marques de méthylation sur la lysine 4 de l’histone H3 (H3K4me) par l’histone déméthylase Lsd2 [
11], en relation avec le caractère répulsif de ces marques envers Dnmt3L [
12]. Des évènements de transcription qui traversent les ICR maternelles pourraient également participer à ce remodelage chromatinien [
13]. En conclusion, le statut de l’empreinte d’un gène résulte de sa proximité avec un centre d’empreinte dont l’identité est dictée par une combinaison complexe de motifs génomiques et épigénétiques influençant l’établissement et le maintien des profils de méthylation parentaux.
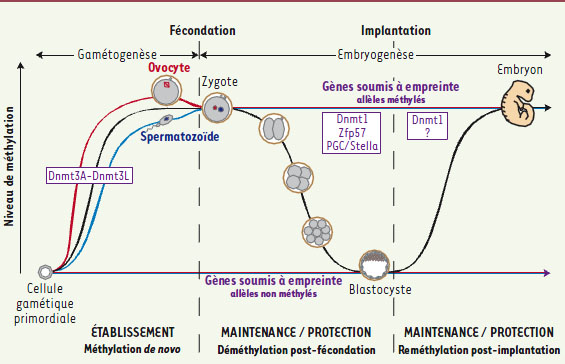
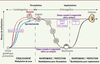 | Figure 1.
Établissement, maintenance et protection des marques de méthylation associées à l’empreinte parentale. Les courbes rouges et bleues représentent respectivement les allèles maternels et paternels des gènes soumis à empreinte et en noir le reste du génome. L’établissement de l’empreinte par apposition de marques de méthylation spécifiques du sexe se fait au cours de la gamétogenèse. La maintenance de ces marques est assurée tout au long de la vie par Dnmt1. La protection de ces marques, par des facteurs identifiés ou non (?), est essentielle face aux vagues de déméthylation post-fécondation et de reméthylation post-implantatoire que subit le génome de l’embryon précoce. |
|
Évolution de l’empreinte parentalechez les mammifères : pas de placenta… pas d’empreinte ! Le mécanisme de répression d’un des deux allèles parentaux soulève la question de comment et surtout pourquoi l’empreinte parentale a évolué, sachant qu’elle nie l’avantage de la diploïdie qui peut compenser l’apparition d’une mutation délétère. La distribution phylogénétique de l’empreinte fournit des arguments fonctionnels à cette ambiguïté. Hétérogénéité de l’empreinte : ornithorynque, marsupiaux, souris et homme Les premiers signes d’empreinte parentale seraient apparus chez les mammifères il y a environ 150 millions d’années, chez l’ancêtre commun des thériens, après la divergence des monotrèmes (Figure 2). La branche des monotrèmes, dont le représentant actuel le plus connu est l’ornithorynque, est apparemment dépourvue de gènes à expression monoparentale. Les thériens comprennent les marsupiaux, dont le kangourou et l’opossum, et les euthériens, comme la souris et l’homme. Tous sont dotés de locus soumis à empreinte. Cependant, le petit nombre de locus observés chez les marsupiaux suggère que l’empreinte a été préférentiellement consolidée chez les euthériens, en fonction des besoins et de l’adaptation des espèces [
14].
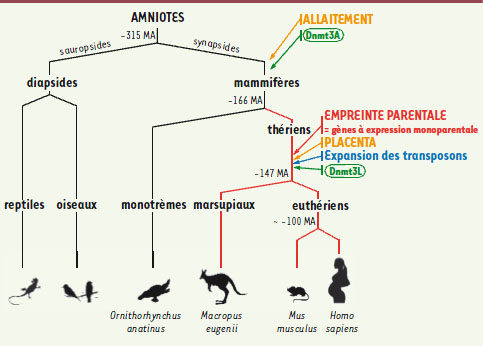
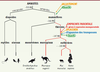 | Figure 2.
Apparition de l’empreinte parentale au cours de l’évolution. Trois groupes de mammifères existent : les monotrèmes et les thériens, composés eux-mêmes des marsupiaux et des euthériens. Tous ont recours à l’allaitement, tandis que le placenta est l’apanage des thériens. L’empreinte parentale est apparue avant la divergence des thériens, coïncidant avec l’acquisition du placenta, de Dnmt3L et de l’expansion des transposons. MA = millions d’années. |
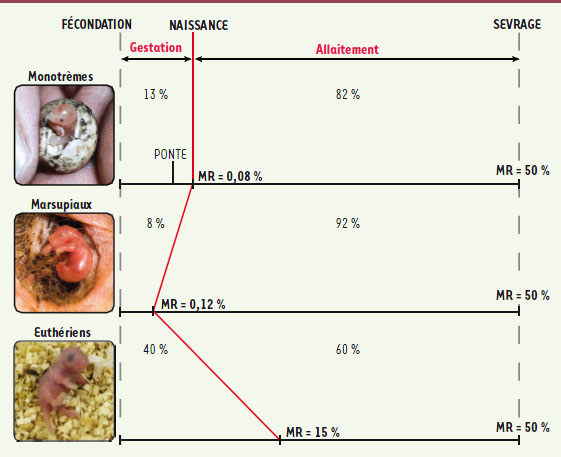
Les trois groupes de mammifères utilisent des modes de reproduction différents et il est possible de corréler l’évolution de l’empreinte parentale à des spécificités physiologiques. L’allaitement, associé à la présence de mamelles chez les femelles, est le dénominateur commun à tous les mammifères. Le développement in utero, ou viviparité, est une autre caractéristique des mammifères, mais n’est cependant pas universelle. Ainsi, les femelles monotrèmes allaitent leurs petits, mais sont ovipares. À la différence des oiseaux et des reptiles, la ponte de l’œuf est ici précédée d’une période relativement longue d’incubation dans le tractus maternel. Les marsupiaux, chez qui l’on retrouve une empreinte moins étendue que chez les euthériens, possèdent un placenta rudimentaire associé à un développement in utero très court. Chez le kangourou par exemple, la gestation dure 26 jours et s’achève par la migration du fœtus dans la poche, où la lactation prend le relais et peut s’étendre jusqu’à 10 mois. Par contraste, le groupe des euthériens arbore un placenta très développé, associé à une période de gestation longue et à un développement beaucoup plus abouti que chez les autres mammifères. Si l’on compare la masse relative des nouveau-nés par rapport à leur mère au moment de la naissance, celle-ci est très inférieure chez les monotrèmes (0,08 %) et les marsupiaux (0,12 %) à ce qu’elle est chez les euthériens (15 %). Ces inégalités sont cependant compensées au moment du sevrage : la masse relative du nouveau-né y est équivalente dans les 3 groupes mammifères, ce qui illustre le rôle prédominant de la lactation pour le transfert de nutriments chez les monotrèmes et les marsupiaux (Figure 3) [
15]. En conclusion, l’émergence de l’empreinte parentale chez les mammifères serait liée à l’apparition du placenta, et son amplification à la sophistication de cet organe.
 | Figure 3.
Différences physiologiques dans la reproduction des mammifères : proportion relative entre développement
in utero
et lactation. La lactation contribue de façon majeure au développement des petits chez les monotrèmes (82 %) et les marsupiaux (92 %) par rapport aux euthériens (60 %). Ces derniers ont en revanche recours à une phase intra-utérine nettement plus importante. MR = masse relative des nouveau-nés par rapport à celle de la mère. |
Le « conflit des génomes parentaux » D’un point de vue fonctionnel, le processus de lactation et les échanges placentaires génèrent chez les mammifères une asymétrie dans le transfert des ressources parentales à la progéniture, au profit d’une implication très largement maternelle. La théorie du « conflit des génomes parentaux », émise par Tom Moore et David Haig en 1991, tente d’expliquer d’un point de vue évolutif l’apparition de l’empreinte au sein du règne mammifère, en tenant compte de cet investissement parental inégal [
16]. Cette théorie se base sur le profil d’expression des premiers gènes soumis à empreinte découverts au début des années 1990, le gène de croissance Igf2 (insulin growth factor 2) à expression paternelle et son régulateur négatif Igf2r à expression maternelle [
17,
18]. Par extrapolation, il a été postulé que les gènes à expression paternelle réguleraient positivement la croissance et l’apport de nutriments au fœtus, l’intérêt paternel étant de favoriser sa progéniture et de transmettre ses gènes. Par opposition, l’intérêt de la mère est de réduire le transfert de nutriments pour répartir ses ressources sur plusieurs portées, et les gènes à expression maternelle réguleraient négativement la croissance et les transferts maternofœtaux. Les gènes soumis à empreinte actuellement décrits (une centaine) ont des fonctions souvent divergentes de la simple régulation de croissance ou du transfert nutritif. Plusieurs sont notamment exprimés dans le cerveau avec des fonctions essentielles au comportement reproductif des mammifères [
19]. Une théorie actuelle moins « belliqueuse » prône ainsi un rôle coadaptateur de l’empreinte parentale, qui agirait pour la coordination de gènes impliqués dans les interactions entre la mère et ses petits, avant et après la naissance [
20]. Le gène Peg3 (paternally expressed gene 3), par exemple, améliore les capacités de reproduction par un double rôle. Son expression chez l’adulte contrôle la production de lait chez la mère, tandis qu’il influence la croissance placentaire et post-natale et le réflexe de succion chez le petit. Cependant, l’absence d’empreinte chez les monotrèmes suggère que le placenta reste la force évolutive dominante et/ou que le génome des monotrèmes n’est pas doté des régulateurs génétiques et épigénétiques nécessaires à l’évolution de l’empreinte. |
Coévolution de l’empreinte parentale avec l’expansion des transposons L’analyse génomique des espèces dotées et dépourvues d’empreinte a fourni des informations clefs quant à l’évolution du processus d’empreinte. Une centaine de gènes soumis à empreinte sont connus chez les euthériens, et seulement 5 sont répertoriés chez les marsupiaux. Seulement 2 de ces gènes sont de plus associés à des ICR porteuses de marques de méthylation gamétique. Ceci suggère un déploiement spécifique des ICR chez les euthériens ou, alternativement, l’utilisation par les marsupiaux d’un mode de régulation indépendant de la méthylation. Le consortium SAVOIR (sequence analysis of vertebrate orthologous imprinted regions) a entrepris depuis plusieurs années l’analyse comparative systématique chez le poulet, le wallaby/kangourou et l’ornithorynque des régions orthologues des locus soumis à empreinte connus [
21]. La publication récente de la séquence du génome de l’ornithorynque a facilité l’identification des régions synténiques et des éléments fonctionnels importants, en relation avec l’évolution des génomes [
22]. L’organisation des locus soumis à empreinte est généralement conservée chez tous les mammifères, avec une expansion des régions chez les euthériens, due à des phénomènes de duplication et de transposition. Cette observation, liée au fait que le génome de l’ornithorynque est relativement pauvre en éléments transposables, suggère une évolution coïncidente de l’empreinte avec la nécessité de contrôler une masse critique d’éléments génomiques parasites chez les mammifères supérieurs [
23]. Le degré de conservation des facteurs impliqués dans la mise en place développementale des marques de méthylation est aussi instructif. Dnmt3A est présent chez tous les mammifères, tandis que Dnmt3L est spécifique des thériens [
24]. Or Dnmt3L est aussi requis pour le contrôle par méthylation des éléments transposables dans les gamètes mâles chez la souris [
25]. Le placenta, l’expansion génomique des transposons, Dnmt3L et l’empreinte parentale ont donc émergé de façon concomitante au cours de l’évolution des mammifères (Figure 2), sans que l’on puisse désigner les relations de cause à effet entre ces évènements. Le scénario le plus accepté est : (1) l’acquisition du gène Dnmt3L comme élément de défense gamétique contre les transposons colonisant le génome des thériens, et (2) la cooptation de ce facteur pour la régulation de l’expression monoparentale d’une poignée de gènes, en relation avec la fonction d’un organe placentaire complexe. Le plus grand nombre de gènes soumis à empreinte dans le placenta murin par rapport à l’homme suggère que la monogamie et la production moyenne d’un seul fœtus par gestation auraient induit une pression de sélection moindre dans l’espèce humaine [
26]. |
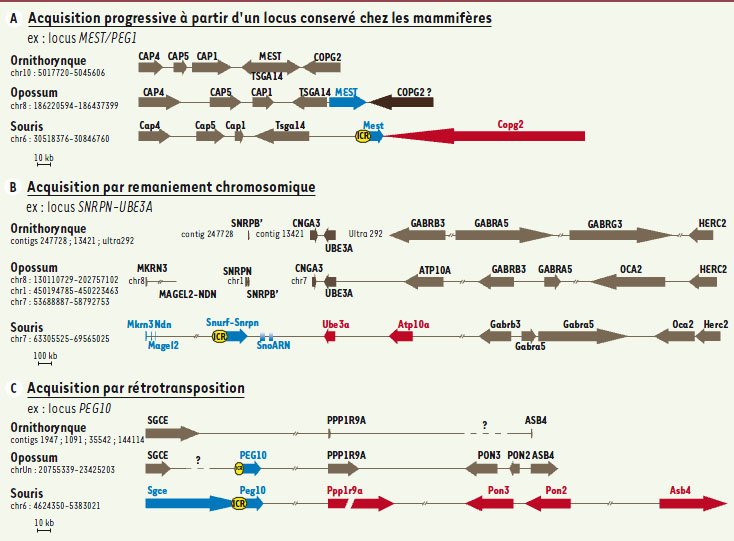
Modus operandi de l’acquisition de nouveaux gènes soumis à empreinte L’empreinte parentale est donc, comme nous l’avons décrit ci-dessus, hétérogène en quantité (nombre de gènes) mais aussi en qualité (mode de régulation) selon les espèces. Elle représente un phénomène en marche, qui évolue par vagues de sélection au cours de la spéciation des mammifères. On peut définir deux modes de recrutement de nouveaux gènes soumis à empreinte. Les gènes de la première catégorie existent chez les monotrèmes, mais n’ont été sélectionnés pour une expression mono-allélique que chez les thériens. L’ICR fonctionnelle riche en motifs CpG et portant les marques de méthylation gamétique n’est souvent acquise que chez les euthériens, comme dans le cas du gène PEG1 (Figure 4A). Des réarrangements chromosomiques ont aussi participé à la construction de larges locus soumis à empreinte, réunissant plusieurs blocs chromosomiques autour d’une même ICR apparue au même moment. L’exemple le plus connu est celui du locus associé aux syndromes de Prader-Willi et d’Angelman3 (PWS-AS), qui regroupe les gènes SNRPN (small nuclear ribonucleoprotein N) et UBE3A (ubiquitin protein ligase E3A) chez les euthériens, alors qu’ils sont situés sur des chromosomes différents chez les autres mammifères (Figure 4B) [
27].
 | Figure 4.
Mécanismes évolutifs d’acquisition d’un locus soumis à empreinte, illustration par analyse comparative chez les trois groupes de mammifères. Rouge : gène à expression maternelle, bleu : gène à expression paternelle, gris : gène à expression biallélique, jaune : ICR. A. Acquisition progressive des caractéristiques au sein d’un locus préexistant chez l’ancêtre commun de tous les mammifères. Exemple du gène PEG1, qui existe chez les monotrèmes, mais n’est exprimé paternellement que chez les thériens. L’ICR n’est présente que chez les euthériens. B. Acquisition de l’empreinte par réarrangement chromosomique. Exemple du locus UBE3A-SNRPN. Chez la souris ce locus est situé sur le chromosome 7 et correspond à des blocs séparés sur les chromosomes 1, 7 et 8 chez l’opossum. C. Acquisition par rétrotransposition. Exemple du locus PEG10. Le rétrotransposon nouvellement inséré chez les marsupiaux est devenu le gène Peg10. L’ICR associée au promoteur du gène est présente chez l’opossum suggérant son apparition au moment de l’insertion du gène. La régulation monoparentale s’est ensuite étendue aux gènes voisins chez les euthériens. |
La seconde catégorie correspond à des gènes qui sont apparus spécifiquement chez les thériens et ont immédiatement acquis un statut soumis à empreinte. Plusieurs de ces gènes dérivent de rétrotransposons, dont la séquence a été domestiquée et utilisée pour développer de nouvelles fonctions cruciales pour la fonction placentaire. C’est notamment le cas des gènes Peg10 (paternally expressed gene 10) et Rtl1, tous deux dérivés de l’insertion d’un rétrotransposon sushi-ichi4 de type LTR (long terminal repeat) et indispensables au développement du placenta murin (Figure 4C) [
28,
29]. On voit encore ici la coïncidence évolutive entre l’empreinte parentale, l’organe placentaire et la défense de l’organisme contre les éléments transposables qui utilise la méthylation de l’ADN pour réprimer la mobilisation de séquences parasites endogènes et maintenir l’intégrité génomique. Dans cette catégorie de « néogènes soumis à empreinte », on trouve également un nombre important de gènes qui ont détourné la machinerie de rétrotransposition pour se mouvoir et se dupliquer à une autre position génomique. Ce mécanisme est notamment à l’origine de l’acquisition des gènes Nap1l5, U2af1-rs1, Inpp5f-v2 et Mcts2, qui, pour une raison encore inconnue, proviennent systématiquement d’un orthologue originaire du chromosome X [
30]. Dans tous les cas de gènes dérivés directement ou indirectement de rétrotransposons, l’apparition d’une ICR fonctionnelle est simultanée ou postérieure à l’événement d’insertion. D’une manière générale, on observe aussi que toute nouvelle ICR tend à étendre son influence dans la région (Figure 4), induisant en cis l’expression mono-allélique d’un nombre croissant de gènes chez les euthériens, comme un recrutement fortuit à la cause de l’empreinte parentale. |
L’empreinte parentale reflète un mécanisme d’expression singulier, appliqué à une centaine de gènes mammifères et dépendant de déterminants génétiques et épigénétiques. L’empreinte est phylogénétiquement liée à la viviparité et s’est répandue avec la complexité de la placentation. Les gènes soumis à empreinte sont généralement exprimés au cours du développement embryonnaire chez les mammifères placentaires, en accord avec une physiologie reproductive centrée sur la gestation in utero. Notre vision souffre cependant de plusieurs biais qui influencent notre interprétation évolutive de ce phénomène : (1) la majorité des études sont réalisées chez la souris et l’Homme, (2) l’ornithorynque est une espèce protégée et seuls des tissus adultes sont disponibles pour étude, (3) l’allaitement joue un rôle majeur dans l’asymétrie de transfert des ressources parentales chez les marsupiaux et les monotrèmes. Il est concevable de proposer qu’une empreinte associée à la lactation puisse exister chez ces espèces. Les organes liés à la fonction de l’allaitement, comme la glande mammaire et l’hypophyse, devraient être étudiés en priorité chez les marsupiaux et les monotrèmes pour l’identification de gènes à expression monoparentale. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Footnotes |
1. Watts PC, Buley KR, Sanderson S, et al. Parthenogenesis in Komodo dragons. Nature 2006; 444 : 1021–2. 2. Barton SC, Surani MA, Norris ML. Role of paternal and maternal genomes in mouse development. Nature 1984; 311 : 374–6. 3. McGrath J, Solter D. Completion of mouse embryogenesis requires both the maternal and paternal genomes. Cell 1984; 37 : 179–83. 4. Bourc’his D, Xu GL, Lin CS, et al. Dnmt3L and the establishment of maternal genomic imprints. Science 2001; 294 : 2536–9. 5. Kaneda M, Okano M, Hata K, et al. Essential role for de novo DNA methyltransferase Dnmt3a in paternal and maternal imprinting. Nature 2004; 429 : 900–3. 6. Li E, Bestor TH, Jaenisch R. Targeted mutation of the DNA methyltransferase gene results in embryonic lethality. Cell 1992; 69 : 915–26. 7. Reik W, Dean W, Walter J. Epigenetic reprogramming in mammalian development. Science 2001; 293 : 1089–93. 8. Nakamura T, Arai Y, Umehara H, et al. PGC7/Stella protects against DNA demethylation in early embryogenesis. Nat Cell Biol 2007; 9 : 64–71. 9. Li X, Ito M, Zhou F, et al. A maternal-zygotic effect gene, Zfp57, maintains both maternal and paternal imprints. Dev Cell 2008; 15 : 547–57. 10. Luedi PP, Hartemink AJ, Jirtle RL. Genome-wide prediction of imprinted murine genes. Genome Res 2005; 15 : 875–84. 11. Ciccone DN, Su H, Hevi S, et al. KDM1B is a histone H3K4 demethylase required to establish maternal genomic imprints. Nature 2009; 461 : 415–8. 12. Ooi SK, Qiu C, Bernstein E, et al. DNMT3L connects unmethylated lysine 4 of histone H3 to de novo methylation of DNA. Nature 2007; 448 : 714–7. 13. Chotalia M, Smallwood SA, Ruf N, et al. Transcription is required for establishment of germline methylation marks at imprinted genes. Genes Dev 2009; 23 : 105–17. 14. Edwards CA, Rens W, Clarke O, et al. The evolution of imprinting: chromosomal mapping of orthologues of mammalian imprinted domains in monotreme and marsupial mammals. BMC Evol Biol 2007; 7 : 157. 15. Renfree MB, Hore TA, Shaw G, et al. Evolution of genomic imprinting: insights from marsupials and monotremes. Annu Rev Genomics Hum Genet 2009; 10 : 241–62. 16. Moore T, Haig D. Genomic imprinting in mammalian development: a parental tug-of-war. Trends Genet 1991; 7 : 45–9. 17. Barlow DP, Stöger R, Herrmann BG, et al. The mouse insulin-like growth factor type-2 receptor is imprinted and closely linked to the Tme locus. Nature 1991; 349 : 84–7. 18. DeChiara TM, Robertson EJ, Efstratiadis A. Parental imprinting of the mouse insulin-like growth factor II gene. Cell 1991; 64 : 849–59. 19. Wang X, Sun Q, McGrath SD, et al. Transcriptome-wide identification of novel imprinted genes in neonatal mouse brain. PLoS One 2008; 3 : e3839. 20. Curley JP, Barton S, Surani A, Keverne EB. Coadaptation in mother and infant regulated by a paternally expressed imprinted gene. Proc Biol Sci 2004; 271 : 1303–9. 21. Smits G, Mungall AJ, Griffiths-Jones S, et al. Conservation of the H19 noncoding RNA and H19-IGF2 imprinting mechanism in therians. Nat Genet 2008; 40 : 971–6. 22. Warren WC, Hillier LW, Marshall Graves JA, et al. Genome analysis of the platypus reveals unique signatures of evolution. Nature 2008; 453 : 175–83. 23. Pask AJ, Papenfuss AT, Ager EI, et al. Analysis of the platypus genome suggests a transposon origin for mammalian imprinting. Genome Biol 2009; 10 : R1. 24. Yokomine T, Hata K, Tsudzuki M, Sasaki H. Evolution of the vertebrate DNMT3 gene family: a possible link between existence of DNMT3L and genomic imprinting. Cytogenet Genome Res 2006; 113 : 75–80. 25. Bourc’his D, Bestor TH. Meiotic catastrophe and retrotransposon reactivation in male germ cells lacking Dnmt3L. Nature 2004; 431 : 96–9. 26. Monk D, Arnaud P, Apostolidou S, et al. Limited evolutionary conservation of imprinting in the human placenta. Proc Natl Acad Sci USA 2006; 103 : 6623–8. 27. Rapkins RW, Hore T, Smithwick M, et al. Recent assembly of an imprinted domain from non-imprinted components. PLoS Genet 2006; 2 : e182. 28. Suzuki S, Ono R, Narita T, et al. Retrotransposon silencing by DNA methylation can drive mammalian genomic imprinting. PLoS Genet 2007; 3 : e55. 29. Sekita Y, Wagatsuma H, Nakamura K, et al. Role of retrotransposon-derived imprinted gene, Rtl1, in the feto-maternal interface of mouse placenta. Nat Genet 2008; 40 : 243–8. 30. Wood AJ, Roberts RG, Monk D, et al. A screen for retrotransposed imprinted genes reveals an association between X chromosome homology and maternal germ-line methylation. PLoS Genet 2007; 3 : e20. |