| |
| Med Sci (Paris). 2010 May; 26(5): 541–543. Published online 2010 May 15. doi: 10.1051/medsci/2010265541.Chroniques génomiques À la recherche de l’héritabilité perdue Bertrand Jordan* Marseille-Nice Génopole, case 901, Parc scientifique de Luminy, 13288 Marseille Cedex 9, France MeSH keywords: Dosage génique, Fréquence d'allèle, Maladies génétiques congénitales, épidémiologie, génétique, Hétérogénéité génétique, Prédisposition génétique à une maladie, Variation génétique, Étude d'association pangénomique, Humains, Risque |
Les GWAS, un investissement qui se révèle peu productif À côté des affections « mendéliennes » dont l’élucidation a marqué les débuts de la nouvelle génétique au cours des années 1980 et 1990, il existe de nombreuses maladies souvent fréquentes qui comportent une composante génétique indiscutable mais complexe, impliquant de nombreux gènes. Le but principal des travaux portant sur ces affections est naturellement d’identifier les gènes (ou au moins les régions d’ADN) dont les variants sont responsables de ces prédispositions : cela devrait faciliter la compréhension des mécanismes pathogéniques à l’œuvre, autoriser le dépistage des personnes à risque, et, à terme, permettre la découverte de nouvelles thérapies ciblées et efficaces. À cet égard, la possibilité ouverte depuis quelques années de pratiquer des études d’association génétique à grande échelle (appelées GWAS pour Genome Wide Association Studies), examinant des centaines de milliers de snip1 chez des milliers d’individus a suscité de grands espoirs. On pensait qu’une bonne partie de l’influence génétique correspondait à des variants relativement fréquents dans le génome, selon l’hypothèse common disease/common variant [
1], et que la conjonction de quelques variants fréquents chez le même individu serait responsable de sa susceptibilité à une affection donnée. Il a rapidement fallu déchanter : comme nous l’avons déjà indiqué ici même [
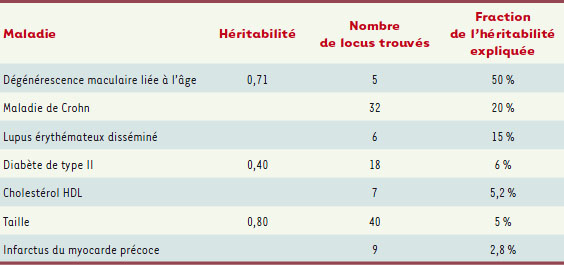
2], des variants communs sont bien identifiés de manière reproductible, mais ils ne rendent compte que d’une très faible partie de l’héritabilité2,. Les chiffres rapportés il y a un an [2] n’ont fait que se confirmer au fil de plus de quatre cents de ces études GWAS lourdes et coûteuses, qui mobilisent à chaque fois de nombreux laboratoires, des dizaines de chercheurs et des millions de dollars. Le Tableau I montre la gravité du problème : on ne connaît à l’heure actuelle que trois maladies pour lesquelles les locus identifiés rendent compte de 10 % ou plus de l’héritabilité. Pour le diabète de type II, ou pour un caractère comme la taille à l’âge adulte, les dizaines de locus désignés par de multiples études GWAS ne sont responsables que de 5 à 6 % d’une héritabilité pourtant très forte (0,40 pour le diabète, 0,80 pour la taille) [
3]…
 | Tableau I.
Les résultats décevants des GWAS (adapté de [ 3], en ajoutant des valeurs de la littérature pour l’héritabilité totale de quelques maladies) |
|
Les CNV, un nouvel espoir ? C’est en ce sens que l’on parle d’héritabilité perdue, ou manquante : 95 % de la vulnérabilité héréditaire au diabète de type II sont liés à des éléments encore non identifiés dans notre ADN. Éléments, il va sans le dire, qui sont activement recherchés aujourd’hui. Pour l’essentiel, les GWAS pratiquées jusqu’à aujourd’hui s’intéressaient aux Snip, variations ponctuelles dans l’ADN. Notons que, par nécessité, les puces à ADN employées sont ciblées sur les Snip fréquents, ceux pour lesquels l’allèle mineur a une fréquence d’au moins 5 %. Si l’on voulait interroger tous les Snip possibles dans notre génome, ce sont six milliards de points qu’il faudrait examiner - c’est-à-dire la séquence complète… C’est hors de portée des puces, mais ce sera bientôt accessible grâce aux progrès fulgurants des techniques de séquençage… [
4]. On pouvait cependant encore espérer sauver l’hypothèse common disease/common variant, grâce à la découverte relativement récente des CNV (copy number variants), duplications ou au contraire délétions de petites régions dans notre génome [
5]. Ces zones dupliquées, tripliquées ou au contraire délétées, au nombre de plusieurs milliers et se retrouvant chez de nombreux individus, allaient peut-être rendre compte d’une bonne partie de l’héritabilité manquante. Les fabricants de puces s’étant empressés d’adapter leurs outils afin qu’ils puissent détecter les dix mille CNV les plus fréquents, de nouvelles études GWAS furent lancées. Les résultats d’un travail monumental signé de plus de deux cents auteurs viennent d’être publiés [
6]. L’article, qui émane du Wellcome Trust Case Control Consortium, examine 19 000 personnes (3 000 individus témoins et 2 000 pour chacune de huit affections majeures3). Les résultats sont « surprenants et décevants », comme l’indique l’auteur senior, Peter Donnelly : cette entreprise herculéenne ne révèle que trois associations confirmées entre locus CNV et maladie - et toutes trois étaient déjà connues… |
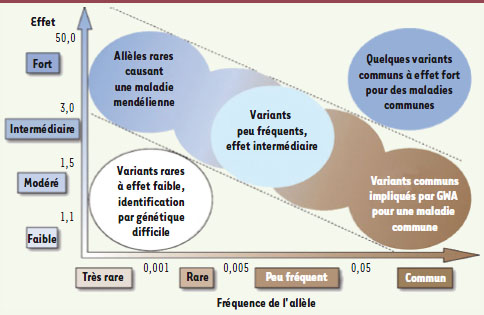
La chasse aux variants rares… et significatifs ! Il ne nous reste donc que les variants rares (ponctuels ou non) pour expliquer ces fortes susceptibilités génétiques. Rappelons que nous ne sommes plus ici dans le cas des affections mendéliennes ou monogéniques, et que les déterminants génétiques des maladies fréquentes sont à coup sûr nombreux, chacun d’eux exerçant une influence modérée (la Figure 1 propose une vision synthétique du paysage des variants à effet pathogène). Pour fixer les idées, la vulnérabilité génétique pour le diabète de type II pourrait être due à une vingtaine d’allèles peu fréquents associés chacun à un risque relatif de 2 ou 3 [3]. Mais la découverte de chacun de ces locus sera difficile : leur effet individuel est trop faible pour permettre de les « pister » par des études familiales - sinon ils auraient déjà été identifiés ! Le séquençage, contrairement aux apparences, n’offre pas une réponse immédiate, même en faisant abstraction des problèmes de coût. N’oublions pas que deux personnes diffèrent en plusieurs millions de points au niveau de leur ADN : il ne sera donc pas trivial de déterminer lesquelles de ces différences sont susceptibles de contribuer à un risque génétique. Pour avancer, il faudra non seulement beaucoup de séquences, mais aussi de grands progrès dans les méthodes d’interprétation et dans la prédiction des conséquences possibles d’une altération donnée dans l’ADN - le fameux passage du génotype au phénotype…
 | Figure 1.
Les différents types de déterminants génétiques, classés selon la fréquence de l’allèle pathogène (en abscisse) et le risque relatif conféré par cet allèle (d’après [ 3] © Nature). |
|
L’affaire se complique encore… Un autre article récent [
7] ajoute encore à la complexité de la question tout en ouvrant, peut-être, la voie vers une solution. Il s’agit d’un travail fondé sur des simulations assez sophistiquées et qui - en simplifiant - indique que des variants rares ayant un effet notable peuvent être, par hasard, « concentrés » chez des personnes qui ont en commun un certain variant fréquent, à cause de la structure de leur généalogie. Les auteurs montrent que ce type d’« association synthétique » doit en fait être très courant : en procédant à une étude GWAS sur une telle population, on a alors l’impression de découvrir un variant fréquent conférant un risque faible à une fraction notable de la population, alors qu’en fait seule une petite partie de cette fraction possède un variant rare conférant un risque élevé… Si ces hypothèses et ces calculs (rapportés dans un article assez ardu mais dont les conclusions sont bien résumées dans un commentaire en ligne de Nature [
8]) sont corrects, même les variants fréquents déjà identifiés ne sont peut-être pas les vrais responsables de l’effet (modeste) observé sur la vulnérabilité. D’un autre côté, ils indiquent la présence de variants rares présents dans leur voisinage élargi, variants rares dont la découverte serait une avancée notable, et peuvent donc servir de guide pour une recherche par séquençage. |
Une toute-puissance… toute relative Force est de reconnaître que, malgré les extraordinaires outils dont elle dispose aujourd’hui, l’approche purement génétique se heurte à de grandes difficultés. Elle est bien loin l’époque des premiers succès de la « génétique inverse » [
9], où, en un raccourci fulgurant, l’étude génétique de quelques familles à l’aide de quelques centaines de marqueurs microsatellites pouvait repérer le gène impliqué dans une maladie héréditaire mendélienne alors même que l’on ignorait tout de la protéine en cause, du mécanisme pathogénique et, bien sûr, de l’architecture générale du génome humain. Les maladies complexes, mi-génétiques, mi-environnementales, se révèlent rétives à une telle approche. L’identification des séquences d’ADN responsables de leur forte héritabilité reste un but d’actualité, car elle est nécessaire tant pour la compréhension des mécanismes pathogéniques que pour l’amélioration des thérapies ; mais la génétique pure, même avec les avancées technologiques en cours comme le séquençage ultra-rapide, aura certainement besoin du secours de la physiologie, des données fonctionnelles, et des analyses de pathways pour aboutir à enfin retrouver l’héritabilité perdue… |
L’auteur déclare n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Footnotes |
1. Reich DE, Lander ES. On the allelic spectrum of human disease. Trends Genet. 2001; 17 : 502–10. 2. Jordan B. Le déclin de l’empire des GWAS.Med Sci (Paris) 2009; 25 : 537–9. 3. Manolio TA, Collins FS, Cox NJ, et al. Finding the missing heritability of complex diseases. Nature 2009; 461 : 747–53. 4. Jordan B. Le boom des séquenceurs nouvelle génération. Med Sci (Paris) 2010; 26 : 325–7. 5. Jordan B. Un génome plein de trous et de bosses. Med Sci (Paris) 2007; 23, 123–4. 6. Wellcome Trust Case Control Consortium. Genome-wide association study of CNVs in 16,000 cases of eight common diseases and 3,000 shared controls. Nature 2010; 464 : 713–20. 7. Dickson SP, Wang K, Krantz I, et al. Rare variants create synthetic genome-wide associations. PLoS Biol 2010; 8 : e1000294. 8. Maher B. Hiding place for missing heritability uncovered. Nature ; 25 janvier 2010 doi: 10.1038/news.2010.33. 9. Jordan B. Grandeur et servitudes de la génétique inverse. Med Sci (Paris) 1988; 4 ; 138–40. |