| |
| Med Sci (Paris). 2010 June; 26(6-7): 579–581. Published online 2010 June 15. doi: 10.1051/medsci/2010266-7579.Points chauds de sélection positive dans les génomes de primates David Enard, Frantz Depaulis, and Hugues Roest Crollius* Institut de biologie de l’École Normale Supérieure (IBENS), CNRS UMR 8197, 46, rue d’Ulm, 75230 Paris Cedex 05, France MeSH keywords: Animaux, Génome, Modèles génétiques, Mutation, Polymorphisme de nucléotide simple, Primates, génétique, Recombinaison génétique, Sélection génétique, Récepteur de type Toll-1 |
Quand la première édition de L’Origine des Espèces est publiée en 1859 [
1], on n’y trouve aucune référence à l’évolution de l’homme et à ses liens de parenté avec les grands singes. Darwin, qui n’en pense pas moins, anticipe ainsi un procès en hérésie et le risque d’une opposition démultipliée à sa théorie de la sélection naturelle. À l’ère génomique, l’homme est devenu « l’espèce modèle » pour l’étude de la sélection naturelle chez les vertébrés. Les projets de génotypage à grande échelle comme le projet HapMap [
11], d’abord dédiés aux études d’associations, ont en effet permis d’identifier plusieurs dizaines voire centaines de gènes impliqués dans l’adaptation des populations humaines à leurs environnements au cours des cent mille dernières années. L’approche gène-candidat utilisée jusquelà n’avait pas permis d’anticiper un nombre si important, qui implique que l’homme a été et reste en constante adaptation. |
Beaucoup d’adaptations, peu d’explications Les « gènes de l’adaptation » humaine sont donc nombreux dans le génome, mais on ne connaît la nature de l’adaptation que dans quelques cas. Un des exemples les plus connus est celui du locus de la lactase, une enzyme de digestion du lactose. Une mutation survenue il y a un peu moins de dix mille ans a permis à ses porteurs de digérer le lactose à l’âge adulte audelà de la période de sevrage [
2]. De négligeable, la fréquence de la mutation avantageuse a augmenté rapidement dans la population européenne, sous l’effet de l’avantage reproducteur lié à l’apport nutritif supplémentaire. Cette fréquence dépasse aujourd’hui 70 % en Europe du Nord. Autre exemple, une mutation du gène SLC24A5 (solute carrier family 24, member 5) associée à une pigmentation plus claire a été sélectionnée dans la population européenne en raison d’une plus grande adaptation des peaux claires aux hautes latitudes [
3]. Ou encore, une mutation au locus G6PD (glucose 6 phosphate déshydrogénase) qui confére une résistance à la malaria a été et est toujours sélectionnée dans des populations africaines là où la maladie est endémique [
4]. |
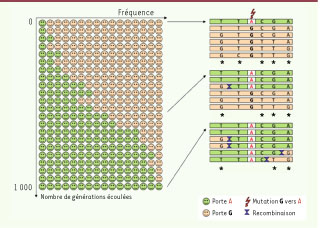
Identifier les gènes sous sélection positive Comment, outre une explication biologique forte, sait-on que ces gènes sont liés à une adaptation ? La sélection naturelle positive est la sélection des mutations conférant à leur porteur un plus grand succès reproducteur. Un gène impliqué dans l’adaptation est donc un gène dans lequel une mutation donnant un phénotype héréditaire et avantageux est survenue et a été sélectionnée jusqu’à remplacer l’état préexistant (Figure 1). Dans les données de génotypage de type HapMap, ce ne sont pas les mutations elles-mêmes qui sont détectées, mais l’empreinte que leur sélection a laissée sur la variabilité environnante; cette empreinte est appelée un balayage sélectif. La fréquence d’une mutation avantageuse augmente rapidement sous l’effet de la sélection. L’haplotype qui porte cette mutation est entraîné dans sa progression, selon l’effet d’autostop génétique, au détriment des autres haplotypes qui disparaissent (Figure 1). On a donc bien eu un balayage : la variabilité génétique en déséquilibre de liaison avec la mutation a été « balayée ».  | Figure 1
Conséquences d’un balayage sélectif. La variabilité (astérisques) diminue autour d’une mutation sélectionnée (partie droite de la figure) au fur et à mesure que la fréquence de cette mutation augmente dans la population sous l’effet de la sélection positive directionnelle (partie gauche de la figure). |
|
Et en dehors de l’homme ? En l’absence de données équivalentes de génotypage à grande échelle chez d’autres vertébrés, l’homme reste la seule espèce de cet embranchement pour laquelle on dispose de cartes génomiques de la sélection positive. Les méthodes existantes de détection de balayages sélectifs nécessitent en effet de génotyper au moins plusieurs dizaines d’individus. On ignore ainsi si la sélection positive a été aussi fréquente au cours de l’évolution récente d’autres Vertébrés (par exemple au cours des 10 000 dernières générations). De plus, on ignore si les mêmes gènes ont été impliqués dans l’adaptation à la fois de l’homme et d’autres espèces, une information qui pourrait s’avérer précieuse pour comprendre l’évolution humaine. En particulier, qu’en est-il de nos proches cousins les grands singes, et plus généralement des primates ? Les génomes diploïdes du chimpanzé [
5], du gorille, de l’orang-outan, du macaque [
6] et du marmouset ont été séquencés à forte couverture, chacun à partir de l’ADN d’un individu unique. Ce qui signifie que, même si l’on ne dispose pas de la variabilité déduite d’un grand nombre d’individus comme pour l’homme, il est tout de même possible d’identifier les positions ponctuelles hétérozygotes entre les deux chromosomes de chaque individu primate séquencé. Le chimpanzé séquencé possède ainsi 0,8 million de positions hétérozygotes, l’orang-outan 1,4 million, et le macaque 2,2 millions. Pour comparaison, les génomes de Craig Venter [
7] et de James Watson [
8] totalisent chacun 1,6 million de positions hétérozygotes. |
Étudier la sélection récente à partir d’individus uniques La théorie en génétique des populations prédit qu’il devrait être possible de détecter les balayages sélectifs survenus indépendamment dans ces espèces seulement à partir de leurs positions hétérozygotes. Il faut toutefois s’attendre à des inférences de sélection positive moins précises que lorsqu’on dispose d’un grand nombre d’individus, ce qui explique que ce type de ressources a jusque-là été ignoré pour l’étude de la sélection récente. Nous avons mis au point une méthode statistique qui permet d’isoler des régions du génome où la concentration en positions hétérozygotes est très faible et où la survenue d’un balayage sélectif (réduction de variabilité) est donc probable [
9]. De nombreux facteurs autres que la sélection peuvent réduire la concentration locale en positions hétérozygotes, et toute la difficulté a été d’en éliminer les effets confondants. En appliquant notre méthode aux génomes de Craig Venter et James Watson, nous avons d’abord pu vérifier que les gènes candidats trouvés recoupent ceux déjà publiés à partir des données HapMap. La méthode a donc pu être utilisée pour rechercher les balayages dans les génomes du chimpanzé, de l’orang-outan et du macaque. Nous avons alors montré par simulations de populations que le pourcentage d’inférences fausses de balayages est assez élevé dans chaque espèce, du fait du faible nombre d’individus. |
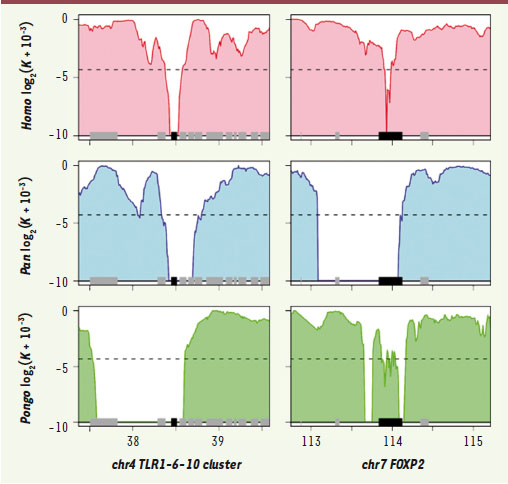
Points chauds de sélection positive Toutefois, ce taux élevé de faux positifs n’est pas un problème pour la question que nous avons ensuite posée : les gènes soumis à une sélection positive récente chez l’homme l’ont-ils aussi été chez le chimpanzé, l’orang-outan et/ou l e macaque ? La réponse est oui puisqu’environ deux fois plus de gènes qu’attendu par hasard avec un balayage sélectif chez l’homme ont aussi un balayage sélectif inféré chez deux des trois autres primates analysés, voire dans les trois. Les gènes concernés peuvent être considérés comme des points chauds de sélection positive récente chez les primates. Par exemple, des acteurs fondamentaux de l’immunité innée, les récepteurs Toll-like 1,6 et 10, ont leurs gènes regroupés dans un balayage sélectif chez l’homme, le chimpanzé et l’orangoutan (Figure 2). Plus surprenant, le locus du gène FOXP2, dont la sélection positive récente chez l’homme est souvent discutée en relation à l’acquisition du langage [
10,
12], semble aussi montrer des signes de balayage chez le chimpanzé et l’orang-outan (Figure 2). Ce dernier exemple illustre bien l’intérêt de savoir si un gène a été sous sélection positive récente uniquement chez l’homme ou aussi chez d’autres primates, voire d’autres mammifères ou d’autres vertébrés. En effet, la nature de l’adaptation (régime alimentaire, résistance aux pathogènes, climat, capacités cognitives, etc.) qui expliquerait ces épisodes de sélection positive à la fois chez l’homme et chez un autre animal a des chances d’être différente dans les deux cas de figures. D’une manière générale, un cas de sélection positive chez l’homme qui a eu lieu indépendamment chez le chimpanzé nous invite à l’expliquer de manière moins anthropocentrique qu’un événement qui aurait eu lieu uniquement dans la lignée humaine.  | Figure 2
Balayages sélectifs inférés aux locus des récepteurs Toll-like 1, 6 et 10 et du gène FOXP2. Chaque graphique représente une région génomique de 2 Mb. Les gènes sont figurés en bas du graphique (noir pour le (s) gène(s) candidat(s) à la sélection, gris pour les autres). Ligne 1 (rouge) : homme. Ligne 2 (bleu) : chimpanzé. Ligne 3 (vert) : orangoutan. Une baisse de la mesure du polymorphisme représentée en ordonnée indique la survenue probable d’un balayage sélectif. |
|
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article. |
1. Darwin C. On the origin of species by means of natural selection, or the preservation of favoured races in the struggle for life. London : John Murray, 1859 : 502 p. 2. Bersaglieri T, Sabeti PC, Patterson N, et al. Genetic signatures of strong recent positive selection at the lactase gene. Am J Hum Genet 2004; 74 : 1111-20. 3. Izagirre N, Garcia I, Junquera C, et al. A scan for signatures of positive selection in candidate loci for skin pigmentation in humans. Mol Biol Evol 2006; 23 : 1697-706. 4. Sabeti PC, Reich DE, Higgins JM, et al. Detecting recent positive selection in the human genome from haplotype structure. Nature 2002; 419 : 832-7. 5. Chimpanzee sequencing and analysis Consortium. Initial sequence of the chimpanzee genome and comparison with the human genome. Nature 2005; 437 : 69-87. 6. Gibbs RA, Rogers J, Katze MG, et al. Evolutionary and biomedical insights from the rhesus macaque genome. Science 2007; 316 : 222-34. 7. Levy S, Sutton G, Ng PC, et al. The diploid genome sequence of an individual human. PLoS Biol 2007; 5 : e254. 8. Wheeler DA, Srinivasan M, Egholm M, et al. The complete genome of an individual by massively parallel DNA sequencing. Nature 2008; 452 : 872-6. 9. Enard D, Depaulis F, Roest Crollius H. Human and nonhuman primate genomes share hotspots of positive selection. PLoS Genet 2010; 6 : e1000840. 10. Enard W, Przeworski M, Fisher SE, et al. Molecular evolution of FOXP2, a gene involved in speech and language. Nature 2002; 418 : 869-72. 11. Montpetit A, Chagnon F. La carte d’haplotype du génome humain : Une révolution en génétique des maladies à hérédité complexe. Med Sci (Paris) 2006; 22 : 1061-7. 12. Rochefort C. FoxP2, un gène qui fait parler l’homme et chanter les oiseaux. Med Sci (Paris) 2008; 24 : 906-7. |