| |
| Med Sci (Paris). 2010 June; 26(6-7): 655–658. Published online 2010 June 15. doi: 10.1051/medsci/2010266-7655.Chroniques génomiques La séquence… et après ? Bertrand Jordan* Marseille-Nice Génopole, case 901, Parc scientifique de Luminy, 13288 Marseille Cedex 9, France MeSH keywords: Séquence nucléotidique, Maladies génétiques congénitales, génétique, Génétique médicale, tendances, Génome humain, Génotype, Humains, Polymorphisme de nucléotide simple, Probabilité |
La séquence, nouvel espoir après les Snip ? Les difficultés des études d’association génétique à grande échelle (WGA, whole genome association studies) [
1] jettent un sérieux doute sur la capacité de telles analyses à fournir des informations cliniques réellement utiles. Dans la mesure où les résultats combinés des meilleures études ne rendent compte que de 5 à 10 % de la composante génétique pour des maladies fréquentes comme le diabète insulino-dépendant, quel crédit accorder aux profils vendus par 23andMe ou Navigenics, et surtout à leur interprétation en termes de vulnérabilité à diverses affections ? Pourtant, selon certains, la « séquence personnelle » [
2] serait en passe de lever ces difficultés et d’offrir l’accès à des informations réellement fiables, prenant notamment en compte les variants rares que ne peuvent révéler pour des raisons logistiques les études de snip pratiquées à l’aide de puces à ADN. Il est vrai que les technologies de séquençage rapide continuent à progresser [
3], et que le coût d’une séquence humaine de bonne qualité baisse de manière continue : déja inférieur à 10 000 dollars, il pourrait s’approcher à brève échéance du mythique « génome à 1 000 dollars ». À l’heure actuelle près de cent génomes humains ont déjà été séquencés et publiés (en incluant les projets portant sur des cellules tumorales), et le nombre total est sûrement bien plus élevé si l’on y ajoute les données encore confidentielles. On peut donc réellement envisager que, dans un futur proche, chacun de nous dispose de sa propre séquence (inscrite sur la puce d’une carte Vitale nouveau modèle ?) et que celle-ci soit utiliséetant pour estimer le risque de telle ou telle affection que pour choisir le médicament le plus adapté en cas de traitement. Cela suppose naturellement que l’interprétation clinique de ces données soit rapide, fiable et compréhensible pour le patient… et c’est ici que le bât blesse, comme en témoigne une publication récemment parue dans The Lancet [
4]. |
De la séquence à la clinique « Clinical assessment incorporating a personal genome », tel est le titre de cet article qui décrit en détail l’interprétation clinique de la séquence d’ADN de Stephen Quake, chercheur au Department of Bioengineering de l’Université de Stanford et cofondateur de l’entreprise Helicos. Cette dernière, qui a mis sur le marché l’appareil Heliscope, premier des séquenceurs de troisième génération1, rencontre actuellement de sérieuses difficultés, au point d’avoir dernièrement licencié la moitié de son personnel. C’est néanmoins grâce à un Heliscope que Stephen Quake a séquencé son propre ADN, publié au milieu de l’année dernière [
5]. Il n’a fallu que 2 millilitres de sang et quelques semaines de travail (l’article, chose rare de nos jours, ne comporte que trois auteurs) pour obtenir 148 gigabases de séquence brute et repérer, par rapport augénome humain de référence2 et en plus de 752 CNV (copy number variations), 2,8 millions de différences ponctuelles avec un taux de faux positifs estimé inférieur à 1 %. L’interprétation en termes cliniques de cette séquence, rapportée dans l’article qui fait l’objet de cette chronique, a en revanche mobilisé trente et un auteurs et nécessité des mois de travail, la consultation des nombreuses bases de données dans lesquelles sont dispersées les informations fonctionnelles et la construction de systèmes informatiques spécifiques… le tout pour un résultat finalement assez mince. Assez mince, surtout lorsqu’on le compare à ce que laissent entendre les sites Internet d’entreprises comme 23and Me ou Navigenics : à leur lecture, on a nettement l’impression que, pour quelques centaines de dollars, la mise en œuvre d’une puce repérant les allèles de cinq cent mille Snip fournira une ample moisson d’informations sur les prédispositions génétiques du client, et que ces indications sont suffisamment solides et précises pour déboucher sur la recommandation d’examens spécifiques ou sur des consignes fermes de mode de vie. |
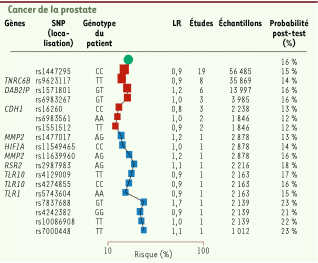
Au terme d’un important travail réunissant bioinformaticiens, biologistes, cliniciens et même éthiciens (le patient étant impliqué dans le processus d’interprétation), les données obtenues peuvent être rangées dans trois catégories. Tout d’abord, les mutations repérées (à l’état hétérozygote) et dont l’effet pathologique (à l’état homozygote) est connu : six au total, impliquées dans l’hémochromatose, l’épidermolyse bulleuse, la cystinurie… On détecte également quelques variants additionnels touchant des gènes associés à des maladies rares, mais ceux-ci sont nouveaux et leur effet éventuel est inconnu. On voit donc - et ce n’est pas une surprise - que ce génome recèle, comme, sans nul doute, pour chacun d’entre nous, des mutations dont l’effet à l’état homozygote serait nettement délétère. S’ajoutent à cela de très nombreux variants (plusieurs milliers) repérés dans des gènes dont l’importance médicale est encore inconnue - ce qui manifeste l’étendue de notre ignorance… Le deuxième type de résultat concerne la pharmacogénétique, la sensibilité individuelle à divers médicaments : une dizaine de variants sont repérés, pour certains à l’état homozygote, qui suggèrent notamment que Stephen Quake devrait être traité avec de faibles doses de warfarine (ou coumadine) en cas de cardiopathie, et que l’emploi de bêta-bloquants est contre-indiqué. Enfin, et c’est sans doute le point le plus intéressant de cet article, les auteurs ont tenté d’évaluer le risque clinique encouru pour une série d’affections fréquentes présentant une composante génétique marquée. Ils ont pour cela évalué la probabilité a priori de survenue de ces affections pour un homme du même âge vivant dans un environnement similaire, grâce à la collation des données de la littérature. Ils ont ensuite multiplié cette probabilité par la valeur du risque relatif (émanant des études WGA) pour chacun des allèles trouvés dans l’ADN analysé (Figure 1).  | Figure 1
Évaluation du risque de cancer de la prostate d’après l’analyse de la séquence d’ADN de S. Quake. L’évaluation a été faite en partant du risque a priori (évalué à 16 %) et en appliquant successivement la contribution génétique (colonne LR, likelihood ratio) pour chaque allèle de Snip trouvé dans le génome de Stephen Quake. Le nombre d’études GWA et le nombre d’échantillons étudiés sont indiqués dans la colonne suivante, la probabilité résultante dans la dernière colonne. Noter que les validités statistiques (nombre d’études, nombre de cas) décroissent très fortement au fur et à mesure que l’on descend sur le diagramme (extrait de la figure 4 de [ 4] © Lancet 2010). |
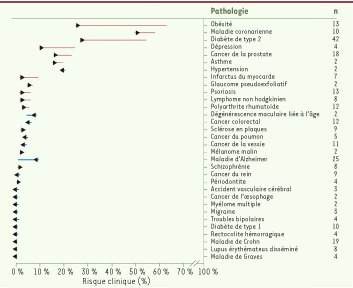
On voit que dans l’exemple choisi (cancer de la prostate) le risque a priori est de 16 %, mais que l’on arrive au final, compte tenu des allèles de Snip détectés, à une valeur de 23 %… Encore faut-il noter que toutes les études WGA n’ont pas la même puissance statistique : sur la Figure 1, les résultats les plus solides, figurés par des carrés rouges, sont utilisés en premier, les études moins significatives (carrés bleus) venant ensuite. Du coup, toujours dans l’exemple choisi, si l’on ne tient compte que des études les plus solides, la valeur finale du risque est de 12 % au lieu de 23 %, il est donc légèrement diminué par rapport au risque a priori au lieu d’être fortement augmenté… Quoi qu’il en soit, le résultat final pour les affections qui ont pu être étudiées de cette manière est représenté sur la Figure 2, montrant que quelques risques sont notablement augmentés et de nombreux autres réduits - tout en gardant à l’esprit les limites de la méthodologie…  | Figure 2
Évaluation du risque clinique encouru par Stephen Quake pour une série d’affections comportant une composante génétique notable. La méthodologie est celle indiquée pour la Figure 1. La partie verticale de chaque flèche indique le risque a priori, la direction de la flèche et la couleur du trait (bleu = protection, rouge = vulnérabilité) montrent le sens de la correction due aux influences génétiques. Le chiffre en dernière colonne indique le nombre de Snip utilisés pour calculer le risque (d’après la figure 3 de [ 4] © Lancet 2010). |
Malgré ces limites, ce travail est néanmoins novateur et intéressant : il propose une méthode rationnelle pour intégrer les résultats de nombreuses études WGA et en établir une traduction clinique3. Il souligne aussi à quel point l’image médiatique de ces analyses (« votre ADN nous indique que vous mourrez d’une attaque cardiaque à 53 ans et 5 mois ») est éloignée de la réalité… Tout cela laisse aussi planer de sérieux doutes sur l’intérêt de prévisions découlant d’une analyse bio-informatique standardisée portant sur quelques centaines d’allèles de Snip, comme le proposent 23andMe et consorts… |
Un bilan mince et pourtant déjà problématique Loin de tout nous dire sur Stephen Quake et sur son destin biologique, cet important travail ne fournit donc que des informations cliniques très partielles et souvent contestables : il témoigne à l’évidence de la difficulté que nous avons encore à passer du génotype au phénotype, de notre compréhension très imparfaite de la manière dont notre organisme met en œuvre notre séquence d’ADN. Il montre néanmoins la voie à suivre pour, peu à peu, progresser dans l’interprétation de telles données pour s’approcher de cette médecine personnalisée dont on a annoncé un peu vite l’avènement imminent. Reste que la gestion de ces résultats, aussi partiels soient-ils, pose plusieurs questions. Leur obtention requiert une somme de travail qui ne pourra à l’évidence pas être investie pour chacune des « séquences à 1 000 dollars » qu’on nous promet pour bientôt : le coût total des études présentées dans cet article est sans doute de l’ordre du million de dollars… Certes, on peut penser que tout cela va s’organiser, s’automatiser et demander de moins en moins d’intervention humaine - mais il ne faut pas oublier que les connaissances évoluent vite : la traduction clinique d’un génome devra intégrer les résultats les plus récents et, sans doute, être reprise périodiquement. Et, comme le détaille un intéressant point de vue paru dans le même numéro du Lancet [
6], qui va expliquer ces résultats au patient ? On sait que la compréhension d’une probabilité, d’un risque relatif, est déjà problématique pour une large partie du public, qu’en sera-t-il quand une séquence d’ADN à 1 000 dollars révèleraune vulnérabilité accrue ou au contraire diminuée pour des dizaines d’affections ? Sans oublier naturellement toute la problématique de l’« intimité génétique » (qui à le droit de consulter ces résultats ?) ou du « droit de ne pas savoir », qui risque fort d’être mise à mal dès lors qu’une séquence fournit un catalogue exhaustif des fêlures de votre patrimoine génétique. De ce point de vue, il est peut-être heureux que nous soyons aussi peu avancés dans une interprétation précise, cela nous laisse un peu de temps pour réfléchir à la manière d’utiliser ces nouvelles connaissances… |
L’auteur déclare n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article. |
Footnotes |
1. Jordan B. À la recherche de l’héritabilité perdue… Med Sci (Paris) 2010 ; 26 : 541-3. 2. Jordan B. Chacun sa séquence ! Med Sci (Paris) 2009 ; 25 : 647-8. 3. Jordan B. Le boom des séquenceurs nouvelle génération. Med Sci (Paris) 2010 ; 26 : 325-7. 4. Ashley EA, Butte AJ, Wheeler MT, et al. Clinical assessment incorporating a personal genome. Lancet 2010 ; 375 : 1525-35. 5. Pushkarev D, Neff NF, Quake SR. Single-molecule sequencing of an individual human genome. Nat Biotechnol 2009 ; 27 : 847-52. 6. Ormond KE, Wheeler MT, Hudgins L, et al. Challenges in the clinical application of whole-genome sequencing. Lancet 2010 ; 375 : 1749-51. |