| |
| Med Sci (Paris). 2010 November; 26(11): 943–949. Published online 2010 November 15. doi: 10.1051/medsci/20102611943.Résistance aux antibiotiques chez Staphylococcus aureus
Les points-clés en 2010 Oana Dumitrescu, Olivier Dauwalder, Sandrine Boisset, Marie-Élisabeth Reverdy, Anne Tristan, and François Vandenesch* Centre national de référence des staphylocoques, Inserm U851, IFR128, Université Lyon 1, rue Guillaume Paradin, 69372 Lyon Cedex 08, France Hospices civils de Lyon, Centre de biologie et de pathologie Est, Institut de microbiologie, Laboratoire de bactériologie, 59, boulevard Pinel, 69677 Bron, France |
Résistance aux antibiotiques et plasticité génétique de S. aureus
Alors qu’il s’agit d’un commensal parmi les plus fréquents de notre flore normale, Staphylococcus aureus est un pathogène redoutable qui a su développer des résistances à chaque nouvel antibiotique introduit depuis un demi-siècle. La plasticité de son génome lui confère la capacité de s’adapter à toutes les conditions environnementales, et notamment d’acquérir des gènes de résistance aux antibiotiques et de développer des mécanismes de régulation pour s’adapter à des concentrations croissantes d’antibiotiques. Ainsi, dès 1941 sont apparus les staphylocoques résistants à la pénicilline, grâce à l’acquisition d’une pénicillinase plasmidique, enzyme dégradant la pénicilline. La résistance à la pénicilline, initialement restreinte au milieu hospitalier, a très vite diffusé en milieu communautaire et concerne actuellement plus de 90 % des souches de S. aureus. Pendant les années 1950 sont apparues les souches de S. aureus multirésistantes : à la résistance à la pénicilline était associée la résistance à la streptomycine, à l’érythromycine, à la tétracycline, au chloramphénicol ainsi qu’aux sulfamides. L’introduction en 1959 de la méticilline, dérivé semi-synthétique de la pénicilline, pour le traitement des infections staphylococciques a soulevé un grand espoir. Mais à peine un an plus tard, les premières souches hospitalières de S. aureus résistantes à la méticilline (SARM) sont apparues dans un hôpital de Grande-Bretagne [
1]. Le secret de ce pouvoir d’adaptation a été partiellement percé par le séquençage du génome de S. aureus effectué par les équipes de Baba et d’Hiramatsu [
2]. Le génome de S. aureus est formé de deux domaines fonctionnels distincts. La majeure partie du chromosome contient les gènes qui assurent la maintenance de la bactérie. La deuxième partie du génome est constituée d’éléments génétiques accessoires et mobiles comme des plasmides, transposons, prophages ou des îlots de pathogénie portant la plupart des gènes associés à des facteurs de virulence et à la résistance aux antibiotiques [
3,
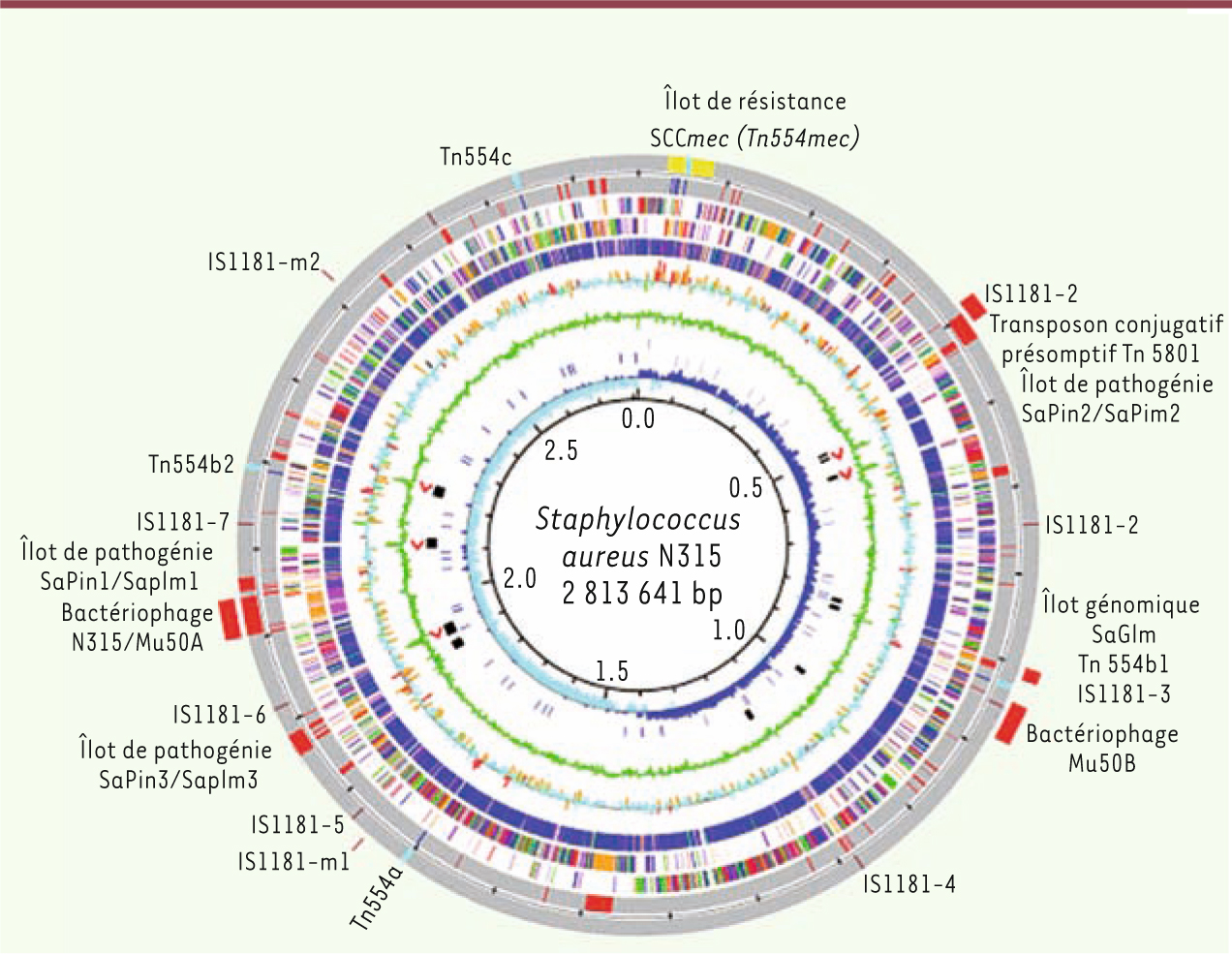
4] (Figure 1). Ainsi, en dehors des mutations spontanées, S. aureus diversifie son génome grâce aux échanges de matériel génétique avec d’autres espèces bactériennes par des phénomènes de transfert horizontal de gènes. Le phénotype de résistance, comme le profil patho génique, semble donc bien être déterminé par les combinaisons de ces éléments génétiques accessoires portés par le chromosome. 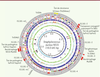 | Figure 1
Génome de S. aureus-souche N315 résistante à la méticilline et souche Mu50 de sensibilité diminuée à la vancomycine. Les éléments spécifiques à Mu50 (96 % d’homologie avec la N315) sont représentés au-delà du cercle extérieur. Les éléments génétiques mobiles sont représentés au niveau du cercle extérieur : la cassette SCC mec, en jaune ; le transposon Tn554, en bleu clair ; les phages et îlots de pathogénie, en rouge ; les séquences d’insertion IS1181, en marron (adapté de [ 2]). |
|
La cassette staphylococcique SCC mec, support de la résistance à la méticilline Le gène mecA
Le temps zéro de l’évolution des SARM est l’acquisition du gène mecA, fragment d’ADN de 2,1 kb codant une protéine liant la pénicilline additionnelle (PLP2a) [ 4]. Cette transpeptidase PLP2a a une affinité faible vis-à-vis des β-lactamines. Les souches de S. aureus possédant le gène mecA sont donc résistantes à toute la famille des β-lactamines, notamment à la méticilline ou à l’oxacilline. Le gène mecA est inclus dans un élément génétique mobile : la cassette staphylococcique (SCC mec, staphylococcal cassette chromosome mec). Cette cassette s’insère au niveau d’un site spécifique du chromosome : le site attB
scc
,situé à l’extrémité 3’ d’une séquence à cadre de lecture ouvert désignée sous le nom de orfX dont la fonction demeure inconnue [
5]. Structure de la cassette SCCmec
La cassette SCC mec comporte deux éléments essentiels : le complexe du gène mecA et un complexe de gènes codant des recombinases ccr (cassette chromosome recombinase) qui assurent les phénomènes d’intégration et d’excision de la cassette. La cassette SCC mec comporte également des éléments dits accessoires comme des séquences d’insertion, des transposons ou des copies de plasmide portant des gènes de résistance à des métaux lourds ainsi qu’à des antibiotiques autres que les β-lactamines [ 4–
6]. Le complexe mec
Le complexe mec est constitué d’une copie intacte du gène mecA, d’une copie de la séquence d’insertion IS431 et des gènes régulateurs du gène mecA : mecI (codant un répresseur transcriptionnel de mecA) et mecR1 (codant la protéine MecR1). MecR1 détecte la présence de β-lactamines grâce à son domaine extracellulaire. Une fois l’antibiotique lié, il y a activation du domaine intracellulaire qui acquiert une activité protéasique et dégrade le répresseur MecI, favorisant ainsi l’expression de mecA. Ces gènes régulateurs peuvent être intacts ou tronqués, les mutations survenant au niveau de ces gènes régulateurs pouvant affecter le niveau de résistance à la méticilline. À ce jour, cinq classes de complexe mec (A à E) ont été décrites chez les staphylocoques [ 6,
9]. Le complexe des gènes des recombinases Les recombinases sont responsables de la mobilité de la cassette. Le complexe des gènes des recombinases est constitué soit d’une paire de gènes, ccrA et ccrB combinés (4 allotypes décrits ccrAB1-4), soit d’un gène unique ccrC retrouvé au niveau de la cassette SCC mec de types V et VII [
7,
8]. Ainsi, à ce jour, cinq types de complexes de recombinases ont été identifiés ( ccrAB1-4, ccrC) et leur nomenclature est régie par les recommandations du groupe international d’études qui travaille actuellement sur la classification des cassettes SCC mec [ 9]. Les différents types SCCmec
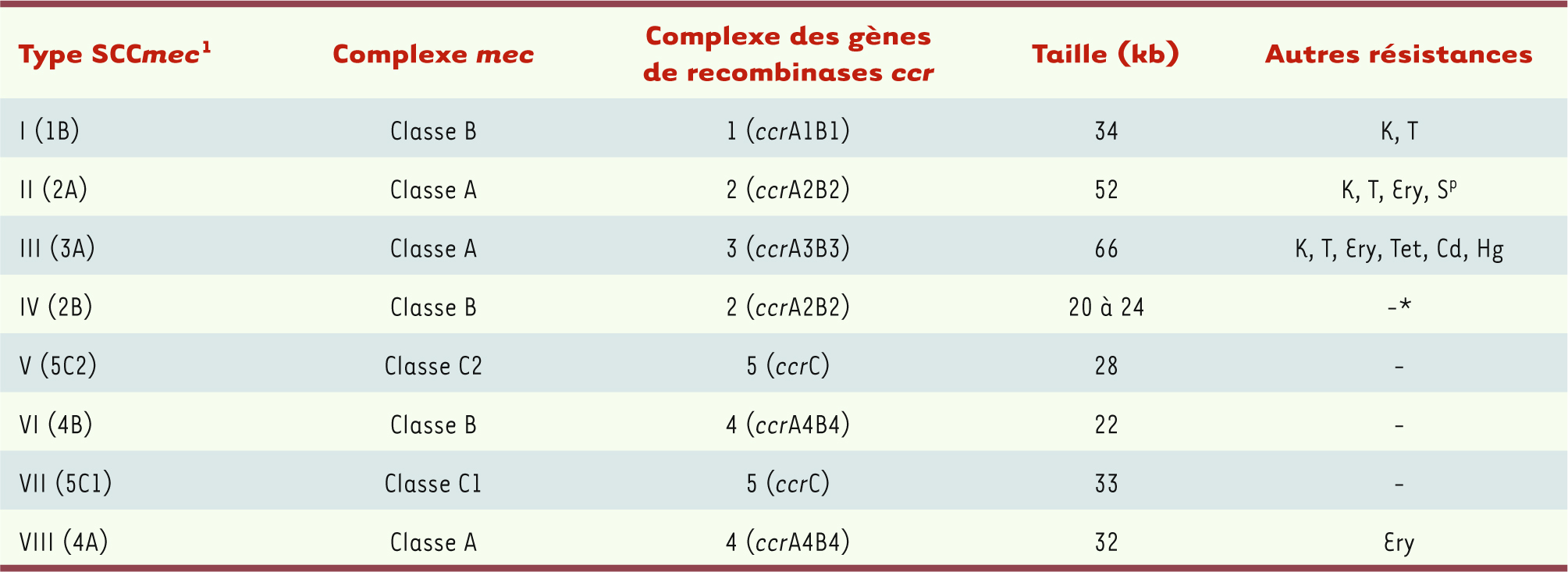
La combinaison des différentes classes de complexe mec et des 5 types de recombinases permet de définir huit types de cassettes (I-VIII). Ces huit types diffèrent, d’une part, par leur structure et leur taille (20 à 66 kb) et, d’autre part, par leur répertoire de résistances aux antibiotiques [ 7, 8,
10] (Tableau I).
 | Tableau I
Les huit types SCCmec. D’après la nouvelle classification et la nomenclature établies par le groupe de travail IWG-SCC (International group on the classification of staphylococcal cassette chromosome element) [ 9]. 1Nomenclature des types SCC mec : chiffre romain suivi par le type de complexe de gènes de recombinases et le type de complexe mec entre parenthèses.
* À l’exception du sous-type IVc qui présente des résistances associées aux aminosides (phénotype KTG). K : kanamycine, T : tobramycine, Ery : érythromycine, Tet : tétracycline, Sp : spectinomycine, Cd : cadmium, Hg : mercure. |
Les variants alléliques SCCmec de types I, II ou III sont plus particulièrement présents dans les souches de SARM d’origine hospitalière (SARM-H), tandis que les nouveaux variants alléliques de SCCmec, les types IV à VIII, ont été identifiés dans des souches de SARM d’origine communautaire (SARM-C) [
11]. Les cassettes SSCmec des SARM-H comprennent, en plus du complexe du gène mecA, des éléments génétiques dits accessoires portant des gènes de résistance à des antibiotiques autres que les β-lactamines, responsables du profil de multirésistance aux antibiotiques des souches hospitalières. Les allèles SCCmec de types IV à VIII portés par les SARM-C sont de plus petite taille et ne contiennent pas de gènes additionnels de multirésistance aux antibiotiques (Tableau I).
Hypothèses sur l’acquisition de la cassette SCCmec
L’origine de la cassette de résistance demeure inconnue. Cependant, différents indices comme la présence d’un gène mecA homologue chez Staphylococcus sciuri ou la présence de la séquence d’insertion IS1272 chez Staphylococcus haemolyticus retrouvée dans les types I et IV de SCC mec orientent vers l’hypothèse d’un échange horizontal entre S. aureus et des staphylocoques à coagulase négative [
12]. Jusqu’à présent la cassette SCC mec n’a été retrouvée que dans le genre Staphylococcus. Cependant le séquençage d’une souche de Macrococcus caseolyticus résistante à la méticilline a révélé l’existence d’un complexe mec, mecIRA m, qui pourrait être un précurseur de l’actuel complexe mec de S. aureus [
13]. |
SARM d’origine communautaire et virulence Les premiers cas d’infection à SARM ont été rapportés il y a plus de trente ans et revêtaient un caractère nosocomial, l’acquisition de SARM étant liée à une hospitalisation récente ou à l’exposition prolongée et récurrente aux antibiotiques. Néanmoins, depuis les années 1990, les infections à SARM se sont multipliées chez des patients n’ayant aucun facteur de risque traditionnel d’acquisition de SARM : il s’agit des infections à SARM-C [3]. Les souches de SARM isolées dans ce contexte ont des caractéristiques permettant de les différencier de souches SARM associées au milieu hospitalier ; notamment, le SARM-C majoritaire en France présente un profil caractéristique de résistance aux antibiotiques (résistant à la pénicilline, à l’oxacilline, à la kanamycine, à la tétracycline et de sensibilité intermédiaire à l’acide fusidique) permettant une identification rapide par tout laboratoire de bactériologie [
14]. En plus de la cassette SCCmec le plus souvent de type IV ou V, les souches de SARM-C présentent une autre particularité, un facteur de virulence rarement retrouvé dans d’autres souches de S. aureus, la leucocidine de Panton Valentine (PVL) [
32]. La PVL appartient à la famille des toxines synergo-hyménotropes ayant un tropisme pour les membranes cellulaires sur lesquelles elles agissent par l’action synergique de deux composés protéiques [
15]. Ces deux composés protéiques indépendants (composés S et F) s’associent à la surface de la membrane des polynucléaires neutrophiles humains pour former des pores. Le locus luk-PV porté par le phage phiSLT est constitué d’un seul opéron qui contient deux gènes, lukS-PV et lukF-PV, codant les protéines LukS-PV et LukF-PV. La PVL exerce des activités cytotoxiques au niveau des leucocytes humains en induisant la libération progressive de médiateurs pro-inflammatoires (leucotriène B4, interleukine 8 et histamine), ce qui conduit à la formation de foyers d’infiltration et de nécrose tissulaire [
16]. Alors que la PVL a été purifiée en 1960, l’intérêt pour cette toxine a été relancé récemment, d’une part à la suite de la description d’infections très sévères dues à des souches sensibles ou résistantes à la méticilline sécrétant la PVL (pneumonies nécrosantes, infections nécrosantes sévères de la peau et des tissus mous, infections ostéo-articulaires graves) [
17], et d’autre part en raison de l’émergence mondiale de souches SARM-C dans lesquelles la présence des gènes codant la PVL est quasi pathognomonique [
18]. Bien que la diffusion des souches SARM-C soit mondiale, leur distribution n’est pas géographiquement uniforme. La situation la plus alarmante prévaut actuellement aux États-Unis, où la proportion de SARM-C représente en moyenne 59 % des S. aureus isolés des infections de la peau et de tissus mous d’origine communautaire. Une situation similaire a été rapportée en Afrique du Nord, où la prévalence des SARM-C est de 48,8 %. En Europe, la distribution n’est pas uniforme : il existe des pays à faible diffusion, tel le Royaume-Uni avec moins de 2 % de SARM isolés dans les infections communautaires à S. aureus, mais aussi des pays à forte diffusion telle la Grèce avec 75 % de souches de SARM circulant dans la communauté. Pour résumer la situation mondiale, la diffusion de SARM-C est hétérogène mais avec une tendance générale à l’augmentation. En France, où la prévalence de ces SARM-C est faible, 3,6 % dans la dernière étude disponible, on retrouve majoritairement le clone ST80 (désigné par l’analyse MultiLocusSequenceType) caractérisé par son profil particulier de résistance aux antibiotiques le rendant reconnaissable par des techniques simples [
19]. On peut également signaler la diffusion d’un autre clone de SARM à la fois communautaire et hospitalier - clone Géraldine -, caractérisé par une cassette de résistance à la méticilline SCCmec type I tronquée, un phénotype de résistance aux antibiotiques caractéristique (résistant à la pénicilline, à l’oxacilline, à la kanamycine, à la tobramycine, et de sensibilité intermédiaire à l’acide fusidique). Il possède le gène codant la toxine du choc toxique staphylococcique (TSST-1) et non pas la PVL. Ces souches de SARM sont responsables d’infections suppuratives et toxiniques [
20]. |
La sensibilité diminuée aux glycopeptides de S. aureus
Historique En mai 1996, la première documentation clinique d’infection par un S. aureus de sensibilité diminuée à la vancomycine a été rapportée chez un jeune japonais [
21]. Depuis, plusieurs cas semblables ont été documentés aux États-Unis, en France, ainsi qu’en Amérique du Sud, en Australie et au Royaume-Uni. Les souches de S. aureus ne répondant pas au traitement par la vancomycine ont une sensibilité diminuée à cet antibiotique. Ainsi, selon les recommandations du Comité de l’antibiogramme de la Société française de microbiologie (CA-SFM), une concentration minimale inhibitrice (CMI) de vancomycine comprise entre 4 et 16 μg/ml définit une souche de sensibilité diminuée ou intermédiaire. Ces micro-organismes sont dénommés VISA pour vancomycin-intermediate S. aureus, ou plus généralement GISA, glycopeptide-intermediate S. aureus, car ils sont également résistants à la teicoplanine. Mécanisme de résistance Le mécanisme de résistance lié à l’apparition de GISA n’est pas connu. Il s’agit d’un mode de résistance hétérogène, c’est-à-dire concernant une fraction seulement de la population bactérienne. Le phénotype de résistance s’amplifie avec les passages successifs sur des milieux contenant des concentrations croissantes en antibiotiques. Une série d’études récentes a permis d’évaluer la prévalence des souches dénommées hétéro-GISA au sein de souches de SARM, la prévalence la plus élevée observée étant de 18 % [
22], ceci en pratiquant une recherche systématique au moyen de tests particulièrement sensibles [
23]. Ces souches hébergent en effet une sous-population résistante aux glycopeptides. Parmi les isolats de GISA responsables d’infections cliniques, aucun ne dispose de gènes tels que vanA, vanB, vanC1, vanC2, vanC3 associés à la résistance aux glycopeptides chez les entérocoques [
33] (→). En effet, l’acquisition du gène vanA à l’origine des S. aureus résistants à la vancomycine (VRSA) est un phénomène très rare et, actuellement, seuls 9 isolats cliniques VRSA ont été décrits dans le monde. Ceci suggère que le transfert de résistance entre espèces, possible en laboratoire, n’est pas pour l’instant un phénomène dont le retentissement clinique est significatif. En revanche, selon les données cliniques dont nous disposons, l’exposition prolongée aux glycopeptides (vancomycine et téicoplanine) semble constituer l’élément essentiel de l’acquisition du phénotype de résistance GISA. Le mécanisme implique très probablement une altération de la liaison des molécules de l’antibiotique à la paroi de la bactérie et une perturbation de la synthèse de certains de ses composés [
24].
(→) Voir l’article de Vincent Cattoir et Roland Leclercq, page 936 de ce numéro
L’émergence de GISA étant secondaire à une exposition prolongée aux glycopeptides chez des patients souffrant d’infections à MRSA, les facteurs favorisants additionnels identifiés à ce jour comprennent le recours aux procédures invasives, en particulier cathéter intravasculaire et dialyse. La présence d’un corps étranger semble avoir joué un rôle important dans la genèse ou dans la persistance de l’infection dans la plupart des cas cliniques rapportés. Difficultés d’identification L’identification des GISA pose problème, car ces micro-organismes échappent généralement aux tests microbiologiques conventionnels effectués en routine au laboratoire [
25]. L’identification des GISA requiert la pratique de techniques de criblage et/ou des techniques d’études de populations bactériennes, les deux étant difficiles à adapter à la routine d’un laboratoire de microbiologie hospitalier. Importance clinique et épidémiologique L’apparition de GISA est inquiétante parce que l’arsenal thérapeutique à disposition est dramatiquement réduit. Il s’agit là d’un phénomène qui doit appeler à la vigilance des microbiologistes, mais aussi de cliniciens qui prennent en charge les patients infectés avec les souches GISA. En effet, au début des années 2000, plusieurs études conduites aux États-Unis et en Asie ont mis en évidence un glissement vers l’augmentation d’environ 1 mg/l des CMI de la vancomycine vis-à-vis de S. aureus et ceci pour des souches classifiées comme sensibles [
26,
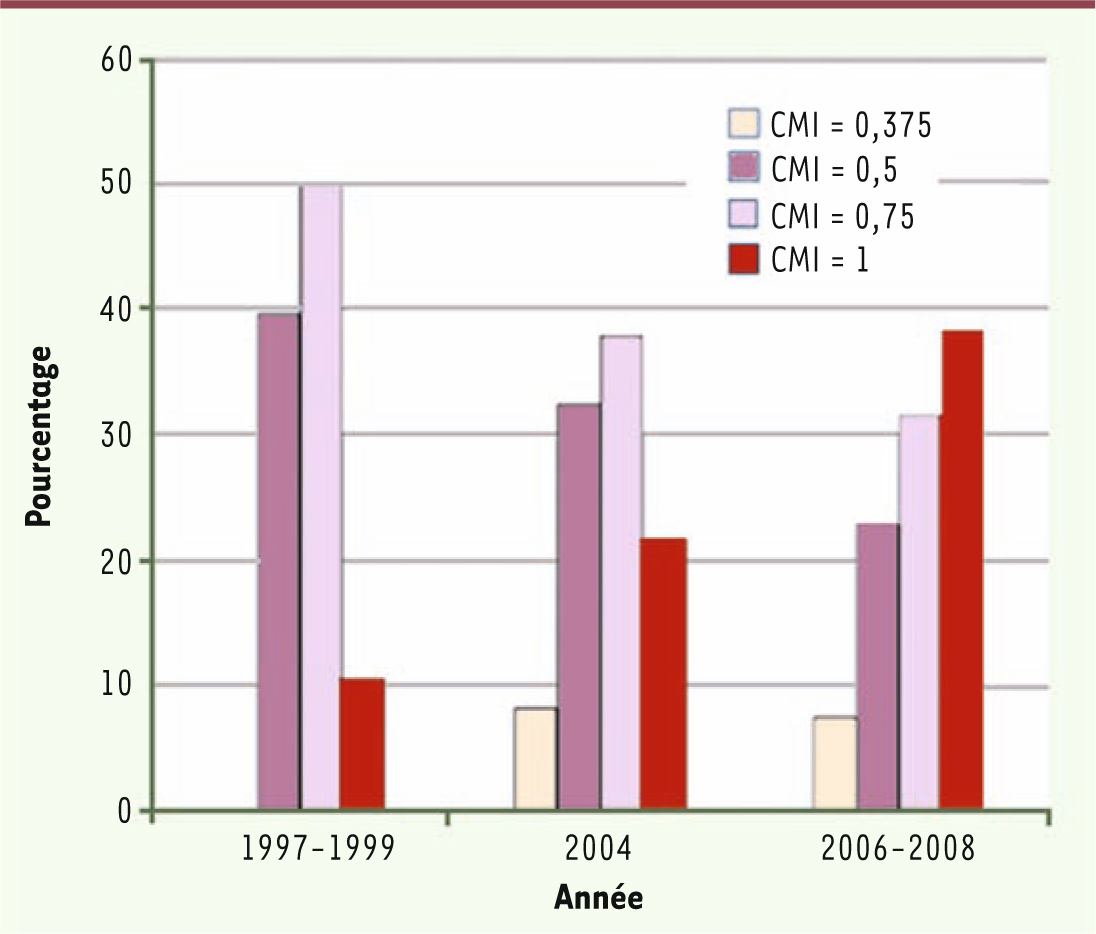
27]. Ce phénomène de dérive semble s’être développé dans un intervalle de temps relativement court, entre 2000 et 2005, et il porte le nom de vancomycin creep (Figure 2). Plusieurs explications ont été invoquées, la plus citée étant l’exposition prolongée des souches à des concentrations sub-inhibitrices de vancomycine en milieu hospitalier. Une autre explication consiste à supposer qu’il s’agirait d’un phénomène ancien, et que les observations récentes se seraient multipliées grâce aux nouvelles méthodes de criblage plus performantes que par le passé [
28].  | Figure 2
Évolution de la sensibilité aux glycopeptides des S. aureus résistants à la méticilline (adapté de Ho et al. [ 27]). |
Plusieurs étapes successives semblent participer au développement des infections à souches GISA : la colonisation, puis l’infection par SARM, l’exposition prolongée à un agent de la classe des glycopeptides, souvent en présence d’un corps étranger, soldé sur le plan clinique par une mauvaise réponse thérapeutique. Ainsi, Sakoulas et al. ont rapporté une efficacité thérapeutique satisfaisante de la vancomycine dans 56 % des cas de bactériémies à S. aureus ayant des CMI de la vancomycine inférieures à 0,5 mg/l et dans seulement 9,5 % des cas pour les souches ayant des CMI de 1 à 2 mg/l [
29]. Chez des patients hémodialysés septicémiques, un taux de mortalité plus élevé a été observé lorsque les souches de S. aureus avaient une CMI de la vancomycine égale à 2 mg/l par rapport aux patients infectés avec des souches ayant une CMI inférieure à 0,5 mg/l. Des souches avec des CMI supérieures à 2 mg/l, mais toujours dans la zone de sensibilité (selon la CA-SFM), ont été associées à des échecs thérapeutiques sans que les taux sériques aient été déterminés [
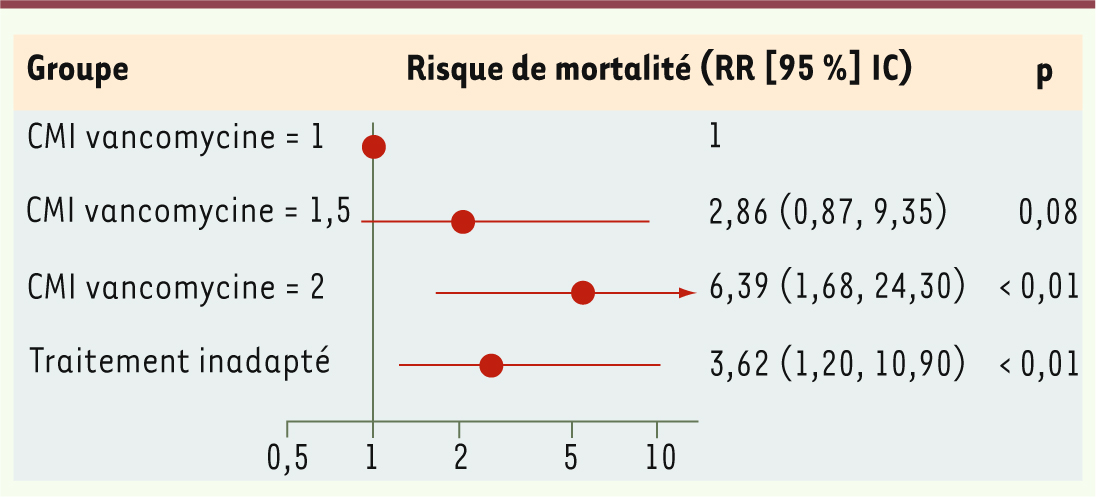
30]. Ces observations cliniques suggèrent une certaine perte d’efficacité thérapeutique de la vancomycine dès lors que les patients sont infectés avec des souches dont les CMI de vancomycine avoisinent la valeur de 2 mg/l (Figure 3)
 | Figure 3
Corrélation entre la CMI vancomycine et le pronostic des infections à S. aureus résistants à la méticilline. CMI en mg/l (adapté de Soriano et al. [
31]). |
Compte tenu de ces observations et dans le but de mieux encadrer la prescription des glycopeptides, l’autorité nord-américaine équivalente de la CA-SFM, le clinical and laboratory standards institute (CLSI) a abaissé en 2006 les bornes de sensibilité aux glycopeptides. Désormais, les souches sont considérées sensibles si la CMI de la vancomycine est inférieure ou égale à 2 mg/l au lieu de 4 mg/l. Dans le sillage du CLSI, l’autorité européenne European committee on antimicrobial susceptibility testing (EUCAST) a émis des recommandations similaires en 2009, ce qui laisse présager un changement imminent des recommandations de la CA-SFM. |
S. aureus est un pathogène dont le fort pouvoir d’adaptation permet la survie grâce à l’acquisition successive de gènes de résistance aux antibiotiques, de mécanismes de régulation de la croissance en présence d’antibiotiques et de facteurs de virulence particuliers. Certains de ces aspects semblent intriqués et leur complexité n’est, à ce jour, pas résolue. La vigilance des cliniciens et microbiologistes est requise afin de signaler l’émergence de phénomènes épidémiologiques nouveaux, ainsi que de veiller au respect des consignes de prévention et à l’utilisation judicieuse des antibiotiques, tant en milieu hospitalier que dans la communauté. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article. |
Footnotes |
1. Oliveira DC, Tomasz A, Lencastre H. Secrets of success of a human pathogen : molecular evolution of pandemic clones of meticillin-resistant Staphylococcus aureus. Lancet Infect Dis 2002 ; 2 : 180-9. 2. Kuroda M, Ohta T, Uchiyama I, et al. Whole genome sequencing of meticillin-resistant Staphylococcus aureus. Lancet 2001 ; 357 : 1225-40. 3. Bukharie HA, Abdelhadi MS, Saeed IA, et al. Emergence of methicillinresistant Staphylococcus aureus as a community pathogen. Diagn Microbiol Infect Dis 2001 ; 40 : 1-4. 4. Ito T, Okuma K, Ma XX, et al. Insights on antibiotic resistance of Staphylococcus aureus from its whole genome : genomic island SCC. Drug Resist Updat 2003 ; 6 : 41-52. 5. Hiramatsu K, Cui L, Kuroda M, et al. The emergence and evolution of methicillin-resistant Staphylococcus aureus. Trends Microbiol 2001 ; 9 : 486-93. 6. Katayama Y, Ito T, Hiramatsu K. A new class of genetic element, staphylococcus cassette chromosome mec, encodes methicillin resistance in Staphylococcus aureus. Antimicrob Agents Chemother 2000 ; 44 : 1549-55. 7. Ito T, Ma XX, Takeuchi F, et al. Novel type V staphylococcal cassette chromosome mec driven by a novel cassette chromosome recombinase, ccrC. Antimicrob Agents Chemother 2004 ; 48 : 2637-51. 8. Oliveira DC, de Lencastre H. Multiplex PCR strategy for rapid identification of structural types and variants of the mec element in methicillin-resistant Staphylococcus aureus. Antimicrob Agents Chemother 2002 ; 46 : 2155-61. 9. International working group on the classification of staphylococcal cassette chromosome elements (IWG-SCC). Classification of staphylococcal cassette chromosome mec (SCCmec) : guidelines for reporting novel SCCmec elements. Antimicrob Agents Chemother 2009 ; 53 : 4961-7. 10. Zhang K, McClure JA, Elsayed S, et al. Novel staphylococcal cassette chromosome mec type, tentatively designated type VIII, harboring class A mec and type 4 ccr gene complexes in a Canadian epidemic strain of methicillin-resistant Staphylococcus aureus. Antimicrob Agents Chemother 2009 ; 53 : 531-40. 11. Hiramatsu K, Katayama Y, Yuzawa H, et al. Molecular genetics of methicillin-resistant Staphylococcus aureus. Int J Med Microbiol 2002 ; 292 : 67-74. 12. Katayama Y, Takeuchi F, Ito T, et al. Identification in methicillin-susceptible Staphylococcus hominis of an active primordial mobile genetic element for the staphylococcal cassette chromosome mec of methicillin-resistant Staphylococcus aureus. J Bacteriol 2003 ; 185 : 2711-22. 13. Tsubakishita S, Kuwahara-Arai K, Baba T, et al. Staphylococcal cassette chromosome mec-like element in Macrococcus caseolyticus. Antimicrob Agents Chemother 2010 ; 54 : 1469-75. 14. Dufour P, Gillet Y, Bes M, et al. Community-acquired methicillin-resistant Staphylococcus aureus infections in France : emergence of a single clone that produces Panton-Valentine leukocidin. Clin Infect Dis 2002 ; 35 : 819-24. 15. Woodin AM. Purification of the two components of leucocidin from Staphylococcus aureus. Biochem J 1960 ; 75 : 158-65. 16. Konig B, Prevost G, Piemont Y, et al. Effects of Staphylococcus aureus leukocidins on inflammatory mediator release from human granulocytes. J Infect Dis 1995 ; 171 : 607-13. 17. Gillet Y, Issartel B, Vanhems P, et al. Association between Staphylococcus aureus strains carrying gene for Panton-Valentine leukocidin and highly lethal necrotising pneumonia in young immunocompetent patients. Lancet 2002 ; 359 : 753-9. 18. Vandenesch F, Naimi T, Enright MC, et al. Community-acquired methicillinresistant Staphylococcus aureus carrying Panton-Valentine leukocidin genes : worldwide emergence. Emerg Infect Dis 2003 ; 9 : 978-84. 19. Dauwalder O, Lina G, Durand G, et al. Epidemiology of invasive methicillinresistant Staphylococcus aureus clones collected in France in 2006 and 2007. J Clin Microbiol 2008 ; 46 : 3454-8. 20. Durand G, Bes M, Meugnier H, et al. Detection of new methicillin-resistant Staphylococcus aureus clones containing the toxic shock syndrome toxin 1 gene responsible for hospital and community-acquired infections in France. J Clin Microbiol 2006 ; 44 : 847-53. 21. Hiramatsu K, Aritaka N, Hanaki H, et al. Dissemination in Japanese hospitals of strains of Staphylococcus aureus heterogeneously resistant to vancomycin. Lancet 1997 ; 350 : 1670-3. 22. Sancak B, Ercis S, Menemenlioglu D, et al. Methicillin-resistant Staphylococcus aureus heterogeneously resistant to vancomycin in a Turkish university hospital. J Antimicrob Chemother 2005 ; 56 : 519-23. 23. Wootton M, Howe RA, Hillman R, et al. A modified population analysis profile (PAP) method to detect hetero-resistance to vancomycin in Staphylococcus aureus in a UK hospital. J Antimicrob Chemother 2001 ; 47 : 399-403. 24. Cui L, Iwamoto A, Lian JQ, et al. Novel mechanism of antibiotic resistance originating in vancomycin-intermediate Staphylococcus aureus. Antimicrob Agents Chemother 2006 ; 50 : 428-38. 25. Swenson JM, Anderson KF, Lonsway DR, et al. Accuracy of commercial and reference susceptibility testing methods for detecting vancomycin-intermediate Staphylococcus aureus. J Clin Microbiol 2009 ; 47 : 2013-7. 26. Steinkraus G, White R, Friedrich L. Vancomycin MIC creep in non-vancomycin-intermediate Staphylococcus aureus (VISA), vancomycin-susceptible clinical methicillin-resistant S. aureus (MRSA) blood isolates from 2001-05. J Antimicrob Chemother 2007 ; 60 : 788-94. 27. Ho PL, Lo PY, Chow KH, et al. Vancomycin MIC creep in MRSA isolates from 1997 to 2008 in a healthcare region in Hong Kong. J Infect 2010 ; 60 : 140-5. 28. Gould IM. Clinical relevance of increasing glycopeptide MICs against Staphylococcus aureus. Int J Antimicrob Agents 2008 ; 31 (suppl 2) : 1-9. 29. Sakoulas G, Moise-Broder PA, Schentag J, et al. Relationship of MIC and bactericidal activity to efficacy of vancomycin for treatment of methicillin-resistant Staphylococcus aureus bacteremia. J Clin Microbiol 2004 ; 42 : 2398-402. 30. Maclayton DO, Suda KJ, Coval KA, et al. Case-control study of the relationship between MRSA bacteremia with a vancomycin MIC of 2 microg/mL and risk factors, costs, and outcomes in inpatients undergoing hemodialysis. Clin Ther 2006 ; 28 : 1208-16. 31. Soriano A, Marco F, Martinez JA, et al. Influence of vancomycin minimum inhibitory concentration on the treatment of methicillin-resistant Staphylococcus aureus bacteremia. Clin Infect Dis 2008 ; 46 : 193-200. 32. Vandenesh F, Lina G, Gillet Y et al. Chronique d’une controverse sur une bactérie équipée pour tuer. Med Sci (Paris) 2009 ; 25 : 984-6. 33. Cattoir V, Leclercq R. Les entérocoques résistants aux glycopeptides. Med Sci (Paris) 2010 ; 26 : 936-42. |