| |
| Med Sci (Paris). 2010 December; 26(12): 1025–1027. Published online 2010 December 15. doi: 10.1051/medsci/201026121025.Quand les éléments génétiques mobiles bondissent entre espèces animales Clément Gilbert,1 Sarah Schaack,1 and Cédric Feschotte1* 1University of Texas, Arlington, 760 10, Texas, États-Unis MeSH keywords: Animaux, Éléments transposables d'ADN, génétique, physiologie, Évolution moléculaire, Transfert horizontal de gène, Génome, Interactions hôte-parasite, Humains, Opossum, Rhodnius, Rodentia, Saimiri, Spécificité d'espèce, Trypanosoma cruzi |
En 1959, une équipe de microbiologistes japonais montrait pour la première fois que la faculté de résister a certains antibiotiques pouvait être transférée d’une espèce de bactérie à une autre lorsque celles-ci étaient cultivées dans le même milieu de culture [
1]. Cinquante ans plus tard, après avoir séquencé et analysé plus de 2 000 génomes bactériens, nous savons maintenant que la plupart des bactéries sont capables d’échanger leur matériel génétique (dont les gènes de résistance aux antibiotiques) de manière horizontale, entre individus d’une même espèce et entre espèces plus ou moins éloignées, sans avoir recours à la reproduction sexuée [
2]. Il est aussi désormais admis que les transferts horizontaux de gènes ont un impact évolutif considérable sur l’évolution des procaryotes, tant ils sont fréquents et communs chez ces organismes. L’importance de ce phénomène est moins claire chez les eucaryotes. Bien qu’un nombre grandissant de transferts horizontaux de gènes de procaryotes à eucaryotes aient été décrits, les cas de transferts entre eucaryotes, et notamment entre eucaryotes multicellulaires, restent rares dans la littérature [
3]. L’influence exercée par le processus de transfert horizontal sur l’évolution des génomes eucaryotes serait-elle donc négligeable ? 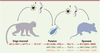 | Figure 1
Transferts horizontaux d’éléments transposables entre des mammifères et leurs parasites. Dans une étude récente [
8], nous montrons qu’une punaise hématophage (Rhodnius prolixus) vectrice du trypanosome responsable de la maladie de Chagas (Trypanosoma cruzi) partage une famille d’éléments transposables (OCl) avec le singe écureuil (Saimiriboliviensis) et trois (OC1, hAT1 et SPIN) avec l’opossum (un marsupial) (Monodelphis domestica), deux de ses hôtes préférés en Amérique du Sud. Malgré les 900 millions d’années séparant les mammifères des insectes, les séquences consensus des transposons de la punaise sont extrêmement similaires à celles du singe et du marsupial ( T). La région codante de ces transposons n’a pas évolué sous contrainte fonctionnelle depuis leur insertion dans les trois génomes. Ainsi l’extrême conservation de ces transposons n’est pas le produit d’une évolution verticale sous un régime de sélection purifiante. Cela suggère donc que les transposons ont été transférés horizontalement entre la punaise et les deux mammifères. Ces transferts ont pu se produire lors des repas sanguins de la punaise, soit de manière directe, soit par l’intermédiaire de virus ou des trypanosomes transmis par la punaise. Les nombres de copies d’éléments transposables (*) et de nucléotides (** en kilobases) générés pas ces transferts horizontaux laissent penser que ce processus a probablement eu un impact substantiel sur les trajectoires évolutives des différentes espèces. |
|
Influence des éléments transposables sur l’évolution des génomes Ce paradigme prévaut probablement pour une majorité de biologistes. Il s’explique en partie par l’importance - peut-être disproportionnée - qui a jusqu’à présent été attribuée aux transferts de gènes. Or, contrairement à ce que l’on observe chez les procaryotes, les gènes (séquences codantes) représentent généralement une fraction mineure et relativement statique des génomes eucaryotes (< 2 % du génome humain). Chez la plupart des eucaryotes multicellulaires, le génome nucléaire est composé principalement d’ADN non codant, qui dérive en grande partie de l’activité réplicative d’éléments génétiques mobiles ou éléments transposables (ET). Les ET sont des segments d’ADN ayant la capacité de se déplacer d’un chromosome à l’autre et de se multiplier via divers mécanismes. Leur amplification au sein des génomes est parfois si prolifique qu’elle a pour résultat la formation d’immenses familles de séquences répétées en centaines de milliers de copies et dispersées sur tous les chromosomes. Par exemple, ces éléments et leurs reliques moléculaires occupent la moitié du génome humain et plus des trois quarts du génome du maïs [
4,
5]. Bien que les ET puissent être simplement perçus comme des parasites génomiques - n’ayant a priori aucune utilité pour leur hôte - ou comme des agents mutagènes délétères, il est indéniable que leur mobilité et leur multiplicité sont à la base de nombreux processus de plasticité génomique. En ce sens, les ET représentent une abondante source de variation et d’innovation génétiques. Les exemples illustrant les multiples mécanismes par lesquels les ET ont contribué à la création de nouveautés génomiques sont maintenant nombreux, que ce soit de manière indirecte, via leur activité recombinogénique (source de réarrangements chromosomiques, de réorganisation d’exons), ou encore de manière directe, via le recyclage (ou « domestication ») de leur séquence pour former de nouvelles régions régulatrices ou de nouveaux gènes [
6]. Considérant la profusion des ET et l’influence qu’ils exercent sur l’évolution des génomes eucaryotes, il semble qu’une évaluation complète et pertinente de l’impact des transferts horizontaux chez ces organismes se doit de considérer les transferts d’ET, en plus des transferts de gènes. |
Transferts horizontaux d’éléments transposables chez les eucaryotes Dans une synthèse récente de la littérature, nous avons comptabilisé plus de 200 cas robustes de transferts horizontaux d’ET décrits chez les eucaryotes multicellulaires [
7]. La liste de génomes complets disponibles (<; 1 000) est encore loin de refléter la diversité des eucaryotes (> 1,5 million d’espèces), mais déjà certaines tendances générales émergent. La première est que tous les types majeurs d’ET peuvent être sujets au transfert horizontal. La seconde est que l’on observe un accroissement constant du nombre de cas de transferts d’ET rapportés chez les opistokontes1, (métazoaires et champignons) et les plantes, les deux groupes d’eucaryotes multicellulaires représentant la part la plus importante des génomes complètement séquencés. Enfin, à plusieurs reprises, de tels transferts se sont produits sur des distances phylogénétiques considérables, avec par exemple 12 transferts impliquant au moins deux taxons qui appartiennent à des phyla animaux séparés par plus de 500 millions d’années (par exemple entre insectes et mammifères [8]). Le nombre et l’étendue de ces transferts sont d’autant plus remarquables que jusqu’à présent, quasiment aucune étude n’a cherché à caractériser ce phénomène de manière systématique. On peut noter que certaines lignées de mammifères (chauves-souris de la famille des vespertilionidés) ont été infiltrées par plusieurs familles d’ET dont l’activité a produit au total plus de 21 millions de paires de bases. De plus, un des ET transférés horizontalement dans la lignée des rongeurs muridés a donné naissance à un nouveau gène codant une protéine dont la fonction cellulaire n’a pas encore été caractérisée mais qui a clairement évolué sous un régime de pression sélective [
9]. L’impact évolutif de ces transferts est donc indéniable, mais il reste à évaluer de manière plus fine et plus quantitative l’ampleur du phénomène. |
Les relations hôtes-parasites à l’origine des transferts horizontaux ? Étant donné leur inhérente mobilité, il n’est peut être pas surprenant de constater que, chez les eucaryotes, les ET semblent être transférés horizontalement plus souvent que les gènes. Cependant, la faculté de transposition n’explique qu’une partie du mécanisme moléculaire par lequel ces segments d’ADN peuvent passer d’une espèce à l’autre. Comment ces éléments peuvent-ils traverser les nombreuses barrières cellulaires et accéder aux cellules reproductrices d’une autre espèce ? Cela reste complètement mystérieux. De nombreuses hypothèses impliquant divers vecteurs bactériens et/ou viraux ont été proposées, souvent sur la base d’expériences conduites en laboratoire, sans qu’aucune n’ait été entièrement validée dans la nature. Récemment, nous avons découvert que les génomes de dix vertébrés, dont huit mammifères, un reptile et un amphibien, contiennent entre une et quatre familles d’ET également présentes dans le génome d’une punaise hématophage (Rhodnius prolixus), un insecte vecteur de trypanosomes responsables de la maladie de Chagas [8]. Les ET de la punaise sont quasiment identiques à ceux des vertébrés et forment un groupe phylogénétique avec ceux du singe écureuil et de l’opossum (un marsupial), deux de ses hôtes préférés en Amérique du Sud (Figure 1). Ces résultats suggèrent que ces ET ont été transférés directement entre la punaise et ses deux hôtes mammifères, sans toutefois révéler la direction de ces transferts. La transmission des trypanosomes entre la punaise et l’homme se produit durant les repas sanguins de la punaise, lorsque la salive de celle-ci entre en contact avec le sang de l’hôte. Une étude récente suggère même que dans certains cas de trypanosomiase chronique, l ‘ADN mitochondrial du trypanosome, dont le cycle de développement se déroule en partie à l’intérieur des cellules humaines, peut être transféré dans le génome de l’homme [
10]. Les ET auraient pu emprunter la même route que les trypanosomes, ou même être véhiculés par l’intermédiaire de ces derniers. Nous n’avons pas trouvé de trace des quatre familles d’ET en question dans le génome complet de Trypanosoma cruzi, un des agents de la maladie de Chagas, mais nous continuons de chercher activement ces éléments dans les génomes d’autres trypanosomes. Il est cependant possible que les trypanosomes aient véhiculé les ET entre la punaise et ses hôtes mammaliens sans que ces ET ne se soient intégrés de manière stable et soient donc détectables dans le génome de ces microparasites. Ici encore le mécanisme moléculaire précis à l’origine de ces transferts ne peut pas être formellement démontré. Néanmoins, c’est à notre connaissance la première fois que des transferts horizontaux d’ET impliquant des vertébrés et un de leurs parasites invertébrés ont été détectés. Il s’agit donc de l’évidence la plus probante supportant l’hypothèse proposée par Margaret Kidwell en 1991 selon laquelle les relations hôtes-parasites pourraient faciliter les transferts horizontaux [
11]. Des datations basées sur l’horloge moléculaire des mammifères nous ont permis de montrer que la plupart de ces transferts horizontaux se sont produits entre 15 et 50 millions d’années avant l’ère actuelle. Afin de caractériser de manière plus directe un vecteur et un mécanisme de transfert horizontal, il serait souhaitable d’identifier un transfert plus récent. Si cette possibilité pouvait paraître irréaliste il y a seulement quelques années, la réduction continue des coûts du séquençage génomique devrait nous permettre de rapidement détecter un tel événement et d’évaluer plus précisément l’impact des transferts horizontaux sur l’évolution des eucaryotes. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Footnotes |
1. Akiba TK, Koyama Y, Ishiki S, et al. On the mechanism of the development of multiple drugresistant clones of Shigella. Japan J Microbiol 1960 ; 4 : 219-27. 2. Frost LS, Leplae R, Summers AO, et al. Mobile genetic elements: the agents of open source evolution. Nat Rev Microbiol 2005 ; 3 : 722-32. 3. Keeling PJ, Palmer JD. Horizontal gene transfer in eukaryotic evolution. Nat Rev Genet 2008 ; 9 : 605-18. 4. Lander ES, Linton LM, Birren B, et al. Initial sequencing and analysis of the human genome. Nature 2001 ; 409 : 860-921. 5. Schnable PS, Ware D, Fulton RS, et al. The B73 maize genome: complexity, diversity, and dynamics. Science 2009; 326 : 1112-5. 6. Feschotte C, Pritham EJ. DNA transposons and the evolution of eukaryotic genomes. Annu Rev Genet 2007 ; 41 : 331-68. 7. Schaack S, Gilbert C, Feschotte C. Promiscous DNA: horizontal transfer of transposable elements and why it matters for eukaryotic evolution. Trends Ecol Evol 2010; 25 : 537-46. 8. Gilbert C, Schaack S, Pace JK, et al. A role for hostparasite interactions in the horizontal transfer of DNA transposons across animal phyla. Nature 2010 ; 464 : 1347-50. 9. Pace JK, Gilbert C, Clark MS, et al. Repeated horizontal transfer of a DNA transposon in mammals and other tetrapods. Proc Natl Acad Sci USA 2008 ; 105 : 17023-8. 10. Hecht MM, Nitz N, Araujo PF, et al. Inheritance of DNA transferred from American trypanosomes to human hosts. PLoS ONE 2010 ; 5 : e9181. 11. Houck MA, Clark JB, Peterson KR, et al. Possible horizontal transfer of Drosophila genes by the mite Proctolaelaps regalis. Science 1991 ; 6 : 1125-9. |


