| |
| Med Sci (Paris). 2011 January; 27(1): 63–69. Published online 2011 February 10. doi: 10.1051/medsci/201127163.Déterminants de la carte génétique Le rôle-clé de la spécification des sites de recombinaison méiotique Corinne Grey,1 Vérane Sommermeyer,2 Valérie Borde,2* and Bernard de Massy1** 1Institut de génétique humaine, UPR1142/CNRS, 141, rue de la Cardonille, 34396 Montpellier Cedex 5, France 2Dynamique des chromosomes et recombinaison, Institut Curie/Centre de recherche, UMR 218, Pavillon Pasteur, 26, rue d’Ulm, 75248 Paris Cedex 05, France |
La reproduction sexuée implique la différenciation de cellules diploïdes en cellules reproductrices haploïdes qui, après fécondation, forment des cellules diploïdes portant chaque chromosome en deux exemplaires, dits homologues, un d’origine maternelle et un d’origine paternelle. Le passage de l’état diploïde à l’état haploïde est réalisé lors de la méiose par un cycle cellulaire modifié constitué d’une phase de réplication suivie de deux divisions successives [
1]. Avant la première division, des échanges entre chromosomes homologues se produisent, par un processus appelé recombinaison méiotique (
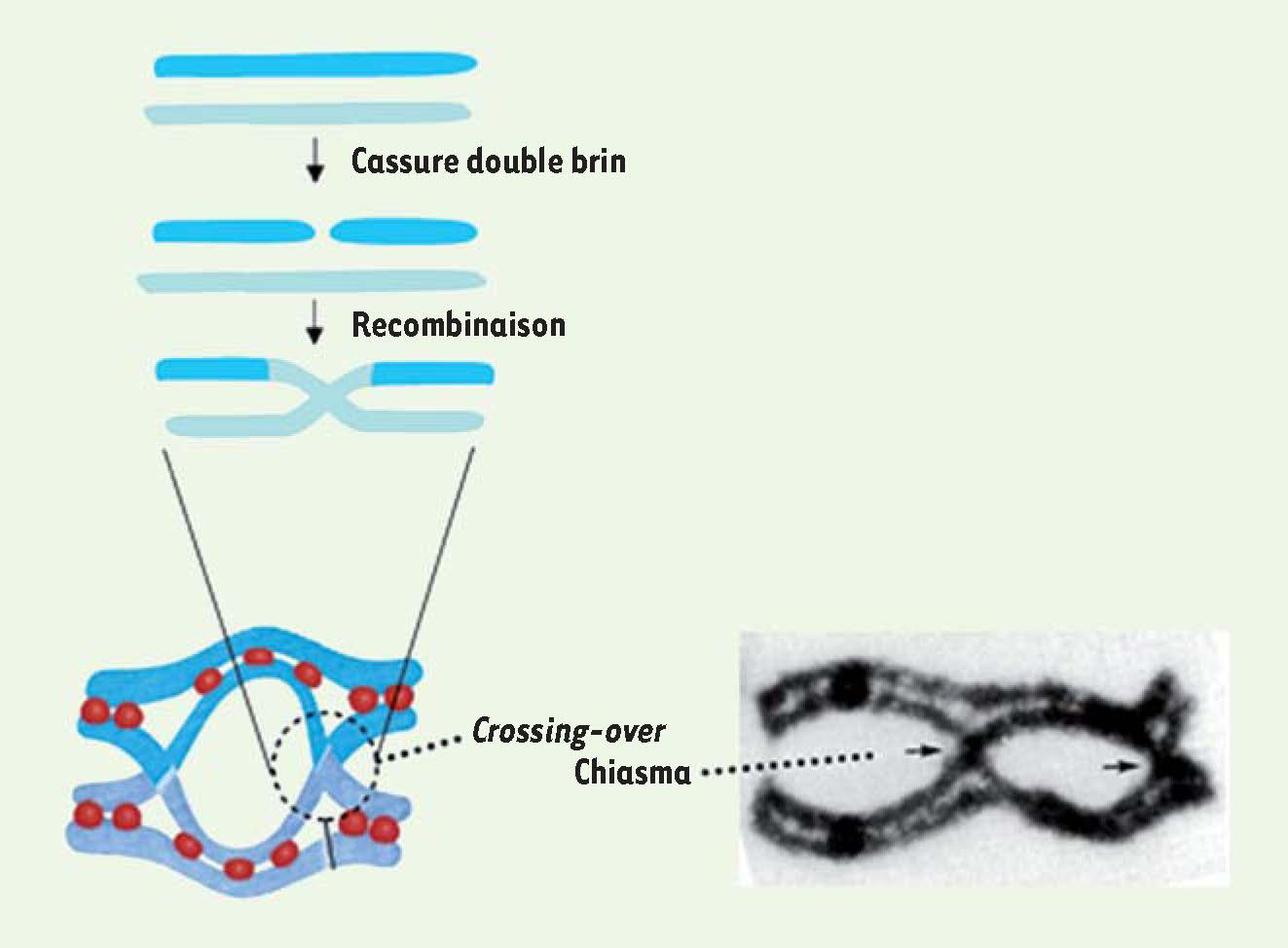
Figure 1
). Ces échanges, ou crossing-over, sont nécessaires pour la ségrégation des chromosomes en première division de méiose et contribuent à augmenter la diversité génétique en créant de nouvelles combinaisons d’allèles. La recombinaison méiotique est ainsi à la fois un mécanisme moléculaire qui assure la formation des gamètes avec un contenu chromosomique normal et donc la fertilité, et un mécanisme qui contribue à l’évolution des génomes. 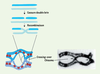 | Figure 1
Le crossing-over. Un événement de crossing-over résulte de la formation d’une cassure double brin (CDB) sur une des chromatides d’un des génomes parentaux (bleu clair et bleu foncé) d’une cellule diploïde en prophase de méiose, qui est réparée par recombinaison avec le chromosome homologue. La recombinaison conduit soit à un échange réciproque ou crossing-over qui crée une connexion entre les homologues dont les chromatides sœurs sont associées par les cohésines (rouge), soit à une réparation sans crossing-over (non montré). Lors de cette réparation, une petite région (quelques centaines de paires de bases) autour de la CDB est remplacée par la séquence de la chromatide homologue. La manifestation cytologique du crossing-over, visible en fin de prophase, est le chiasma. |
Les premières mises en évidence de crossing-over datent du début du xx
e siècle alors que la théorie chromosomique des gènes n’était encore qu’une hypothèse. La première carte génétique a été établie par A.H. Sturtevant dans le laboratoire de T. Morgan en 1913 [
2]. Ces travaux ont permis de proposer que les gènes étaient positionnés le long des chromosomes et pouvaient être réassociés par des événements d’échanges se produisant à des fréquences variables selon leur distance et selon d’autres facteurs non déterminés [
3]. La théorie chromosomique des gènes a été validée par la suite, ainsi que la correspondance entre les réassociations de gènes et les échanges chromosomiques [
4]. L’étude des événements de recombinaison méiotique s’est développée chez de nombreux organismes par des approches diverses qui ont révélé une forte conservation évolutive des mécanismes impliqués : les crossing-over résultent d’une induction programmée de cassures double brin de l’ADN (CDB) catalysées par la protéine Spo11 [
5], suivie de leur réparation par recombinaison avec le chromosome homologue (
Figure 1
). La localisation des événements de recombinaison peut être analysée soit par la cartographie moléculaire des extrémités des CDB (chez les levures), soit par la cartographie des crossing-over (chez les mammifères notamment). Une des questions majeures restée sans réponse était de comprendre ces cartes, caractérisées par une localisation non aléatoire le long des chromosomes des événements de recombinaison préférentiellement situés dans de petites régions appelées points chauds [
6]. Nous présentons ici les résultats récents obtenus chez les levures Saccharomyces cerevisiae et Schizosaccharomyces pombe et chez les mammifères, qui ont permis d’identifier des éléments de contrôle importants dans la localisation des points chauds et donc dans la distribution des crossing-over le long des chromosomes. |
Les points chauds de recombinaison chez les levures Chez S. cerevisiae, les CDB sont situées dans leur très grande majorité dans des régions promotrices [
7]. Chez S. pombe, les CDB sont préférentiellement localisées dans de longues régions intergéniques, très espacées les unes des autres, sans corrélation avec la présence d’un promoteur [
8]. Chez ces deux espèces il n’a pas été trouvé de séquence consensus spécifique aux sites de CDB. Ouverture de la chromatine Tous les sites de CDB chez S. cerevisiae sont situés dans des zones dites ouvertes de la chromatine, caractérisées par l’hypersensibilité à la DNaseI ou à la nucléase micrococcale [
9,
10]. À quelques locus, on observe une augmentation de l’accessibilité de la chromatine au niveau du site de CDB, juste avant la formation de la cassure [ 9]. De même, chez S. pombe, une structure ouverte de la chromatine est nécessaire pour la formation des CDB. Aux points chauds ade6-M26 et tdh1, on observe un remodelage de la chromatine spécifique aux cellules en méiose et nécessaire à la fixation des facteurs impliqués dans la formation des CDB [
11]. Au niveau de deux autres points chauds, la chromatine est constitutivement ouverte [ 11,
12]. Facteurs de transcription et de remodelage de la chromatine Le fait que les CDB aient lieu plus particulièrement dans les promoteurs des gènes chez S. cerevisiae pose immédiatement la question du rôle des facteurs de transcription. Un lien direct entre l’activité transcriptionnelle, mesurée par la détection d’ARN messagers stables, et la présence d’une CDB dans le promoteur n’a pas été observé, que ce soit dans les études à l’échelle du génome [
13] ou à des points chauds définis [
14]. Néanmoins, l’activité du point chaud situé dans la région promotrice du gène HIS4 de S. cerevisiae requiert la fixation de quatre facteurs de transcription (Rap1, Bas1, Bas2, Gcn4). Ceux-ci pourraient agir directement ou indirectement en recrutant des remodeleurs de la chromatine tels que le complexe SAGA ( Spt-Ada-Gcn5-acetyltransferase complex) ou Swi/Snif. Par exemple, la mutation de Bas1 diminue l’activité de 85 points chauds, sans lien évident avec des modifications du niveau de transcription des gènes adjacents [
15]. Nombre des cibles de Gcn4, dont la fixation dépend de l’état nutritionnel des cellules, figurent parmi les points chauds les plus actifs du génome [ 13]. Ceci suggère que les taux de recombinaison pourraient être en partie influencés par l’état métabolique et l’environnement de la cellule [
16]. Chez S. pombe, le point chaud ade6-M26 a été découvert suite à l’apparition d’une mutation à proximité du gène ade6. Cette mutation crée un site ATGACGTCA reconnu par le facteur de transcription Atf1-Pcr1. La fixation de ce dernier recrute plusieurs facteurs de remodelage de la chromatine comme Snf22, Gcn5 et Ada2 [11, 12,
17]. Plusieurs des sites naturels reconnus par ce facteur sont également des points chauds [
18]. Toutefois, de nombreux points chauds identifiés dans le génome ne contiennent pas le site de fixation d’Atf1-Pcr1 [8]. L’identification d’autres motifs reconnus par d’autres facteurs de transcription et dont la présence stimule l’activité de recombinaison suggère que le mécanisme moléculaire mis en évidence au locus ade6-M26 pourrait s’appliquer à une grande partie du génome de S. pombe et impliquer une ou plusieurs familles de facteurs de transcription [
19]. Modifications des histones Un autre aspect de la fonction de la chromatine dans la formation des CDB méiotiques est l’ensemble des modifications post-traductionnelles des histones. Parmi celles-ci deux types distincts ont été décrits : celles qui sont associées à un état de la chromatine « ouvert » (méthylation d’H3K4, acétylation d’H3 et H4) et celles qui sont associées à un état de la chromatine « fermé » (méthylation d’H3K9). Ces modifications sont souvent impliquées dans le recrutement de facteurs de remodelage de la chromatine ou de facteurs de transcription [
20]. Chez S. cerevisiae, la recombinaison à HIS4 est plus élevée en absence de deux facteurs impliqués dans la déacétylation des histones (Rpd3, une désacétylase de l’histone H4 et Set2, une histone méthyltransférase capable de recruter Rpd3 sur la chromatine) [
21]. De même, la délétion du gène SIR2, codant pour une histone désacétylase spécifique d’H4K16, entraîne une augmentation des CDB dans les régions subtélomériques et les gènes ribosomiques, qui sont normalement les cibles de Sir2 [
22]. La stimulation de la recombinaison chez ce mutant est probablement due à l’augmentation locale de l’acétylation des histones, qui rend la chromatine plus accessible. Chez S. pombe, le rôle des membres des complexes de remodelage de la chromatine SAGA et Mst2 dans l’activité du point chaud M26 met aussi en jeu leur fonction d’acétylation des histones [12, 17]. Plusieurs études convergent vers un rôle important de la méthylation de la lysine 4 de l’histone H3 (H3K4) dans la formation des CDB méiotiques chez S. cerevisiae. Cette modification est associée à la transcription et se situe en général sur le premier ou les deux premiers nucléosomes des gènes activement transcrits. Elle est assurée par une histone méthyltransférase, Set1 chez S. cerevisiae, recrutée sur la chromatine par l’ARN polymérase II en début d’élongation. Cette marque peut être lue par des protéines dites effectrices [
23] qui participent au maintien de la structure ouverte de la chromatine pendant la transcription. Dans des souches set1∆, dont la triméthylation d’H3K4 est absente, on observe une réduction des CDB méiotiques [
24]. Cette analyse a été récemment étendue à l’ensemble du génome [
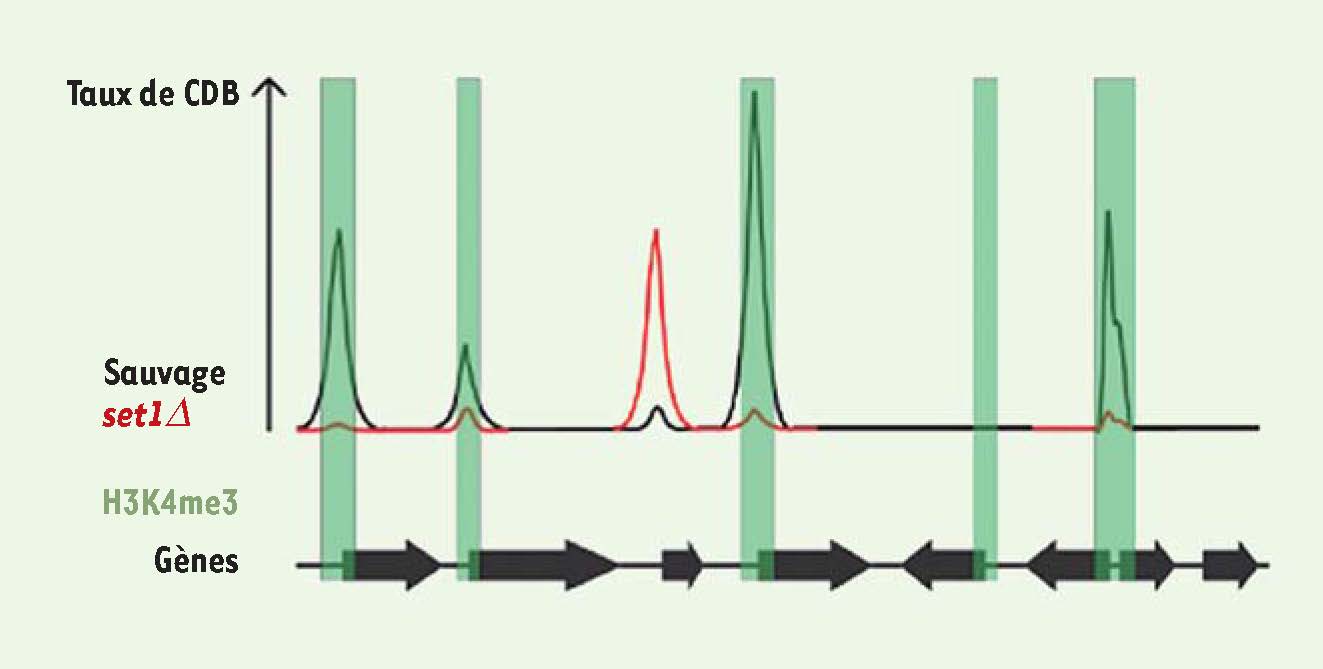
25] : par immunoprécipitation de la chromatine d’une souche sauvage et révélation sur puce à ADN (ChIP-chip, chromatin immunoprecipitation on chip), un enrichissement de la triméthylation d’H3K4 est observé près des sites de CDB, indépendamment de la formation des CDB par Spo11, même avant l’entrée en méiose. De plus, dans le mutant set1∆, la formation des CDB est réduite dans 84 % des points chauds par rapport à une souche sauvage (
Figure 2
). On observe en revanche dans le mutant set1∆ de nouveaux points chauds à des sites très faiblement triméthylés dans la souche sauvage, ce qui suggère l’existence (ou l’activation) d’un mécanisme indépendant de Set1 (
Figure 2
). Il est à noter que S. pombe ne semble pas utiliser la triméthylation d’H3K4 pour la formation des CDB puisqu’un mutant set1∆ n’a pas de défaut de sporulation [
26]. 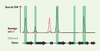 | Figure 2
Association des points chauds de cassure double brin (CDB) avec la triméthylation de la lysine 4 de l’histone H3 chez la levure S. cerevisiae. La triméthylation de la lysine 4 de l’histone H3 (H3K4me3, rectangles verts) est associée aux promoteurs des gènes (flèches noires) transcriptionnellement actifs. Chez une levure sauvage, les points chauds de CDB (pics noirs) sont associés à cette triméthylation. Dans une souche invalidée pour Set1 (set1Δ), la seule H3K4 méthyltransférase, la fréquence de CDB est diminuée dans la majorité des points chauds. Cependant, certaines régions, froides pour les CDB et très faiblement triméthylées chez une souche sauvage, se révèlent devenir des points chauds chez le mutant set1Δ (pics rouges). |
|
Les points chauds de recombinaison chez les mammifères Au cours des dernières années, un grand effort a été déployé pour établir chez les mammifères des cartes génétiques précises. Ces études ont principalement consisté à effectuer des génotypages extensifs d’un grand nombre de familles d’origines diverses afin de cartographier les crossing-over le long des bras chromosomiques. Chez l’homme et la souris, ces cartes montrent que les événements de recombinaison ne sont pas répartis de manière homogène [
27,
28]. En outre, l’analyse de la diversité génétique dans des populations humaines (projet HapMap [
29,
45]) a permis d’estimer le taux de crossing-over à haute résolution dans l’ensemble du génome, et donc de connaître la position des points chauds de recombinaison. Chez l’homme, environ vingt-cinq mille points chauds ont ainsi été identifiés [
30]. Ceux-ci ont une longueur d’environ deux kilobases et sont séparés par des régions de quelques dizaines de kilobases en moyenne (
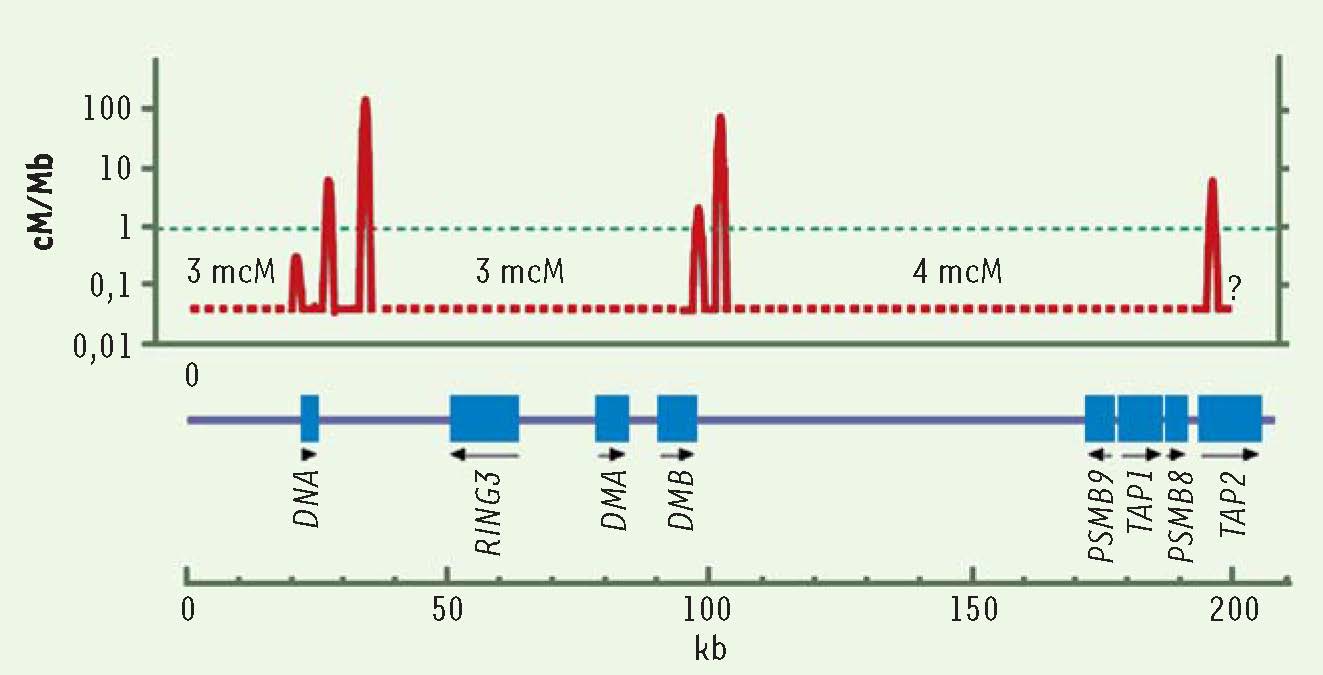
Figure 3
). L’analyse de leur localisation dans les génomes de mammifères n’a toutefois pas permis de mettre en évidence de relation forte avec des éléments fonctionnels du génome, mis à part une tendance à être situés en dehors des gènes, en opposition apparente avec les données obtenues chez S. cerevisiae.  | Figure 3
Points chauds de crossing-over chez l’homme. Six points chauds de crossing-over d’activités variables (en cM/Mb) ont été détectés dans une région de 200 kb sur le chromosome 6. Ils sont localisés dans des intervalles de 1 à 2 kb. Le taux de crossing-over moyen dans le génome est de 1 cM/Mb (ligne pointillée verte) [
43]. |
En revanche, il a pu être montré qu’une proportion importante (41 %) des points chauds identifiés chez l’homme est associée à un motif dégénéré de séquence d’ADN (le 13-mère CCNCCNTNNCCNC) [
31]. De manière intéressante, la localisation des points chauds dans les génomes de mammifères peut varier de façon importante entre des espèces proches ou entre des individus d’une même espèce [
32–
34]. L’homme et le chimpanzé, par exemple, n’utilisent pas les mêmes sites de recombinaison [
35,
36] et de manière surprenante, le point chaud humain MSTM1a n’est actif que chez trois hommes sur vingt-six analysés, malgré le fait que leurs séquences d’ADN dans et autour du point chaud soient identiques [34]. Ces observations ont contribué à poser la question de l’état chromatinien des points chauds et à émettre l’hypothèse de la présence d’un (ou plusieurs) facteur polymorphe qui se situerait en dehors des points chauds et qui influerait en trans sur leurs activités et/ou localisations. |
La localisation des points chauds des mammifères est déterminée par la protéine PRDM9 À la suite de plusieurs observations indépendantes, la protéine PRDM9 (PR domain zinc finger protein 9) a semblé pouvoir être un facteur intéressant influant en trans la localisation des points chauds [
44]. Prdm9 est exprimé spécifiquement lors de la prophase I en méiose et code pour une histone méthyltransférase qui catalyse, grâce à son domaine PR/SET, la triméthylation de la lysine 4 de l’histone H3 (H3K4me3). De façon intéressante, il a récemment été montré chez la souris que les points chauds Psmb9 et Hlx1, localisés dans des régions intergéniques, sont fortement enrichis en cette marque quand ils sont actifs [
37]. D’autre part, Prdm9 murin est codé dans une région génomique qui influe sur la localisation des points chauds le long des chromosomes [
38] et est essentielle pour le déroulement de la méiose : les souris mutantes pour le gène Prdm9 sont stériles et présentent une altération de la recombinaison méiotique avec un arrêt de la spermatogenèse et de l’ovogenèse [
39]. PRDM9 est une protéine à doigts de zinc, ce qui lui permet de reconnaître des motifs spécifiques d’ADN et de s’y fixer. En effet l’identité des acides aminés présents à trois positions importantes sur chaque doigt de zinc joue un rôle majeur pour déterminer les séquences d’ADN reconnues. De façon remarquable, dans le cas de PRDM9, ces trois acides aminés montrent un taux de polymorphisme élevé entre sous-espèces (chez la souris) et individus (chez l’homme), tandis que le reste de la séquence protéique est hautement conservé [
40,
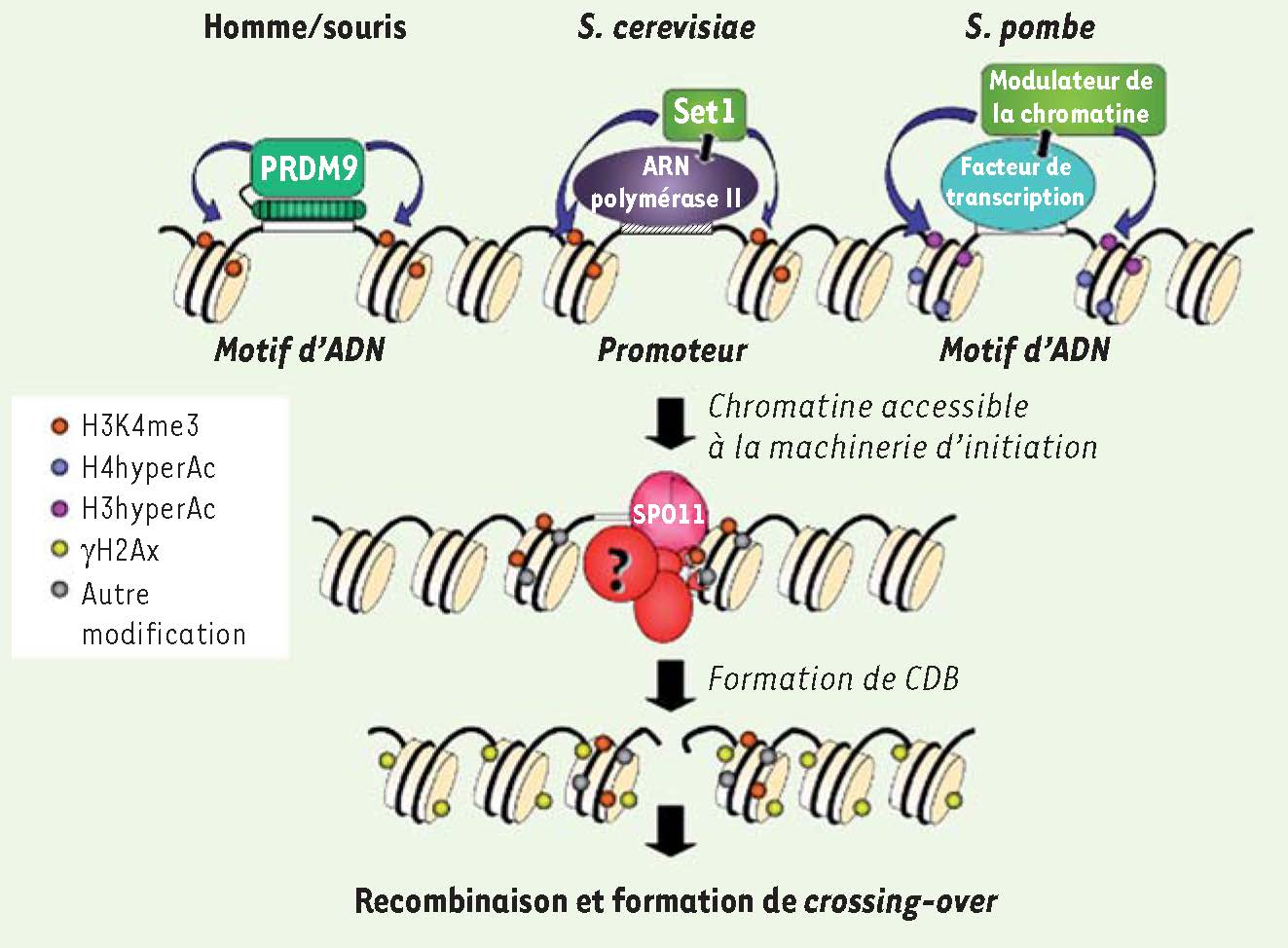
41]. De plus, plusieurs travaux récents montrent que l’allèle majoritaire de PRDM9 humain code pour une protéine qui contient des doigts de zinc capables de reconnaître le motif 13-mère associé aux points chauds [40, 41]. Par une analyse d’association génétique dans des familles, chez les individus porteurs de cet allèle majoritaire à l’état homozygote, environ 60 % des crossing-over ont lieu au niveau de points chauds déjà identifiés par l’analyse de la diversité génétique. Quelques individus portent également un allèle minoritaire codant pour une protéine ayant une série de doigts de zinc distincte et qui reconnaît une séquence d’ADN différente. Chez ces individus, seuls 20 % des crossing-over ont lieu dans les points chauds identifiés par l’analyse de la diversité génétique [40]. Cette différence spectaculaire dans le choix des sites de crossing-over permet de conclure que les doigts de zinc de PRDM9 jouent un rôle majeur pour déterminer la position des crossing-over chez l’homme et vraisemblablement chez la souris. Ces travaux permettent de proposer un modèle dans lequel la fixation de PRDM9 sur des motifs d’ADN spécifiques permet la triméthylation de H3K4 sur les nucléosomes avoisinants, favorisant localement l’initiation de la recombinaison méiotique (
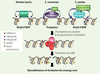
Figure 4
).  | Figure 4
Modèles moléculaires de spécification des points chauds. Chez la souris et chez l’homme, la protéine PRDM9 se lie, grâce à ses doigts de zinc, à un motif d’ADN (rectangle blanc) et triméthyle la lysine 4 de l’histone H3 (pastille orange). Chez S. cerevisiae, la triméthylation de la lysine 4 de H3 est réalisée grâce à la fixation de l’ARN polymérase II aux promoteurs de transcription (rectangle hachuré) suivi par le recrutement de l’histone-méthyltransferase Set1. Chez S. pombe, les sites de recombinaison sont déterminés par des motifs d’ADN sur lesquels se fixent des facteurs de transcription capables de recruter des modulateurs de la chromatine qui induisent entre autre l’hyperacétylation des histones H3 (pastille rose) et H4 (pastille bleue). Chez tous ces organismes la chromatine aux sites de recombinaison devient alors accessible à un complexe protéique (rouge) non identifié à ce jour (marqué « ? ») capable d’interpréter les différentes modifications d’histones et d’interagir de manière directe ou indirecte avec la machinerie d’initiation de la recombinaison dont fait partie la protéine Spo11 (rose). Spo11 catalyse la formation de cassure double brin (CDB), ce qui induit un signal de phosphorylation du variant d’histone H2AX (pastille jaune). Ce signal est interprété par la machinerie de recombinaison homologue qui répare les cassures et forme les crossing-over. |
|
Conclusions et implications Stratégies alternatives pour cibler l’activité de Spo11 La protéine Spo11 n’a pas ou peu de spécificité de séquence et son activité requiert que l’ADN soit accessible et qu’elle soit éventuellement ciblée vers des sites ou régions spécifiques. Les données actuelles montrent que plusieurs stratégies distinctes sont mises en œuvre dans différentes espèces pour réaliser cette étape-clef de la recombinaison (
Figure 4
). Le point commun entre ces stratégies semble être un système moléculaire qui introduit en des points précis du génome des modifications de la chromatine et permet le recrutement de protéines qui elles-mêmes recrutent Spo11. Le choix de la localisation de ces points du génome, ainsi que leurs propriétés spécifiques pour l’initiation de la recombinaison varient selon les espèces et sans doute également au sein d’un même organisme. Le choix des sites résulte d’une combinaison de facteurs en partie élucidée : chez S. cerevisiae, les sites sont apparemment choisis en raison de leur caractéristique de promoteur de la transcription, mais cette seule propriété n’est pas suffisante. Chez l’homme, les sites sont choisis en partie par la séquence d’ADN, c’est-à-dire s’ils possèdent une séquence reconnue par la protéine PRDM9, mais cette propriété est également loin d’être suffisante puisqu’une majorité des séquences du génome portant le motif 13-mère ne correspond pas à des points chauds. Chez S. pombe, sur la base de quelques exemples, la fixation de facteurs de transcription sur leurs motifs détermine la localisation de points chauds. Il est possible que ce mécanisme existe également chez S. cerevisiae et les mammifères. D’autres facteurs épigénétiques ou liés à la structure des chromosomes contribuent sans doute également à l’activité des points chauds et aux différences des cartes génétiques mâles et femelles chez les mammifères. Une des propriétés de la chromatine identifiées au niveau des points chauds concerne la triméthylation d’H3K4 détectée chez S. cerevisiae et la souris, et dont le rôle fonctionnel pour l’initiation de la recombinaison méiotique a été démontré chez S. cerevisiae. La présence d’histones possédant cette marque n’est cependant qu’une pièce d’un ensemble de propriétés encore non défini. En effet, chez la souris par exemple, les promoteurs de transcription sont enrichis en H3K4me3 mais ne sont pas des sites de recombinaison. D’autres modifications de la chromatine ou le recrutement d’autres protéines sont vraisemblablement impliqués dans le processus qui permet à Spo11 d’induire une CDB. Les mécanismes mis en jeu au sein d’un même organisme sont peut-être multiples. En effet, la détection de CDB chez S. cerevisiae set1Δ et chez les souris Prdm9
-/-
suggèrent le ciblage de Spo11 par des voies alternatives, actives en l’absence des protéines Set1 ou PRDM9, et peut-être également dans une méiose normale en contexte sauvage. Un système plastique et évolutif Une des caractéristiques étonnantes des points chauds, documentée par diverses approches chez les mammifères, est leur dynamique évolutive. Ceci est illustré par une variabilité interindividuelle des points chauds chez l’homme [
33], par la non-conservation des points chauds entre l’homme et le chimpanzé [ 41] et par l’évolution rapide du gène Prdm9 [
42]. Cette dynamique est au moins en partie liée au « paradoxe des points chauds » : si une mutation au niveau d’un point chaud diminue la formation de CDB en cis, alors dans des hétérozygotes les CDB auront lieu plus fréquemment sur l’autre chromosome et leurs réparations par recombinaison homologue recopieront la mutation située près de la CDB (
Figure 1
). Ce processus a pour résultat la transmission préférentielle de la mutation, et en absence d’autres contraintes, doit conduire à terme à la disparition des points chauds. Les propriétés de PRDM9 permettent en théorie, par l’apparition d’une mutation à une position codant pour un des résidus critiques des doigts de zinc, de modifier sa spécificité de séquence, de créer ainsi d’autres sites de CDB et donc d’apporter une réponse à ce paradoxe. Chez les levures, l’activité des points chauds pourrait être maintenue par sélection sur la fonction de ces séquences pour la cellule, dans la mesure où elles ont un autre rôle fonctionnel, par exemple pour l’expression des gènes adjacents. Les propriétés fascinantes de ces mécanismes de contrôle de la recombinaison méritent d’être approfondies avec entre autres des analyses chez d’autres organismes modèles pour lesquels il serait intéressant d’avoir des données complémentaires afin d’intégrer les mécanismes moléculaires et leurs implications évolutives. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
1.
Petronczki
M
,
Siomos
MF
,
Nasmyth
K
. Un ménage à quatre : the molecular biology of chromosome segregation in meiosis . Cell.
2003; ; 112 : :423.-440. 2.
Sturtevant
AH
. A third group of linked genes in drosophila ampelophila . Science.
1913; ; 37 : :990.-992. 3.
Morgan
TH
. Localization of the hereditary material in the germ cells . Proc Natl Acad Sci USA.
1915; ; 1 : :420.-429. 4.
Creighton
HB
,
McClintock
B
. A correlation of cytological and genetical crossing-over in zea mays . Proc Natl Acad Sci USA.
1931; ; 17 : :492.-497. 5.
Baudat
F
,
De Massy
B
. SPO11 : une activité de coupure de l’ADN indispensable à la méiose . Med Sci (Paris).
2004; ; 20 : :213.-218. 6.
Buard
J
,
de Massy
B
. Playing hide and seek with mammalian meiotic crossover hotspots . Trends Genet.
2007; ; 23 : :301.-309. 7.
Baudat
F
,
Nicolas
A
. Clustering of meiotic double-strand breaks on yeast chromosome III . Proc Natl Acad Sci USA.
1997; ; 94 : :5213.-5218. 8.
Cromie
GA
,
Hyppa
RW
,
Cam
HP
, et al. A discrete class of intergenic DNA dictates meiotic DNA break hotspots in fission yeast . PLoS Genet.
2007; ; 3 : :e141.. 9.
Ohta
K
,
Shibata
T
,
Nicolas
A
. Changes in chromatin structure at recombination initiation sites during yeast meiosis . EMBO J.
1994; ; 13 : :5754.-5763. 10.
Wu
T-C
,
Lichten
M
. Meiosis-induced double-strand break sites determined by yeast chromatin structure . Science.
1994; ; 263 : :515.-518. 11.
Hirota
K
,
Steiner
WW
,
Shibata
T
,
Ohta
K
. Multiple modes of chromatin configuration at natural meiotic recombination hot spots in fission yeast . Eukaryot Cell.
2007; ; 6 : :2072.-2080. 12.
Yamada
T
,
Mizuno
KI
,
Hirota
K
, et al. Roles of histone acetylation and chromatin remodeling factor in a meiotic recombination hotspot . EMBO J.
2004; ; 23 : :1792.-1803. 13.
Gerton
JL
,
DeRisi
J
,
Shroff
R
, et al. Inaugural article: global mapping of meiotic recombination hotspots and coldspots in the yeast Saccharomyces cerevisiae
. Proc Natl Acad Sci USA.
2000; ; 97 : :11383.-11390. 14.
White
MA
,
Detloff
P
,
Strand
M
,
Petes
TD
. A promoter deletion reduces the rate of mitotic, but not meiotic, recombination at the HIS4 locus in yeast . Curr Genet.
1992; ; 21 : :109.-116. 15.
Mieczkowski
PA
,
Dominska
M
,
Buck
MJ
, et al. Global analysis of the relationship between the binding of the Bas1p transcription factor and meiosis-specific double-strand DNA breaks in Saccharomyces cerevisiae
. Mol Cell Biol.
2006; ; 26 : :1014.-1027. 16.
Abdullah
MF
,
Borts
RH
. Meiotic recombination frequencies are affected by nutritional states in Saccharomyces cerevisiae
. Proc Natl Acad Sci USA.
2001; ; 98 : :14524.-14529. 17.
Hirota
K
,
Mizuno
K
,
Shibata
T
,
Ohta
K
. Distinct chromatin modulators regulate the formation of accessible and repressive chromatin at the fission yeast recombination hotspot ade6-M26 . Mol Biol Cell.
2008; ; 19 : :1162.-1173. 18.
Steiner
WW
,
Smith
GR
. Natural meiotic recombination hot spots in the Schizosaccharomyces pombe genome successfully predicted from the simple sequence motif M26 . Mol Cell Biol.
2005; ; 25 : :9054.-9062. 19.
Steiner
WW
,
Steiner
EM
,
Girvin
AR
,
Plewik
LE
. Novel nucleotide sequence motifs that produce hotspots of meiotic recombination in Schizosaccharomyces pombe
. Genetics.
2009; ; 182 : :459.-469. 20.
Kouzarides
T
. Chromatin modifications and their function . Cell.
2007; ; 128 : :693.-705. 21.
Merker
JD
,
Dominska
M
,
Greenwell
PW
, et al. The histone methylase Set2p and the histone deacetylase Rpd3p repress meiotic recombination at the HIS4 meiotic recombination hotspot in Saccharomyces cerevisiae
. DNA Repair (Amst).
2008; ; 7 : :1298.-1308. 22.
Mieczkowski
PA
,
Dominska
M
,
Buck
MJ
, et al. Loss of a histone deacetylase dramatically alters the genomic distribution of Spo11p-catalyzed DNA breaks in Saccharomyces cerevisiae
. Proc Natl Acad Sci USA.
2007; ; 104 : :3955.-3960. 23.
Ruthenburg
AJ
,
Allis
CD
,
Wysocka
J
. Methylation of lysine 4 on histone H3: intricacy of writing and reading a single epigenetic mark . Mol Cell.
2007; ; 25 : :15.-30. 24.
Sollier
J
,
Lin
W
,
Soustelle
C
, et al. Set1 is required for meiotic S-phase onset, double-strand break formation and middle gene expression . EMBO J.
2004; ; 23 : :1957.-1967. 25.
Borde
V
,
Robine
N
,
Lin
W
, et al. Histone H3 lysine 4 trimethylation marks meiotic recombination initiation sites . EMBO J.
2009; ; 28 : :99.-111. 26.
Noma
K
,
Grewal
SI
. Histone H3 lysine 4 methylation is mediated by Set1 and promotes maintenance of active chromatin states in fission yeast . Proc Natl Acad Sci USA.
2002; ; 99
(suppl 4) : :16438.-16445. 27.
Matise
TC
,
Chen
F
,
Chen
W
, et al. A second-generation combined linkage physical map of the human genome . Genome Res.
2007; ; 17 : :1783.-1786. 28.
Shifman
S
,
Bell
JT
,
Copley
RR
, et al. A high-resolution single nucleotide polymorphism genetic map of the mouse genome . PLoS Biol.
2006; ; 4 : :e395.. 29.
Frazer
KA
,
Ballinger
DG
,
Cox
DR
, et al. A second generation human haplotype map of over 3.1 millions SNP . Nature.
2007; ; 449 : :851.-861. 30.
Myers
S
,
Bottolo
L
,
Freeman
C
, et al. A fine-scale map of recombination rates and hotspots across the human genome . Science.
2005; ; 310 : :321.-324. 31.
Myers
S
,
Freeman
C
,
Auton
A
, et al. A common sequence motif associated with recombination hot spots and genome instability in humans . Nat Genet.
2008; ; 40 : :1124.-1129. 32.
Baudat
F
,
de Massy
B
. Cis- and trans-acting elements regulate the mouse Psmb9 meiotic recombination hotspot . PLoS Genet.
2007; ; 3 : :e100.. 33.
Coop
G
,
Wen
X
,
Ober
C
, et al. High-resolution mapping of crossovers reveals extensive variation in fine-scale recombination patterns among humans . Science.
2008; ; 319 : :1395.-1398. 34.
Neumann
R
,
Jeffreys
AJ
. Polymorphism in the activity of human crossover hotspots independent of local DNA sequence variation . Hum Mol Genet.
2006; ; 15 : :1401.-1411. 35.
Ptak
SE
,
Roeder
AD
,
Stephens
M
, et al. Absence of the TAP2 human recombination hotspot in chimpanzees . PLoS Biol.
2004; ; 2 : :E155..
36.
Winckler
W
,
Myers
SR
,
Richter
DJ
, et al. Comparison of fine-scale recombination rates in humans and chimpanzees . Science.
2005; ; 308 : :107.-111. 37.
Buard
J
,
Barthes
P
,
Grey
C
,
de Massy
B
. Distinct histone modifications define initiation and repair of meiotic recombination in the mouse . EMBO J.
2009; ; 28 : :2616.-2624. 38.
Grey
C
,
Baudat
F
,
de Massy
B
. Genome-wide control of the distribution of meiotic recombination . PLoS Biol.
2009; ; 7 : :e35.. 39.
Hayashi
K
,
Yoshida
K
,
Matsui
Y
. A histone H3 methyltransferase controls epigenetic events required for meiotic prophase . Nature.
2005; ; 438 : :374.-378. 40.
Baudat
F
,
Buard
J
,
Grey
C
, et al. PRDM9 is a major determinant of meiotic recombination hotspots in humans and mice . Science.
2010; ; 327 : :836.-840. 41.
Myers
S
,
Bowden
R
,
Tumian
A
, et al. Drive against hotspot motifs in primates implicates the PRDM9 gene in meiotic recombination . Science.
2010; ; 327 : :876.-879. 42.
Oliver
PL
,
Goodstadt
L
,
Bayes
JJ
, et al. Accelerated evolution of the Prdm9 speciation gene across diverse metazoan taxa . PLoS Genet.
2009; ; 5 : :e1000753.. 43.
Jeffreys
AJ
,
Kauppi
L
,
Neumann
R
. Intensely punctate meiotic recombination in the class II region of the major histocompatibility complex . Nat Genet.
2001; ; 29 : :217.-222. 44.
Baudat
F
,
Buard
J
,
Grey
C
,
de Massy
B
. Identification d’une protéine-clé pour le contrôle des sites de recombinaison méiotique . Med Sci (Paris).
2010; ; 26 : :468.-470. 45.
Montpetit
A
,
Chagnon
F
. La carte d’haplotype du génome humain : une révolution en génétique des maladies à hérédité complexe . Med Sci (Paris).
2006; ; 22 : :1061.-1067. |