| |
| Med Sci (Paris). 2011 April; 27(4): 443–446. Published online 2011 April 28. doi: 10.1051/medsci/2011274023.Les cellules souches pluripotentes humaines Un outil-clé pour décrypter les mécanismes physiopathologiques Yves Maury,1 Morgane Gauthier,1 Marc Peschanski,1 and Cécile Martinat1* 1Inserm/UEVE UMR-861, ISTEM, AFM, Institut des cellules souches pour le traitement et l’étude des maladies monogéniques, 5, rue Henri Desbruères, 91030Évry Cedex, France MeSH keywords: Adulte, Différenciation cellulaire, effets des médicaments et des substances chimiques, Lignée cellulaire, Techniques de coculture, Maladies génétiques congénitales, génétique, anatomopathologie, Protéines Hedgehog, pharmacologie, Humains, Protéines membranaires, déficit, physiologie, Modèles biologiques, Motoneurones, Dystrophie myotonique, Myotonin-protein kinase, Protéines de tissu nerveux, Neurites, Cellules souches pluripotentes, cytologie, Protein-Serine-Threonine Kinases, Plan de recherche, Création d'embryons à des fins de recherche, Cellules stromales, Synapses, ultrastructure, Trétinoïne, Expansion de trinucléotide répété |
Les cellules souches pluripotentes humaines sont communément associées à des applications de médecine régénératrice pour des maladies dégénératives. Elles présentent toutefois un tout autre potentiel qui peut offrir de nombreuses possibilités en santé publique : être utilisées pour permettre une meilleure compréhension des mécanismes de certaines pathologies humaines et, consécutivement, le développement de stratégies thérapeutiques adaptées. Grâce à leurs capacités d’autorenouvellement et de pluripotence, les cellules souches pluripotentes humaines représentent une source illimitée de cellules dont on peut induire la différenciation « à la demande » dans l’ensemble des types cellulaires de l’organisme. L’utilisation de cellules souches pluripotentes porteuses de la mutation causale d’une maladie humaine peut donc permettre de suivre l’apparition et la progression des processus physiopathologiques, en favorisant notamment l’identification de nouveaux acteurs cellulaires et moléculaires. |
Modélisation de maladies humaines : deux approches cellulaires Bien que des travaux aient déjà apporté des résultats prometteurs en montrant que l’on pouvait répliquer dans des cellules dérivées de lignées de cellules souches pluripotentes des signes pathologiques connus, l’identification de mécanismes pathologiques jusqu’alors inconnus n’avait pas encore été démontrée. Notre équipe vient de relever doublement ce défi en révélant les mécanismes moléculaires à l’origine d’anomalies fonctionnelles jusque-là ignorées de la maladie de Steinert, à l’aide de cellules souches embryonnaires humaines (CSEh) porteuses de la mutation causale [
1]. L’obtention de ces cellules repose actuellement sur deux méthodes : (1) la dérivation de CSEh à partir d’embryons obtenus par fécondation in vitro et écartés du programme de procréation à la suite d’un diagnostic pré-implantatoire (DPI) car porteurs de la mutation causale d’une maladie monogénique; (2) la conversion de cellules somatiques en cellules souches pluripotentes (hiPS, pour induced pluripotent stem cells) par manipulation génétique. En 2007, Nissim Benvenisty et ses collaborateurs ont été les premiers à démontrer que certains mécanismes moléculaires du syndrome de l’X fragile (X-Fra) pouvaient être reproduits dans les CSEh portant la mutation responsable [
2]. À la suite de l’identification des techniques de reprogrammation génique [
3], plusieurs équipes ont tenté de reproduire de la même façon les mécanismes de pathologies monogéniques à l’aide de lignées hiPS (Tableau I). Les hiPS partagent les propriétés cardinales des CSEh, l’immortalité et la pluripotence, mais des travaux récents démontrent que le processus de reprogrammation entraîne des modifications génétiques distinguant ces deux types cellulaires [
4–
6], ce qui complique parfois l’interprétation des données. L’équipe de Benvenisty en a donné un exemple précis en révélant l’incapacité d’iPS provenant de patients X-Fra à répliquer les mécanismes pathologiques pourtant présents dans les cellules dérivées de CSEh [
7].
Tableau I.
| Pathologie/syndrome (Sd) |
Phénotype pathologique présent |
Références |
| Déficit immunitaire combiné sévère par déficit en adénosine désaminase |
Non vérifié |
[
13] |
| Sd de Shwachman-Bodian-Diamond |
Non vérifié |
[13] |
| Maladie de Gaucher type III |
Non vérifié |
[13] |
| Maladie de Duchenne |
Absence de dystrophine dans des tératomes dérivés d’iPS |
[13,
14] |
| Dystrophie musculaire de Becker |
Non vérifié |
[13] |
| Parkinson |
Non vérifié |
[13,
15,
16] |
| Huntington |
Non vérifié |
[13] |
| Diabète de type 1 |
Inconnu |
[13,
17] |
| Sd de Down - trisomie 21 |
Densité microvasculaire plus faible |
[13,
18] |
| Sd de Lesch-Nyhan |
Non vérifié |
[13] |
| Amyotrophie spinale infantile |
Diminution de SMN (survival motor neuron) et défaut d’épissage de l’exon 7 Le nombre et la taille des motoneurones diminuent au cours de la culture et de la maturation |
[
19] |
| Dysautonomie infantile |
Défaut d’épissage alternatif du gène IKBKAP (inhibitor of kappa light polypeptide gene enhancer in B-cells, kinase complex-associated protein). Atteinte de la différenciation et de la migration neuronales |
[
20] |
| Sclérose latérale amyotrophique |
Inconnu |
[
21] |
| Syndrome de Rett |
Mutation du gène MeCP2 (methyl-CpG-binding protein 2), défauts (morphologiques et électrophysiologiques) des cellules neurales |
[
22–
24] |
| β-thalassémie |
Non vérifié |
[
25,
26] |
| Hutchinson–Gilford-progeria |
Mutation du gène LMNA (codant les lamines de types A et C)Dans les cellules du lignage mésodermique issues de ces iPS, présence de progérine et LMNA, augmentation des dommages de l’ADN et défaut morphologique des noyaux, apparition du phénotype sénescent dans des cellules du muscle lisse |
[
27,
28] |
| Sd du QT long |
Mutation du gène KCNQ1 (code pour un voltage-gated potassium channel, sous-famille Q) : défaut de conduction dans les cardiomyocytes dérivés d’iPSMutation du gène KCNH2 (code pour un voltage-gated potassium channel, sous-famille H) : défaut de conduction des cardiomyocytes dérivés d’iPS. Potentiel d’action prolongéMutation du canal calcium de type L Cav1.2 : contraction irrégulière des cardiomyocytes dérivés d’iPS. Influx excessif de calcium et potentiel d’action prolongé |
[
29–
32] |
Liste des lignées de cellules souches pluripotentes (iPS) humaines générées depuis 2007 à partir de cellules de patients. |
Nos études fondées sur des cellules dérivées d’embryons issus du DPI s’inscrivent dans ce cadre général du développement du champ de la modélisation pathologique in vitro. |
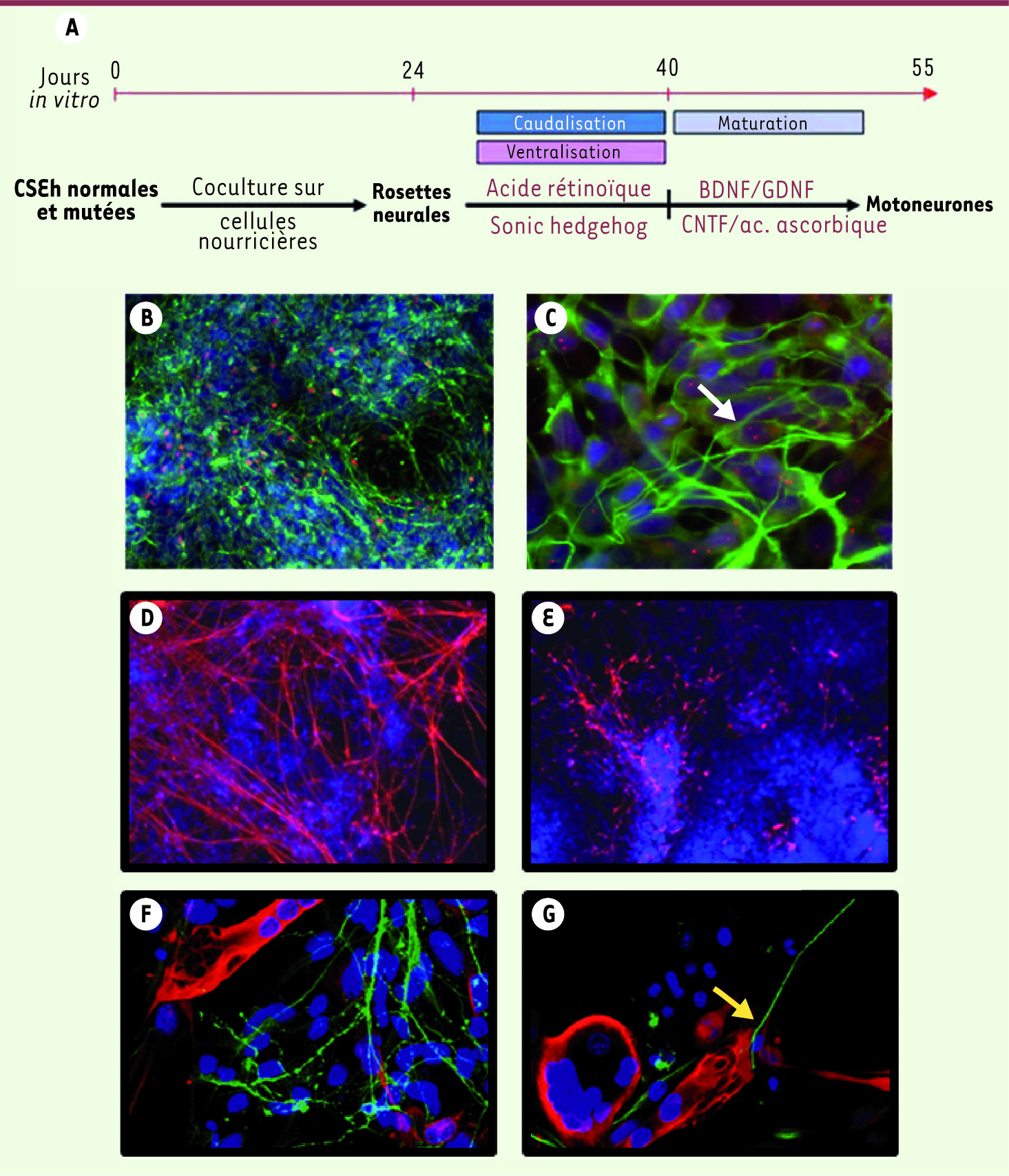
Preuve de concept de l’utilisation des cellules CSEh pour déchiffrer de nouveaux mécanismes physiopathologiques La maladie de Steinert ou dystrophie myotonique de type 1 (DM1) est la plus fréquente des dystrophies musculaires héréditaires de l’adulte, avec une prévalence estimée à 1/8 000. Elle se caractérise principalement par un retard à la décontraction musculaire après stimulation nerveuse (myotonie), souvent associé à une atrophie musculaire, des troubles cardiaques, endocriniens, cognitifs, etc. À l’origine de cette maladie, on trouve une répétition anormale de triplets CTG dans la région 3’ transcrite non traduite du gène codant pour la myotonine ou DMPK (dystrophic myotonic protein kinase). Cette mutation est responsable de la formation d’inclusions intranucléaires ribonucléoprotéiques qui séquestrent des protéines normalement impliquées dans les processus de maturation d’ARNm. Ainsi, des défauts de transcription, d’épissage et de stabilité de certains ARNm ont été associés à plusieurs signes cliniques [
8]. Cependant, la physiopathologie de cette maladie reste encore mal comprise, en particulier en ce qui concerne les mécanismes impliqués dans le syndrome myotonique. Nous avons pu utiliser deux lignées CSEh dérivées à l’AZ-VUB (Academisch Ziekenhuis van de Vrije Universiteit, Bruxelles) à partir d’embryons distincts porteurs de la mutation causale de DM1. Nous avons, dans un premier temps, différencié les cellules CSEh en motoneurones grâce à un protocole d’induction neurale par coculture des CSEh avec des cellules stromales, suivi d’une spécification motoneuronale à l’aide d’agents induisant un phénotype caudal (acide rétinoïque) et ventral (sonic hedgehog). Dans ces conditions, nous avons obtenu 30 % à 40 % de neurones présentant une morphologie et un phénotype de motoneurones exprimant le marqueur nucléaire Hb9 (Figure 1A-B). Deux signes moléculaires caractéristiques de DM1, la présence des inclusions ribonucléiques et une anomalie d’épissage du gène codant pour le récepteur au glutamate de type NMDA, étaient présents tout au long de ce processus de différenciation [
9]. Outre cette réplication de caractéristiques connues, nous avons fait une observation originale : une croissance anormalement profuse des prolongements neuronaux (Figure 1D). L’utilisation d’un système de coculture entre ces motoneurones et des cellules musculaires humaines a révélé de plus que cette arborisation exubérante des neurites s’accompagnait d’une diminution nette de leur capacité à former des synapses (Figure 1F). Il est intéressant de noter que ce phénotype anormal rappelle quelques observations anatomo-pathologiques réalisées chez des patients DM1 [
10–
12].
 | Figure 1
Anomalies phénotypiques des motoneurones issus des cellules CSEh DM1. On observe une augmentation anormale de l’arborisation neuritique entraînant des défauts dans la capacité des motoneurones à interagir avec leur cible musculaire. A. Représentation schématique du protocole de différenciation des CSEh en motoneurones. La première étape de ce protocole se base sur une coculture entre les CSEh et des cellules stromales permettant l’induction neurale. Les précurseurs neuraux ainsi obtenus, sous la forme de rosettes neurales, sont traités par de l’acide rétinoïque ou Sonic hedgehog, deux facteurs connus pour leur rôle dans la différenciation cholinergique. La maturation terminale des motoneurones est ensuite induite, pendant 1 à 2 semaines, en maintenant les cellules en présence de facteurs de survie tels que le BDNF (brain-derived neurotrophic factor), le GDNF (growth-derived neurotrophic factor) et l’acide ascorbique. B. Une proportion (30 %) des neurones (détectés par immunomarquage pour TuJI) expriment le marqueur nucléaire spécifique des motoneurones Hb9 (en rouge). C. Détection par hybridation in situ des inclusions ribonucléoprotéiques (en rouge) au niveau des neurones détectés par immunomarquage pour le marqueur TuJI (en vert). Un exemple est illustré par la flèche blanche. D. La détection du marqueur pan neuronal MAP2 (en rouge) permet de révéler l’augmentation de l’arborisation neuritique dans les cultures neuronales issues des cellules CSEh DM1 par rapport aux CSEh contrôles (E). F. Les neurites des motoneurones DM1 (en vert, marqueur TuJI) se connectent moins bien aux cellules musculaires (en rouge, marqueur desmine) que des motoneurones contrôles (G). La flèche jaune illustre un exemple d’interaction neurone-cellule musculaire. |
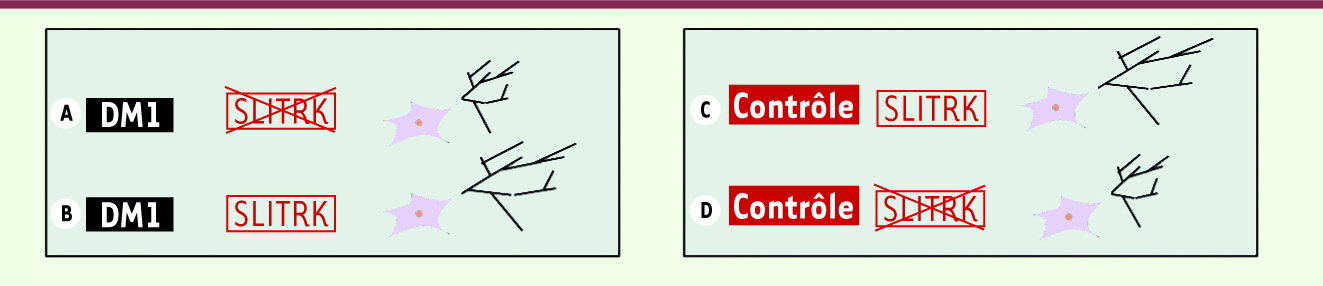
Afin de comprendre les mécanismes moléculaires associés à ces phénotypes, nous avons identifié les gènes différentiellement exprimés entre les cellules mutées et contrôles. Nous avons ainsi identifié deux gènes codant pour des membres de la famille SLITRK1, connue pour son rôle dans la croissance neuritique, dont l’expression était totalement effondrée. Nous avons retrouvé ce déficit, jusque-là inconnu, dans des prélèvements de tissu cérébral provenant de patients. Nous avons établi le lien causal entre la perte d’expression des SLITRK et l’anomalie neuritique en restaurant une activité normale de ces protéines dans les cellules DM1, ce qui a normalisé le défaut fonctionnel, l’arborisation neuritique excessive (Figure 2). À l’inverse, l’extinction des gènes à l’aide de shARN dans des cellules sauvages provoquait la pousse neuritique exubérante typique de la pathologie (Figure 2).
 | Figure 2
Lien causal entre le défaut d’arborisation neuritique et le défaut d’expression de deux gènes de la famille SLITRK dans les progéniteurs neuraux issus de cellules CSEh DM1. A. Dans les cultures de neurones issus de CSEh DM1, il y a absence d’expression des gènes SLITRK2 et SLITRK4, aboutissant à une augmentation du réseau neuritique. B. La surexpression à l’aide de vecteurs lentiviraux des gènes SLITRK2 et SLITRK4 dans les cultures neuronales DM1 aboutit à une diminution du réseau neuritique similaire à celui observé dans les cultures contrôles (C). D. L’extinction, par shARN, de l’expression des gènes SLITRK2 et SLITRK4 aboutit dans des cultures neuronales contrôles à une augmentation de l’arborisation neuritique. |
|
De la recherche fondamentale à la recherche appliquée Ces résultats démontrent ainsi le potentiel offert par les CSEh pour disséquer des mécanismes jusque-là inconnus de pathologies humaines. Ils soulignent en particulier la possibilité qu’offrent ces cellules d’identifier des anomalies aussi fines que celles pouvant affecter la communication intercellulaire. Le développement de nombreux protocoles de différenciation permettant d’obtenir un nombre important de phénotypes cellulaires différenciés ouvre largement ce champ d’exploration des maladies monogéniques, notamment de celles qui bénéficient d’un diagnostic pré-implantatoire. Au-delà de cette recherche fondamentale, l’application directe de cette modélisation pathologique est la recherche de composés capables de corriger, ou de compenser, les anomalies ainsi identifiées. Chacune de ces anomalies représentatives des pathologies et que l’on retrouve in vitro dans la descendance des cellules souches pluripotentes est une cible potentielle pour le criblage des millions de composés amassés par les industriels de la pharmacie, à la recherche des hits à partir desquels pourront être développés les traitements appropriés à ces maladies génétiques qui, pour la plupart, n’ont pas jusqu’alors bénéficié de ces approches pharmacologiques à haut débit. Human pluripotent stem cells: opening key for pathological modeling conflit d’intérêts
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Ce travail a été soutenu par l’Institut national de la santé et de la recherche médicale (Inserm), l’Association française contre les myopathies (AFM), la Région Île-de-France, le Génopole, Médicen (Programme IngeCell).
|
Footnotes |
1.
Marteyn
A
,
Maury
Y
,
Gauthier
MM
, et al.
Mutant human embryonic stem cells reveal neurite and synapse formation defects in Type 1 myotonic dystrophy . Cell Stem Cell.
2011 ; online.. 2.
Eiges
R
,
Urbach
A
,
Malcov
M
, et al.
Developmental study of fragile X syndrome using human embryonic stem cells derived from preimplantation genetically diagnosed embryos . Cell Stem Cell.
2007; ; 5 : :568.-577. 3.
Takahashi
K
,
Tanabe
K
,
Ohnuki
M
, et al.
Induction of pluripotent stem cells from adult human fibroblasts by defined factors . cell.
2007; ; 5 : :861.-872. 4.
Gore
A
,
Li
Z
,
Fung
HL
, et al.
Somatic coding mutations in human induced pluripotent stem cells . Nature.
2011; ; 471 : :63.-67. 5.
Hussein
SM
,
Batada
NN
,
Vuoristo
S
, et al.
Copy number variation and selection during reprogramming to pluripotency . Nature.
2011; ; 471 : :58.-62. 6.
Lister
R
,
Pelizzola
M
,
Kida
YS
, et al.
Hotspots of aberrant epigenomic reprogramming in human induced pluripotent stem cells . Nature.
2011; ; 471 : :68.-73. 7.
Urbach
A
,
Bar-Nur
O
,
Daley
GQ
,
Benvenisty
N.
. Differential modeling of fragile X syndrome by human embryonic stem cells and induced pluripotent stem cells . Cell Stem Cell.
2007; ; 5 : :407.-411. 8.
Ranum
LP
,
Cooper
TA.
. RNA-mediated neuromuscular disorders . Annu Rev Neurosci.
2006; ; 29 : :259.-277. 9.
Jiang
H
,
Mankodi
A
,
Swanson
MS
, et al.
Myotonic dystrophy type 1 is associated with nuclear foci of mutant RNA, sequestration of muscleblind proteins and deregulated alternative splicing in neurons . Hum Mol Genet.
2004; ; 24 : :3079.-3088. 10.
Stranock
SD
,
Davis
JN.
. Ultrastructure of the muscle spindle in dystrophia myotonica. II. The sensory and motor nerve terminals . Neuropathol Appl Neurobiol.
1978; ; 5 : :407.-418. 11.
Panaite
PA
,
Gantelet
E
,
Kraftsik
R
, et al.
Myotonic dystrophy transgenic mice exhibit pathologic abnormalities in diaphragm neuromuscular junctions and phrenic nerves . J Neuropathol Exp Neurol.
2008; ; 8 : :763.-772. 12.
Nagao
M
,
Kato
S
,
Hayashi
H
,
Misawa
H.
. Hyperproliferation of synapses on spinal motor neurons of Duchenne muscular dystrophy and myotonic dystrophy patients . Acta Neuropathol.
2003; ; 6 : :557.-560. 13.
Park
IH
,
Arora
N
,
Huo
H
, et al.
Disease-specific induced pluripotent stem cells . Cell.
2008; ; 134 : :877.-886. 14.
Kazuki
Y
,
Hiratsuka
M
,
Takiguchi
M
, et al.
Complete genetic correction of ips cells from Duchenne muscular dystrophy . Mol Ther.
2009; ; 2 : :386.-393. 15.
Soldner
F
,
Hockemeyer
D
,
Beard
C
, et al.
disease patient-derived induced pluripotent stem cells free of viral reprogramming factors . Cell.
2009; ; 5 : :964.-977. 16.
Hargus
G
,
Cooper
O
,
Deleidi
M
, et al.
Differentiated Parkinson patient-derived induced pluripotent stem cells grow in the adult rodent brain and reduce motor asymmetry in Parkinsonian rats . Proc Natl Acad Sci USA.
2010; ; 107 : :15921.-15926. 17.
Maehr
R
,
Chen
S
,
Snitow
M
, et al.
Generation of pluripotent stem cells from patients with type 1 diabetes . Proc Natl Acad Sci USA.
2009; ; 37 : :15768.-15773. 18.
Baek
KH
,
Zaslavsky
A
,
Lynch
RC
, et al.
Down’s syndrome suppression of tumour growth and the role of the calcineurin inhibitor DSCR1 . Nature.
2009; ; 7250 : :1126.-1130. 19.
Ebert
AD
,
Yu
J
,
Rose
FF
Jr
, et al.
Induced pluripotent stem cells from a spinal muscular atrophy patient . Nature.
2009; ; 7227 : :277.-280. 20.
Lee
G
,
Papapetrou
EP
,
Kim
H
, et al.
Modelling pathogenesis and treatment of familial dysautonomia using patient-specific iPSCs . Nature.
2009; ; 7262 : :402.-406. 21.
Dimos
JT
,
Rodolfa
KT
,
Niakan
KK
, et al.
Induced pluripotent stem cells generated from patients with ALS can be differentiated into motor neurons . Science.
2008; ; 5893 : :1218.-1221. 22.
Hotta
A
,
Cheung
AY
,
Farra
N
, et al.
Isolation of human iPS cells using EOS lentiviral vectors to select for pluripotency . Nat Methods.
2009; ; 5 : :370.-376. 23.
Marchetto
MC
,
Carromeu
C
,
Acab
A
, et al.
A model for neural development and treatment of Rett syndrome using human induced pluripotent stem cells . Cell.
2010; ; 4 : :527.-539. 24.
Cheung
AY
,
Horvath
LM
,
Grafodatskaya
D
, Isolation of MECP2-null Rett Syndrome patient hiPS cells and isogenic controls through X-chromosome inactivation . Hum Mol Genet.
2011;, (17 mars) ( online.). 25.
Ye
L
,
Chang
JC
,
Lin
C
, et al.
Induced pluripotent stem cells offer new approach to therapy in thalassemia and sickle cell anemia and option in prenatal diagnosis in genetic diseases . Proc Natl Acad Sci USA.
2009; ; 24 : :9826.-9830. 26.
Wang
Y
,
Jiang
Y
,
Liu
S
, et al.
Generation of induced pluripotent stem cells from human beta-thalassemia fibroblast cells . Cell Res.
2009; ; 9 : :1120.-1123. 27.
Zhang
J
,
Lian
Q
,
Zhu
G
, et al.
A human iPSC model of Hutchinson-Gilford progeria reveals vascular smooth muscle and mesenchymal stem cell defects . Cell Stem Cell.
2010; ; 1 : :31.-45. 28.
Liu
GH
,
Barkho
BZ
,
Ruiz
S
, et al.
Recapitulation of premature ageing with iPSCs from Hutchinson-Gilford progeria syndrome . Nature.
2011;, 23 février
(online).. 29.
Moretti
A
,
Bellin
M
,
Welling
A
, et al.
Patient-specific induced pluripotent stem-cell models for long-QT syndrome . N Engl J Med.
2010; ; 15 : :1397.-1409. 30.
Itzhaki
I
,
Maizels
L
,
Huber
I
, et al.
Modelling the long QT syndrome with induced pluripotent stem cells . Nature.
2011; ; 7337 : :225.-229. 31.
Matsa
E
,
Rajamohan
D
,
Dick
E
, et al.
Drug evaluation in cardiomyocytes derived from human induced pluripotent stem cells carrying a long QT syndrome type 2 mutation . Eur Heart J.
2011; ; (2 mars) ( online.). 32.
Yazawa
M
,
Hsueh
B
,
Jia
X
, et al.
Using induced pluripotent stem cells to investigate cardiac phenotypes in Timothy syndrome . Nature.
2011; ; 7337 : :230.-234. |