| |
| Med Sci (Paris). 2011 June; 27(6-7): 626–631. Published online 2011 July 1. doi: 10.1051/medsci/2011276015.La déubiquitinase CYLD Une cible récurrente dans les processus oncogéniques Marion Bonnet1* and Gilles Courtois2** 1Institute of Genetics, University of Cologne, Cologne, Allemagne 2Inserm U781, Hôpital Necker - Enfants malades, Université Paris Descartes, 149, rue de Sèvres, 75015Paris, France |
La cylindromatose familiale (OMIM # 132700) est une maladie génétique rare de transmission autosomique dominante qui se caractérise par l’apparition durant la vie adulte de tumeurs bénignes appelées cylindromes [
1]. Ces tumeurs se développent exclusivement à partir des annexes cutanées (glandes sudoripares ou sébacées, follicules pileux) et sont majoritairement localisées au niveau de la face et du cuir chevelu. Il a été montré que le gène causant la cylindromatose était localisé sur le chromosome 16, dans la région 16q12.1, et qu’il codait pour une protéine agissant probablement comme un suppresseur de tumeur. En effet, une perte d’hétérozygotie due à l’inactivation de l’allèle sain est détectée, la plupart du temps, dans les cellules formant le cylindrome. En 2000, l’équipe de G.R. Bignell a identifié le gène responsable de la cylindromatose et montré qu’il codait pour une nouvelle protéine que les chercheurs ont nommée CYLD [
2]. De manière intéressante, l’analyse histologique de tissus de patients isolés souffrant de cylindromatose ou de patients membres de familles dont plusieurs membres sont touchés a révélé, en de nombreuses occasions, l’apparition de tumeurs distinctes du cylindrome, homogènes ou sous forme de mélange, mais toutes dérivées du follicule pileux ou des glandes cutanées [
3]. En outre, l’analyse génétique de patients souffrant de trichoépithéliome familial (OMIM # 601606), une maladie qui présente des similitudes avec la cylindromatose, mais est considérée jusqu’à récemment comme s’en distinguant, a également révélé des mutations du gène CYLD [
4]. Cela suggère fortement que les mutations de CYLD affectent le compartiment folliculo-sébacé qui contient les cellules souches donnant naissance au follicule pileux et à ses glandes associées, générant des tumeurs appartenant à des sous-types différents. Une question majeure subsiste néanmoins. Alors que CYLD est exprimée dans tout l’organisme et que les patients souffrant de cylindromatose sont porteurs de mutations hétérozygotes du gène dans toutes les cellules somatiques, la sélectivité de la perte d’hétérozygotie, qui touche exclusivement le compartiment folliculo-sébacé et finalement aboutit à l’apparition d’un cylindrome ou d’un trichoépithéliome, reste mystérieuse. L’exposition au rayonnement UV représente une explication possible, mais la sensibilité particulière d’un réservoir de cellules souches en contact proche avec d’autres produits de l’environnement ne peut être exclue. |
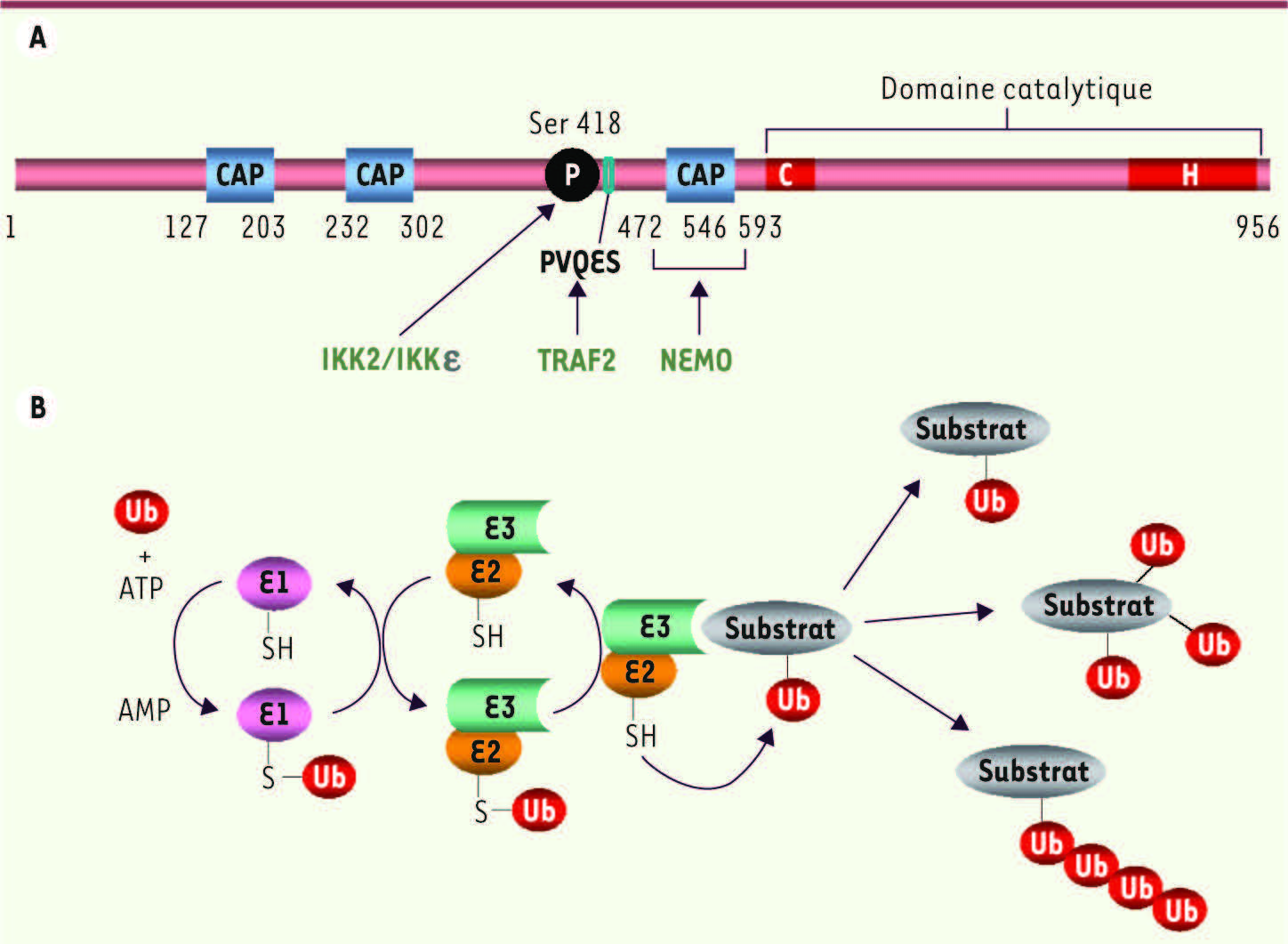
CYLD a initialement été décrite comme un membre potentiel de la famille des déubiquitinases (DUB). En effet, elle contient du côté carboxy-terminal un domaine présentant des similitudes avec le domaine catalytique des DUB (Figure 1A). La plupart des mutations responsables de la cylindromatose ou du trichoépitheliome correspondent à des mutations non-sens, des mutations qui affectent le cadre de lecture ou des mutations d’épissage qui se traduisent par une délétion de cette région.  | Figure 1
La protéine CYLD et le processus d’ubiquitination.A. Structure de la protéine CYLD. Les divers domaines, motifs et résidus fonctionnels sont indiqués. Dans la moitié amino-terminale sont localisés trois domaines CAP-Gly (CAP), un résidu sérine phosphorylé par IKK2 et IKKε (Ser 418), le motif PVQES reconnu par TRAF2 et le site d’interaction avec NEMO, qui correspond au troisième domaine CAP-Gly. Dans la moitié carboxy-terminale est localisé le domaine catalytique. Les deux sous-domaines qui le composent et qui contiennent les résidus actifs, une cystéine et une histidine, sont indiqués. B. Le processus d’ubiquitination. La séquence de réactions aboutissant à l’ubiquitination d’un substrat est montrée ainsi que les composants impliqués. Ub : ubiquitine ; E1 : enzyme activant l’ubiquitine ; E2 : enzyme conjuguant l’ubiquitine ; E3 : enzyme liant l’ubiquitine à son substrat. |
Le processus d’ubiquitination est une modification post-traductionnelle qui consiste en l’addition covalente d’un polypeptide de 76 aa, l’ubiquitine, à une protéine [
5]. Elle fait intervenir trois protéines distinctes (Figure 1B). La première, une E1, active l’ubiquitine. La seconde, une E2, catalyse l’addition d’ubiquitine au substrat en formant une liaison de type isopeptidique avec un résidu lysine. La spécificité de cette étape est assurée par une E3 qui reconnaît le substrat. Une seule molécule d’ubiquitine peut être ajoutée (mono-ubiquitination), mais comme l’ubiquitine contient elle-même sept lysines, des chaînes peuvent être formées (poly-ubiquitination). De manière intéressante, le type de chaîne qui est formé dicte le destin de la protéine ubiquitinée [
6]. Si les chaînes utilisent les résidus lysines 48 (K48) ou 11 (K11), la protéine sera reconnue par le protéasome, complexe protéasique de haut poids moléculaire, et dégradée. En revanche, si les chaînes utilisent le résidu lysine 63 (K63), la protéine ne représentera pas un substrat pour le protéasome, mais son activité (affinité pour d’autres protéines, trafic cellulaire, etc.) sera modifiée. Il en est de même dans le cas d’une polyubiquitination qui fait intervenir des chaînes linéaires d’ubiquitine. Le processus d’ubiquitination est contrôlé négativement par des DUB qui peuvent hydrolyser spécifiquement les chaînes K48 ou K63. Nous verrons ci-dessous que CYLD présente une affinité particulière pour les chaînes K63. En dehors de cette appartenance à la famille des déubiquitinases, la séquence de CYLD contient trois domaines ressemblant au domaine CAP-Gly qui a été identifié dans des protéines qui interagissent avec les microtubules [
7]. Néanmoins, l’interaction de CYLD avec les microtubules reste controversée. |
Les multiples fonctions de CYLD Lors de criblages par double-hybride, deux laboratoires ont montré que CYLD présentait une affinité pour NEMO (NF-κB essential modulator), la sous-unité régulatrice de la kinase IKK (IκB kinase) qui régule le processus d’activation de NF-κB [
8,
9,
30]. De manière concomitante, utilisant une stratégie basée sur l’interférence par ARN, Bernards et collaborateurs [
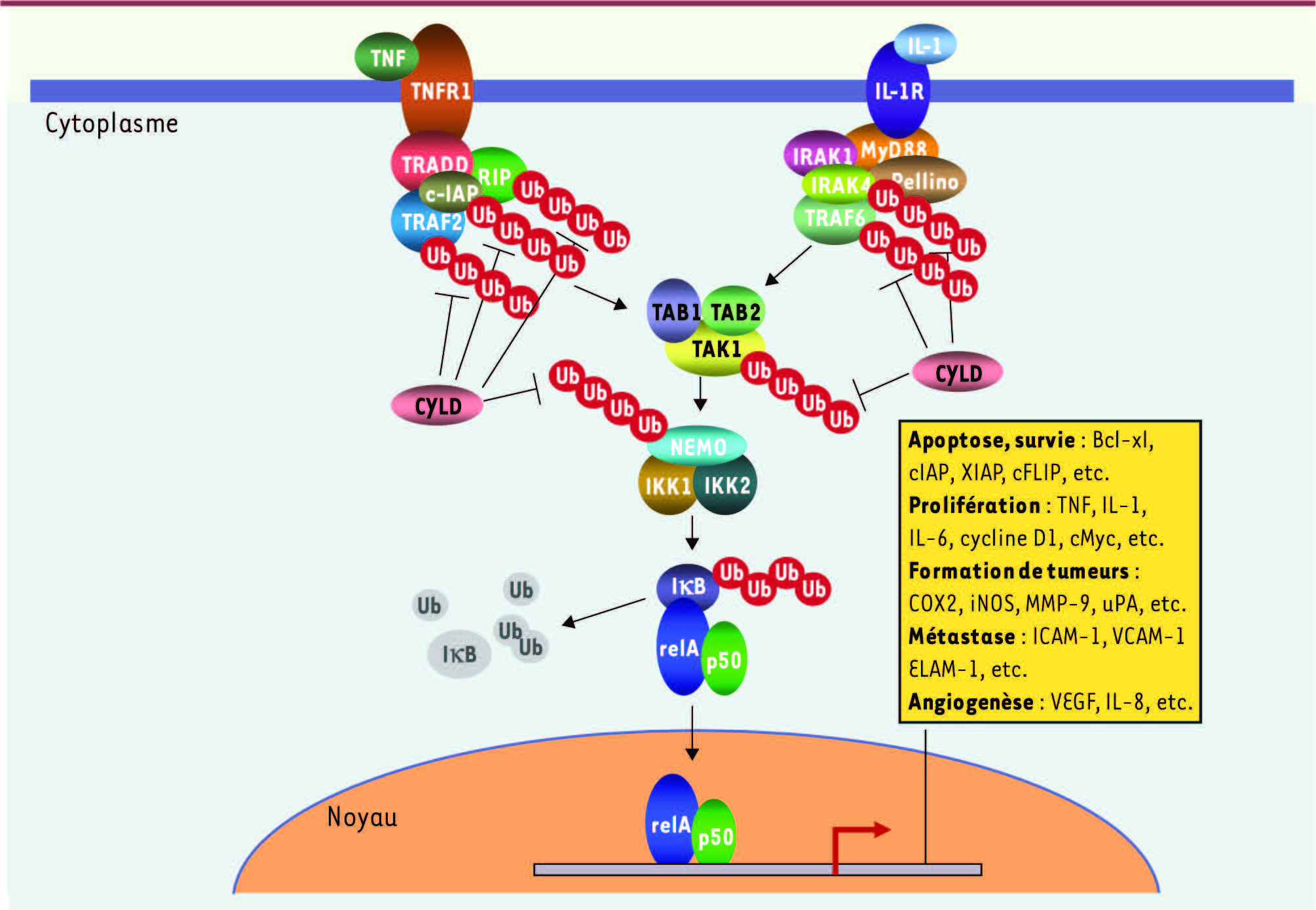
10] ont observé que l’inhibition de l’expression de CYLD se traduisait par une augmentation de l’activation de NF-κB induite par le TNF (tumor necrosis factor), indiquant que CYLD exerce un effet négatif sur l’activation de NF-κB. Ce processus nécessitant non seulement une étape d’ubiquitination de type K48 qui conduit à la dégradation de l’inhibiteur IκB, mais aussi des étapes d’ubiquitination de type K63 (Figure 2), le niveau auquel CYLD pouvait agir a été étudié. Il a été montré que CYLD agissait en amont d’IKK, au niveau de NEMO et des molécules TRAF (TNF receptor associated factor) en hydrolysant spécifiquement les chaînes de type K63.
 | Figure 2
Les étapes d’ubiquitination dans les voies canoniques d’activation de NF-κ B. Les voies de signalisation activées par le TNF-α et l’IL-1β sont représentées (pour revue voir [
29]). Les composants modifiés par des chaînes d’ubiquitine de type K63 sont indiqués en utilisant des chaînes droites alors que les chaînes de type K48, induisant la dégradation d’IκB, sont indiquées en utilisant des chaînes en zigzag. Les substrats potentiels ou avérés (voir Tableau I) de CYLD sont également indiqués ainsi que les principales cibles transcriptionnelles pouvant participer à un processus de cancérogenèse. RelA/p50 représente l’espèce dimérique NF-κB la plus fréquente. TRADD : TNF receptor-associated via death domain ; c-IAP : cellular inhibitor of apoptosis protein ; MyD88 : myeloid differentiation primary response gene 88 ; IRAK : interleukin-1-associated kinase ; TAB : TAK-1 binding protein ; IκBα: inhibitor of NF-κ B α ; Bcl-xl : B-cell lymphoma extra large ; XIAP : X-linked inhibitor of apoptosis protein ; cFLIP : cellular FLICE inhibitory protein ; COX2 : cyclo-oxygenase 2 ; iNOS : inducible nitric oxide synthase ; MMP-9 : matrix metalloproteinase-9 ; uPA : urokinase-type plasminogen activator ; ICAM-1 : intercellular adhesion molecule-1 ; VCAM-1 : vascular cell adhesion molecule-1 ; ELAM-1 : endothelial-leukocyte adhesion molecule-1 ; VEGF : vascular endothelial growth factor. |
Tableau I.
|
Bcl3 (K63)
|
Prolifération/cycle cellulaire |
|
HDAC6
|
|
|
PLK1
|
|
|
TRPA1
|
Signalisation canal calcique |
|
TRAF2 (K63)
|
|
|
TRAF6 (K63)
|
|
|
TRAF7
|
Voies pro-inflammatoires/apoptose (NF-κB, MAPK) |
|
RIP1 (K63)
|
|
|
TAK1 (K63)
|
|
|
NEMO (K63)
|
|
|
TRIP
|
|
|
RIG1(K63)
|
Voies anti-virales (IRF) |
|
Lck (K48)
|
Voie du TCR |
|
Dvl (K63)
|
Voie Wnt/β-caténine |
Les substrats de CYLD.
Les divers substrats de CYLD, qui interagissent avec cette DUB, sont indiqués ainsi que les processus/voies de signalisation auxquels ils participent. Dans la plupart des cas, il a été montré qu’ils sont modifiés par des chaînes de type K63. Bcl3 : B cell lymphoma 3 ; HDAC6 : histone deacetylase 6 ; TRPA1 : transient receptor potential A1 ; RIP1 : receptor interacting protein 1 ; TAK1 : TGFb-associated kinase 1 ; TRIP : TNF receptor interacting protein ; RIG1 : regulation of igh-1b 1 ; Lck : lymphocyte-specific protein tyrosine kinase ; Dvl : dishevelled ; IRF : interferon response factor ; TCR : T cell receptor (modifié d’après [
28]). |
Depuis ces observations, qui indiquent une fonction majeure de CYLD dans la régulation de la voie NF-κB, cette déubiquitinase a aussi été montrée comme contrôlant le processus d’activation des MAPK (mitogen activated protein kinases) [
11], l’activation de PLK-1 (polo-like kinase-1) durant la mitose [
12], la nécroptose [
13] et, plus récemment, l’activation de la voie Wnt/β-caténine [
14] (Tableau I). Dans pratiquement tous les cas, le substrat ciblé apparaît modifié par des chaînes d’ubiquitine de type K63. La préparation de souris invalidées pour CYLD a confirmé, in vivo, son rôle de régulateur négatif des voies NF-κB et JNK (c-JuN kinase) et démontré que l’absence de CYLD se traduisait par une augmentation du processus inflammatoire lorsque des conditions inflammatoires étaient induites, ou le développement accru de tumeurs quand un traitement procarcinogénique était appliqué [
15]. De manière notable, aucune de ces lignées invalidées n’a développé jusqu’à ce jour des tumeurs de type cylindrome, ou d’autres tumeurs, de façon spontanée. |
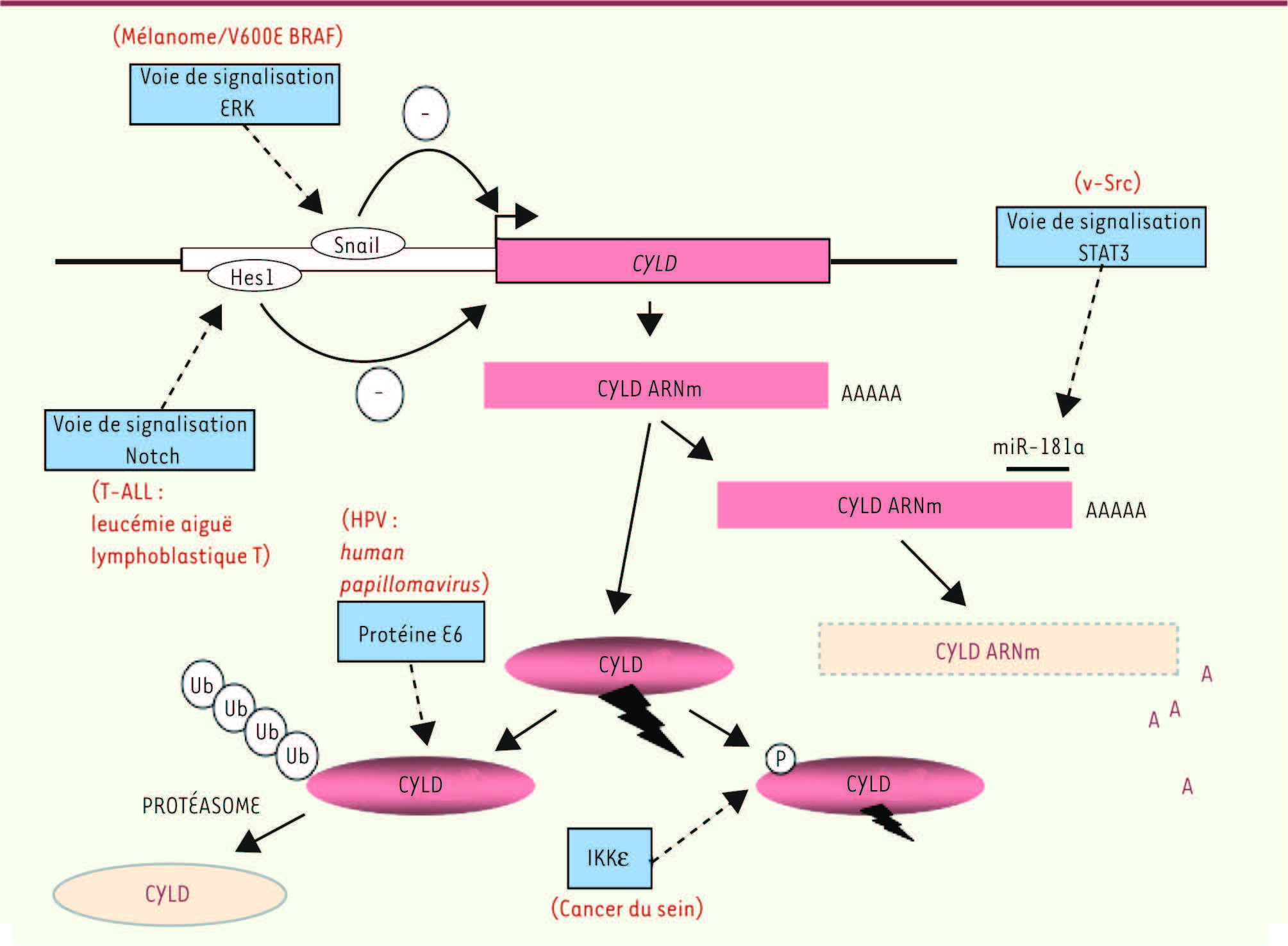
L’inhibition de CYLD dans les processus cancéreux Durant les deux dernières années, une série de publications a révélé un lien étroit entre l’inactivation de CYLD et l’oncogenèse, et cela dans des situations très diverses. CYLD est une cible de Snail1 dans le mélanome La plupart des mélanomes présentent une surexpression du répresseur transcriptionnel Snail1 [
16]. Ceci est dû à l’activation constitutive de B-Raf qui est souvent mutée dans ce type de pathologie (la mutation V600E est observée dans 60 % des cas). R. Massoumi et collaborateurs [
17] ont récemment montré que dans les mélanomes, le niveau d’expression de CYLD est très réduit dans la plupart des cas, et, plus remarquablement, que c’est Snail1, en se fixant au niveau de la région promotrice du gène CYLD, qui en est responsable (Figure 3). En effet, l’inhibition de Snail1 par interférence ARN se traduit par une augmentation de la synthèse de CYLD dans des cellules issues de mélanomes et une réduction de la croissance tumorale. L’expression réduite de CYLD entraînerait une accumulation de la protéine BCL-3 sous forme polyubiquitinée (chaînes K63), induisant ainsi sa translocation dans le noyau et l’activation de gènes qui participent au cycle cellulaire ou à la migration tels que ceux codant pour la cycline D1 ou la N-cadhérine. Il est à noter que BCL-3 est une molécule coactivatrice qui interagit avec plusieurs membres de la famille de facteurs de transcription NF-κB, tels que p50 et p52, renforçant l’idée qu’une dérégulation de la voie NF-κB est impliquée dans le développement du mélanome [
18]. 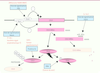 | Figure 3
Le ciblage de CYLD dans les processus oncogéniques. Les différents mécanismes inhibiteurs de CYLD au niveau du gène, de l’ARN messager ou de la protéine sont indiqués, ainsi que les types de cancer ou les modes de transformation cellulaire les induisant. Voir le texte pour plus de détails. Erk : extracellular signal-regulated kinase. |
Notch active NF-κB en agissant sur CYLD dans la leucémie aiguë lymphoblastique de type T Il a été montré qu’une dérégulation des voies Notch et NF-κB était responsable du développement de leucémies aiguës lymphoblastiques de type T (T-ALL) [
31] mais jusqu’à une date très récente la relation entre ces deux voies de signalisation restait énigmatique. A. Bigas et collaborateurs ont montré que l’activation constitutive de NF-κB causée par l’activation de Notch faisait en fait intervenir CYLD [
19] (Figure 3). En effet, comme dans le cas de Snail pour la voie B-Raf, une régulation négative du promoteur CYLD par Hes1, un coactivateur/répresseur recruté par Notch, a lieu. Cette inhibition de CYLD se traduit par une augmentation de l’activité de IKK, dépendante de NEMO. Il en résulte qu’un traitement de cellules leucémiques T-ALL avec un inhibiteur de la voie NF-κB représente une option thérapeutique. La boucle d’amplification NF-κB/IL-6/Stat3 induite par v-Src requiert l’inactivation de CYLD Utilisant un élégant système cellulaire basé sur la transformation induite par v-Src de cellules épithéliales mammaires, K. Struhl et collaborateurs ont identifié une boucle d’amplification inflammatoire/cancéreuse qui fait intervenir, successivement dans un premier temps, NF-κB, IL-6 ( interleukin-6) et STAT-3 ( signal transducer and activator of transcription-3) [
20]. L’établissement de cette boucle nécessite ensuite la synthèse de deux miARN (miR), miR-21 et miR-181b-1, par STAT-3 et leur action en retour sur NF-κB [
21]. Or, la recherche des ARNm ciblés par miR-21 et miR-181b-1 a identifié ceux codant pour PTEN ( phosphatase and tensin homolog) et CYLD, respectivement (Figure 3). L’inactivation de PTEN se traduit par une activation d’Akt, un activateur de NF-κB, alors que l’inactivation de CYLD affecte directement un inhibiteur de NF-κB. La protéine E6 du papillomavirus induit la dégradation de CYLD Une induction de NF-κB dans des conditions d’hypoxie peut survenir mais les mécanismes impliqués restent mal compris. M.B. Rettig et collaborateurs ont montré récemment que dans les cellules cancéreuses, l’activation constitutive de NF-κB par l’hypoxie est observée uniquement quand les cellules sont infectées par le papillomavirus humain (HPV) [
22]. Ils ont identifié l’agent responsable, la protéine E6, et montré qu’elle était nécessaire et suffisante pour induire NF-κB en conditions d’hypoxie. La cible d’E6 est CYLD, qui est ubiquitinée avec des chaînes K48 et dégradée par le protéasome (Figure 3). Bien que le mécanisme exact conduisant à la protéolyse de CYLD par l’infection HPV reste incomplètement caractérisé, il en résulte une activation d’IKK. Ce processus pourrait impliquer TRAF6 ( TNF receptor-associated factor), une protéine qui s’auto-ubiquitine avec des chaînes de type K63 et représente un substrat de CYLD. CYLD est un substrat pour la kinase IKKe La kinase IKKε, qui présente des similitudes avec les kinases IKK-1 et IKK-2, sous-unités catalytiques d’IKK, a été identifiée comme jouant un rôle oncogénique dans le cancer du sein [
23,
32] (). Une recherche de ses substrats potentiels, en déterminant tout d’abord la séquence de phosphorylation privilégiée avec une banque de peptides, a identifié CYLD [
24]. De manière intéressante, il a été montré que la phosphorylation de CYLD par IKKε au niveau de la sérine 418 a un effet inhibiteur sur son activité catalytique, une observation réminiscente du rétrocontrôle négatif que pourrait exercer IKK-2 sur CYLD durant le processus d’activation de NF-κB [
25] (Figure 3). Dans le cas de IKKε, cette inactivation apparaît essentielle au processus de transformation puisque l’expression d’une forme mutée de CYLD (CYLD S418A) ralentit la croissance avec perte d’inhibition de contact induite par IKKε. Dans ce système encore, le contrôle de la voie NF-κB par CYLD apparaît important puisqu’une inhibition de cette voie inhibe également le pouvoir transformant d’IKKε. |
Considérant la liste des processus oncogéniques impliquant CYLD que nous avons développée ci-dessus, il ne fait plus de doute maintenant que CYLD remplit effectivement une fonction de suppresseur de tumeur in vivo. Plus remarquable encore est la diversité des stratégies mises en place par les cellules cancéreuses pour inactiver cette enzyme, comme l’illustre la
Figure 3
. L’expression de l’ARNm codant pour CYLD peut être inhibée en agissant directement sur le promoteur (c’est le cas avec Snail1 [mélanome] ou Hes1 [T-ALL]). Alternativement, cet ARNm peut représenter une cible pour un miR et être dégradé (par exemple avec miR-181b-1 [transformation par v-Src]). En outre, deux mécanismes distincts affectant l’activité de la protéine CYLD elle-même ont été mis en lumière : la dégradation par le protéasome (c’est le cas avec E6 [cancers avec papillomavirus]) ou l’inhibition de son activité déubiquitinase (exemple avec IKKε [cancer du sein]). Des mutations de CYLD qui ont été observées dans le myélome multiple [
26], la maladie de Hodgkin [
27], etc., ne sont pas mentionnées ci-dessus. Dans tous ces cas, un effet de l’inactivation de CYLD sur la voie de signalisation NF-κB est observé, confirmant l’idée qu’une des fonctions essentielles de cette enzyme est bien de réguler le niveau d’activation de NF-κB. Il reste néanmoins à étudier dans ces divers systèmes si les autres mécanismes contrôlés par CYLD jouent également un rôle. Or, curieusement, la maladie qui a permis l’identification de CYLD, la cylindromatose, reste à ce jour très mal caractérisée. Bien qu’elle implique la formation de tumeurs, il n’est pas sûr qu’un dérèglement de la voie NF-κB en soit la cause principale. Considérant les autres fonctions de CYLD déjà identifiées, une analyse plus fine de son activité in vivo s’impose ainsi que l’invalidation de cyld dans le compartiment à partir duquel les tumeurs sont supposées se développer. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article. |
1.
Takahashi
M
,
Rapley
E
,
Biggs
PJ
, et al.
Linkage and LOH studies in 19 cylindromatosis families show no evidence of genetic heterogeneity and refine the CYLD locus on chromosome 16q12–q13 . Hum Genet.
2000; ; 106 : :58.-65. 2.
Bignell
GR
,
Warren
W
,
Seal
S
, et al.
Identification of the familial cylindromatosis tumour-suppressor gene . Nat Genet.
2000; ; 25 : :160.-165. 3.
Poblete Gutiérrez
P
,
Eggermann
T
,
Höller
D
, et al.
Phenotype diversity in familial cylindromatosis: a frameshift mutation in the tumor suppressor gene CYLD underlies different tumors of skin appendages . J Invest Dermatol.
2002; ; 119 : :527.-531. 4.
Zheng
G
,
Hu
L
,
Huang
W
,
LD
CY
, et al.
CYLD mutation causes multiple familial trichoepithelioma in three Chinese families.
Hum Mutat.
2004; ; 23 : :400.. 5.
Pickart
CM
,
Eddins
MJ
. Ubiquitin: structures, functions, mechanisms . Biochim Biophys Acta.
2004; ; 1695 : :55.-72. 6.
Winget
JM
,
Mayor
T
. The diversity of ubiquitin recognition : hot spots and varied specificity . Mol Cell.
2010; ; 38 : :627.-635. 7.
Steinmetz
MO
,
Akhmanova
A
. Capturing protein tails by CAP-Gly domains . Trends Biochem Sci.
2008; ; 33 : :535.-545. 8.
Trompouki
E
,
Hatzivassiliou
E
,
Tsichritzis
T
, et al.
CYLD is a deubiquitinating enzyme that negatively regulates NF-κB activation by TNFR family members . Nature.
2003; ; 424 : :793.-796. 9.
Kovalenko
A
,
Chable-Bessia
C
,
Cantarella
G
, et al.
The tumour suppressor CYLD negatively regulates NF-κB signalling by deubiquitination . Nature.
2003; ; 424 : :801.-805. 10.
Brummelkamp
TR
,
Nijman
SM
,
Dirac
AM
, et al.
Loss of the cylindromatosis tumour suppressor inhibits apoptosis by activating NF-κB . Nature.
2003; ; 424 : :797.-801. 11.
Reiley
W
,
Zhang
M
,
Sun
SC
. Negative regulation of JNK signaling by the tumor suppressor CYLD . J Biol Chem.
2004; ; 279 : :55161.-55167. 12.
Stegmeier
F
,
Sowa
ME
,
Nalepa
G
, et al.
The tumor suppressor CYLD regulates entry into mitosis . Proc Natl Acad Sci USA.
2007; ; 104 : :8869.-8874. 13.
Hitomi
J
,
Christofferson
DE
,
Ng
A
, et al.
Identification of a molecular signaling network that regulates a cellular necrotic cell death pathway . Cell.
2008; ; 135 : :1311.-1323. 14.
Tauriello
DV
,
Haegebarth
A
,
Kuper
I
, et al.
Loss of the tumor suppressor CYLD enhances Wnt/β-catenin signaling through K63-linked ubiquitination of Dvl . Mol Cell.
2010; ; 37 : :607.-619. 15.
Sun
SC
. CYLD: a tumor suppressor deubiquitinase regulating NF-κB activation and diverse biological processes . Cell Death Differ.
2010; ; 17 : :25.-34. 16.
Poser
I
,
Domínguez
D
,
de Herreros
AG
, et al.
Loss of E-cadherin expression in melanoma cells involves up-regulation of the transcriptional repressor Snail . J Biol Chem.
2001; ; 276 : :24661.-24666. 17.
Massoumi
R
,
Kuphal
S
,
Hellerbrand
C
, et al.
Down-regulation of CYLD expression by Snail promotes tumor progression in malignant melanoma . J Exp Med.
2009; ; 206 : :221.-232. 18.
Amiri
KI
,
Richmond
A
. Role of NF-κB in melanoma . Cancer Metastasis Rev.
2005; ; 24 : :301.-313. 19.
Espinosa
L
,
Cathelin
S
,
D’Altri
T
, et al.
The Notch/Hes1 pathway sustains NF-κB activation through CYLD repression in T cell leukemia . Cancer Cell.
2010; ; 18 : :268.-281. 20.
Hirsch
HA
,
Iliopoulos
D
,
Joshi
A
, et al.
A transcriptional signature and common gene networks link cancer with lipid metabolism and diverse human diseases . Cancer Cell.
2010; ; 17 : :348.-361. 21.
Iliopoulos
D
,
Jaeger
SA
,
Hirsch
HA
, et al.
STAT3 activation of miR-21 and miR-181b–1 via PTEN and CYLD are part of the epigenetic switch linking inflammation to cancer . Mol Cell.
2010; ; 39 : :493.-506. 22.
An
J
,
Mo
D
,
Liu
H
, et al.
Inactivation of the CYLD deubiquitinase by HPV E6 mediates hypoxia-induced NF-κB activation . Cancer Cell.
2008; ; 14 : :394.-407. 23.
Boehm
JS
,
Zhao
JJ
,
Yao
J
, et al.
Integrative genomic approaches identify IKBKE as a breast cancer oncogene . Cell.
2007; ; 129 : :1065.-1079. 24.
Hutti
JE
,
Shen
RR
,
Abbott
DW
, et al.
Phosphorylation of the tumor suppressor CYLD by the breast cancer oncogene IKKε promotes cell transformation . Mol Cell.
2009; ; 34 : :461.-472. 25.
Reiley
W
,
Zhang
M
,
Wu
X
, et al.
Regulation of the deubiquitinating enzyme CYLD by IκB kinase γ-dependent phosphorylation . Mol Cell Biol.
2005; ; 25 : :3886.-3895. 26.
Annunziata
CM
,
Davis
RE
,
Demchenko
Y
, et al.
Frequent engagement of the classical and alternative NF-κ pathways by diverse genetic abnormalities in multiple myeloma . Cancer Cell.
2007; ; 12 : :115.-130. 27.
Schmidt
A
,
Schmitz
R
,
Giefing
M
, et al.
Rare occurrence of biallelic CYLD gene mutations in classical Hodgkin lymphoma . Genes Chromosomes Cancer.
2010; ; 49 : :803.-809. 28.
Massoumi
R
. Ubiquitin chain cleavage: CYLD at work . Trends Biochem Sci.
2010; ; 35 : :392.-399. 29.
Hayden
MS
,
Ghosh
S
. Shared principles in NF-κB signaling . Cell.
2008; ; 132 : :344.-362. 30.
Gautheron
J
,
Courtois
G
. New functions of NEMO, the regulatory subunit of IKK . Med Sci (Paris).
2008; ; 24 : :954.-959. 31.
Kastner
P
,
Chan
S
. Notch activation during T-cell leukemogenesis in Ikaros-deficient mice . Med Sci (Paris).
2006; ; 22 : :708.-710. 32.
Grandvaux
N
. La kinase IKKε : de l’oncogenèse à la résistance au traitement du cancer du sein . Med Sci (Paris).
2011; ; 27 : :619.-625. |