| |
| Med Sci (Paris). 2011 June; 27(6-7): 651–655. Published online 2011 July 1. doi: 10.1051/medsci/2011276018.Représentation en sciences du vivant (9) - Émergence d’une spécialité médicale nouvelle : la pathologie Pierre Brousset1* and Georges Delsol1 1Laboratoire d’anatomie cytologie pathologiques, TSA 40031 CHU Purpan, place Baylac, 31059Toulouse Cedex 09, France |
L’anatomie pathologique au xxi
e siècle Pour une spécialité morphologique, qui s’attache à décrire des images microscopiques de cellules et de tissus, l’anatomie pathologique (ou anatomie et cytologie pathologiques) a une image négative auprès du grand public voire des autorités administratives ou hospitalo-universitaires de tutelle. Ce constat bien ironique est une réalité quotidienne pour tous les praticiens de cette discipline. De manière simple, quand nous voulons expliquer quelle est la nature exacte de notre travail en tant que pathologistes, nous sommes obligés d’utiliser un argumentaire métaphorique, en expliquant que notre activité consiste à examiner, sous le microscope, des fragments de tissus qui ont été prélevés afin de vérifier s’il n’existe pas de cellules cancéreuses ou de cellules inflammatoires. Ayant dit cela, nous n’avons pas beaucoup éclairé notre interlocuteur qui continue de penser que nous exerçons une spécialité très bizarre et le raccourci pour lui sera tout trouvé de nous considérer comme une sorte de biologiste. Plus sérieusement, l’anatomie pathologique, que nous nommerons volontairement « pathologie » au cours de cet exposé, devient une discipline majeure de la médecine car elle reste basée sur une expertise longue à obtenir, mais surtout parce qu’elle est perméable à tous les développements des sciences biomédicales mais aussi des sciences fondamentales. Elle a toujours eu pour vocation d’analyser des fragments de tissus ou des cellules à des fins diagnostiques, contribuant ainsi à une meilleure prise en charge de l’homme malade et à une meilleure compréhension des processus pathologiques dans leur globalité. La partie « anatomique » de cette spécialité médicale prend sa source dans les nombreuses études nécropsiques qui étaient sous la responsabilité des pathologistes et qui ont permis à des générations de médecins d’appréhender, au niveau anatomique, les lésions qu’ils allaient examiner au niveau microscopique. Nos prédécesseurs dans la discipline ont commencé leur carrière en pratiquant de très nombreuses autopsies et la question qui se pose aujourd’hui est de savoir si cette pratique peut encore avoir une valeur didactique pour la formation des jeunes internes. Probablement pas. La formation du pathologiste du xxi
e siècle peut faire l’économie d’analyses nécropsiques. En effet, nous avons tous constaté que le nombre d’autopsies scientifiques avait considérablement diminué et cette diminution est imputable à l’amélioration des techniques d’imagerie médicale, précédemment abordée dans cette série [
1–
3]. La somme de connaissances qu’un jeune étudiant devra acquérir pour devenir un pathologiste de bon niveau est devenue considérable. Il va devoir « faire son œil » sur une multitude de lésions qui s’accompagnent aujourd’hui de prises en charge très protocolaires, extrêmement contrôlées. Il va devoir maîtriser les techniques qui lui permettront d’affiner le diagnostic morphologique, qui sont issues de l’immunologie ou de la biologie moléculaire. Il va enfin devoir se familiariser avec les nouvelles techniques d’imagerie cellulaire et tissulaire, mais aussi apprendre à interpréter des dossiers sur des lames digitalisées dites virtuelles. Enfin, il va devoir se tenir en permanence informé des évolutions techniques et conceptuelles qui constitueront une source permanente de remise en question. La prise en charge en pathologie d’un cancer colorectal, d’un cancer du sein ou du poumon n’est plus la même en 2011 qu’en 2000. Ainsi, l’apport de la partie anatomique de la discipline se trouve considérablement réduit pour ne pas dire quasi nul, si l’on écarte bien sûr l’étape macroscopique de prise en charge des pièces opératoires. Le diagnostic morphologique, ou pourrait-on dire histopathologique, a, ces dernières années, bénéficié de l’apport considérable d’outils dérivés de l’immunologie, de la biologie moléculaire et de l’imagerie photonique et laser. La première des révolutions a été l’apparition des anticorps monoclonaux, utilisables sur coupes tissulaires (sur matériel inclus en paraffine), assortis de systèmes de détection enzymatiques. Ces techniques ont littéralement révolutionné la discipline, tant sur le plan nosologique que sur le plan thérapeutique. Grâce à ces outils, ce qui pouvait autrefois être classé comme tumeur indifférenciée à cellules rondes devenait soit un mélanome, soit un lymphome, soit un carcinome ou un sarcome, et c’est toute la force de ces techniques qui aujourd’hui demeurent incontournables dans la prise en charge diagnostique de routine. Le nombre d’anticorps évolue de manière exponentielle, le panel immunohistochimique le plus restreint nécessite souvent quatre ou cinq anticorps pour un seul échantillon. À l’extrême, dans le cas des hémopathies malignes ou des tumeurs indifférenciées, il n’est pas rare de recourir à une vingtaine d’immunomarquages. |
Techniques d’imagerie cellulaire et tissulaire Lames virtuelles : la fin du microscope optique conventionnel ? Cette approche va révolutionner notre manière d’appréhender l’image microscopique et peut bien marquer progressivement la fin des techniques de microscopie photonique conventionnelles [
4]. En d’autres termes, on est en train d’assister à la disparition du microscope optique conventionnel de l’arsenal du pathologiste. Qui l’aurait dit ? La lecture basée sur les lames virtuelles permet d’organiser son travail et d’opérer une gestion la plus efficace possible de ses activités. Il est en effet possible de scanner l’intégralité d’une lame et de consulter les images à distance sur n’importe quel ordinateur (voire même sur un téléphone portable de type smartphone ou sur un Ipad), de zoomer et de faire des commentaires à distance, et éventuellement de faire des comptes-rendus, alors que l’échantillon peut se trouver à plusieurs milliers de kilomètres [ 4]. Cette approche diagnostique est extrêmement prometteuse et va très probablement se développer de manière exponentielle. On pourrait établir un parallèle avec les images de la terre obtenues par Google Earth ou Géoportail, avec lesquelles on peut passer de la vision globale d’un continent à l’observation de sa maison et de son jardin. Ici, on fait la même chose avec une lame histologique pour visualiser in fine des détails cellulaires et subcellulaires. Les seuls facteurs limitants aujourd’hui sont le dimensionnement de l’outil informatique et la capacité de stockage des images qui sont souvent très volumineuses. La solution à ce problème n’est l’affaire que de quelques mois. En recherche, cette technique peut être utilisée pour effectuer des quantifications et peut s’appliquer aux tissue microarrays (TMA) qui permettent d’obtenir des images successives de dizaines d’échantillons sur une même lame (Figure 1). Plusieurs sociétés ont développé des logiciels qui permettent d’exécuter diverses fonctions d’analyse d’images sur les lames virtuelles produites. Le logiciel prend en charge la préparation des images pré-analyse et la récupération des résultats post-analyse (image de segmentation et données). Les fonctions peuvent être de la morphométrie avec une segmentation de l’image basée sur la luminance, la teinte et des critères morphomathématiques. D’autres fonctions sont associées à la description exhaustive des objets comme dans le cas des TMA. Les spots sont ainsi segmentés et une grille de score paramétrable est utilisée.
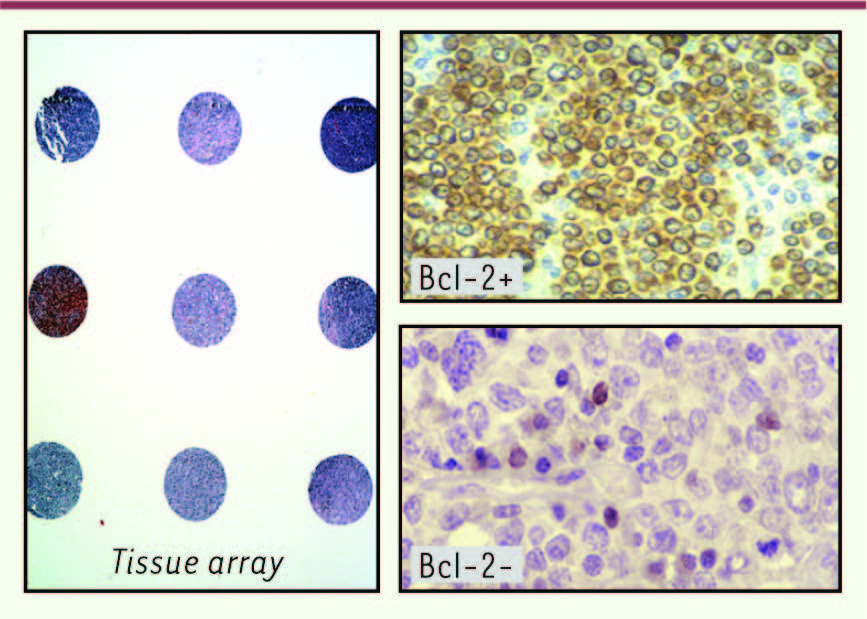
 | Figure 1
Tissue microarray de lymphome B diffus à grandes cellules. Immunodétection de l’oncoprotéine BCL2. |
Le diagnostic histopathologique du futur : une vision dynamique et en 3D de l’image cellulaire Il est un paradoxe que les pathologistes ont du mal à expliquer qui est leur relative incompétence pour toutes les techniques d’imagerie microscopique modernes (basées sur le laser). Il n’est pas rare d’observer dans les centres de recherche des microscopes confocaux ou biphotoniques sous la responsabilité de scientifiques non-médecins, qui savent manier ces outils d’imagerie et obtenir des informations cellulaires et subcellulaires que la microscopie photonique ne peut pas donner. Il est important que le pathologiste se réapproprie ces techniques et les exploite dans le champ du diagnostic histopathologique. En effet, la microscopie laser permet d’avoir une vision dynamique de l’image cellulaire. On peut voir là un des tournants importants des stratégies diagnostiques des dix prochaines années. Ces techniques permettent non seulement une pénétration tissulaire mais surtout d’effectuer une reconstitution en 3D des structures [
5,
6]. Ainsi, on peut non seulement visualiser les interactions cellulaires mais aussi obtenir une image dynamique du métabolisme (FRET, BRET 1). L’idéal serait bien évidemment de pouvoir obtenir des images microscopiques sur des structures tissulaires vivantes. Cela semble possible grâce à la microscopie à feuille de lumière qui consiste à réaliser des coupes optiques d’un échantillon en l’illuminant latéralement avec une feuille de lumière [
7,
8]. Cette technique est basée sur le selective plane illumination microscope (SPIM). Ces développements laissent entrevoir les progrès qui seront faits ces prochaines années. Non seulement on pourra identifier une cellule ou un tissu, mais il sera possible en parallèle de donner des informations précises sur leur métabolisme, sur l’état fonctionnel ou inactivé de telle ou telle voie de signalisation essentielle au phénotype tumoral. On pourrait même, en temps réel, mesurer l’efficacité des drogues anticancéreuses délivrées aux patients. On peut aller plus loin avec la microscopie CARS ( coherent anti-stokes raman scattering), ou microscopie par effet Raman, qui reste encore une technique en plein développement [
9]. Par rapport aux autres techniques de microscopie, son intérêt majeur réside, d’une part, dans l’absence de marquage et, d’autre part, dans son pouvoir de combiner information spectrale et imagerie, et cela à partir de liaisons chimiques endogènes à l’échantillon [ 9]. L’intérêt un peu futuriste de cette technique serait de voir une molécule unique se déplacer dans l’espace cellulaire ou tissulaire sans la marquer [ 9]. Les évolutions à prévoir devront aussi prendre en compte l’émergence des nanoparticules pour l’imagerie cellulaire et tissulaire [
10,
11]. D’ores et déjà les quantum-dots ( semiconductor nanocrystals) sont utilisés par certaines entreprises pour remplacer les composés fluorescents traditionnels ainsi que les silica-based nanoparticles, les gold nanoparticles et les nanophosphores [ 10, 11]. Ces composés sont souvent plus résolutifs que les fluorophores classiques mais sont surtout beaucoup plus stables dans le temps sans extinction du signal. Il convient aussi de mentionner l’apport des techniques de spectrométrie de masse dans l’imagerie moléculaire et tissulaire [
12,
13]. Les techniques de type MALDI-TOF (matrix assisted laser desorption ionization time of flight) et TOF-SIMS (time of flight secondary ion mass spectrometry) sont applicables à des composés différents, ce qui les rend complémentaires pour l’analyse et la cartographie protéique d’une coupe tissulaire. En effet, le MALDI-TOF permet de cartographier des protéines et peptides à la surface d’une coupe alors que le TOF-SIMS, qui ne possède pas de matrice, permet l’analyse des lipides. Ces techniques restent difficiles à mettre en place mais pourraient subir des évolutions permettant d’obtenir des informations qui échappent aux techniques immunomorphologiques. Leur défaut actuel, mais peut-être perfectible, est la très modeste résolution morphologique. L’utilisation de toutes les techniques mentionnées dans ce chapitre ne peut s’envisager que selon des indications précises. Il n’est pas concevable qu’elles puissent être appliquées en routine pour tous les échantillons mais plutôt dans des circonstances ciblées. Par exemple, il paraît inéluctable que l’étude d’une cible (récepteur, enzyme, etc.) nécessite une analyse de son état fonctionnel. Pour savoir si une voie de signalisation est active, il faudra en étudier les acteurs mais aussi se renseigner sur les modifications chimiques qui déterminent leur fonction (phosphorylation, méthylation, etc.) et les éventuels partenaires ou substrats avec lesquels cette molécule interagit. On pourra parler d’imagerie fonctionnelle et dynamique à orientation diagnostique mais surtout théranostique. |
Biologie et pathologie moléculaires Depuis une quinzaine d’années, des techniques issues de la biologie moléculaire sont apparues dans le champ d’action du pathologiste. Ces techniques, un peu balbutiantes à leur début, ont permis de mettre au point l’hybridation in situ pour typer des virus (papillomavirus, virus d’Epstein Barr) ou pour mettre en évidence des réarrangements chromosomiques sur noyau interphasique (technique de FISH). Le pathologiste est aujourd’hui confronté à des techniques de biologie moléculaire beaucoup plus globales, basées sur l’extraction des acides nucléiques, la PCR et le séquençage, et qui vont lui permettre indifféremment d’affirmer la clonalité d’une prolifération lymphoïde, de mettre en évidence un gène de fusion dans une tumeur ou de déterminer une mutation qui, si elle est présente, aura un impact thérapeutique immédiat pour le patient (théranostic) [
14–
16]. À partir d’échantillons tissulaires lésionnels, des techniques de génomique à haut débit (puces ADN) ont permis d’obtenir des profils d’expression géniques définissant ainsi des signatures moléculaires. Ces signatures, basées sur le transcriptome, ont abouti à des remembrements nosologiques importants (exemples : lymphomes, sarcomes, etc.) [16,
17]. De fait, le pathologiste moderne devra devenir un pathologiste moléculaire. Il devra connaître la structure des acides nucléiques, la thermodynamique des molécules d’ADN, les conséquences cellulaires de mutations, d’amplifications géniques, de réarrangements chromosomiques; il devra parfaitement maîtriser les outils qui permettent ces analyses moléculaires afin d’éviter les nombreux pièges liés à leur maniement parfois très délicat. La position du pathologiste est stratégique car c’est lui qui qualifie l’échantillon qui sera soumis à une analyse moléculaire [
15, 16]. Quand on ne sait pas ce qu’on analyse (présence ou pas de tissu tumoral, importance de la nécrose, etc.), on ne peut pas faire une analyse critique du résultat. En clair, le pathologiste a un rôle-clé car il devra resituer des événements moléculaires dans leur contexte tissulaire. Dès lors, il est tout à fait logique, dans une continuité d’exécution, qu’il prenne aussi en charge les analyses moléculaires spécifiques, dès l’instant où il aura fait l’effort d’acquérir les compétences nécessaires. Comme il a réussi à apprivoiser les techniques d’immunodétection, le pathologiste pourra très aisément maîtriser les techniques moléculaires, de plus en plus nombreuses, et qui contribuent quotidiennement au théranostic. Enfin, il devra être à même de transposer en routine les résultats des études transcriptomiques. L’exemple le plus flagrant est celui des lymphomes B à grandes cellules où l’analyse transcriptomique a révélé l’existence de deux sous-populations, l’une issue des centres germinatifs et l’autre dérivant de cellules activées [
18]. Cette dichotomie peut être reproduite simplement sur coupes tissulaires grâce à trois anticorps seulement (anti-CD10, anti-BCL6, anti-IRF4/MUM1). De plus, la validation de nouveaux marqueurs issus du transcriptome peut s’exécuter à haut débit grâce aux tissue microarrays (TMA) qui contiennent plusieurs centaines de fragments tumoraux sur une même lame, provenant de patients dont la réponse au traitement et l’évolution sont connues (Figure 1). |
La pathologie est un des éléments-clés de la recherche biomédicale La pathologie est une discipline universelle. Le pathologiste est un médecin, clinicien, morphologiste et, du fait de cette position, se trouve tout naturellement orienté vers la recherche. De plus en plus de pathologistes en France et en Europe coordonnent des travaux de recherche et s’impliquent à divers niveaux de la recherche médicale. Certains font de la recherche translationnelle et d’autres mettent leur talent en jeu pour une recherche plus cognitive et plus fondamentale. Du fait de sa position stratégique, le pathologiste fait aussi l’objet de toutes les attentions de la part des chercheurs qui sont intéressés par les tissus humains pathologiques (pathologie dégénérative, cancers, etc.). C’est lui qui va gérer et alimenter les centres de ressources biologiques avec des échantillons tissulaires extrêmement importants pour la recherche médicale. Ces banques de tissus et ces centres de ressources biologiques n’ont d’intérêt que s’ils répondent aux normes de qualité de préservation des échantillons (NF S96900) mais surtout si les échantillons sont accompagnés d’annotations cliniques dynamiques qui font toute leur valeur. Ces échantillons attisent aussi la convoitise des industriels qui recherchent de plus en plus souvent des partenariats pour leurs études et le développement de nouvelles drogues et autres thérapeutiques ciblées. |
Quel avenir pour la pathologie ? Il est toujours difficile de prévoir l’évolution des disciplines et les impacts que pourront avoir les innovations qui aujourd’hui sont à l’état expérimental. Certains ont pensé que les analyses transcriptomiques à haut débit allaient menacer la pathologie et remplacer le diagnostic morphologique [18]. La crainte était la même avec l’apparition de la cytométrie de flux, il y a quelques années. Ces techniques ne sont pas concurrentes mais largement complémentaires de l’examen histopathologique. Il existe un problème toujours difficile à résoudre et qui protège le pathologiste dans sa spécificité, c’est l’automatisation de la lecture des lames histologiques. Pourtant, cette automatisation qui semble envisageable en imagerie macroscopique paraît encore inconcevable en imagerie microscopique. Ce problème non résolu d’automatisation implique que la pathologie, bien qu’elle devienne de plus en plus scientifique et qu’elle se rapproche des sciences exactes, reste une forme d’art (Figure 2). Les exigences de prise en charge clinique des patients sont telles que le recours à des hyperspécialistes aujourd’hui de plus en plus demandé. Il est difficile de dire s’il s’agit d’une bonne ou d’une mauvaise tendance pour l’évolution de la discipline, mais l’expérience acquise dans un domaine et cette hyperspécialisation garantissent une plus grande fiabilité du diagnostic et de l’évaluation pronostique et théranostique des lésions ainsi observées.
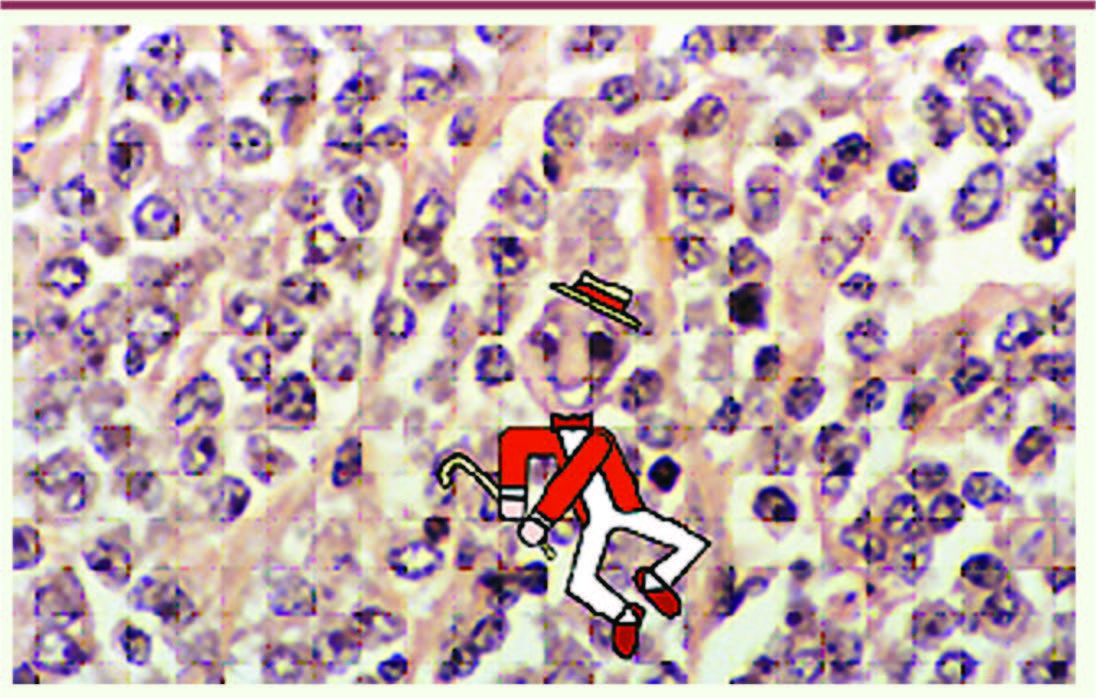
 | Figure 2
Lymphome B diffus à grandes cellules. Certaines images sont amusantes, témoignant de l’extrême diversité morphologique des cellules cancéreuses. Cette hétérogénéité peut être étendue à d’autres modèles lésionnels de sorte que le même type de tumeur chez deux patients distincts peut avoir des critères morphologiques parfois très différents. |
|
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Footnotes |
1.
Moulin
AM
,
Baulieu
JL
. L’imagerie et la nouvelle « fabrique » du corps humain . Med Sci (Paris).
2010; ; 26 : :993.-998. 2.
Coatrieux
JL
,
Velut
J
,
Dillenseger
JL
,
Toumoulin
C
. De l’imagerie médicale à la thérapie guidée par l’image . Med Sci (Paris).
2010; ; 26 : :1103.-1109. 3.
Izeddin
I
,
Darzacq
X
,
Dahan
M
. Microscopies cellulaires à l’échelle de la molécule individuelle . Med Sci (Paris).
2011; ; 27 : :547.-552. 4.
Jara-Lazaro
AR
,
Thamboo
TP
,
The
M
,
Tan
PH
. Digital pathology: exploring its applications in diagnostic surgical pathology practice . Pathology.
2010; ; 42 : :512.-518. 5.
Dushek
O
,
Mueller
S
,
Soubies
S
, et al. Effects of intracellular calcium and actin cytoskeleton on TCR mobility measured by fluorescence recovery . PLoS One.
2008; ; 3 : :e3913.. 6.
Nava
RG
,
Li
W
,
Gelman
AE
, et al. Two-photon microscopy in pulmonary research . Semin Immunopathol.
2010; ; 32 : :297.-304. 7.
Weninger
WJ
,
Mohun
T
. Phenotyping transgenic embryos: a rapid 3-D screening method based on episcopic fluorescence image capturing . Nat Genet.
2002; ; 30 : :59.-65. 8.
Huisken
J
,
Swoger
J
,
Del Bene
F
, et al. Optical sectioning deep inside live embryos by selective plane illumination microscopy . Science.
2004; ; 305 : :1007.-1009. 9.
Djaker
N
,
Marguet
D
,
Rigneault
H
. Microscopie Raman stimulée (CARS) : Principes et applications . Med Sci (Paris).
2006; ; 10 : :853.-858. 10.
Choi
HS
,
Frangioni
JV
. Nanoparticles for biomedical imaging: fundamentals of clinical translation . Mol Imaging.
2010; ; 9 : :291.-310. 11.
Ohulchanskyy
TY
,
Roy
I
,
Yong
KT
, et al. High-resolution light microscopy using luminescent nanoparticles . Wiley Interdiscip Rev Nanomed Nanobiotechnol.
2010; ; 2 : :162.-175. 12.
Wisztorski
M
,
Lemaire
R
,
Stauber
J
, et al. Imagerie MALDI : une nouvelle technologie pour découvrir et valider de nouveaux biomarqueurs . Med Sci (Paris).
2007; ; 23 : :S31.-S36. 13.
Com
E
,
Hondermarck
H
. Protéomique fonctionnelle en oncologie : comprendre plus de décrire . Med Sci (Paris).
2007; ; 23 ((hs1)) : :27.-30. 14.
Sandberg
AA
,
Meloni-Ehrig
AM
. Cytogenetics and genetics of human cancer: methods and accomplishments . Cancer Genet Cytogenet.
2010; ; 203 : :102.-126. 15.
Coindre
JM
. Molecular biology of soft-tissue sarcomas . Bull Cancer.
2010; ; 97 : :1337.-1345. 16.
Delsol
G
. Molecular abnormalities in lymphomas . Bull Cancer.
2010; ; 97 : :1347.-1364. 17.
Ladanyi
M
,
Chan
W
,
Triche
TJ
,
Gerald
WL
. Expression profiling of human tumors. The end of surgical pathology?
J Mol Diag.
2001; ; 3 : :92.-97. 18.
Hans
CP
,
Weisenburger
DD
,
Greiner
TC
, et al. Confirmation of the molecular classification of diffuse large B-cell lymphoma by immunohistochemistry using a tissue microarray . Blood.
2004; ; 103 : :275.-282. |