| |
| Med Sci (Paris). 2011 December; 27(12): 1069–1071. Published online 2011 December 23. doi: 10.1051/medsci/20112712011.ADAM et migration cellulaire Rôle inattendu du domaine cytoplasmique Hélène Cousin1* and Dominique Alfandari1 1Veterinary and Animal Sciences department, University of Massachusetts, ISB room 427N, 661 North Pleasant Street, Amherst, MA 01003, États-Unis MeSH keywords: Protéines ADAM, composition chimique, génétique, métabolisme, physiologie, Animaux, Mouvement cellulaire, Cytoplasme, Embryon de mammifère, Embryon non mammalien, Développement embryonnaire, Humains, Mammifères, embryologie, Souris, Modèles biologiques, Crête neurale, Structure tertiaire des protéines, Xenopus |
La famille ADAM (a disintegrin and metalloprotease) est composée d’une trentaine de protéines transmembranaires contenant un domaine métalloprotéase qui n’est actif que chez la moitié d’entre elles (Figure 1). Parmi ces ADAM protéolytiques, ADAM 10 et 17 sont les plus étudiées. Leur rôle est essentiel pour activer ou inhiber une grande variété de voies de signalisation comme celles activées par l’EGF (epidermal growth factor), le TNFα (tumor necrosis factor) et Notch [
1,
11]. Un rôle moins connu d’ADAM 10 consiste à cliver les N- et E-cadhérines, diminuant l’adhésion intercellulaire et favorisant ainsi les transitions épithélio-mésenchymateuses [
2,
3]. Le sous-groupe des meltrines, composé des meltrines α, β, γ, ε (ADAM 12, 19, 9 et 35 respectivement) et de ADAM 13/33, est moins étudié. Il se distingue par un domaine cytoplasmique de 150 à 200 résidus contenant plusieurs domaines de liaison riches en proline et un point isoélectrique basique.
 | Figure 1
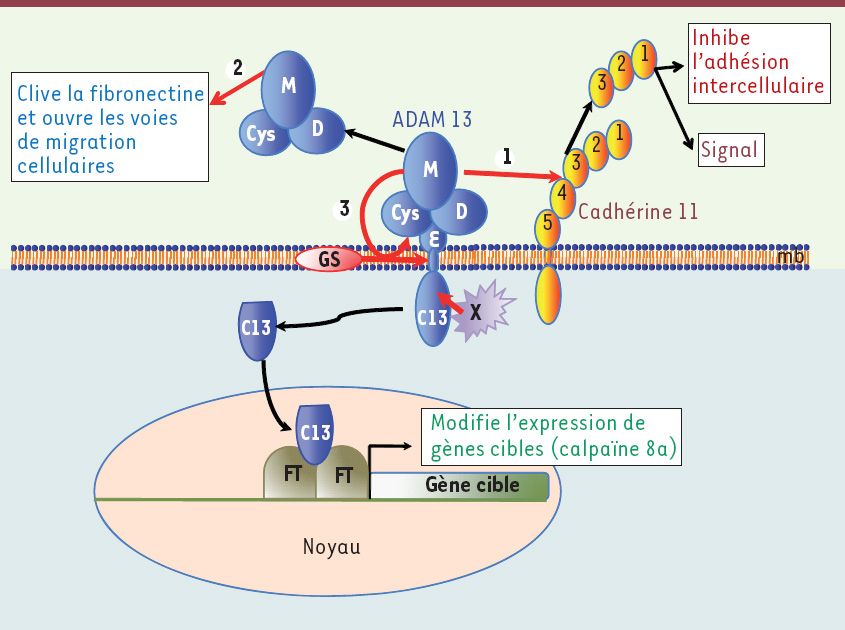
Représentation schématique des fonctions d’ADAM 13 nécessaires à la migration des cellules de la crête neurale céphalique (CNCC). ADAM 13 est constituée de cinq domaines : métalloprotéase (M), disintégrine (D), riche en cystéine (Cys), EGF (E), transmembranaire et cytoplasmique (C13). Le domaine métalloprotéase clive plusieurs substrats (flèches rouges). 1. Le clivage de la cadhérine 11 libère un fragment contenant les domaines EC1, 2 et 3 dans le milieu extracellulaire. Ce fragment joue probablement deux rôles : inhibition de l’adhésion intercellulaire et modulation de la signalisation cellulaire. 2. ADAM 13 clive la fibronectine, soit pour faciliter la migration des CNCC, soit pour libérer des facteurs de croissance retenus dans la matrice extracellulaire. 3. ADAM13 s’autoclive en CIS. Ce clivage permet une action longue distance du domaine métalloprotéase. Cet autoclivage marque le début d’une cascade protéolytique incluant un clivage dans le domaine transmembranaire par la γ-sécrétase (GS) et un clivage dans le domaine cytoplasmique par une protéase inconnue (X). Au final, le domaine C13 est libéré dans le cytosol et migre dans le noyau où il modifie la transcription de certains gènes impliqués dans la migration des CNCC (exemple : calpaïne 8a). C13 ne possède pas de domaine de liaison à l’ADN évident, suggérant que la modification de l’expression génique qu’il induit fait intervenir l’activité de facteurs de transcription (FT). mb : membrane plasmique. |
|
Meltrines et migration de la crête neurale céphalique de xénope Au cours du développement du xénope, ADAM 13 et 19 sont exprimées dans les cellules de la crête neurale céphalique (CCNC). Lors de la neurulation, ces cellules émergent de la plaque neurale, migrent ventralement et dirigent le développement des structures craniofaciales des vertébrés. Chez le xénope, la migration des CCNC peut être étudiée in vitro en cultivant des explants sur un substrat approprié [
4] ou in vivo en marquant les CCNC avec un traceur cellulaire (GFP, green fluorescence protein) (Figure 2). En injectant des morpholinos antisens pour bloquer la traduction des transcrits ADAM, nous avons pu étudier le rôle de ces derniers dans les CCNC. Nos résultats montrent qu’ADAM 13 et 19 sont nécessaires à la migration des CCNC et que leurs fonctions sont partiellement redondantes [
5]. Afin de déterminer les domaines d’ADAM 13 nécessaires à la migration des CCNC, nous avons remplacé la protéine ADAM 13 endogène par des formes mutantes. Nos résultats montrent que l’activité métalloprotéase et le domaine cytoplasmique sont essentiels. ADAM 13 est capable de cliver plusieurs substrats extracellulaires dont la fibronectine et la cadhérine 11 [
6–
8]. Le clivage de la cadhérine 11 produit un fragment soluble (EC1-3) nécessaire à la migration des cellules in vivo (Figure 1).
 | Figure 2
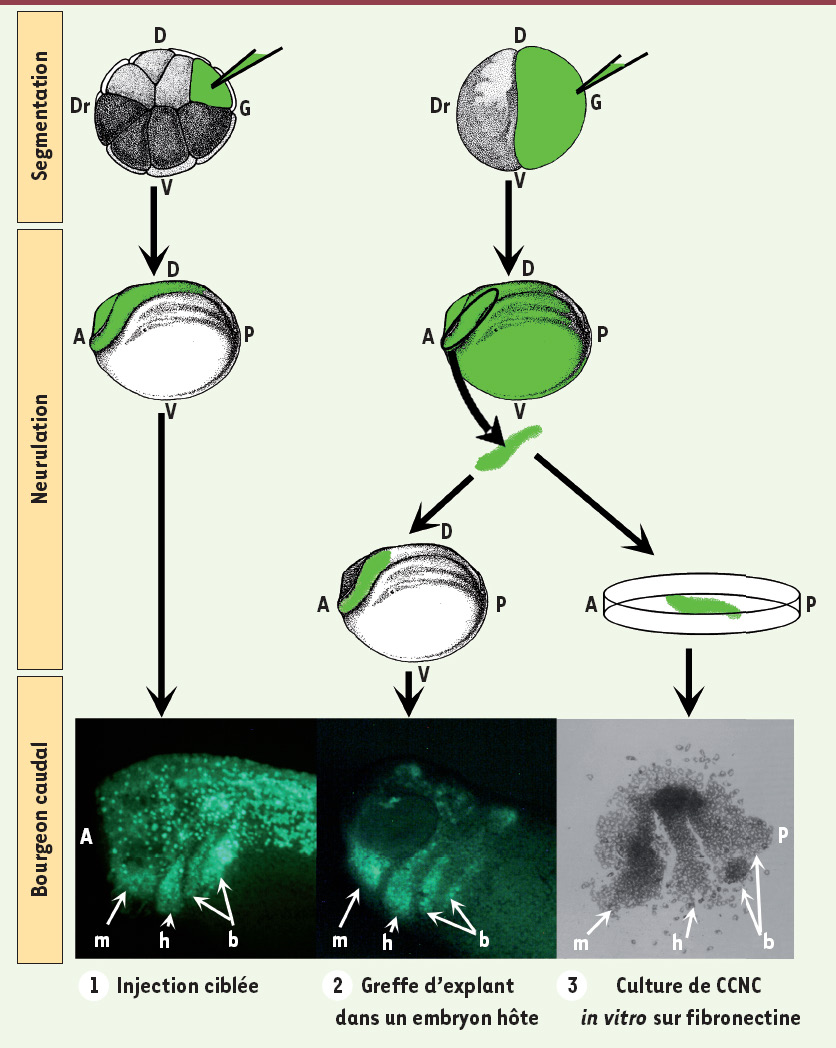
Méthodes d’étude de la migration des CCNC chez le xénope. La migration des CCNC peut être étudiée de trois manières. 1. Injection ciblée. Cette méthode consiste à injecter au stade 8-cellules les composés chimiques désirés (morpholinos, transcrits, etc.) avec un marqueur de lignage cellulaire comme la GFP dans la cellule dorso-animale (territoire présomptif de la crête neurale). Les embryons sont cultivés jusqu’au stade du bourgeon caudal et la présence ou l’absence de migration des CCNC est analysée dans les voies de migration : mandibulaire (m), hyoïde (h) ou branchiales (b). 2. Greffe. Cette méthode consiste à injecter les mêmes composés chimiques dans une cellule au stade 2-cellules. Au stade jeune neurula, les CCNC d’embryons contrôles sont remplacées par les CCNC des embryos injectés. Ces embryons greffés sont élevés jusqu’au stade bourgeon caudal et la présence ou l’absence de migration des CCNC est analysée. 3. Culture de CCNC. La migration des CCNC peut être étudiée in vitro en cultivant les CCNC d’embryons injectés au stade 2-cellules dans une boîte de Pétri sur laquelle de la fibronectine a été préalablement adsorbée. La migration des CCNC peut être analysée soit par imagerie vidéo en time-lapse, soit en analysant la migration au bout de 15 heures de culture. Les photos représentent un agrandissement de la tête d’un embryon au stade bourgeon caudal ou d’un explant de CCNC en culture. A : antérieur, P : postérieur, D : dorsal, V : ventral, G : gauche, Dr : droite. |
|
Rôle surprenant du domaine cytoplasmique d’ADAM 13 Le domaine cytoplasmique d’ADAM 13 (C13) est l’un des plus longs de la famille des ADAM (197 résidus, 21 kDa) et contient des motifs de liaison aux protéines à domaines SH3. Nous avons montré que la protéine adaptatrice PACSIN2 (protein kinase C and casein kinase substrate in neurons protein 2) lie le domaine cytoplasmique d’ADAM 13 et régule son activité [
9]. Récemment, nous avons montré qu’ADAM 13 faisait l’objet d’une série de clivages protéolytiques incluant un autoclivage dans son domaine riche en cystéine [8] et un clivage par la γ-sécrétase. Ces clivages successifs libèrent dans le cytoplasme un fragment de 17 kDa [5] qui migre par la suite dans le noyau. Cette localisation nucléaire est essentielle à la fonction de ADAM 13 dans la migration des CCNC (Figure 1). En analysant l’expression génique des CCNC exposées aux morpholinos ou sauvages par microarray et PCR quantitative (Q-PCR), nous avons découvert que le domaine cytoplasmique d’ADAM 13 modifie l’expression d’un grand nombre de gènes avec une faible amplitude, indiquant que ce domaine est un régulateur d’expression et non pas un activateur ou un inhibiteur de transcription. Parmi les gènes dont l’expression est augmentée par C13, nous avons identifié le gène codant pour la protéase cytoplasmique calpaïne 8a (Capn8a), protéase qui est nécessaire à la migration des CCNC chez le xénope; la réexpression de Capn8a chez les embryons dépourvus de C13 est capable de restaurer partiellement la migration des CCNC. Ceci montre que le rôle d’ADAM 13 dans le noyau est physiologique et qu’une faible variation de l’expression de certains gènes peut avoir des conséquences importantes pour le développement embryonnaire. |
Évolution d’ADAM 13 : l’exception des mammifères placentaires L’identification des orthologues d’ADAM 13 dans les autres espèces est sujette à controverse. Les taux de similitude globale les plus élevés ont été obtenus en alignant les séquences ADAM 13 de xénope avec les séquences ADAM 33 murine et humaine. Par conséquent, ADAM 13 de xénope a été renommée ADAM 33 par les bioinformaticiens du NIH (National institutes of health). Cependant, l’analyse des séquences protéiques des domaines cytoplasmiques des ADAM 33/13 murine et d’amphibien ne montre aucune homologie et aucune conservation du point isoélectrique si typique des meltrines. En revanche, le degré d’homologie des domaines cytoplasmiques d’ADAM 13 de xénope, nématode, poisson zèbre et opossum est similaire au degré d’homologie des autres domaines. Cela remet en question l’orthologie entre les formes d’ADAM 13 chez les mammifères placentaires et chez les autres animaux, incluant les marsurpiaux. En utilisant des tests de complémentation (coexpression, dans les embryons exposés aux morpholinos, de la forme d’ADAM 13 dépourvue de domaine cytoplasmique avec le domaine cytoplasmique cytosoluble), nous avons montré : (1) que les domaines cytoplasmiques d’ADAM 13 d’opossum, poisson zèbre et nématode sont capables de restaurer la migration des CNC, alors qu’ADAM 33 murine en est incapable; (2) que le domaine cytoplasmique d’ADAM 33 murine ne migre pas dans le noyau des cellules. Des analyses génomiques ont montré que les marqueurs chromosomiques en 5’ de la séquence d’ADAM 13 de X. tropicalis et d’ADAM 33 humain sont conservés alors que ceux présents en 3’ sont différents [
10]. Ces observations suggèrent qu’au cours de la divergence des mammifères placentaires et des marsurpiaux, un remaniement chromosomique a probablement éliminé le domaine cytoplasmique. Malgré cette divergence de fonctionnalité d’ADAM 33 chez les mammifères placentaires, nous pensons que les meltrines jouent un rôle clé dans le dévelopemment craniofacial de ces animaux. Au cours de l’évolution, la meltrine originelle (ADAM 2 chez le nématode) a été quadruplée. Nos expériences de complémentation ont montré que les domaines cytoplasmiques des ADAM 19 du xénope et de souris sont capables de restaurer la migration des CCNC en l’absence du domaine cytoplasmique d’ADAM 13. Par ailleurs, ADAM 9, comme ADAM 13, clive la cadhérine 11 chez le xénope. Ces données suggèrent qu’ADAM 9 et ADAM 19 peuvent compenser la perte d’ADAM 13. Notre hypothèse est que chez les mammifères placentaires, ADAM 9 et 19 remplissent séparément les fonctions qu’ADAM 13 assure chez les autres vertébrés. |
Le problème des analyses quantitatives chez les animaux non consanguins Lors de nos analyses de Q-PCR et de microarray, nous avons pu observer que la variation de l’expression des gènes était plus importante d’un lot d’embryon à l’autre que d’un cas expérimental à un autre pour un même lot. En d’autres termes, la différence d’expression naturelle d’un gène d’un groupe d’embryons à un autre est plus importante que celle induite par la perte d’ADAM13. Étant donné que le xénope est une espèce non consanguine, cette observation n’est pas très surprenante mais pose un problème pour l’analyse des résultats de microarray et de Q-PCR. Au lieu de faire une analyse globale des différents microarray et laisser les analyses statistiques identifier les gènes d’intérêt, nous avons dû réaliser des analyses couplées pour chaque expérience (provenant d’une seule fécondation) et comparer les résultats de plusieurs expériences indépendantes pour identifier les gènes dont l’expression changeait significativement. Cette observation a deux conséquences. D’une part, lorsqu’une étude est faite avec des animaux non consanguins, les analyses moléculaires doivent être réalisées entre cas contrôles et expérimentaux strictement issus d’une même ponte. D’autre part, les outils et l’analyse moléculaire doivent être adaptés à l’organisme modèle. En effet, bien que les populations syngéniques telles que la souris et le poisson zèbre présentent de nombreux avantages, le xénope nous rappelle que, comme pour les humains, les variations entres individus sont souvent plus importantes que celles induites par un état pathologique. |
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
Les auteurs remercient Jessica Escoffier et Ludovic Giloteaux pour leurs commentaires sur ce manuscrit. Ce travail a été subventionné par le NIH (DE016289).
|
1.
Alfandari
D
,
McCusker
C
,
Cousin
H
. ADAM function in embryogenesis . Semin Cell Dev Biol.
2009; ; 20 : :153.–163. 2.
Maretzky
T
,
Reiss
K
,
Ludwig
A
, et al.
ADAM10 mediates E-cadherin shedding and regulates epithelial cell-cell adhesion, migration, and beta-catenin translocation . Proc Natl Acad Sci USA.
2005; ; 102 : :9182.–9187. 3.
Reiss
K
,
Maretzky
T
,
Ludwig
A
, et al.
ADAM10 cleavage of N-cadherin and regulation of cell-cell adhesion and beta-catenin nuclear signalling . EMBO J.
2005; ; 24 : :742.–752. 4.
Alfandari
D
,
Cousin
H
,
Gaultier
A
, et al.
Integrin alpha5beta1 supports the migration of Xenopus cranial neural crest on fibronectin . Dev Biol.
2003; ; 260 : :449.–464. 5.
Cousin
H
,
Abbruzzese
G
,
Kerdavid
E
, et al.
Translocation of the cytoplasmic domain of ADAM13 to the nucleus is essential for Calpain8-a expression and cranial neural crest cell migration . Dev Cell.
2011; ; 20 : :256.–263. 6.
Alfandari
D
,
Cousin
H
,
Gaultier
A
, et al.
Xenopus ADAM 13 is a metalloprotease required for cranial neural crest- cell migration . Curr Biol.
2001; ; 11 : :918.–930. 7.
McCusker
C
,
Cousin
H
,
Neuner
R
,
Alfandari
D
. Extracellular cleavage of cadherin-11 by ADAM metalloproteases is essential for Xenopus cranial neural crest cell migration . Mol Biol Cell.
2009; ; 20 : :78.–89. 8.
Gaultier
A
,
Cousin
H
,
Darribere
T
,
Alfandari
D
. ADAM 13 disintegrin and cysteine-rich domains bind to the Hep II domain of fibronectin . J Biol Chem.
2002; ; 27 : :23336.–23344. 9.
Cousin
H
,
Gaultier
A
,
Bleux
C
, et al.
PACSIN2 is a regulator of the metalloprotease/disintegrin ADAM13 . Dev Biol.
2000; ; 227 : :197.–210. 10.
Wei
S
,
Whittaker
CA
,
Xu
G
, et al.
Conservation and divergence of ADAM family proteins in the Xenopus genome . BMC Evol Biol.
2010; ; 10 : :211.. 11.
Peiretti
F
,
Canault
M
,
Morange
P
,
Alessi
MC
,
Nalbone
G
. Les deux visages d’ADAM17 dans l’inflammation : implications dans l’athérosclérose et l’obésité . Med Sci (Paris).
2009; ; 25 : :45.–50. |