Microscopie de fluorescence traditionnelle : les limites du contraste et de la résolution Développée au début du xx
e siècle, la microscopie de fluorescence a connu une véritable explosion depuis une trentaine d’années avec l’avènement des ordinateurs, l’apparition de nouvelles technologies de détection, et surtout avec l’émergence d’une multitude de sondes fluorescentes et de méthodes de marquage spécifiques [
1]. Elle est devenue un outil indispensable à la recherche biologique et médicale pour la localisation in situ d’une ou, simultanément, de plusieurs structures intracellulaires, leur suivi spatiotemporel et la détection de leurs interactions [
2]. Le contraste et la résolution de la microscopie de fluorescence conventionnelle - dite plein champ - se dégradent fortement pour des échantillons épais (à partir de 10 μm) et/ou diffusants, en raison du signal parasite provenant de la fluorescence émise hors du plan focal. Pour s’affranchir de celle-ci, trois principales approches de sectionnement optique ont été développées pour observer des sections minces dans des échantillons volumiques : (a) la déconvolution d’images enregistrées en microscopie de fluorescence traditionnelle [
3] ; (b) la microscopie confocale à balayage laser [
4] ; et (c) l’excitation biphotonique ou multiphotonique [
5,
6]. Le sectionnement optique par déconvolution Il peut être réalisé sur des données provenant d’un microscope traditionnel muni d’un positionneur piézoélectrique (1 nm de résolution) monté sur l’objectif. Après acquisition, les images 2D des différents plans sont traitées numériquement afin d’éliminer la contribution hors plan focal. Toutes les techniques de déconvolution consistent à inverser la relation objet-image après avoir déterminé théoriquement ou expérimentalement la réponse impulsionnelle PSF ( point spread function) du microscope [ 4]. Cependant, elles reposent sur de nombreuses hypothèses à propos des propriétés optiques de l’échantillon et des performances du microscope qui, en raison des effets d’absorption et d’aberrations, ne sont pas forcément vérifiées. La microscopie confocale à balayage laser CLSM (confocal laser scanning microscopy) Elle utilise un diaphragme placé devant le photodétecteur qui permet de rejeter la lumière parasite émise par les fluorophores des plans situés hors du plan focal [ 4]. Le diamètre réglable de ce diaphragme conditionne en grande partie l’épaisseur de la coupe optique et donc la résolution axiale. Le CLSM est devenu la technique de référence pour l’imagerie 3D haute résolution de systèmes biologiques allant de la protéine aux tissus et a trouvé de nombreuses applications cliniques. Toutefois, malgré des progrès récents, la microscopie confocale connaît encore des limites majeures. En effet, le diaphragme étant de taille très inférieure au champ d’observation, il faut effectuer un balayage point par point du champ pour reconstruire une image de l’objet étudié. Cette détection sélective et séquentielle limite considérablement la résolution temporelle (≥ 1 μs/pixel) et induit une dégradation rapide de l’échantillon par photoblanchiment, phototoxicité, ou effet thermique. Par ailleurs, la plupart des composants biologiques ayant des coefficients d’absorption élevés dans le domaine du visible et de l’ultraviolet, la profondeur de pénétration reste très limitée (de 50 à 100 μm) [
7]. Enfin, bien que les photodiodes à avalanches offrent une excellente sensibilité (rendement quantique élevé et faible courant de fuite), le rapport signal/bruit est limité par le très faible nombre de photons émis et atteignant le détecteur (de 20 à 100 photons/pixel). Une solution pour améliorer la résolution temporelle (jusqu’à 500 images/s) est de remplacer le diaphragme par un disque rotatif (> 1 000 tours/s) percé d’environ 20 000 trous (appelé disque de Nipkow) qui permet d’observer simultanément avec un capteur CCD plusieurs volumes d’excitation : c’est le principe de microscopes confocaux multipoints (ou spinning disk). La transmission de la lumière peut être augmentée significativement par l’adjonction d’une matrice de microlentilles introduite par Yokogawa [
8]. Comme pour toute méthode « multiplexée », un soin particulier doit être apporté à l’alignement afin d’éviter la diaphonie entre trous voisins. La microscopie de fluorescence sous excitation biphotonique (ou multiphotonique) Basée sur des interactions non linéaires lumière-matière, elle a la particularité d’effectuer des coupes optiques en confinant l’excitation au volume focal, restreignant ainsi la photodégradation uniquement à ce volume. Puisque tous les photons émis contiennent des informations utiles, aucun filtrage spatial par un diaphragme n’est requis, ce qui augmente considérablement le rapport signal/bruit. L’excitation biphotonique provient de l’absorption quasi simultanée de deux photons par un fluorophore pour atteindre son état excité [ 5]. La section efficace d’absorption à deux photons de la plupart des fluorophores étant très faible, l’excitation à deux photons nécessite une intensité instantanée élevée qui n’a été rendue possible que par le développement des sources laser à impulsions ultra brèves (< 1 ps) [
9]. Un des avantages majeurs de cette microscopie non linéaire est l’utilisation de photons infrarouges (typiquement 700-1 100 nm) qui sont moins diffusés et absorbés (10 fois moins) par les tissus que ceux du domaine visible ou UV, ce qui permet une meilleure pénétration de la lumière dans les tissus (jusqu’à 700 μm) [
10]. Mais, les longueurs d’onde utilisées étant plus grandes qu’en excitation à un photon, la résolution spatiale est moins bonne qu’en microscopie confocale. Par ailleurs, bien que réduits au volume focal, les dommages engendrés au milieu cellulaire suite à une excitation biphotonique sont difficiles à caractériser. Certaines études ont, toutefois, pu montrer que les très fortes densités de puissance utilisées en régime femtoseconde (quelques TW/cm 2) induisent des dommages photothermiques principalement dus à l’échauffement du solvant, ou des dommages photochimiques qui peuvent compromettre la viabilité des cellules et endommager les tissus [
11]. Mais, ce qui freine le plus sa démocratisation dans les laboratoires est le coût élevé et la relative complexité de sa mise en œuvre. Les différentes techniques d’imagerie tomographique optique OPT ( optical projection tomography) [
12], OCT ( optical coherence tomography) [
13] et leurs variantes permettent également de produire des images en coupe axiale à des profondeurs de quelques millimètres avec une résolution spatiale isotrope de quelques μm et un champ de vision très large (> 1 mm), ce qui en fait d’excellentes techniques pour l’imagerie de tissus épais. Toutefois, l’OCT n’utilise pas la fluorescence de l’échantillon et souffre d’une résolution temporelle modérée (< 100 images/s). Quant à l’OPT, le sectionnement optique n’est pas en temps réel, car il est obtenu après fusion des données tomographiques en une image 3D qui nécessite un long traitement d’images. D’autres méthodes de sectionnement optique Elles ont été récemment développées comme les microscopies interférentielles 4Pi et I 5M ( incoherent interference illumination image interference microscopy), le STED ( stimulated emission depletion), ou les diverses formes de microscopie d’illumination structurée (SIM ou structured illumination microscopy) (pour revue voir [
14]). Ces techniques permettent en outre de dépasser la limite de diffraction mais elles requièrent un équipement sophistiqué et de lourdes procédures d’alignement, et elles sont moins adaptées à l’imagerie d’échantillons vivants épais en raison de leur faible résolution temporelle, leur faible profondeur de pénétration et leur effet de photodégradation. Basée sur la microscopie confocale thêta [
15], la microscopie de fluorescence à feuille de lumière (MFL) utilise une approche totalement différente. En effet, des coupes optiques non invasives à hautes résolutions spatiale et temporelle sont obtenues en illuminant avec une feuille de lumière un échantillon sous différents angles et en observant la lumière émise avec un microscope de fluorescence traditionnelle suivant un axe perpendiculaire au plan de cette feuille. Dans les paragraphes qui suivent, nous passerons en revue les principes qui sont à la base de la MFL, les différents éléments qui la composent et nous verrons comment l’adapter pour des applications spécifiques. |
Une approche différente : la microscopie de fluorescence à feuille de lumière (MFL) Principe Le principe des techniques de MFL est d’éclairer latéralement, à l’aide d’une feuille de lumière, une tranche d’un échantillon perpendiculairement à un objectif de détection qui produit une image en champ lointain du plan illuminé. Les systèmes d’illumination et de détection sont alors complètement indépendants l’un de l’autre. Le sectionnement optique est obtenu en superposant le plan focal du système de détection avec la feuille de lumière. Même s’il existe différentes implémentations de cette idée (pour revue voir [
17]), le principe général reste le même et est illustré dans la
Figure 1
. La configuration la plus répandue, appelée SPIM ( selective plane illumination microscopy), consiste à utiliser une lentille cylindrique couplée avec un objectif pour créer la feuille de lumière (Figure 2) [
16]. Celle-ci peut aussi être générée en focalisant un faisceau laser et en le balayant verticalement pendant toute la durée de l’acquisition. Ce concept fut récemment introduit avec le DSLM ( digital scanned laser light-sheet fluorescence microscope) [
18,
19]. Un des intérêts du DSLM est d’uniformiser le profil d’intensité de la feuille de lumière sans utiliser de système de mise en forme du faisceau et de contrôler la hauteur du balayage pour améliorer au maximum l’efficacité de l’illumination.
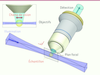 | Figure 1.
Principe de la microscopie à feuille de lumière. Les systèmes d’illumination (en bleu) et de détection (en vert), placés à la perpendiculaire (ou proche de la perpendiculaire) l’un de l’autre, sont complètement indépendants. L’échantillon (en jaune) est illuminé latéralement en focalisant un faisceau laser collimaté en une feuille de lumière à l’aide d’une lentille cylindrique [ 16]. Cette feuille de lumière coïncide avec le plan focal de l’objectif du système de détection. La fluorescence provenant de la tranche illuminée de l’échantillon est alors enregistrée par une caméra CCD. Dans l’insert est représentée une vue du dessus de la feuille de lumière au niveau de l’échantillon. |
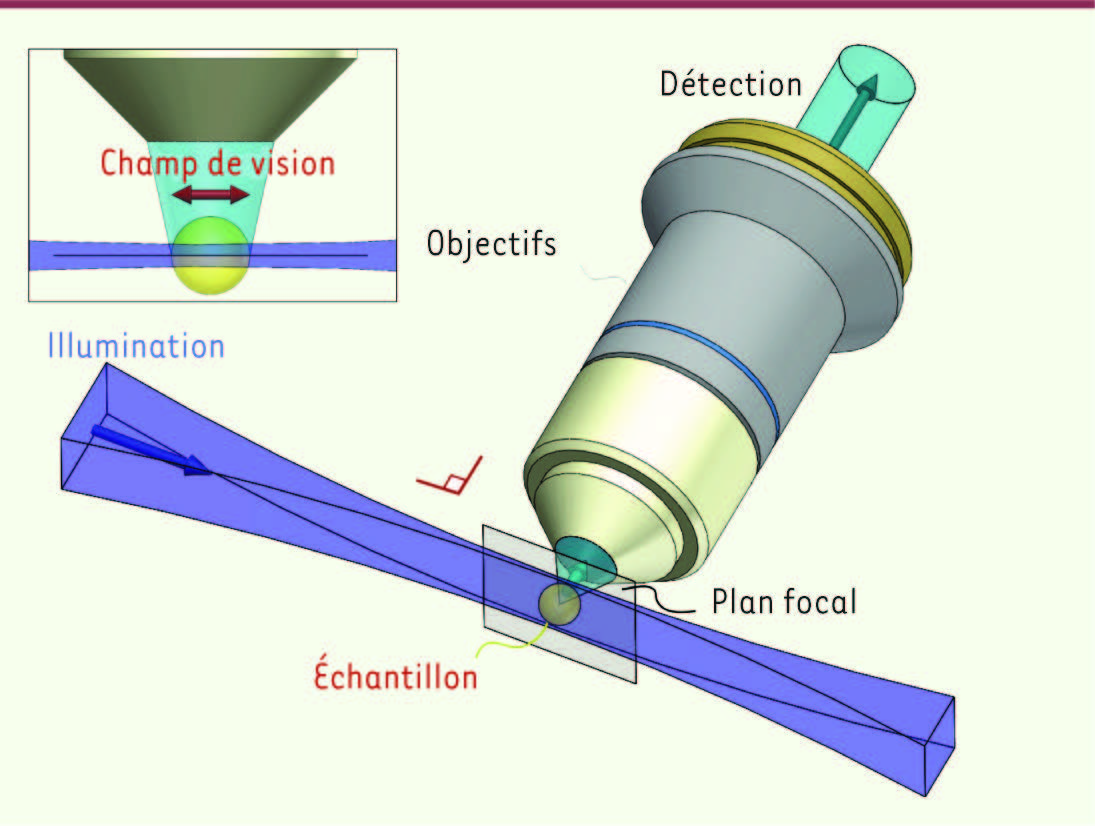
 | Figure 2.
Vue schématique du SPIM. A. Le faisceau de lumière provenant de un ou plusieurs lasers est mis en forme (AF = agrandisseur de faisceau, FR = fente rectangulaire ajustable) et focalisé (LC = lentille cylindrique, Obj1 = objectif) pour générer une feuille de lumière dans le plan focal de l’objectif de détection. Un filtre acousto-optique (AOTF) permet de sélectionner la longueur d’onde d’excitation provenant d’une ou de plusieurs sources laser et de contrôler précisément l’exposition de l’échantillon. Le signal de fluorescence de l’échantillon est ensuite focalisé par un second objectif Obj2 puis filtré (F1 et F2) pour être enregistré par un ou deux capteurs matriciels CCD (ou CMOS). Un système laser de photomanipulation (FRAP, photoactivation ou microdissection) peut être utilisé, de manière optionnelle, en parallèle à travers l’objectif de détection Obj2 et focalisé sur l’échantillon. LT : lentille de tube ; MD = miroir dichroïque, MFF = système de mise en forme du faisceau. Les pointillés indiquent les différentes options possibles. B. L’échantillon est en général enfermé dans un gel mou aqueux comme l’agarose et le tout est placé dans une chambre à perfusion (vue en coupe) remplie d’une solution aqueuse (eau, milieu cellulaire) dont on peut contrôler les paramètres physiologiques (température, pH, CO2). C. Pour l’imagerie en volume, l’échantillon est déplacé par rapport à la feuille de lumière grâce à trois moteurs de translation (X, Y, Z) et un moteur de rotation (θ). |
Dans une MFL, le système de détection consiste en un microscope à épifluorescence classique : un objectif, un filtre d’émission et une lentille de tube qui forme l’image sur une caméra CCD. Deux fluorophores peuvent être imagés de manière synchrone si l’on utilise un miroir dichroïque qui sépare spectralement la fluorescence et la redirige sur deux caméras, ou de manière séquentielle en utilisant une roue à filtre devant une seule caméra (Figure 2). L’échantillon est généralement immergé dans une chambre d’observation remplie d’un milieu approprié (eau, tampon, etc.) et dont les paramètres physiologiques (T, pH, CO2, etc.) peuvent être contrôlés (Figure 2B). La préparation de l’échantillon est conditionnée par sa nature (cellules, embryons, organes, etc.) et ses dimensions, mais également par l’implémentation de la MFL choisie. Dans le cas du SPIM/DSLM, l’échantillon est le plus souvent enfermé dans un gel aqueux (agarose, collagène, gomme gellane, etc.) [
20], ou dans une poche transparente (en plastique ou moulée dans un gel) [
21], afin de limiter les mouvements de l’échantillon tout en permettant son développement (Figure 2C). Imagerie multivues Une des caractéristiques essentielles de la MFL est de pouvoir obtenir une image d’un échantillon sous différents angles. Des piles d’images 3D, enregistrées suivant deux ou plusieurs directions du même échantillon, sont ensuite fusionnées pour aboutir au volume final représentant l’objet (Figure 3). L’acquisition est généralement réalisée par de multiples objectifs disposés autour de l’échantillon qui reste fixe [
22,
23] ou en tournant l’échantillon pour l’imager sous différentes directions avec un seul objectif [ 20]. L’imagerie multivues présente deux intérêts majeurs : (1) dans des tissus diffusants et absorbants, la qualité des images est améliorée et devient plus uniforme (Figure 4B, D ; (2) dans les échantillons transparents, la résolution devient isotrope. Différentes techniques de reconstruction de multivues ont été développées (voir comme exemples [
24,
25]). De plus, l’excellent rapport signal/bruit de la MFL permet l’utilisation simultanée de techniques de traitement d’images, comme la déconvolution, et la fusion des différentes vues [
26]. Ces techniques de reconstruction 3D ont été utilisées avec succès sur des embryons de medaka [ 20], des embryons de poisson zèbre (Figure 4B), des follicules (Figure 4A et
Vidéo 1
1,
) et des embryons de drosophile [ 19, 24, 25], des drosophiles adultes (Figure 4D et
Vidéo 2
1
) et de cellules épithéliales sous forme de kystes ou de sphéroïdes [
27]. Bien que ces techniques nécessitent des ressources et des puissances importantes de calcul, les développements constants et rapides des outils tant matériels que logiciels les rendent aujourd’hui plus accessibles.
 | Figure 3.
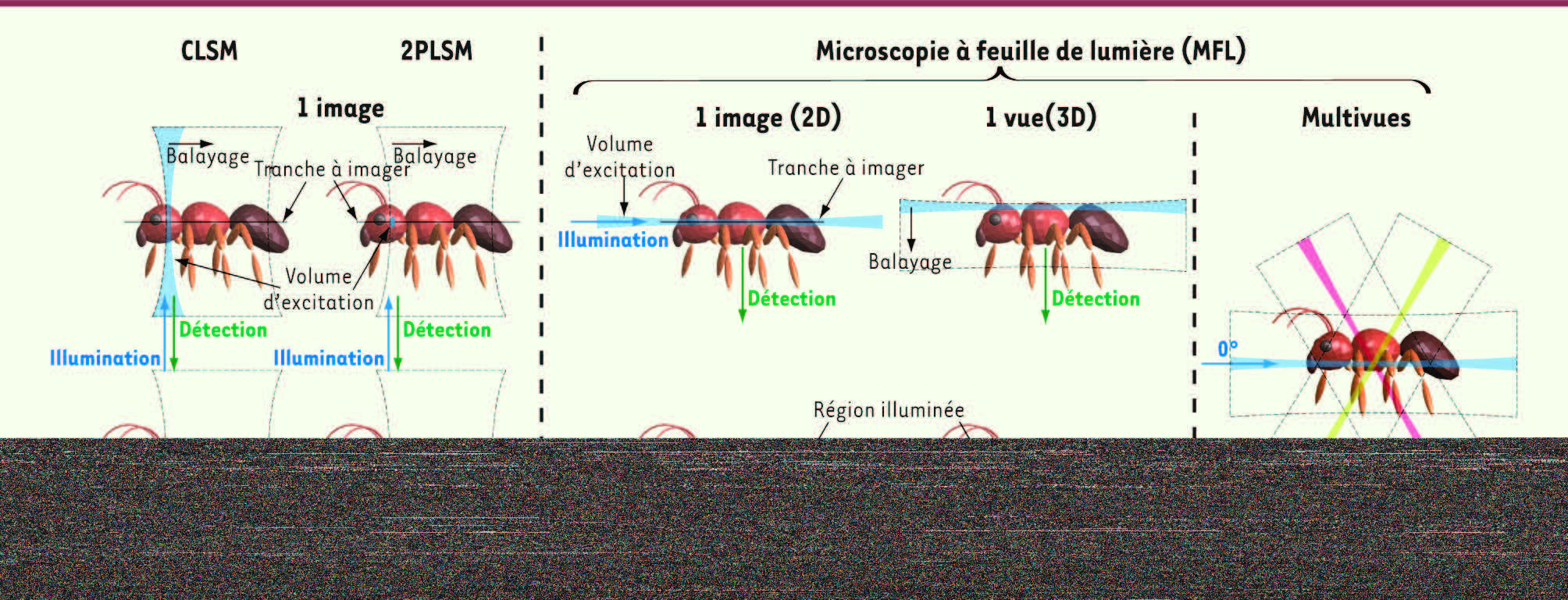
Différence d’illumination en microscopies confocale, biphotonique et à feuille de lumière. En microscopie confocale (CLSM), l’illumination (en bleu) par un diaphragme oblige à effectuer un balayage de la surface de la tranche optique d’intérêt pour reconstruire une image de l’objet étudié (en haut, à gauche). Puisque l’illumination et la détection (en vert) se font par le même objectif, tout l’échantillon est pratiquement illuminé (zone noire en bas), bien qu’un seul plan de l’échantillon soit réellement imagé. En microscopie biphotonique à balayage laser (2PLSM), le volume d’excitation (en bleu clair) est réduit à la région autour du plan d’intérêt, mais les puissances d’excitation souvent élevées peuvent compromettre la viabilité des échantillons. En microscopie à feuille de lumière, l’illumination latérale permet d’exciter uniquement le plan de détection et aucun photo-dommage n’est alors infligé en dehors de ce plan. Une vue de l’échantillon est réalisée en déplaçant l’échantillon latéralement à travers la feuille de lumière et en enregistrant à chaque étape une image. En imagerie multivues (à droite), le même volume est enregistré suivant plusieurs directions. Les informations provenant des différentes vues sont ensuite combinées pour former une seule pile d’images. |
 | Figure 4.
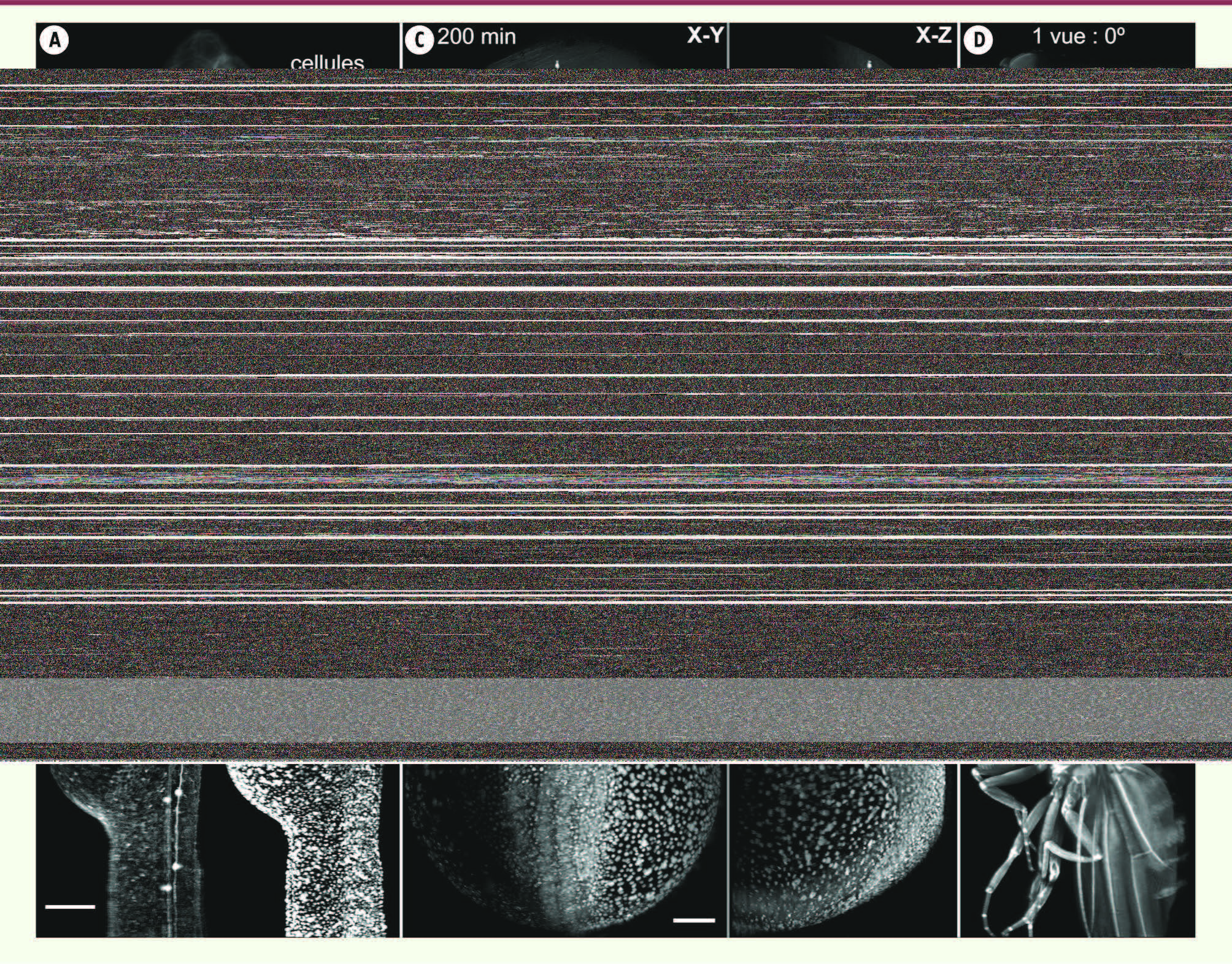
Images obtenues en microscopie à feuille de lumière. A. Follicule de drosophile melanogaster marqué actine-GFP (préparé par A. Ephrussi et E. Reynaud, EMBL-Heidelberg, Allemagne) : vue 3D en projection d’intensité maximale du volume reconstruit à partir de 12 piles de 120 images (voxel = 0,3225 μm × 0,3225 μm × 1,29 μm). Encart : vue d’un pont cytoplasmique reliant 2 cellules nourricières. Vidéo 1 2. B. Embryon d’un poisson zèbre Danio rerio marqué GFP-Claudine B (à gauche) et mCherry-H2B (à droite) (préparé par U. Schulze et D. Gilmour, EMBL-Heidelberg, Allemagne) : vues 3D en projection d’intensité maximale du volume reconstruit à partir de 16 piles de 576 images prises à 22,5 unités d’intervalle (voxel = 0,74 μm × 0,74 μm × 1,48 μm). C. Embryon d’un poisson zèbre Danio rerio marqué H2B-eGFP à plusieurs stades du développement embryonnaire : t = 200, 650 et 1 200 min après fertilisation. Images adaptées de [ 18]. Animation complète sur http://www.embl.de/digitalembryo/fly.html. D. Drosophile melanogaster adulte obtenue par autofluorescence. Comparaison entre 3 vues obtenues en projection d’intensité maximale du volume reconstruit à partir d’une pile de 778 images prises à l’angle 0 o (en haut), d’une pile de 778 images prises à l’angle 180 o (au milieu) et de 32 piles de 778 images prises à 11,25 o d’intervalle (en bas) (voxel = 0,74 μm × 0,74 μm × 2,96 μm). Dans les vues 0 o et 180 o, la drosophile est éclairée par la gauche mais en raison de son opacité, la partie de droite apparaît manquante. Ces parties manquantes sont révélées en illuminant l’échantillon suivant différentes directions. En additionnant toutes ces informations, il devient possible de reconstruire une vue 3D de l’échantillon avec une excellente résolution spatiale (en bas). Vidéo 2 2. Objectifs d’illumination : A. Zeiss Epiplan 10x/0,2 ; B-D. Zeiss Plan-Apochromat 5x/0,16. Objectifs de détection : A. Zeiss Achroplan W 20x/0,5 ; B-C. Zeiss C-Apochromat W 10x/0,45 ; D. Zeiss Fluar 5x/0,25. Barres d’échelle : A. 20 μm, B. 50 μm, C. 100 μm, D. 400 μm. |
Dimensions de la feuille de lumière et résolution L’épaisseur W de la feuille de lumière, et donc de la tranche optique, est inversement proportionnelle à l’ouverture numérique (ON) de l’objectif d’illumination (Figure 5). Elle est typiquement de 1 à 10 μm et doit être ajustée de façon à ce que l’extension L du champ de vision corresponde au plus à deux longueurs de Rayleigh. Au-delà, l’élargissement de la feuille de lumière devient non négligeable (Figure 5). En imagerie simple vue, l’épaisseur W définit aussi la résolution axiale du système (voir Encadré). Cette résolution sera donc d’autant plus fine que l’ON de l’objectif d’illumination sera grande. La résolution axiale d’une MFL est meilleure que celle d’un microscope à épifluorescence ou que celle d’un microscope biphotonique. Pour des objectifs d’illumination ayant une ON inférieure à 0,6-0,8, elle est également meilleure qu’en microscopie confocale [
28]. De plus, l’utilisation d’un objectif d’illumination avec une ON considérablement plus faible que l’ON de l’objectif de détection assure une meilleure profondeur de pénétration. Théoriquement, la résolution latérale en MFL est équivalente à celle d’un microscope à épifluorescence. En pratique, elle est cependant souvent meilleure, grâce à l’amélioration du contraste par le sectionnement optique. De plus, la résolution peut être améliorée de manière significative par la propriété tomographique de la MFL. Dans ce cas, la fusion des différentes vues obtenues sous différents angles conduit à une résolution isotrope égale à la résolution latérale [ 24, 28].
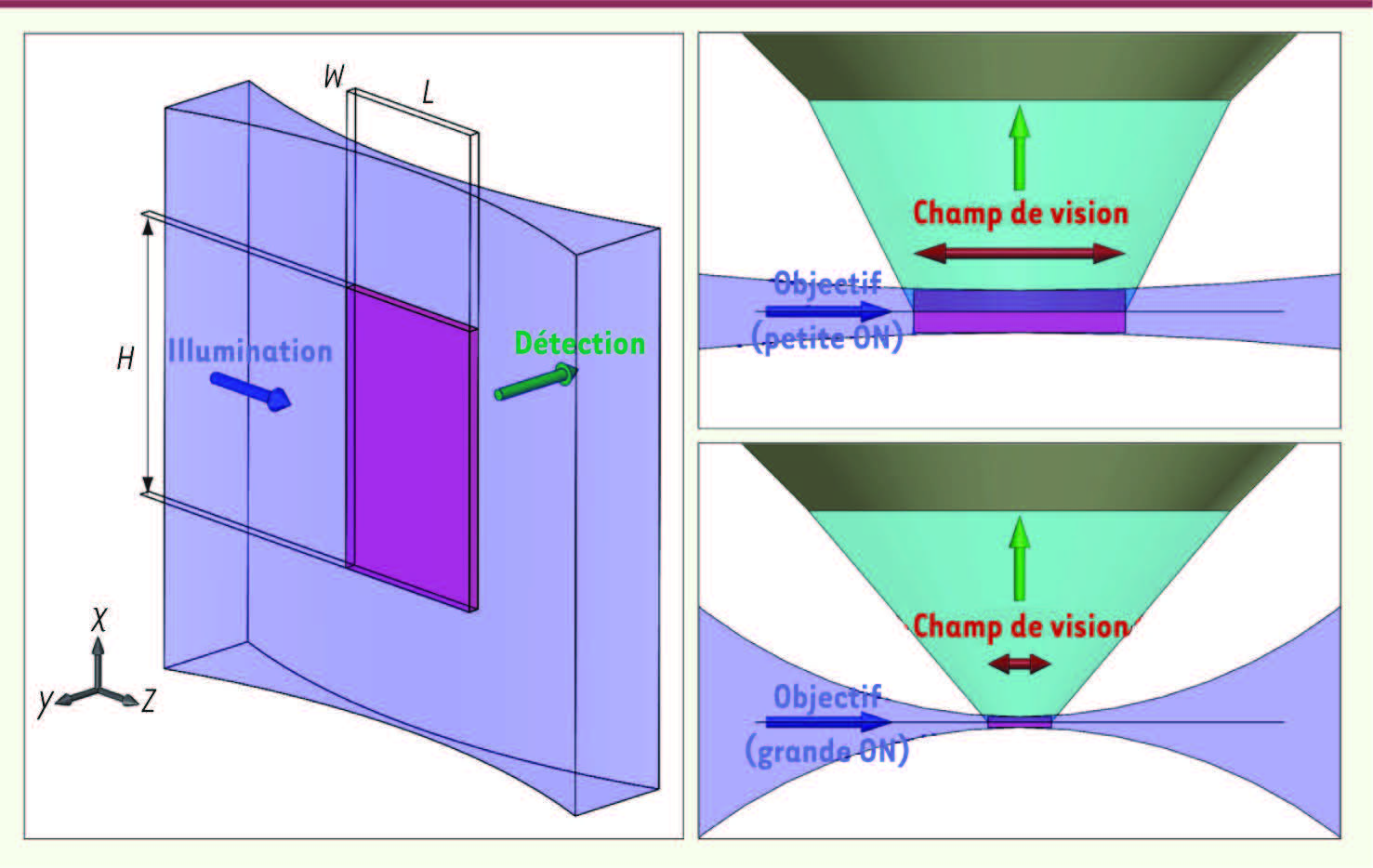
 | Figure 5.
Dimensions de la feuille de lumière dans un MFL. La feuille de lumière a un profil gaussien dans les deux directions X et Y (à gauche). Elle doit être suffisamment large suivant X pour illuminer la hauteur H du champ de vision. Pour le SPIM, l’extension de la feuille de lumière suivant X est > 10 fois plus grande que H et seule la moitié de la puissance totale d’illumination est utilisée. Dans le cas du DSLM, l’extension de la feuille de lumière suivant X est choisie pour être du même ordre de grandeur que H et la quasi-totalité de la puissance d’illumination est utilisée. Dans la direction Y, la feuille de lumière est fine au centre du champ de vision du système de détection (objectif + caméra) et s’élargit aux bords. En raison de cet élargissement, la résolution se dégrade lorsque l’on s’éloigne du centre. Il faut donc ajuster les ouvertures numériques (ON) des systèmes d’illumination et/ou de détection de telle manière que les bords du champ de vision soient au plus à une longueur de Rayleigh du centre, longueur au-delà de laquelle cette dégradation devient non négligeable et qui correspond à un faisceau  fois plus épais qu’au centre. La longueur L du champ de vision doit alors vérifier la condition : L 2 W
2/λ avec W l’épaisseur de la feuille de lumière définie comme la largeur du profil gaussien suivant Y. Pour vérifier cette condition, les objectifs d’illumination à grandes ON (en bas, à droite) ont une épaisseur W et une longueur L (et donc un volume détecté) plus faibles que les objectifs à petites ON (en haut, à droite). |
|
Résolution spatiale d’un microscope Une propriété déterminante de la qualité d’un microscope est la résolution spatiale qui est caractérisée par la réponse impulsionnelle PSF : il s’agit de l’image formée à travers le microscope d’un point source lumineux. En effet, en raison de la diffraction de la lumière due à l’ouverture limitée de l’objectif, l’image d’un point vue au travers d’un instrument optique n’est pas un point mais une tache d’autant plus grande que l’ouverture sera limitée et le grandissement recherché élevé. Les extensions de la PSF dans les directions transversale et axiale permettent alors de quantifier le pouvoir de résolution du système optique. La résolution est définie comme la distance minimale séparant deux objets ponctuels, afin qu’ils soient discernables, ce qui correspond à la largeur à mi-hauteur (full width at half-maximum ou FWHM) de la PSF [
41]. La théorie de Stelzer-Grill-Heisenberg (ou SGH) [4, 28] permet d’estimer les résolutions latérale σxy et axiale σz d’une lentille sphérique qui sont également valables pour les microscopes classiques : où λ est égale à la longueur d’onde de détection λdet pour les microscopes traditionnels à épifluorescence tandis que  pour les microscopes confocaux (avec λill la longueur d’onde d’illumination), n l’indice du milieu dans lequel la lumière se propage et α le demi-angle d’ouverture du cône lumineux produit par l’objectif. En microscopie à feuille de lumière, l’éclairage par un plan n’a aucun effet sur la résolution latérale σxy qui est alors décrite par l’expression précédente tandis que la résolution axiale σz est gouvernée par la séparation du système de détection et de l’illumination par une feuille de lumière. Dans ce cas, la contribution du système de détection reste celle d’une lentille sphérique, soit pour les microscopes confocaux (avec λill la longueur d’onde d’illumination), n l’indice du milieu dans lequel la lumière se propage et α le demi-angle d’ouverture du cône lumineux produit par l’objectif. En microscopie à feuille de lumière, l’éclairage par un plan n’a aucun effet sur la résolution latérale σxy qui est alors décrite par l’expression précédente tandis que la résolution axiale σz est gouvernée par la séparation du système de détection et de l’illumination par une feuille de lumière. Dans ce cas, la contribution du système de détection reste celle d’une lentille sphérique, soit  où a est le demi-angle d’ouverture de l’objectif de détection et où l’ouverture numérique vaut ON = nsinα. Quant à la contribution du système d’illumination, elle est décrite par la théorie SGH d’une lentille cylindrique de faible ouverture numérique où a est le demi-angle d’ouverture de l’objectif de détection et où l’ouverture numérique vaut ON = nsinα. Quant à la contribution du système d’illumination, elle est décrite par la théorie SGH d’une lentille cylindrique de faible ouverture numérique  où L est l’extension horizontale du champ de vision et W l’épaisseur de la feuille de lumière
(Figure 5)
. Au final, la résolution axiale totale d’une MFL est donnée par la relation : où L est l’extension horizontale du champ de vision et W l’épaisseur de la feuille de lumière
(Figure 5)
. Au final, la résolution axiale totale d’une MFL est donnée par la relation :
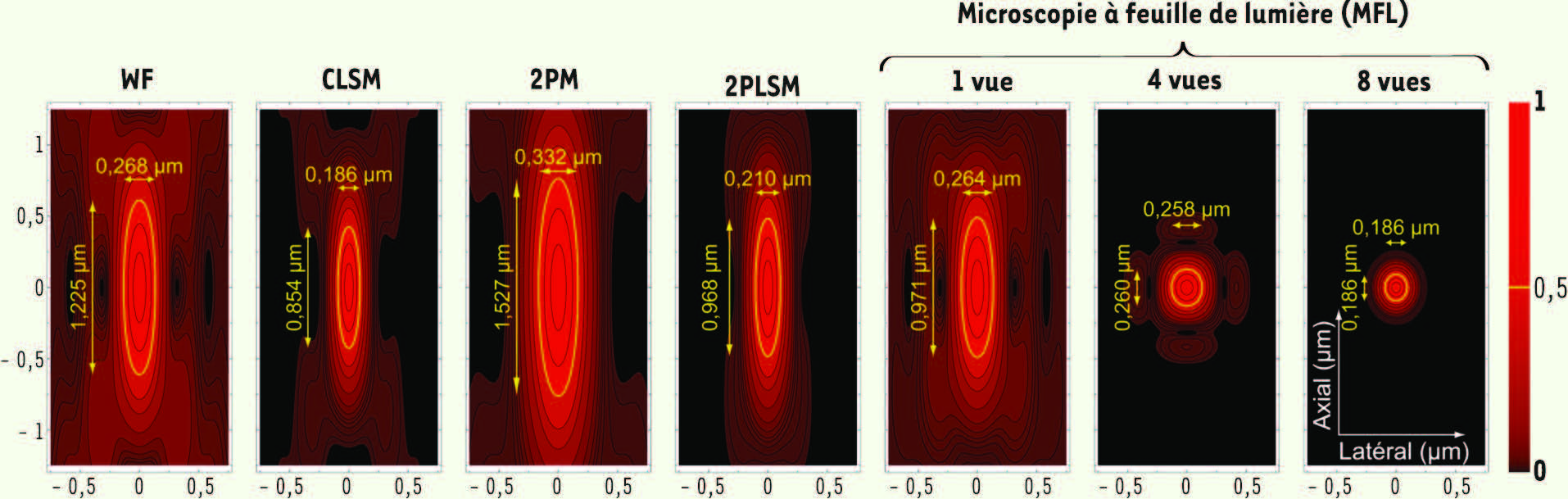
Les PSF théoriques pour différents types de microscope sont représentées sur la
Figure S1
. En imagerie simple vue, la microscopie confocale CLSM possède les meilleures résolutions axiale et latérale. Cependant, dès la fusion de 8 vues, la résolution de la MFL devient identique à la résolution latérale du CLSM mais elle est, en plus, isotrope.
 | Figure S1.
PSF théoriques calculées pour différents types de microscopes. WD : microscope traditionnel à épifluorescence; CLSM : microscope confocal à balayage laser; 2PM : microscope biphotonique; 2PLSM : microscope biphotonique à balayage laser ; MFL : microscope à feuille de lumière (1, 4 et 8 vues). Toutes les PSF sont calculées, dans les approximations de symétrie sphérique et de polarisation circulaire, avec la longueur d’onde d’illumination λill = 488 nm sauf pour 2PM et 2PLSM λ2p_ill = 900 nm, la longueur d’onde de détection λdet = 520 nm, un objectif à immersion à eau (n = 1,33) avec une ouverture numérique ON = 1,0 et pour l’illumination du LSM un objectif à air avec une ouverture numérique ON = 0,16. Les lignes de niveau représentées en jaune correspondent à une intensité réduite d’un facteur 2 par rapport à l’intensité maximale, ce qui correspond à la FWHM. |
|
Signal et acquisition La MFL bénéficie des avancées technologiques des capteurs matriciels (CCD, CMOS), en particulier leur très grande sensibilité. En raison du flux de photons atteignant le détecteur, la MFL permet une acquisition avec un très bon rapport signal/bruit (de l’ordre de 1 000:1 en mode rapide), bien plus élevé que pour les microscopes confocaux ou biphotoniques. Un des autres avantages de la MFL est l’imagerie à grande gamme dynamique qui permet d’effectuer de la déconvolution pour accroître la résolution spatiale [ 26] et de quantifier de manière très précise la distribution de la fluorescence d’une protéine marquée. Par exemple, dans les expériences sur les embryons de poisson zèbre, les variations de l’intensité des noyaux cellulaires, dont l’ADN était marqué, ont permis de déterminer le niveau de compaction de l’ADN sans aucune sonde fluorescente supplémentaire [ 18] (Figure 4C). Ces capteurs permettent également une très grande vitesse d’acquisition (typiquement 30 mégapixels/s). Les microscopes confocaux ne peuvent atteindre une telle rapidité qu’en mode multipoints ou en mode à balayage par lignes, mais au détriment du rapport signal/bruit et du sectionnement optique. En ce qui concerne les techniques de MFL elles-mêmes, les techniques sans balayage fournissent une vitesse d’acquisition intrinsèque avantageuse par rapport à la technique de balayage du DSLM. On peut choisir un temps d’irradiation en SPIM inférieur à 1 ms pour, par exemple, suivre le mouvement rapide du cœur et du transport des globules rouges [
29]. Photoblanchiment et phototoxicité En MFL, comme les fluorophores sont excités seulement dans la tranche illuminée, le photoblanchiment et la phototoxicité sont limités au volume de cette tranche et de ce fait fortement réduits [
30]. Cet avantage peut être exprimé comme une réduction d’un facteur n de la charge énergétique. Ce facteur correspond à la profondeur de la série de tranches enregistrées divisée par l’épaisseur de la feuille de lumière. Étant donné que les microscopes à fluorescence traditionnels et confocaux illuminent l’échantillon, y compris hors du plan focal, la charge énergétique et le taux de photoblanchiment sont au moins plus grands d’un facteur n par rapport aux techniques de MFL. Ce facteur est même généralement plus élevé, puisque les microscopes confocaux fonctionnent souvent avec des puissances laser situées dans le régime non linéaire du photoblanchiment. Quant aux microscopes biphotoniques, il est difficile de réaliser une comparaison quantitative de la phototoxicité du fait que les processus d’excitation sont différents. Toutefois, des mesures comparatives et qualitatives ont été réalisées en imagerie in vivo du poisson zèbre et ont révélé une charge énergétique plus élevée de six ordres de grandeur comparée à la MFL [ 18]. Développements et implémentations Bien que la microscopie à feuille de lumière ait été introduite en 1903 par Siedentopf et Zsigmondy [
31], il faut attendre 1993 avec l’OPFOS ( orthogonal-plane fluorescence optical sectioning) pour que le premier objet biologique marqué soit illuminé par une feuille de lumière [
32]. Mais ce n’est que durant la dernière décennie que le développement de cette technique a pris un essor considérable avec le SPIM qui est, par sa simplicité, devenu l’implémentation de la MFL la plus répandue [ 20]. Dans le SPIM, des tranches successives de l’échantillon sont enregistrées en le déplaçant par rapport à la feuille de lumière le long de l’axe de détection. Les vibrations produites par ce déplacement peuvent limiter la vitesse d’acquisition des données 3D. La microscopie OCPI ( objective-coupled planar illumination) a permis de surmonter ce problème en fixant directement le système d’illumination composé d’une fibre optique à l’objectif de détection [
33]. Cet arrangement peut être déplacé rapidement par rapport à l’échantillon. Dans le cas d’échantillons diffusants, le faisceau de lumière s’atténue fortement avec la profondeur. La technique UM ( ultramicroscopy) permet de compenser cette atténuation en utilisant deux feuilles de lumière pour illuminer l’échantillon dans des directions opposées. Cette technique a permis d’imager des embryons de souris, des drosophiles adultes et d’autres tissus fixés de quelques millimètres d’épaisseur [ 23]. Des travaux récents ont aussi montré qu’il était possible d’utiliser le même objectif pour illuminer un échantillon avec une tranche de lumière et pour collecter la fluorescence. Dans cette technique dénommée HILO ( highly inclined and laminated optical sheet) [
34], la feuille de lumière est inclinée d’un angle légèrement inférieur à l’angle de réflexion totale et le plan focal de l’objectif n’est alors illuminé que sur une ligne, limitant ainsi le champ de vision. De plus, en combinant cette technique avec une imagerie oblique, l’OPM ( oblique plane microscopy) permet d’incliner le plan focal pour l’aligner avec la feuille de lumière [
35]. Dans la plupart de ces techniques, des effets d’ombre et de traînée apparaissent dans la direction de l’illumination, dus à l’absorption de la lumière et à l’élargissement de la feuille de lumière par diffusion. Ces effets peuvent être considérablement réduits par deux méthodes : soit par imagerie multivues, soit par illumination multidirectionnelle en faisant pivoter une ou deux feuilles de lumière comme dans le mSPIM (multi-directional SPIM) [22]. Par ailleurs, l’utilisation d’une feuille de lumière structurée spatialement par le mouvement d’une grille (SPIM-SI) [
36], ou par modulation de l’intensité laser (DSLM-SI) [19], permet de diminuer les dégradations dues à la diffusion et les aberrations optiques. La MFL peut être combinée avec des systèmes de manipulations optiques tels que les pincettes optiques et la nanochirurgie laser [
37], ou bien avec des techniques de fluorescence telles que le FCS (fluorescence correlation spectroscopy) [
38], le FRAP (fluorescence recovery after photobleaching) ou l’illumination structurée [19, 36]. De plus, l’illumination avec une source biphotonique permet d’améliorer la profondeur sur laquelle la feuille de lumière garde une épaisseur homogène [
39]. Limitations de la MFL Malgré les approches évoquées pour en limiter l’effet, la diffusion et les interférences qui en découlent freinent l’utilisation de la MFL. L’imagerie d’échantillons multicellulaires de plusieurs millimètres d’épaisseur ou présentant une forte hétérogénéité d’indices souffre d’une dégradation significative de la résolution et du contraste. De tels effets ne sont pas spécifiques aux techniques de MFL mais sont produits par tous les types de microscopes de fluorescence. Toutefois, la MFL possède la configuration la plus adaptée pour analyser ces effets et, du coup, devrait être capable de les réduire par un traitement d’image approprié [
40]. Les données obtenues avec une MFL ne sont pas uniquement en 3D, multicouleurs et multivues, mais correspondent aussi à des séquences temporelles. Dans ce cas, avec l’énorme quantité de données à analyser, il devient particulièrement difficile d’extraire et de représenter la dynamique de l’objet observé [ 18]. |
1.
Javois
LC
. Immunocytochemical methods and protocols (methods in molecular biology) . Totowa, New Jersey: : Humana Press; , 1999; : vol. 115, :466 p.. 2.
Sullivan
KF
. Fluorescent proteins (methods in cell biology) . Oxford: : Academic Press; , 2008; : vol. 85, :660 p.. 3.
McNally
JG
,
Karpova
T
,
Cooper
J
,
Conchello
JA
. Three-dimensional imaging by deconvolution microscopy . Methods.
1999; ; 19 : :373.-385. 4.
Pawley
JB
. Handbook of biological confocal microscopy . New York: : Springer Science-Business Media, LLC; , 2006 : :988 p.. 5.
Dufour
P
,
Dufour
S
,
Castonguay
A
, et al.
Microscopie à deux photons pour l’imagerie cellulaire fonctionnelle : avantages et enjeux ou « un photon c’est bien… mais deux c’est mieux ! » . Med Sci (Paris).
2006; ; 22 : :837.-844. 6.
Débarre
D
,
Pena
AM
,
Supatto
W
, et al.
Microscopies multiharmoniques pour l’imagerie structurale de tissus intacts . Med Sci (Paris).
2006; ; 22 : :845.-850. 7.
Wimmer
VC
,
Möller
A
. High-resolution confocal imaging in tissue . In :
Hewitson
TDD
,
Darby
IAA
, eds.
Histology protocols (methods in molecular biology) . Totowa, New Jersey: : Humana Press; , 2010; : vol. 611, :183.-191. 8.
Tanaami
T
,
Otsuki
S
,
Tomosada
N
, et al.
High-speed 1-frame/ms scanning confocal microscope with a microlens and nipkow disks . Appl Opt.
2002; ; 41 : :4704.-4708. 9.
Denk
W
,
Strickler
JH
,
Webb
WW
. Two-photon laser scanning fluorescence microscopy . Science.
1990; ; 248 : :73.-76. 10.
Helmchen
F
,
Denk
W
. Deep tissue two-photon microscopy . Nat Methods.
2005; ; 2 : :932.-940. 11.
De
AK
,
Goswami
D
. Exploring the nature of photo-damage in two-photon excitation by fluorescence intensity modulation . J Fluoresc.
2009; ; 19 : :381.-386. 12.
Boot
MJ
,
Westerberg
CH
,
Sanz-Ezquerro
J
, et al.
In vitro whole-organ imaging : 4D quantification of growing mouse limb buds . Nat Methods.
2008; ; 5 : :609.-612. 13.
Bouma
BE
,
Yun
SH
,
Vakoc
BJ
, et al.
Fourier-domain optical coherence tomography : recent advances toward clinical utility . Curr Opin Biotechnol.
2009; ; 20 : :111.-118. 14.
Huang
B
,
Bates
M
,
Zhuang
X
. Super-resolution fluorescence microscopy . Annu Rev Biochem.
2009; ; 78 : :993.-1016. 15.
Stelzer
E
,
Lindek
S
. Fundamental reduction of the observation volume in far-field light microscopy by detection orthogonal to the illumination axis: confocal theta microscopy . Opt Commun.
1994; ; 111 : :536.-547. 16.
Greger
K
,
Swoger
J
,
Stelzer
EHK
. Basic building units, properties of a fluorescence single plane illumination microscope . Rev Sci Instrum.
2007; ; 78 : :023705.. 17.
Huisken
J
,
Stainier
DYR
. Selective plane illumination microscopy techniques in developmental biology . Development.
2009; ; 136 : :1963.-1975. 18.
Keller
PJ
,
Schmidt
AD
,
Wittbrodt
J
,
Stelzer
EHK
. Reconstruction of zebrafish early embryonic development by scanned light sheet microscopy . Science.
2008; ; 322 : :1065.-1069. 19.
Keller
PJ
,
Schmidt
AD
,
Santella
A
, et al.
Fast, high-contrast imaging of animal development with scanned light sheet-based structured-illumination microscopy . Nat Methods.
2010; ; 7 : :637.-642. 20.
Huisken
J
,
Swoger
J
,
Del Bene
F
, et al.
Optical sectioning deep inside live embryos by selective plane illumination microscopy . Science.
2004; ; 305 : :1007.-1009. 21.
Keller
PJ
,
Pampaloni
F
,
Stelzer
EHK
. Life sciences require the third dimension . Curr Opin Cell Biol.
2006; ; 18 : :117.-124. 22.
Huisken
J
,
Stainier
DY
. Even fluorescence excitation by multidirectional selective plane illumination microscopy (mSPIM) . Opt Lett.
2007; ; 32 : :2608.-2610. 23.
Becker
K
,
Jährling
N
,
Kramer
ER
, et al.
Ultramicroscopy: 3D reconstruction of large microscopical specimens . J Biophotonics.
2008; ; 1 : :36.-42. 24.
Swoger
J
,
Verveer
P
,
Greger
K
, et al.
Multi-view image fusion improves resolution in three-dimensional microscopy . Opt Express.
2007; ; 15 : :8029.-8042. 26.
Verveer
PJ
,
Swoger
J
,
Pampaloni
F
, et al.
High-resolution three-dimensional imaging of large specimens with light sheet-based microscopy . Nat Methods.
2007; ; 4 : :311.-313. 27.
Pampaloni
F
,
Reynaud
EG
,
Stelzer
EHK
. The third dimension bridges the gap between cell culture and live tissue . Nat Rev Mol Cell Biol.
2007; ; 8 : :839.-845. 28.
Engelbrecht
CJ
,
Stelzer
EHK
. Resolution enhancement in a light-sheet-based microscope (SPIM) . Opt Lett.
2006; ; 31 : :1477.-1479. 29.
Scherz
PJ
,
Huisken
J
,
Sahai-Hernandez
P
,
Stainier
DYR
. High-speed imaging of developing heart valves reveals interplay of morphogenesis and function . Development.
2008; ; 135 : :1179.-1187. 30.
Keller
PJ
,
Pampaloni
F
,
Stelzer
EHK
. Three-dimensional preparation and imaging reveal intrinsic microtubule properties . Nat Methods.
2007; ; 4 : :843.-846. 31.
Siedentopf
H
,
Zsigmondy
R
. Uber sichtbarmachung und grössenbestimmung ultramikroskopischer teilchen, mit besonderer anwendung auf goldrubingläser . Ann Phys.
1903; ; 10 : :1.-39. 32.
Voie
AH
,
Burns
DH
,
Spelman
FA
. Orthogonal-plane fluorescence optical sectioning : three-dimensional imaging of macroscopic biological specimens . J Microsc.
1993; ; 170 : :229.-236. 33.
Holekamp
TF
,
Turaga
D
,
Holy
TE
. Fast three-dimensional fluorescence imaging of activity in neural populations by objective-coupled planar illumination microscopy . Neuron.
2008; ; 57 : :661.-672. 34.
Tokunaga
M
,
Imamoto
N
,
Sakata-Sogawa
K
. Highly inclined thin illumination enables clear single-molecule imaging in cells . Nat Methods.
2008; ; 5 : :159.-161. 35.
Dunsby
C
. Optically sectioned imaging by oblique plane microscopy . Opt Express.
2008; ; 16 : :20306.-20316. 36.
Breuninger
T
,
Greger
K
,
Stelzer
EHK
. Lateral modulation boosts image quality in single plane illumination fluorescence microscopy . Opt Lett.
2007; ; 32 : :1938.-1940. 37.
Engelbrecht
CJ
,
Greger
K
,
Reynaud
EG
, et al.
Three-dimensional laser microsurgery in light-sheet based microscopy (SPIM) . Opt Express.
2007; ; 15 : :6420.-6430. 38.
Wohland
T
,
Shi
X
,
Sankaran
J
,
Stelzer
EHK
. Single plane illumination fluorescence correlation spectroscopy (SPIM-FCS) probes inhomogeneous three-dimensional environments . Opt Express.
2010; ; 18 : :10627.. 39.
Palero
J
,
Santos
SI
,
Artigas
D
,
Loza-Alvarez
P
. A simple scanless two-photon fluorescence microscope using selective plane illumination . Opt Express.
2010; ; 18 : :8491.. 40.
Rohrbach
A
. Artifacts resulting from imaging in scattering media: a theoretical prediction . Opt Lett.
2009; ; 34 : :3041.-3043. 41.
Izeddin
I
,
Darzacq
X
,
Dahan
M
. Microscopies cellulaires à l’échelle de la molécule individuelle . Med Sci (Paris).
2011; ; 27 : :547.-555. |


 fois plus épais qu’au centre. La longueur L du champ de vision doit alors vérifier la condition : L 2W
2/λ avec W l’épaisseur de la feuille de lumière définie comme la largeur du profil gaussien suivant Y. Pour vérifier cette condition, les objectifs d’illumination à grandes ON (en bas, à droite) ont une épaisseur W et une longueur L (et donc un volume détecté) plus faibles que les objectifs à petites ON (en haut, à droite).
fois plus épais qu’au centre. La longueur L du champ de vision doit alors vérifier la condition : L 2W
2/λ avec W l’épaisseur de la feuille de lumière définie comme la largeur du profil gaussien suivant Y. Pour vérifier cette condition, les objectifs d’illumination à grandes ON (en bas, à droite) ont une épaisseur W et une longueur L (et donc un volume détecté) plus faibles que les objectifs à petites ON (en haut, à droite). pour les microscopes confocaux (avec λill la longueur d’onde d’illumination), n l’indice du milieu dans lequel la lumière se propage et α le demi-angle d’ouverture du cône lumineux produit par l’objectif. En microscopie à feuille de lumière, l’éclairage par un plan n’a aucun effet sur la résolution latérale σxy qui est alors décrite par l’expression précédente tandis que la résolution axiale σz est gouvernée par la séparation du système de détection et de l’illumination par une feuille de lumière. Dans ce cas, la contribution du système de détection reste celle d’une lentille sphérique, soit
pour les microscopes confocaux (avec λill la longueur d’onde d’illumination), n l’indice du milieu dans lequel la lumière se propage et α le demi-angle d’ouverture du cône lumineux produit par l’objectif. En microscopie à feuille de lumière, l’éclairage par un plan n’a aucun effet sur la résolution latérale σxy qui est alors décrite par l’expression précédente tandis que la résolution axiale σz est gouvernée par la séparation du système de détection et de l’illumination par une feuille de lumière. Dans ce cas, la contribution du système de détection reste celle d’une lentille sphérique, soit  où a est le demi-angle d’ouverture de l’objectif de détection et où l’ouverture numérique vaut ON = nsinα. Quant à la contribution du système d’illumination, elle est décrite par la théorie SGH d’une lentille cylindrique de faible ouverture numérique
où a est le demi-angle d’ouverture de l’objectif de détection et où l’ouverture numérique vaut ON = nsinα. Quant à la contribution du système d’illumination, elle est décrite par la théorie SGH d’une lentille cylindrique de faible ouverture numérique  où L est l’extension horizontale du champ de vision et W l’épaisseur de la feuille de lumière
où L est l’extension horizontale du champ de vision et W l’épaisseur de la feuille de lumière