| |
| Med Sci (Paris). 2012 March; 28: 7–13. Published online 2012 April 9. doi: 10.1051/medsci/2012281s104.Les réseaux de l’expression génique Émergence et développement d’un domaine clé de la génomique Jean-Philippe Cointet,1,2* Andrei Mogoutov,1** Pascale Bourret,3,4*** Rim El Abed,4**** and Alberto Cambrosio5§ 1INRA-SenS (sciences en société), Institut francilien recherche innovation société/université Paris-Est Marnela-Vallée, cité Descartes, 5, boulevard Descartes, Champs-sur-Marne, 77454Marne-la-Vallée Cedex 2, France 2Institut des systèmes complexes - Paris Île-de-France (ISC-PIF), 57-59, rue Lhomond, 75005Paris, France 3Aix-Marseille Univ, Faculté des Sciences Économiques, 13621Aix-en-Provence, France 4UMR Inserm/IRD/Aix-Marseille Univ, SESSTIM, Institut Paoli- Calmettes, 232, boulevard Sainte-Marguerite, 13273Marseille Cedex 9, France 5Department of Social Studies of Medicine, McGill University, 3647 Peel, Montréal, QC, H3A 1X1Canada |
Le champ de recherche centré sur l’analyse de l’expression génique (gene expression profiling ou GEP) est un des domaines clés de la génomique. Il se caractérise par le recours à des techniques comme la RT-PCR, les microarrays et, plus récemment, les outils de séquençage de dernière génération (next generation sequencing). Plusieurs des techniques associées au GEP étaient disponibles dès la fin des années 1980, mais la croissance véritable du domaine n’a eu lieu qu’à partir de la fin des années 1990. Il ne s’agit cependant pas d’un domaine strictement technique, dans la mesure où les plates-formes technologiques sont étroitement imbriquées avec des hypothèses de recherche et des notions relevant de la biologie moléculaire et, en particulier, de la recherche translationnelle. Mais comment ce domaine a-t-il émergé et comment s’est-il développé ? Sa croissance fulgurante a bien sûr été accompagnée par la publication régulière d’articles de synthèse, mais ces articles reflètent le point de vue forcément sélectif d’un nombre limité d’auteurs confrontés à un nombre sans cesse croissant de publications et à la complexité des initiatives de recherche dont elles découlent, initiatives gérées par des réseaux et consortiums de plus en plus étendus et entrecroisés. Est-il possible d’obtenir un portrait plus complet du développement du domaine, s’appuyant sur la prise en compte d’un corpus étendu de la littérature scientifique pertinente ? Au cours des dernières années, les chercheurs à l’interface entre les sciences sociales et les sciences de l’information ont développé des méthodes performantes d’analyse par ordinateur des réseaux hétérogènes de scientifiques, techniques, institutions, thématiques et entités biomédicales qui définissent un domaine d’activité (voir par exemple [1] et la littérature citée dans cet article). Dans cet article, nous appliquons certaines de ces techniques au domaine du GEP. |
PubMed demeure l’outil bibliographique par excellence dans le domaine biomédical, mais dans le cadre de ce texte nous lui avons préféré Web of Science car, même si cet outil couvre de façon moins exhaustive ce domaine, chaque référence inclut la liste des publications citées, donnant ainsi accès au réseau de citations qui caractérise un champ donné. Nous avons utilisé la requête suivante :
TS=((((“gen* expression profil*”) OR (“gen* signat*”)) OR (“gen* expression signat*”)) OR (“prognostic signat*”))
Elle produit un corpus de 16 348 références sur le GEP (dont 89 % avec le premier terme de la requête). Un tel corpus peut être analysé de différentes façons dans la mesure où il contient des informations sur les auteurs des articles, leurs adresses, le contenu des textes (titres, résumés et mots-clés), les lieux et dates de publication, ainsi que les références citées par chaque article. Nous avons privilégié deux pistes d’analyse : l’analyse des liens de co-citation et l’analyse du contenu des articles (titres et résumés) grâce aux techniques de traitement automatique de la langue (TAL). Deux références sont co-citées si elles apparaissent ensemble dans la liste des références d’un article. De façon similaire, mais à un niveau d’agrégation supérieur, deux journaux A et B ont des liens de co-citation si un article dans un journal C contient parmi ses références des articles publiés dans les journaux A et B. Dans les deux cas, l’hypothèse est que les références ou journaux co-cités appartiennent à un même domaine thématique, ce qui permet d’identifier et cartographier les publications perçues comme étant les contributions majeures du domaine et les différents sous-champs qui le constituent. Quant aux méthodes de TAL, elles donnent un accès plus direct au contenu du domaine. Dans les deux cas, on visualise les résultats sous forme de réseaux de co-citations ou de termes associés : l’interprétation de ces réseaux s’appuie sur la présence d’ensembles de nœuds densément connectés qui forment des clusters. Selon le type de réseau, chaque cluster peut correspondre à une thématique donnée, à un domaine disciplinaire (ou, selon le type de cartes, à un groupe d’auteurs ou d’institutions qui collaborent étroitement). Il s’agit donc d’identifier les différents clusters présents sur une carte et les relations qu’ils entretiennent entre eux. Pour cet article, nous avons utilisé la plateforme d’analyse informatisée CorTexT (www.cortext.fr) constituée d’un ensemble de modules qui permettent de traiter, visualiser et analyser (grâce à des algorithmes de détection automatique des clusters) l’information extraite du corpus (pour plus de détails techniques voir [1]). |
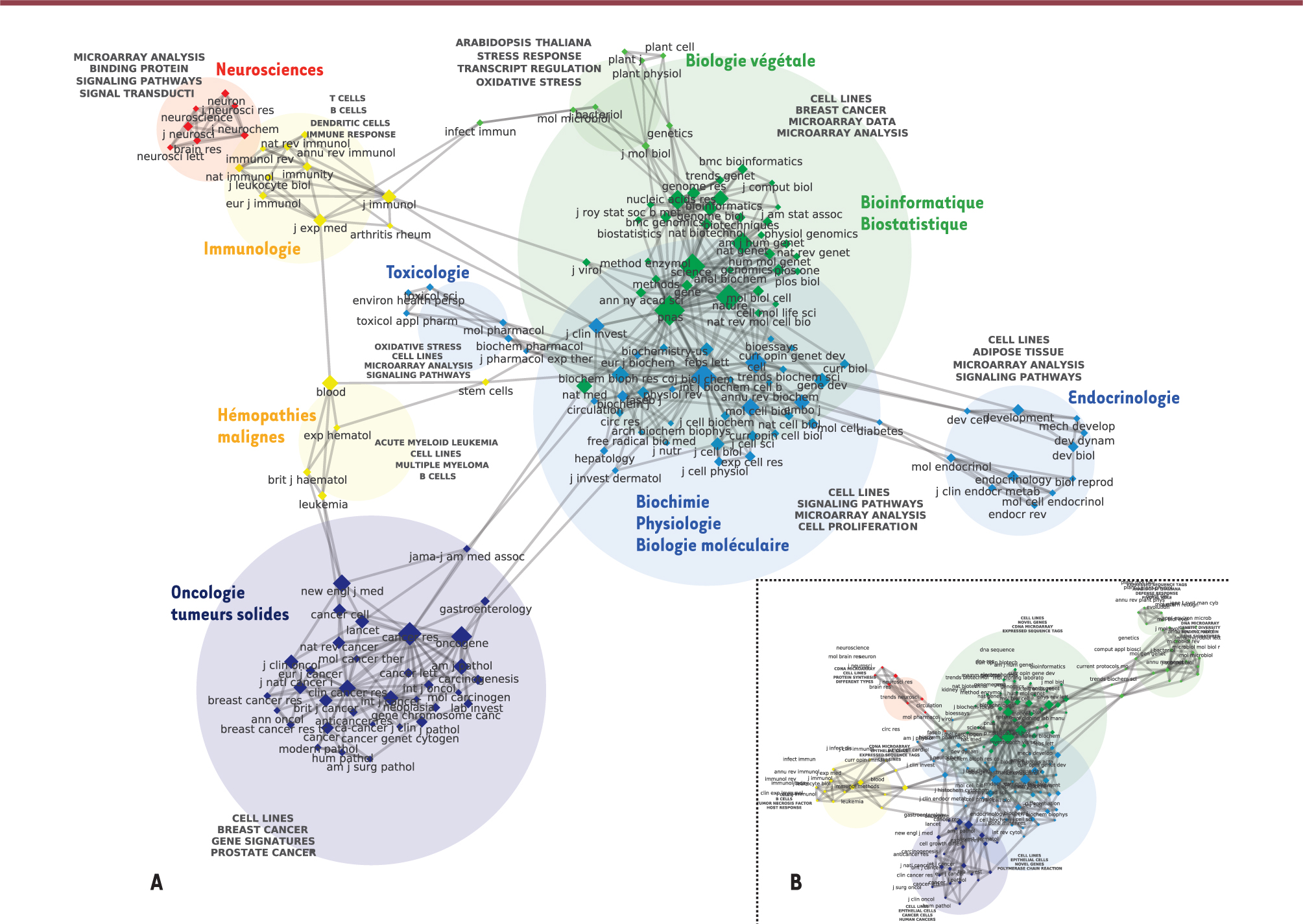
Les sous-domaines du GEP Nous avons d’abord cartographié les sous-domaines du GEP grâce à l’analyse des co-citations entre journaux (Figure 1). Pour ce faire, les 16 348 références ont été divisées en deux groupes correspondant à la période d’émergence du GEP (1991-2000) et à sa période de maturité (2001-2010). Chaque nœud correspond à un titre de journal, sa taille étant proportionnelle au nombre de citations reçues. La couleur des nœuds et cercles différencie les clusters de journaux co-cités. Pour faciliter l’interprétation, CorTexT ajoute automatiquement les trois termes (tirés des titres et des résumés des articles) les plus spécifiquement reliés à chaque cluster. Les deux cartes comportent grosso modo les mêmes clusters, avec cependant quelques différences significatives qui relèvent de l’évolution du domaine.
 | Figure 1.
Réseau de co-citation des journaux obtenu à partir d’articles publiés au cours des périodes 2001-2010 (A) et 1991-2000 (B).
|
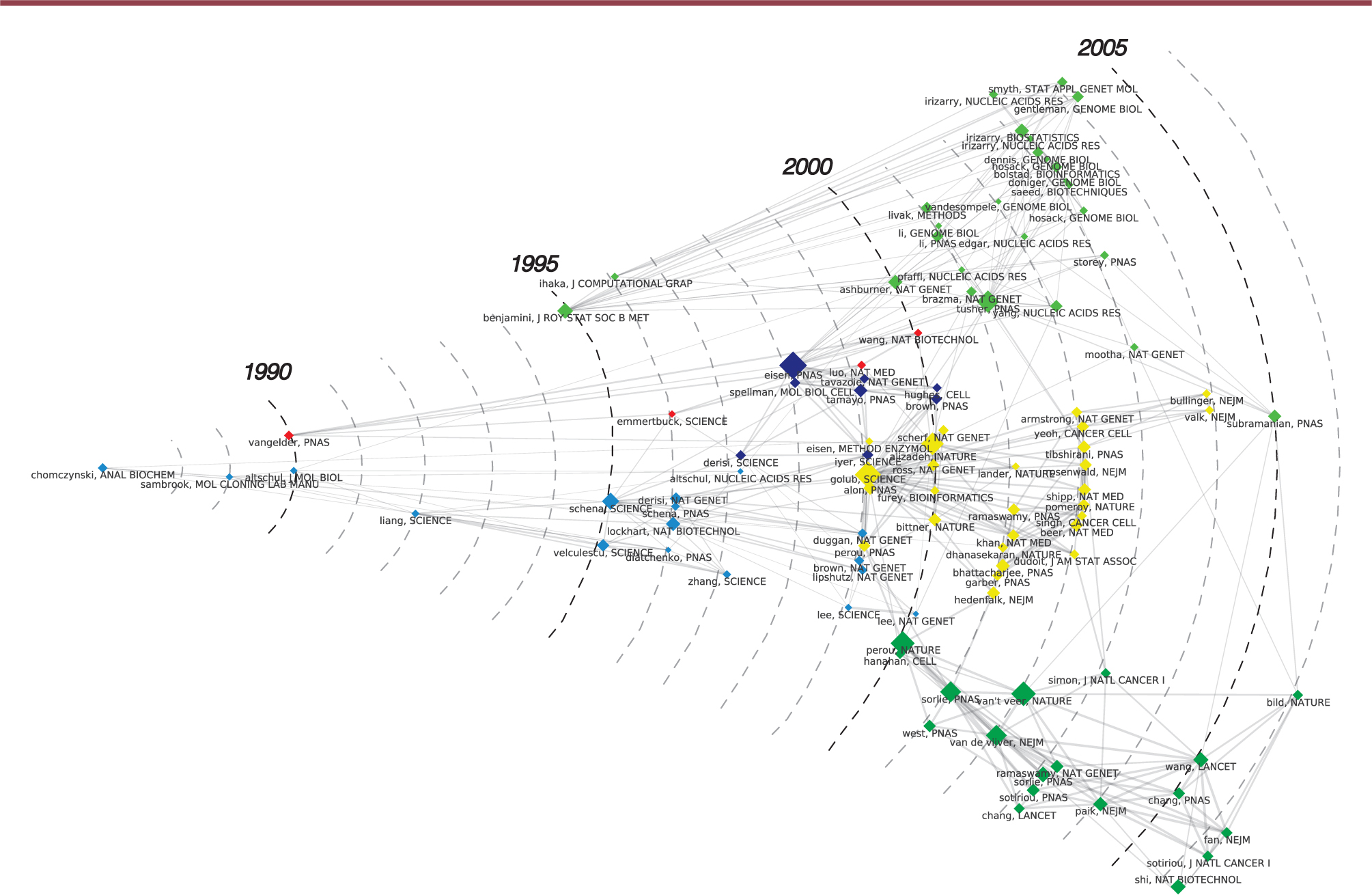
En commençant par la période la plus récente (Figure 1A), on voit en bas à gauche (couleur bleu foncé) un cluster qui renvoie aux journaux d’oncologie, plus particulièrement au cancer du sein et autres cancers solides. Il occupe une place plus importante que le cluster équivalent de la période initiale (Figure 1B). Au-dessus de ce cluster et relié à celui-ci, on retrouve un cluster (couleur jaune) d’onco-hématologie, à son tour relié à un cluster de revues d’immunologie situé au-dessus (en haut à gauche, également en jaune). Au cours de la période initiale, ces deux clusters ne faisaient qu’un (d’où la même couleur), ce qui nous incite à conclure que le cluster initial d’immunologie fondamentale et clinique s’est ensuite divisé, la partie oncologique prenant de l’ampleur. Deux grands clusters (vert et bleu pâle) occupent le centre du réseau. Le cluster bleu contient des revues de recherche fondamentale en biochimie, physiologie et biologie moléculaire, avec, à sa droite et à sa gauche deux satellites consacrés, d’une part, à l’endocrinologie et à la biologie du développement, et, de l’autre, à la toxicologie et à la pharmacologie. Ces trois clusters partagent une même couleur car dans la période initiale, ils étaient étroitement imbriqués. On observe donc ici également un processus de différenciation, avec des revues comme Diabetes servant de pont entre l’endocrinologie et les autres domaines de recherche fondamentale, et la pharmacologie moléculaire reliant la toxicologie au cluster central. Le cluster central en haut (couleur verte) regroupe surtout des revues de bioinformatique, de biostatistique et de méthodologie. Ce cluster, qui au cours de la période initiale occupait une place encore plus importante, recoupe un cluster plus petit de revues de biologie végétale, dont la présence s’explique par le fait que les premières expériences avec des microarrays ont souvent été réalisées sur un organisme modèle végétal (Arabidopsis thaliana). Ceci est confirmé par le fait qu’au cours de la période initiale le cluster de biologie végétale est directement relié à un cluster de génétique et de biologie moléculaire, à son tour rattaché au cluster central. Enfin, on note un cluster des neurosciences en haut à gauche (couleur rouge), relié au cluster de recherche fondamentale dans la période initiale, mais qui s’est autonomisé au cours de la deuxième période. Au total, ces cartes de co-citation des revues scientifiques permettent d’observer l’origine du GEP et sa différenciation progressive, notamment avec l’émergence d’un cluster oncologique qui constitue désormais un de ses principaux domaines d’application. Émergence et développement du GEP La Figure 2 visualise également un réseau de co-citations, mais avec deux différences importantes par rapport aux Figures 1A et 1B. Il s’agit, d’abord, de co-citations entre références, et non plus entre journaux ; cette carte donne ainsi accès de façon plus précise au contenu thématique du domaine. En deuxième lieu, la technique de visualisation inclut une dimension temporelle. En effet, en plus d’être agrégées par cluster, les références sont distribuées selon un axe temporel allant des références les plus anciennes, à gauche, aux plus récentes, à droite (pour mieux visualiser les résultats, nous avons utilisé une représentation radiale). Plutôt qu’une chronologie objective d’un domaine, cette approche permet de visualiser sa généalogie telle qu’elle est perçue par les auteurs des articles.
 | Figure 2.
Réseau chronologique de co-citation des références citées dans les articles du domaine GEP publiés entre 1990 et 2010.
|
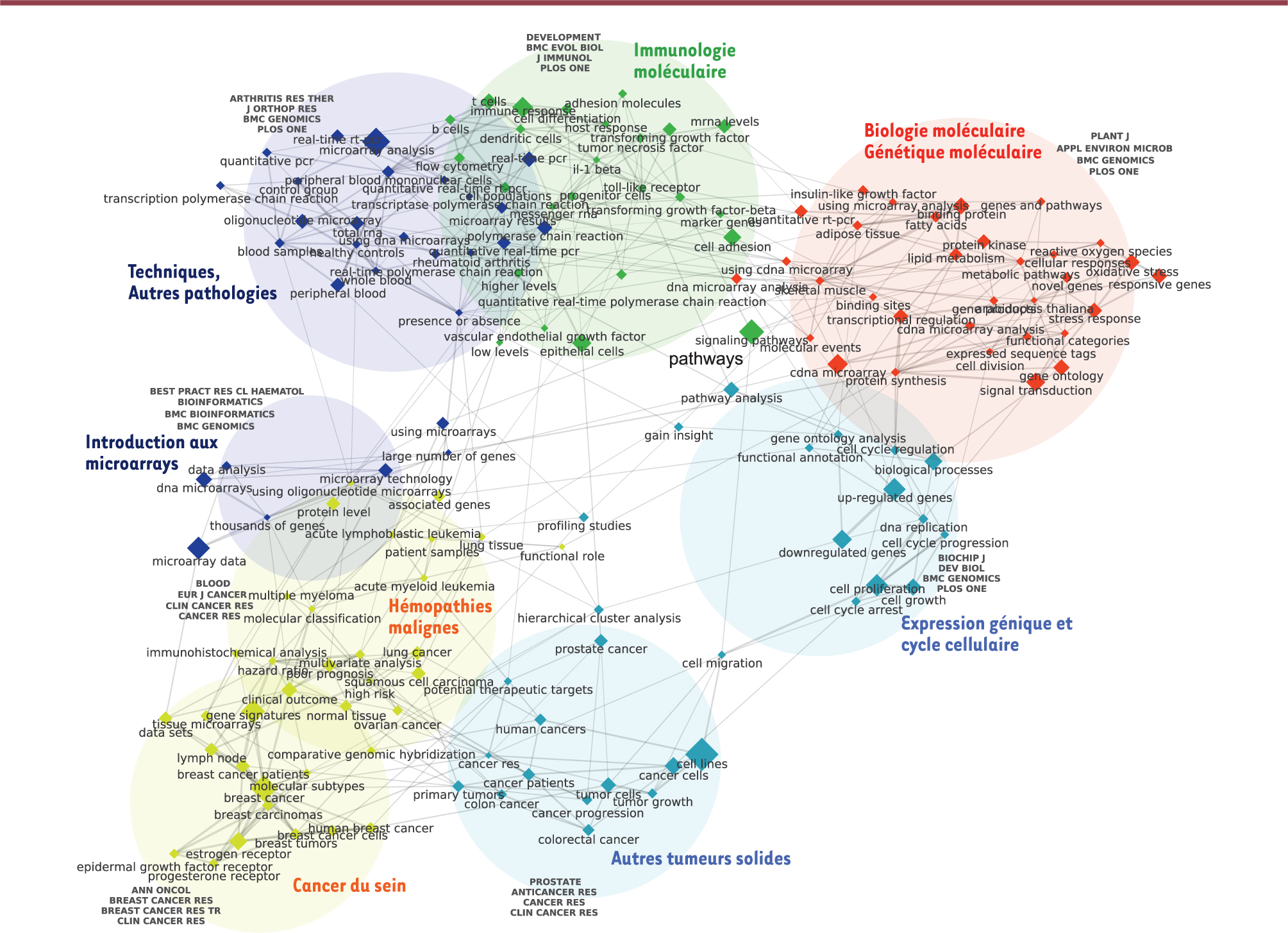
Les articles les plus anciens correspondent à ce qui est vu aujourd’hui comme les fondements des techniques et des méthodologies qui ont présidé à l’émergence du domaine : la méthode d’extraction et de purification de l’ARN [2], le manuel de clonage moléculaire publié par le laboratoire de Cold Spring Harbor que d’aucuns considèrent comme la bible des méthodes de biologie moléculaire [3], la méthode BLAST de comparaison des séquences [4], ou encore l’application de la technique de PCR à l’ARN messager [5]. Une autre lignée généalogique (couleur rouge) remonte à un article concernant l’amplification de l’ARN d’échantillons neurologiques [6], et conduit à des articles qui portent également sur l’amplification de l’ARN et la microdissection par laser des neurones [7]. Plus proche de la période récente, et en nous limitant aux nœuds les plus importants (les plus cités), on observe un article du laboratoire de Patrick Brown à Stanford qui correspond à la première mise en pratique expérimentale des microarrays sur un organisme modèle (Arabidopsis thaliana). Nous sommes ici au cœur de ce premier cluster (bleu) où cette nouvelle technologie, après une période de mise au point technique, débouche sur un domaine d’application. On trouve dans ce même cluster un autre article du laboratoire de Patrick Brown [9], qui applique les microarrays aux cancers humains, ainsi que des articles faisant état d’innovations comme la technique SAGE d’analyse sérielle de l’expression génique et, surtout, le texte de la compagnie Affymetrix [10] qui exemplifie l’utilisation des microarrays développés par cette start-up californienne (qui deviendra un des principaux acteurs industriels du domaine) et qui reposent sur une technologie différente de celle utilisée par Patrick Brown. Le centre de la période la plus récente est occupé par un cluster d’articles (couleur bleu foncé) qui correspondent à la mise au point de méthodes de bioinformatique permettant l’analyse du déluge de données produites par les techniques de GEP. Le cluster est dominé par un autre article du laboratoire de Stanford [11], qui détaille les algorithmes de clustering à l’origine des graphiques colorés (les heat maps) qui vont devenir une des caractéristiques des articles utilisant des microarrays. Les autres articles de ce cluster concernent le développement de méthodes de classification hiérarchique. Ce cluster comprend des articles qui constituent une des deux origines du cluster vert situé en haut à droite de la carte et qui regroupe des contributions plus récentes au développement des méthodes d’analyse. Les autres deux contributions « historiques » ayant participé à l’émergence de ce cluster méthodologique sont deux articles de biostatistique portant sur la technique du false discovery rate [12] et sur le langage informatique R, un des plus utilisés en biostatistique [13]. On aperçoit ici la double racine, biostatistique et bioinformatique, des méthodes actuelles d’analyse des résultats du GEP, ce qui n’a pas été sans provoquer de controverses [14]. Le nœud le plus important de ce cluster [15] concerne une autre technique très répandue (SAM ou significance analysis of microarrays), des contributions renvoyant à la standardisation du domaine - définition des informations minimales qui devraient accompagner chaque article décrivant les expériences de GEP [16], et développement d’un vocabulaire standardisé (gene ontology [17]) - des articles introduisant d’autres logiciels (par exemple Bioconductor) et plusieurs autres articles de biostatisticiens (notamment Irizarry et collègues [30]). Le centre de la période récente est occupé par un cluster (couleur jaune) renvoyant à des articles sur l’application du GEP aux cancers. Les plus anciennes références correspondent à des démonstrations de principe de l’utilité du GEP pour l’oncologie. Ainsi, parmi les nœuds principaux, on retrouve un article sur la mise au point de méthodes automatiques permettant d’identifier de nouveaux sous-groupes de leucémies (class discovery) et d’en classer d’autres dans des catégories existantes (class prediction) [18], ainsi qu’un article sur la découverte de différentes classes moléculaires de lymphomes [19]. Un article sur la classification moléculaire des cancers du sein [20] est à l’origine, avec un autre texte [21], d’un cluster sur cette pathologie (couleur verte, en bas de la carte) sur lequel nous allons revenir. Les autres références du cluster jaune portent, notamment, sur les lignées cellulaires de cancers humains et leur réponse aux chimiothérapies, ou encore sur d’autres tumeurs solides (mélanome, poumon, prostate). Le cluster qui occupe le bas de la carte est consacré au cancer du sein, révélant le rôle unique que cette maladie a joué dans le domaine du GEP. Comme nous l’avons déjà mentionné, on retrouve à l’origine de ce cluster les travaux de Perou et collègues [21, 22] sur la classification moléculaire des cancers du sein, qui ont provoqué une minirévolution en redéfinissant les principaux sous-types de cette pathologie. Le cluster regroupe ensuite des travaux portant sur l’utilisation de signatures génomiques pour le pronostic, la prédiction de la réponse thérapeutique ou la détermination du grade des tumeurs : on y retrouve notamment des publications qui renvoient à des tests commercialisés, respectivement MammaPrint et Oncotype DX [23–25]. Ce cluster contient également un texte de statisticiens du National Cancer Institute [26] mettant en garde contre les pièges et écueils statistiques liés au développement de marqueurs pronostiques et prédictifs, ce qui renvoie au caractère controversé de certains résultats [14]. Le contenu des recherches L’interprétation de la Figure 2 s’appuyait sur une analyse manuelle du contenu des références au sein de chaque cluster. L’analyse des titres et résumés à l’aide d’un algorithme de TAL nous permet d’accéder directement au contenu du domaine. Nous avons distingué trois périodes (1991-2000, 2001-2005, 2006-2010) : le réseau sémantique de chaque période montre l’existence de clusters de mots associés qui correspondent aux thématiques de recherche. Les 150 termes figurant sur chaque carte ont été choisis sur la base de leur fréquence (la taille des nœuds est proportionnelle à celle-ci) mais aussi de leur degré d’association spécifique, ce qui permet de préciser le contenu des thématiques. CorTexT indique près de chaque cluster les titres des journaux qui y sont le plus spécifiquement associés. Les labels que nous avons ajoutés pour aider le lecteur à explorer la carte sont indicatifs et n’ont pas la prétention de capturer finement le contenu de chaque cluster. La Figure 3 correspond à la période 2006-2010 (pour des raisons de contraintes d’espace, nous n’examinerons que celle-ci). En bas, au centre, et à gauche de la carte, on trouve des clusters qui renvoient à l’utilisation du GEP en oncologie. Un cluster cancer du sein est étroitement imbriqué avec un cluster hémopathies malignes, l’interface entre les deux contenant des termes qui renvoient au cancer de l’ovaire et aux myélomes. À leur droite on aperçoit un cluster qui regroupe des termes relevant d’autres cancers solides. En plus de confirmer le rôle de l’oncologie comme domaine d’application privilégié du GEP, la présence de ces clusters montre la place centrale occupée par le cancer du sein (avec des termes renvoyant aux différents récepteurs, aux signatures pronostiques et aux degrés de risque) et par les leucémies et lymphomes, les leçons tirées de l’emploi des techniques de GEP circulant rapidement entre ces deux domaines, notamment pour celles concernant les classifications moléculaires de ces cancers.
 | Figure 3.
Réseau sémantique des concepts extraits des titres et des résumés des articles du domaine GEP publiés au cours de la période 2006-2010.
|
Au-dessus des clusters d’oncologie on trouve deux clusters consacrés aux techniques : le plus petit contient des termes peu spécifiques renvoyant à des présentations générales des microarrays dans des revues de bioinformatique et de génomique, le second, au-dessus, est consacré à des applications techniques dans le cas de pathologies autres que le cancer. En haut, au centre et à droite de la carte on trouve des clusters de recherche fondamentale, avec notamment, au centre, un cluster qui contient des termes relevant de l’immunologie : cellules T et B, mais aussi molécules d’adhésion, cytokines, etc. Une série de termes relevant de l’analyse des voies de signalisation (pathways) relie ce cluster à un autre de biologie/génétique moléculaire situé à sa droite. Celui-ci contient des termes concernant l’organisme modèle Arabidopsis, ainsi que des entités (kinases) et des processus (régulation de la transcription, etc.) caractérisant les travaux récents dans ce champ. Enfin, au-dessous de ce cluster, on trouve un cluster de termes concernant plus spécifiquement les processus génétiques directement reliés à la technologie des microarrays, en particulier les processus de sur- ou sous-expression des gènes, et des termes renvoyant à l’étude du cycle cellulaire, ce qui explique le positionnement intermédiaire de ce cluster entre les clusters de la génétique moléculaire et des cancers solides. Conclusion Cet article vise à contribuer à l’analyse de la dynamique socio-épistémique du GEP. Celle-ci se caractérise par une première étape de confluence de plusieurs méthodes existantes de biologie moléculaire (extraction/purification de l’ARN, etc.) pour donner naissance aux techniques de GEP au milieu des années 1990. Elle est rapidement suivie d’un ensemble de démonstrations de principe de l’intérêt et de la viabilité de cette approche. Cette étape se poursuit par la traduction très rapide de ces nouvelles techniques dans le domaine de l’oncologie, qui apparaît comme un domaine de choix pour l’application du GEP. Tout comme ce fut le cas pour l’émergence de la chimiothérapie [ 27], cette traduction, centrée sur la mise en évidence de sous-types moléculaires, transite d’abord par les hémopathies malignes et débouche ensuite sur les tumeurs solides, surtout sur le cancer du sein qui fédère une grande part des efforts dans ce domaine. Mais une telle dynamique de recherche translationnelle n’est pas limitée aux interactions laboratoire-clinique. Notre analyse fait ressortir le développement en parallèle de méthodes de bioinformatique et de biostatistique (ainsi que des initiatives de standardisation qu’elles suscitent [28]) en tant que conditions de possibilité pour l’analyse et la validation des millions de données générées. Ces deux disciplines continuent de jouer un rôle majeur dans ce domaine, au point qu’on devrait plutôt parler d’une triangulation entre laboratoire, clinique et bioinformatique, ce que d’aucuns appellent désormais les 3B (bench, bedside, bytes) [29]. Conflit d’intérêts
Les auteurs déclarent n’avoir aucun conflit d’intérêts concernant les données publiées dans cet article.
|
1. Jones D, Cambrosio A, Mogoutov A. Detection characterization of translational research in cancer, cardiovascular medicine . J Transl Med. 2011; ; 9 : :57.. 2. Chomczynski P, Sacchi N. 1987 Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction . Anal Biochem. 1987; ; 162 : :156.–159. 3. Sambrook J, Fritsch EF, Maniatis T. Molecular cloning : a laboratory manual . Cold Spring Harbor NY: : Cold Spring Harbor Laboratory Press; , 1989. 4. Altschul SF, Gish W, Miller W, et al. Basic local alignment search tool . J Mol Biol. 1990; ; 215 : :403.–410. 5. Liang P, Pardee AB. Differential display of eukaryotic messenger RNA by means of the polymerase chain reaction . Science. 1992; ; 257 : :967.–971. 6. Van Gelder RN, von Zastrow ME, Yool A, et al. Amplified RNA synthesized from limited quantities of heterogeneous cDNA . Proc Natl Acad Sci USA. 1990; ; 87 : :1663.–1667. 7. Emmert-Buck MR, Bonner RF, Smith PD, et al. Laser capture microdissection . Science. 1996; ; 274 : :998.–1001. 8. Schena M, Shalon D, Davis RW, et al. Quantitative monitoring of gene expression patterns with a complementary DNA microarray . Science. 1995; ; 270 : :467.–470. 9. DeRisi J, Penland L, Brown PO, et al. Use of a cDNA microarray to analyse gene expression patterns in human cancer . Nat Genet. 1996; ; 14 : :457.–460. 10. Lockhart DJ, Dong H, Byrne MC, et al. Expression monitoring by hybridization to high-density oligonucleotide arrays . Nat Biotechnol. 1996; ; 14 : :1675.–1680. 11. Eisen MB, Spellman PT, Brown PO, Botstein D. Cluster analysis and display of genome-wide expression patterns . Proc Natl Acad Sci USA . 1998; ; 95 : :14863.–14868. 12. Benjamini Y, Hochberg Y. Controlling the false discovery rate: a practical and powerful approach to multiple testing . J R Statist Soc B. 1995; ; 57 : :289.–300. 13. Ihaka R, Gentleman RR. A language for data analysis and graphics . J Comput Graph Stat. 1996; ; 5 : :299.–314. 14. Keating P, Cambrosio A. Too many numbers: microarrays in clinical cancer research . Stud Hist Philos Biol Biomed Sci. 2012; ; 43 : :37.–51. 15. Tusher VG, Tibshirani R, Chu G. Significance analysis of microarrays applied to the ionizing radiation response . Proc Natl Acad Sci USA. 2001; ; 98 : :5116.–5121. 16. Brazma A, Hingamp P, Quackenbush J, et al. Minimum information about a microarray experiment (MIAME)-toward standards for microarray data . Nat Genet. 2001; ; 29 : :365.–371. 17. Ashburner M, Ball CA, Blake JA, et al. Gene ontology: tool for the unification of biology. The gene ontology consortium . Nat Genet. 2000; ; 25 : :25.–29. 18. Golub TR, Slonim DK, Tamayo P, et al. Molecular classification of cancer: class discovery and class prediction by gene expression monitoring . Science. 1999; ; 286 : :531.–537. 19. Alizadeh AA, Eisen MB, Davis RE, et al. Distinct types of diffuse large B-cell lymphoma identified by gene expression profiling . Nature. 2000; ; 403 : :503.–511. 20. Perou CM, Jeffrey SS, van de Rijn M, et al. Distinctive gene expression patterns in human mammary epithelial cells and breast cancers . Proc Natl Acad Sci USA. 1999; ; 96 : :9212.–9217. 21. Perou CM, Sorlie T, Eisen MB, et al. Molecular portraits of human breast tumours . Nature. 2000; ; 406 : :747.–752. 22. Sorlie T, Perou CM, Tibshirani R, et al. Gene expression patterns of breast carcinomas distinguish tumor subclasses with clinical implications . Proc Natl Acad Sci USA. 2001; ; 98 : :10869.–10874. 23. van ‘t Veer LJ, Dai H, van de Vijver MJ, et al. Gene expression profiling predicts clinical outcome of breast cancer . Nature. 2002; ; 415 : :530.–536. 24. Paik S, Shak S, Tang G, et al. A multigene assay to predict recurrence of tamoxifen-treated, node-negative breast cancer . N Engl J Med. 2004; ; 351 : :2817.–2826. 25. Kohli-Laven N, Bourret P, Keating P, Cambrosio A. Cancer clinical trials in the era of genomic signatures: biomedical innovation, clinical utility, and regulatory-scientific hybrids . Soc Stud Sci. 2011; ; 41 : :487.–513. 26. Simon R, Radmacher MD, Dobbin K, McShane LM. Pitfalls in the use of DNA microarray data for diagnostic and prognostic classification . J Natl Cancer Inst. 2003; ; 95 : :14.–18. 27. Keating P, Cambrosio A. Cancer on trial: oncology as a new style of practice . Chicago: : University of Chicago Press; , 2012 : :424. p. 28. Rogers S, Cambrosio A. Making a new technology work: the standardization and regulation of microarrays . Yale J Biol Med. 2007; ; 80 : :165.–178. 30. Irizzary RA, Hobbs B, Collin F, et al. Exploration, normalization, and summaries of high density oligonucleotide array probe level data . Biostatistics. 2003; ; 4 : :249.–264. |