| |
| Med Sci (Paris). 2012 May; 28(5): 551–554. Published online 2012 May 30. doi: 10.1051/medsci/2012285023.Recombinaisons suicides du locus IgH Quand le lymphocyte B dépose les armes ! Sophie Péron,1 Brice Laffleur,1 Nicolas Denis-Lagache,1 Jeanne Cook-Moreau,1 Matthieu Filloux,1 and Michel Cogné1* 1Université de Limoges ; CNRS UMR 7276, contrôle de la réponse immune B et lymphoproliférations (CRIBL), 2, rue Marcland, 87025Limoges Cedex, France MeSH keywords: Animaux, Apoptose, génétique, immunologie, Lymphocytes B, métabolisme, physiologie, Cytidine deaminase, Fréquence d'allèle, Locus génétiques, Humains, Chaines lourdes des immunoglobulines, Modèles biologiques, Recombinaison génétique, Hypermutation somatique des gènes des immunoglobulines |
Commutation de classe et hypermutation somatique, événements majeurs contrôlés par AID Lors d’une réponse immune, les lymphocytes B mûrs activés par l’antigène recombinent les gènes codant leur immunoglobuline (Ig) membranaire (B cell receptor ou BCR). Cette recombinaison de class switch (CSR) - ou commutation de classe - délète le gène Cµ du locus codant la chaîne lourde d’Ig sous l’action de l’enzyme AID (activation-induced deaminase) et permet de diversifier la fonction des Ig par commutation d’IgM (chaîne lourde µ) vers IgG, IgA ou IgE1 [
1–
3]. AID attaque l’ADN au niveau des cytosines et peut ainsi déclencher soit le processus d’hypermutation somatique (SHM) des régions variables (V)2 pour améliorer leur affinité pour l’antigène, soit la recombinaison de CSR, grâce à des cassures d’ADN double-brin au voisinage de régions cibles très répétitives du locus IgH (H pour heavy chain ou chaîne lourde), les régions switch (S). Ces évènements diversifient dans les deux cas les effecteurs de la réponse immunitaire. Les deux événements, SHM comme CSR, nécessitent la transcription des régions cibles sous le contrôle d’activateurs situés à l’extrémité 3’ du locus IgH, dans la région 3’ régulatrice (3’RR) [
4,
5]. Des activateurs (enhancers) transcriptionnels s’y trouvent, au nombre de 4 chez la souris (hs3a, hs1-2, hs3b et hs 4) et de 3 chez l’homme (hs3, hs1-2 et hs4, mais avec deux copies de la 3’RR en aval du gène Cα1 et du gène Cα2) (Figure 1). Alors que ces activateurs sont individuellement faibles et en partie redondants, il existe entre eux une forte synergie [
6]. Ensemble, ils donnent à la 3’RR une spécificité B très stringente et un potentiel activateur surpassant celui des éléments situés en amont sur le locus (régions intergéniques V-D, enhancer DQ52, enhancer Eµ, etc.). On peut aussi considérer que les éléments 5’ ont une action qui est restreinte aux étapes précoces du développement B et au contrôle des réarrangements VDJ. À l’inverse, la 3’RR se cantonne (mais avec un rôle dominant) à la régulation des événements génétiques tardifs de la maturation B, lors des réponses immunes [4].
 | Figure 1.
Mise en évidence par un diagramme dot plot de séquences répétitives. Les séquences LS (de structure voisine de celle des régions S du locus IgH) parsèment la région 3’ régulatrice (3’RR) en aval du gène Cα2 humain. La séquence (incluant la région Sα, le gène Cα et la totalité de la 3’RR) est analysée par blocs successifs de 49 bases sur l’axe horizontal, et comparée avec ellemême sur l’axe vertical. Chaque point noir dans le diagramme marque la présence d’un bloc de 49 bases, répété avec une homologie ≥ 80 %, les séquences hautement répétitives prennent donc l’aspect de carrés. La position du gène codant la chaîne lourde de l’IgA2 est indiquée (en jaune), de même que celle des 3 activateurs de la 3’RR (cercles rouges). |
|
Une nouvelle cible de AID : les régions LS de la région 3’RR entraînant des recombinaisons suicides du locus IgH De façon inattendue, nous avons récemment observé que les activateurs transcriptionnels de la 3’RR sont euxmêmes transcrits et mutés par l’enzyme AID dans les cellules B activées [
7]. Cette notion mérite d’être considérée au regard du caractère très particulier de la 3’RR : les activateurs 3’ du locus IgH sont flanqués de séquences répétitives ressemblant aux régions S [
8,
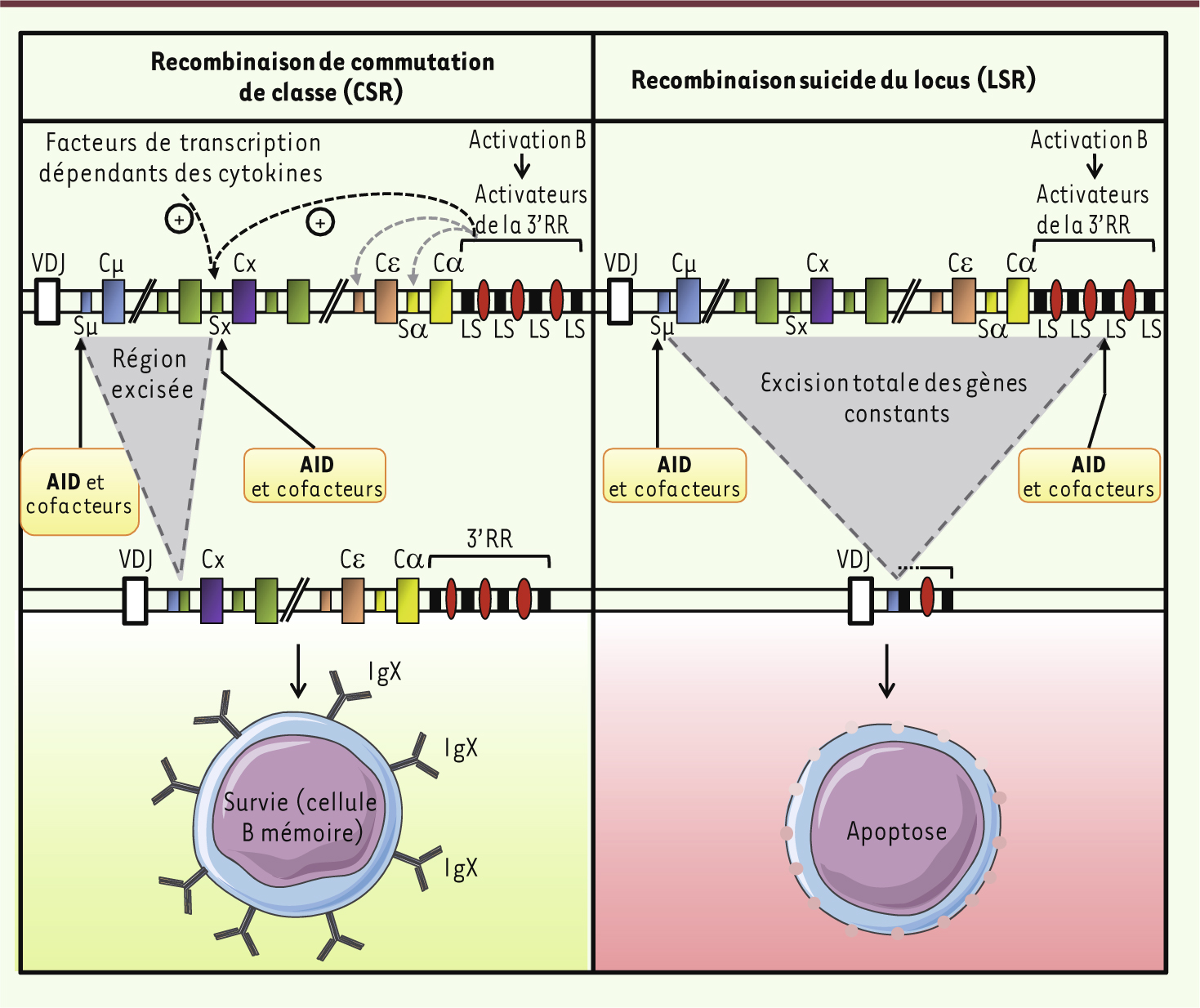
9] (que nous avons dénommées régions LS), présentes à des positions phylogénétiquement conservées chez les espèces de mammifères pour lesquelles la séquence du locus IgH est disponible. Finalement, la triade « transcription + structure répétitive + ciblage par AID » correspond à trois pré-requis de l’accessibilité des régions S aux recombinaisons. En effet, nous avons pu mettre en évidence au niveau de la 3’RR l’occurrence de cassures double brin au voisinage des séquences LS selon un processus voisin de la CSR [7]. Les recombinaisons ainsi suscitées associent alors la région S la plus en amont du locus (région Sµ en amont du gène Cµ) aux sites LS situés au sein de la 3’RR, complètement en aval du locus. Ces recombinaisons provoquent donc une délétion complète de l’ensemble des gènes constants qui revient à inactiver le locus IgH. L’expression membranaire du BCR et le « signal tonique » de survie qu’il induit étant indispensables au lymphocyte B, une telle inactivation du locus IgH fonctionnel a pour corollaire la mort de la cellule B concernée. Nous avons donc dénommé ces délétions géniques « recombinaisons suicides du locus IgH » (locus suicide recombination ou LSR). Ces évènements sont détectables dans les cellules B seulement lors de leur activation, et sont absents chez des animaux déficients pour l’enzyme AID, ou dont la région Sµ a été remplacée par une séquence non répétitive [7]. Ces pré-requis confirment la parenté entre deux recombinaisons contrôlées par AID : CSR assurant le changement de classe du BCR et ainsi la survie de lymphocytes B mémoires, et LSR induisant l’élimination vraisemblable des cellules B qu’elle affecte (Figure 2).
 | Figure 2.
Recombinaisons initiées par AID sur le locus des chaînes lourdes d’immunoglobulines. Les recombinaisons de commutation de classe et les « recombinaisons suicides » du locus ont les mêmes pré-requis avec la transcription des régions cibles de structure répétitive, puis avec une attaque de ces régions initiée par la déméthylation des cytosines par AID. Leur issue est cependant totalement différente : la commutation de classe est notamment associée à la sélection des cellules B mémoires, tandis que les recombinaisons suicides conduisent à l’élimination des cellules concernées. |
|
Fréquence des recombinaisons suicides Dans une première approche de détection directe des recombinaisons LSR sur le locus IgH lui-même, les jonctions LSR nous sont apparues comme 10 à 100 fois plus rares que les jonctions CSR. Cependant, il était clair que les cellules activées étudiées ne bénéficient pas du même destin selon qu’elles subissent une commutation de classe autorisant survie et prolifération ou bien une recombinaison suicide probablement rapidement fatale. Nous avons donc cherché à quantifier non pas le produit final, mais plutôt un sous-produit transitoire de ces recombinaisons : l’épisome circularisé sous la forme duquel sont excisées les séquences délétées sous l’action d’AID. Dans cette deuxième approche, nous observons, en étudiant des dilutions successives d’ADN de différentes populations de cellules B activées, que des épisomes de LSR sont souvent détectables dans des quantités d’ADN de l’ordre du nanogramme (l’équivalent d’environ 200 cellules), c’est-à-dire à une fréquence approchant celle des épisomes de CSR au sein des mêmes échantillons d’ADN. Les méthodes de PCR à longue distance nous ont aussi permis de cloner et séquencer les jonctions de LSR : elles sont observées surtout entre les régions Sµ et 3’RR. Seule une faible fraction des évènements observés correspond à des jonctions complexes de Sµ sur une région Sγ, puis de cette région Sγ sur la région 3’RR. Certains hybridomes révèlent des évènements de LSR qui coexistent avec l’expression d’une Ig fonctionnelle et qui impliquent donc un allèle non fonctionnel. Cependant, de même que la CSR cible le plus souvent le même gène codant la région constante symétriquement sur les 2 allèles IgH, il est probable que la LSR soit, elle aussi, majoritairement bi-allélique. In vivo, nous avons objectivé des événements LSR aussi bien dans les cellules B folliculaires que dans celles de la zone marginale3, et nous avons aussi observé que l’activation B se traduit, in vivo comme in vitro, par l’apparition fugitive d’une population de cellules ayant perdu l’expression du BCR et, de ce fait, engagées dans un processus d’apoptose. |
Conclusion : comment se fait le choix entre CSR et recombinaison suicide ? Au final, ces observations montrent que deux acteurs majeurs du switch et de l’hypermutation somatique, l’enzyme AID comme facteur « trans-activateur » et la 3’RR comme facteur « cis-activateur », sont non seulement capables de partenariat pour agir sur les régions S ou V, mais aussi d’interactions directes qui définissent la 3’RR comme une nouvelle cible d’AID. La transcription de séquences enhancers est, pour sa part, un phénomène potentiellement général et des eARN issus d’autres enhancers ont été récemment caractérisés. Ceci révèle sans doute simplement la parenté entre promoteurs et activateurs transcriptionnels et le fait que la transcription peut n’être qu’un témoin de l’accessibilité d’une région chromatinienne [
10]. Au contraire, le contexte particulier du locus IgH, son exposition à l’action d’AID dans les centres germinatifs et la conservation interespèces de régions LS suggèrent ensemble une fonction des eARN de la 3’RR, et posent de nombreuses questions : comment est régulé le choix entre CSR et LSR ? Existe-t-il, comme c’est le cas pour la CSR vers les divers gènes codant les régions constantes des chaînes lourdes, un contrôle T-dépendant des interactions entre les régions ciblées par AID et susceptibles de former « une synapse » pour recombiner entre elles [
11] ? La qualité de la reconnaissance par le BCR de l’antigène présenté au niveau des cellules folliculaires dendritiques pourrait-elle plutôt entraîner les cellules B les plus affines vers des événements de CSR et d’hypermutation somatique ? Une reconnaissance sous-optimale de l’antigène et/ou une costimulation sous-optimale par le microenvironnement du centre germinatif conduirait-elle plutôt d’autres cellules B vers la LSR, ce qui aboutirait à leur élimination ? Clairement, ces questions méritent maintenant d’être étudiées. Alors que les modalités de contrôle du répertoire préimmun des cellules B sont aujourd’hui bien connues, la description du phénomène de LSR offre une nouvelle piste pour la régulation potentielle du répertoire secondaire des cellules B, au-delà du centre germinatif. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Ce travail a été soutenu par l’ARC (Association pour la recherche sur le cancer), la Ligue nationale contre le cancer, le cancéropôle Grand Sud-Ouest, l’INCa (Institut national du cancer) et la région Limousin.
|
Footnotes |
1.
Goodnow
CC
,
Vinuesa
CG
,
Randall
KL
, et al.
Control systems and decision making for antibody production . Nat Immunol.
2010; ; 11 : :681.–688. 2.
Stavnezer
J
,
Guikema
JEJ
,
Schrader
CE
. Mechanism and regulation of class switch recombination . Annu Rev Immunol.
2008; ; 26 : :261.–292. 3.
Muramatsu
M
,
Kinoshita
K
,
Fagarasan
S
, et al.
Class switch recombination and hypermutation require activation-induced cytidine deaminase (AID), a potential RNA editing enzyme . Cell.
2000; ; 102 : :553.–563. 4.
Pinaud
E
,
Marquet
M
,
Fiancette
R
, et al.
The IgH locus 3’ regulatory region: pulling the strings from behind . Adv Immunol.
2011; ; 110 : :27.–70. 5.
Vincent-Fabert
C
,
Fiancette
R
,
Pinaud
E
, et al.
Genomic deletion of the whole IgH 3’ regulatory region (hs3a, hs1,2, hs3b, and hs4) dramatically affects class switch recombination and Ig secretion to all isotypes . Blood.
2010; ; 116 : :1895.–1898. 6.
Chauveau
C
,
Pinaud
E
,
Cogne
M
. Synergies between regulatory elements of the immunoglobulin heavy chain locus and its palindromic 3’ locus control region . Eur J Immunol.
1998; ; 28 : :3048.–3056. 7.
Péron
S
,
Laffleur
B
,
Denis-Lagache
N
, et al.
AID-Driven deletion causes immunoglobulin heavy chain “locus suicide recombination” in B cells . Science.
2012 ;
avril 26; (online). DOI: 10.1126/science.1218692. 8.
Chauveau
C
,
Cogné
M
. Palindromic structure of the IgH 3’locus control region . Nat Genet.
1996; ; 14 : :15.–16. 9.
Sepulveda
MA
,
Garrett
FE
,
Price-Whelan
A
, et al.
Comparative analysis of human and mouse 3’ Igh regulatory regions identifies distinctive structural features . Mol Immunol.
2005; ; 42 : :605.–615. 10.
Kim
TK
,
Hemberg
M
,
Gray
JM
, et al.
Widespread transcription at neuronal activity-regulated enhancers . Nature.
2010; ; 465 : :182.–187. 11.
Wuerffel
R
,
Wang
L
,
Grigera
F
, et al.
S-S synapsis during class switch recombination is promoted by distantly located transcriptional elements and activation-induced deaminase . Immunity.
2007; ; 27 : :711.–722. 12.
Catalan
N
,
Imai
K
,
Revy
P
, et al.
Deux ans après, l’activation induced cytidine deaminase n’a pas livré tous ses secrets . Med Sci (Paris).
2003; ; 19 : :139.–141. 13.
Reynaud
CA
,
Delbos
F
,
Aoufouchi
S
, et al.
Hypermutation des gènes des immunoglobulines et polymérases mutagènes : quand l’erreur devient une qualité . Med Sci (Paris).
2007; ; 23 : :470.–472. |