| |
| Med Sci (Paris). 2012 August; 28(8-9): 695–698. Published online 2012 August 22. doi: 10.1051/medsci/2012288007.Les transferts horizontaux de gènes et l’arbre de la vie Vincent Daubin1* and Sophie Abby2 1UMR CNRS 5558-LBBE Biométrie et biologie évolutive, équipe bioinformatique et génomique évolutive, UCB Lyon 1, bâtiment Grégor Mendel, 43 boulevard du 11 novembre 1918, 69622Villeurbanne Cedex, France 2Microbial evolutionary genomics, Institut Pasteur, 25, rue du Docteur Roux, 75724Paris Cedex 15, France MeSH keywords: Bactéries, génétique, Évolution moléculaire, Extinction biologique, Transfert horizontal de gène, Spéciation génétique, Viabilité microbienne, Phylogénie, Sélection génétique |

Photo : le grand arbre de la vie, réalisée par Samuel Laganier (plasticien designer), Lenke Sifko (graphiste), Sylvain Charlat et Vincent Daubin (expertise scientifique), exposée à l’université Lyon 1. Dans les années 1960, la phylogénie, discipline qui cherche à reconstruire l’histoire évolutive du vivant et les liens généalogiques entre espèces en les comparant, entra dans une nouvelle ère, celle du « moléculaire ». Basée sur la recherche de caractères dérivés partagés entre organismes, elle trouva en l’ADN une matière abondante dont l’évolution se prêtait bien à la modélisation. Particulièrement chez les micro-organismes, où les critères de comparaisons manquaient cruellement pour comprendre leur histoire, la phylogénie moléculaire fut une révolution qui permit notamment la découverte des archées, le troisième grand domaine du vivant après les eucaryotes et les bactéries [
1]. Pour reconstruire l’histoire des espèces, il suffisait ainsi de choisir une (ou plusieurs) famille(s) de gènes, considérée comme « marqueur moléculaire » de l’histoire des espèces, et de reconstruire l’arbre qui expliquait au mieux les différences observées entre organismes. Enfin, idéalement… Car si les gènes conservent les traces des évènements qui ont marqué l’histoire des organismes qui les portent, ils enregistrent surtout leur propre histoire qui est bien plus complexe (Figure 1). Outre les événements de spéciations, les gènes subissent bien d’autres événements. Le mieux documenté d’entre eux est sans doute la duplication : au gré des remaniements des chromosomes qui les portent, certains gènes peuvent se retrouver en plusieurs copies au sein d’un génome. L’histoire du gène reconstruite est alors un mélange intriqué de deux types d’évènements, duplication et spéciation, dont l’identification n’est pas toujours aisée, ce qui complique significativement leur interprétation. Mais déduire les liens de parenté entre espèces semble encore possible dans ce cas. C’est la découverte du transfert horizontal de gènes, et surtout de son ampleur, qui vint ébranler la confiance des phylogénéticiens en leurs méthodes.
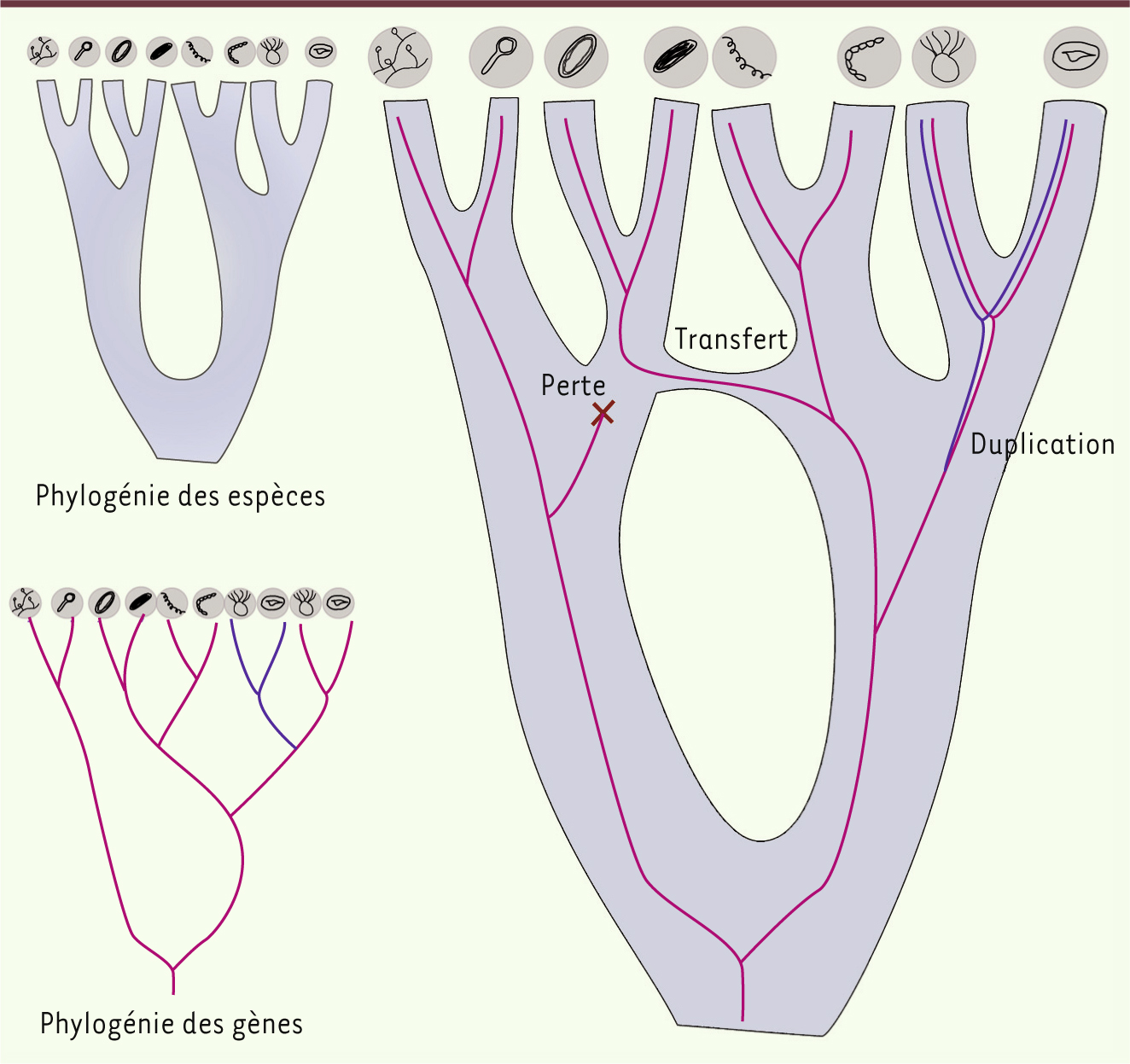
 | Figure 1.
Arbre des espèces et arbre de gènes. Les gènes sont portés par des espèces et suivent donc leur histoire, notamment les événements de spéciation qui font que les gènes d’une espèce ancestrale se retrouvent dans les lignées descendantes. Cependant, les gènes subissent aussi des événements d’évolution qui leur sont propres. Ils peuvent donc, dans certains cas, avoir une histoire plus complexe que celle des espèces. Ils peuvent être dupliqués, perdus, ou transférés d’une espèce à une autre. Ainsi, un arbre de gène reconstruit peut différer de l’arbre des espèces, car il est le résultat de différents mécanismes qu’il est nécessaire de prendre en compte. Mais si ces mécanismes complexifient les arbres de gènes, on peut aussi les considérer comme des marqueurs de l’évolution. Si deux espèces sont issues d’un ancêtre qui a reçu un gène par transfert, leurs génomes vont garder une trace de ce transfert commun à leur ancêtre, et donc conserver une preuve d’une ascendance partagée. |
|
La phylogénie des gènes à l’épreuve des transferts de gènes horizontaux Connue depuis les premières heures de la biologie moléculaire (l’expérience de Griffith [
2] puis celle d’Avery [
3] sur la transformation des pneumocoques sont dans tous les livres de génétique), la capacité de nombreux microorganismes à intégrer de l’ADN étranger dans leur génome, parfois « emprunté » à des espèces très éloignées, est devenue évidente avec le séquençage de génomes complets. Le rôle adaptatif et l’abondance de ce transfert horizontal de gènes apparaissent comme fondamentaux : que ce soit pour l’adaptation à des niches écologiques particulières, l’acquisition de facteurs de virulence ou de résistance aux antibiotiques, ou simplement la réparation de fonctions endommagées, la plupart des bactéries, archées et eucaryotes unicellulaires semblent utiliser ce mécanisme. En fait, l’importance de ces transferts est telle qu’il est possible qu’ils concernent toutes les histoires de gènes. Ainsi, lorsque l’on choisit l’un ou l’autre des milliers de marqueurs moléculaires disponibles pour reconstruire l’histoire d’un groupe de bactéries, les relations décrites sont presque toujours différentes. En effet, lorsqu’un arbre de gène contient des transferts, ceux-ci vont positionner l’espèce receveuse du gène étranger à côté de l’espèce donneuse, et non pas à côté de ses espèces sœurs, ce qui crée une incohérence entre la phylogénie du gène et celle des espèces (Figure 1). Mais alors, à qui faire confiance ? Si tous les gènes sont potentiellement affectés, quel crédit accorder à cet arbre du vivant reconstruit sur des caractères moléculaires ? Et si, chez les microorganismes, tout le monde échange des gènes avec tout le monde, au mépris du concept d’espèce, cette idée d’arbre a-t-elle seulement un sens ? Ainsi, on pourrait se représenter la diversité du vivant comme un grand réseau d’organismes connectés par des transferts de gènes [
4]. Cependant, si cette idée est attirante, elle n’aide pas vraiment à replacer cette diversité dans une perspective d’évolution. Il est crucial de garder à l’esprit que parmi les relations décrites par une phylogénie de gènes, certaines représentent des transmissions de parents à descendants et d’autres des transferts horizontaux [
5]. Et c’est dans cette nuance que résident les traces de l’histoire du vivant. |
Transfert horizontal de gène : un outil précieux pour la reconstitution de l’histoire du vivant Finalement, si l’on revient à la notion de marqueurs moléculaires, on réalise que la phylogénie moléculaire ne raconte que des histoires… de gènes. On peut cependant voir le transfert horizontal comme un évènement évolutif classique. Et en cela, un caractère utile pour la phylogénie. Si des espèces actuelles partagent un ancêtre commun qui a reçu un gène par transfert, elles gardent la trace de cet événement dans leurs génomes. Et ce type de caractères partagés peut donc être utilisé pour reconstruire les liens entre espèces. Afin de mettre en pratique cette idée, nous avons tout d’abord développé une méthode efficace permettant d’identifier les transferts horizontaux dans une histoire de gène, étant donné une histoire hypothétique des espèces [
6]. Cette méthode bioinformatique permet d’analyser les séquences d’un grand nombre de génomes afin de rechercher, pour chacun des gènes qu’ils contiennent, le scénario de transfert le plus parcimonieux permettant d’expliquer les différences entre l’arbre des espèces et l’arbre des gènes. L’originalité de cette méthode est qu’elle s’appuie non seulement sur les différences de formes de ces arbres, mais également sur la confiance statistique que nous pouvons avoir dans ces différences. Le résultat est, pour chaque gène, la description complète de la manière dont il a été transmis au cours de son histoire, soit de parents à descendants, soit par transfert horizontal. En se basant donc sur les phylogénies de milliers de familles de gènes reconstruites par des méthodes de phylogénie moléculaire, nous avons pu tester la capacité de différentes histoires d’espèces candidates à expliquer ces histoires de gènes par des scénarios de transferts [
7]. Le résultat est alors sans appel : les scénarios de transferts montrent une forte discrimination entre les possibles histoires d’espèces, c’est-à-dire que selon l’histoire des espèces considérée, le nombre de transferts horizontaux qu’il faut invoquer pour expliquer le contenu en gènes des espèces diffère fortement. Ceci démontre qu’il existe une histoire des espèces qui permet de minimiser le nombre de transferts horizontaux, et donc de mieux expliquer les répertoires de gènes des génomes actuels. Ainsi, dans la plupart des grands groupes bactériens et archéens testés, il est possible, grâce au transfert horizontal, de reconstruire simultanément une histoire des espèces et des scénarios de transferts permettant d’expliquer au mieux l’ensemble des histoires de gènes présents dans les génomes. Le transfert horizontal se révèle donc être non pas un obstacle, mais un outil précieux pour la reconstruction de l’histoire du vivant. |
Aspects quantitatifs et qualitatifs des transferts horizontaux Notre méthode a cet avantage que tout en permettant de reconstruire un arbre de la vie, elle permet l’identification des évènements de transfert qui ont marqué l’évolution du vivant. Il est alors possible d’étudier le rôle du transfert horizontal dans l’évolution des grandes lignées bactériennes. Le taux de transfert global sur l’arbre du vivant reste relativement faible : nous estimons que dans une histoire de gènes, en moyenne 5 % des branches correspondent à des transferts horizontaux, le reste correspondant à de la transmission verticale, de parents à descendants. Il est donc faux de considérer que la plupart des branches d’un arbre de gène sont affectées par le transfert. Mais ce taux de transfert peut varier de manière importante entre lignées : la
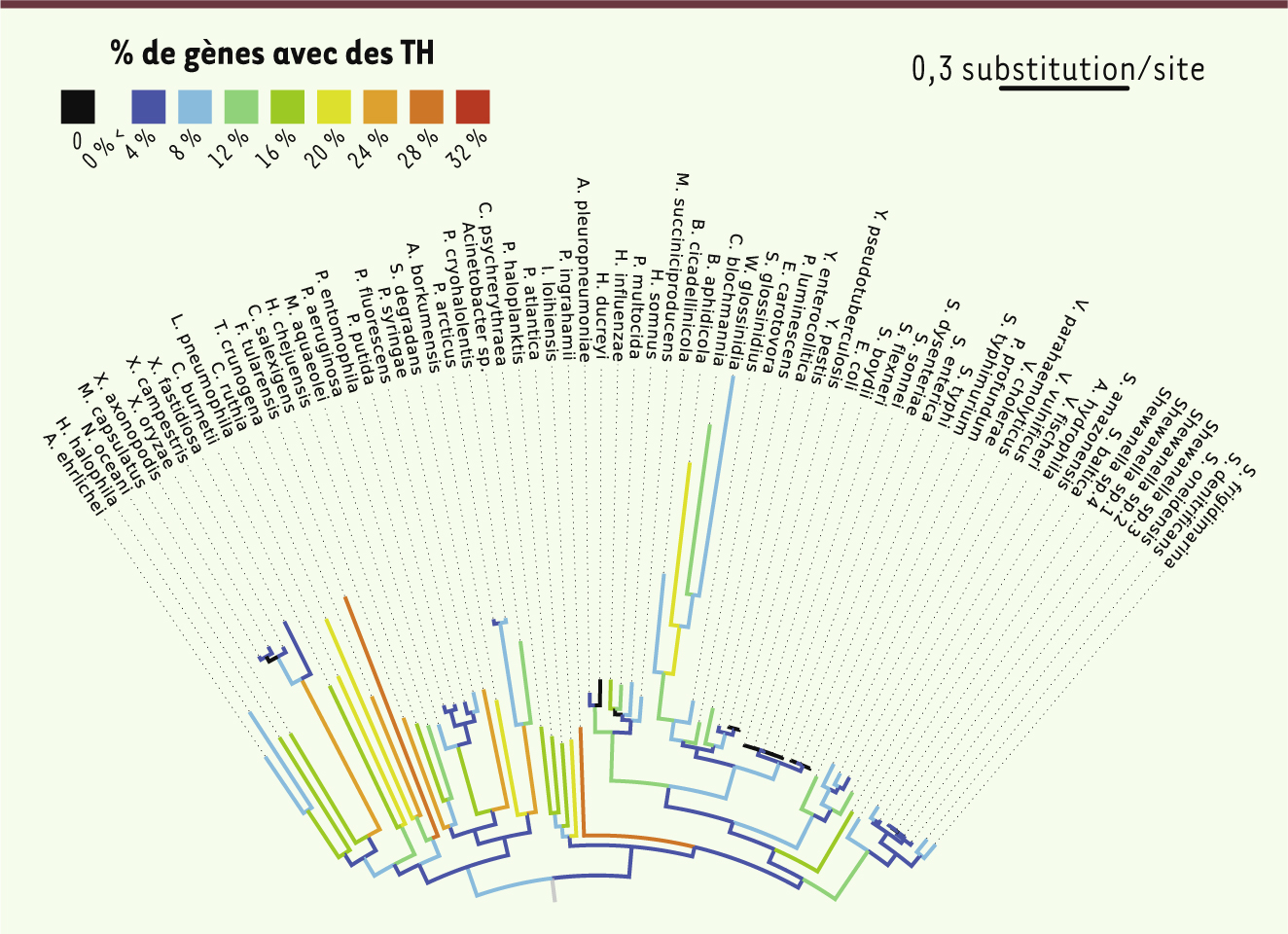
Figure 2
représente une partie de l’arbre du vivant, les gamma-protéobactéries, où les variations de taux de transfert sont particulièrement marquantes. On y voit que sur certaines branches, 20 à 30 % des gènes ont pu être transférés ! Ces taux extrêmes représentent essentiellement des évènements de transferts récents, les branches internes de l’arbre présentant des taux beaucoup plus faibles. Ceci suggère que si l’évolution fait l’expérience de nombreux transferts, ceux qui sont conservés à long terme et qui deviennent indispensables à la vie de grands groupes d’organismes restent rares.
 | Figure 2.
Les transferts horizontaux chez les gamma-protéobactéries. Cet arbre, choisi grâce à l’information des transferts horizontaux (TH) de gènes, représente les relations de parenté supposées entre les espèces de gamma-protéobactéries analysées. La couleur des branches reflète le pourcentage de familles pour lesquelles un transfert a été inféré au niveau de celles-ci. |
Il est également intéressant de regarder quelles fonctions sont particulièrement affectées ou au contraire résistantes au transfert. Nos résultats confirment que tous les types de fonctions cellulaires sont affectés par des transferts. Cependant, les gènes dont les fonctions sont liées à des processus centraux de la vie de la cellule, notamment la transmission et la traduction de l’information génétique montrent des taux nettement plus faibles que les gènes aux fonctions plus périphériques, tels que des facteurs de virulence par exemple. Enfin, au-delà de son intérêt pour reconstruire les liens de parentés entre espèces, notre étude démontre également le potentiel du caractère « transfert horizontal » pour résoudre la difficile question du positionnement de la racine, c’est-à-dire de la position de l’ancêtre le plus ancien dans l’arbre. En effet, la plupart des changements moléculaires classiquement modélisés en phylogénie ne sont pas orientés dans le temps, c’est-à-dire qu’ils permettent de révéler la forme d’un arbre (quelles espèces doivent être regroupées avec quelles autres), mais n’indiquent pas dans quel sens le temps s’est écoulé. Au contraire, le transfert horizontal peut aisément être orienté dans le temps car il implique que les espèces donneuse et receveuse du transfert étaient contemporaines. Ainsi, tout scénario de transfert est relatif à la manière dont les événements de spéciation de l’arbre des espèces sont ordonnés dans le temps. Ces résultats suggèrent ainsi qu’en modélisant le phénomène de transfert horizontal, on pourra bientôt utiliser ces caractères pour reconstruire une chronologie précise de l’histoire du vivant. Autrefois cauchemar des phylogénéticiens, le transfert horizontal doit aujourd’hui être considéré non seulement comme un moteur essentiel de l’évolution, mais aussi comme une clé pour sa compréhension [
8]. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Woese
CR
,
Fox
GE.
Phylogenetic structure of the prokaryotic domain: the primary kingdoms . Proc Natl Acad Sci USA.
1977; ; 74 : :5088.–5090. 2.
Griffith
F.
The significance of pneumococcal types . J Hyg (Lond).
1928; ; 27 : :113.–159. 3.
Avery
OT
,
Macleod
CM
,
McCarty
M.
Studies on the chemical nature of the substance inducing transformation of pneumococcal types: induction of transformation by a desoxyribonucleic acid fraction isolated from Pneumococcus type Iii . J Exp Med.
1944; ; 79 : :137.–158. 4.
Doolittle
WF.
Phylogenetic classification and the universal tree . Science.
1999; ; 284 : :2124.–2129. 5.
Gribaldo
S
,
Brochier
C.
Phylogeny of prokaryotes: does it exist and why should we care?
Res Microbiol.
2009; ; 160 : :513.–521. 6.
Abby
SS
,
Tannier
E
,
Gouy
M
,
Daubin
V.
, Detecting lateral gene transfers by statistical reconciliation of phylogenetic forests . BMC Bioinformatics.
2010; ; 11 : :324.. 7.
Abby
SS
,
Tannier
E
,
Gouy
M
,
Daubin
V.
Lateral gene transfer as a support for the tree of life . Proc Natl Acad Sci USA.
2012; ; 109 : :4962.–4967. 8.
Boussau
B
,
Daubin
V.
Genomes as documents of evolutionary history . Trends Ecol Evol.
2010; ; 25 : :224.–232. |