| |
| Med Sci (Paris). 2012 November; 28(11): 929–931. Published online 2012 November 12. doi: 10.1051/medsci/20122811009.Des mutations épigénétiques spécifiques aux enhancers sont associées à la carcinogenèse colorectale Mylène Brunelle,1 Nicolas Gévry,1 and Mathieu Lupien2* 1Département de biologie, faculté des sciences, université de Sherbrooke, Sherbrooke, Québec, Canada 2Ontario Cancer Institute, University Health Network and Department of Medical Biophysics, University of Toronto, Toronto, Ontario, Canada. Toronto Medical Discovery Tower, 9th Floor Room 9-314, 101 College Street, M5G 1L7Toronto, Canada MeSH keywords: Adénocarcinome, génétique, Chromatine, ultrastructure, Tumeurs colorectales, Éléments activateurs (génétique), Épigenèse génétique, Analyse de profil d'expression de gènes, Régulation de l'expression des gènes tumoraux, Étude d'association pangénomique, Humains, Jumonji Domain-Containing Histone Demethylases, physiologie, Mutation, Nucléosomes, Polymorphisme de nucléotide simple, Transcription génétique |
Les cellules cancéreuses sont caractérisées par l’expression aberrante d’un programme transcriptionnel. Aussi, une meilleure compréhension des mécanismes responsables de cette dérégulation permettrait de développer des stratégies de prévention, de détection et de traitement plus efficaces. À ce jour, des associations ont été identifiées entre des perturbations génétiques et/ou épigénétiques, majoritairement situées dans les régions codantes ou promotrices proximales, et la tumorigenèse. Elles ont permis de mieux appréhender les mécanismes transcriptionnels complexes sous-jacents au cancer. Cependant, le moteur principal de l’expression génique est situé dans les régions non codantes du génome dont l’analyse était limitée avant l’avènement des techniques de séquençage à haut débit. Dans un article récemment publié dans la revue Science, B. Akhtar-Zaidi et al. [
1] ont, grâce à ces technologies émergentes, cartographié, à l’échelle du génome, la signature épigénomique des régions régulatrices distales, les enhancers, dans des lignées cellulaires isolées de tumeurs colorectales primaires. Cette signature, qui n’est pas retrouvée dans les cellules épithéliales normales du côlon, dirigerait ainsi un programme transcriptionnel unique, participant à la carcinogenèse colorectale. Le cancer colorectal (CCR) résulte d’une accumulation de modifications génétiques et épigénétiques (facteurs non génétiques, héritables ou dynamiques) qui influencent l’expression des gènes et transforment les cellules épithéliales de la crypte en adénocarcinomes [
2]. Bien que certaines de ces modifications aient été étudiées en détail, notamment la méthylation aberrante de l’ADN ou les mutations d’oncogènes et de suppresseurs de tumeurs, d’autres demeurent inexplorées, par exemple les changements apportés à la structure et à la composition de la chromatine. De plus, il reste à déterminer les implications mutuelles des dérégulations génétiques et épigénétiques dans la pathogenèse du CCR. La chromatine, par sa structure et sa composition, permet la régulation complexe de l’expression génique en contrôlant de manière dynamique l’accessibilité des facteurs de transcription aux éléments régulateurs, tels que le promoteur et les enhancers qui sont souvent éloignés du gène qu’ils contrôlent (Figure 1) [
3,
4]. Récemment, il a été démontré que le type d’élément régulateur est déterminé par le niveau de méthylation de la lysine 4 de l’histone H3 (H3K4). Les enhancers sont ainsi marqués par la mono et/ou la diméthylation (H3K4me1/me2), et les promoteurs par la di- et la triméthylation (H3K4me2/me3) [
5,
6]. De plus, l’acétylation de la lysine 27 de l’histone H3 (H3K27ac) est associée à l’activité de ces éléments régulateurs [6,
7]. La comparaison de la localisation de cette signature chromatinienne dans différents types cellulaires a permis de confirmer la nature spécifique des enhancers pour une cellule donnée [6–
8]. Ceux-ci sont donc le déterminant majeur de l’expression du programme transcriptionnel responsable de l’identité cellulaire et, de ce fait, leur dérégulation pourrait être une composante clé du développement du cancer.
 | Figure 1.
La chromatine et les éléments régulateurs. La chromatine, un complexe nucléoprotéique responsable du compactage de l’ADN, possède plusieurs niveaux d’organisation structurale [ 3, 4]. Le premier niveau est le nucléosome, formé de l’enroulement de l’ADN autour de l’octamère d’histones, constitué de deux molécules de chacune des histones H2A, H2B, H3 et H4. Les queues amino-terminales des histones, surgissant à l’extérieur du nucléosome, sont sujettes à plusieurs modifications post-traductionnelles, notamment la méthylation (me) et l’acétylation (ac), prenant place, entre autres, sur les résidus lysine (K). Des complexes protéiques possédant une activité enzymatique sont chargés d’ajouter ou d’enlever ces modifications. Ces changements épigénétiques influencent le niveau de compaction de la chromatine et/ou le recrutement d’autres protéines ce qui, en contrôlant temporellement l’accessibilité pour la liaison des facteurs de transcription (TF) aux éléments régulateurs (le promoteur et les enhancers), permet une régulation fine de l’expression génique. Le promoteur est situé autour du site d’initiation de la transcription (SIT) et fournit, entre autres, les indications à l’assemblage du complexe de pré-initiation de la transcription et de l’ARN polymérase II (Pol II). Les enhancers, contenant des sites de liaison pour les facteurs de transcription, sont souvent situés à plusieurs milliers de paires de bases du promoteur, et peuvent activer la transcription de façon indépendante de leur localisation ou orientation vis-à-vis du gène régulé. Les éléments régulateurs possèdent une signature chromatinienne unique permettant de les identifier. Ainsi, H3K4me1/me2 définit les enhancers et H3K4me3 les promoteurs, alors que la présence combinée de ces marques avec H3K27ac révèle leur activité. |
|
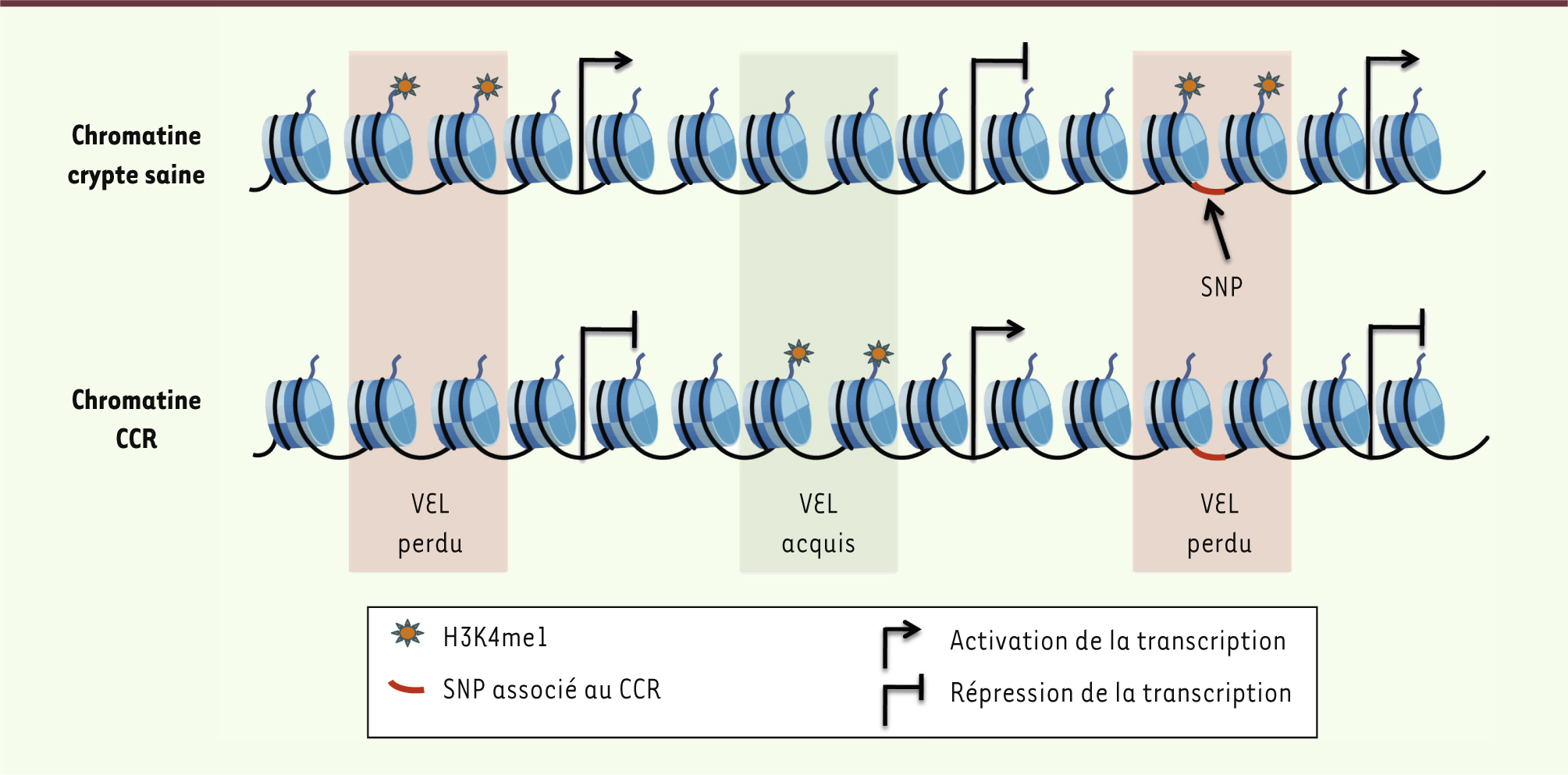
La cartographie épigénomique fonctionnelle des enhancers associés à la carcinogenèse colorectale B. Akhtar-Zaidi et al. [1] ont comparé les régions génomiques enrichies en H3K4me1 dans des cellules épithéliales saines de la crypte et dans des lignées de cellules de CCR. De cette façon, des milliers de régions génomiques différentiellement enrichies en H3K4me1, les VEL (variant enhancer locus), ont été mises en évidence. Cette variation, gain ou perte de la marque d’enhancer, ne serait pas due à une instabilité génétique, telle que la variation du nombre de copies de la séquence d’ADN, suggérant l’implication d’évènements purement épigénétiques. Par ailleurs, l’étude de l’enrichissement de H3K27ac permet de distinguer les enhancers actifs et inactifs [6, 7,
9,
10]. Elle met en évidence le fait que certains des VEL deviennent actifs dans les cellules CCR, alors que la majorité des VEL perdus ne présentent plus cette marque d’enhancers actifs, bien que ces régions soient marquées par H3K27ac dans les cellules normales. Ces résultats témoignent de multiples changements dans l’état de la chromatine dans les cellules cancéreuses. Leur modèle récapitulatif de la tumorigenèse colorectale permet à ces auteurs de déterminer si les changements dans l’épigénome des cellules CCR évoquent un mécanisme général et reproductible de la carcinogenèse colorectale ou sont le résultat d’évènements aléatoires. Ainsi, 197 VEL sont communs à toutes les lignées CCR, alors que 2 604 VEL acquis et 2 047 VEL perdus sont détectés de façon significative dans les lignées étudiées. De façon intéressante, les auteurs révèlent que ces VEL acquis ou perdus durant la carcinogenèse reflétent une caractéristique universelle des cancers : la perte du caractère de différenciation des cellules. En effet, les VEL perdus sont caractéristiques des cellules de la crypte, alors que les VEL acquis sont non spécifiques. Ainsi, les cellules cancéreuses ont acquis un réseau d’enhancers habituellement présents dans d’autres types cellulaires, et ont perdu la signature chromatinienne associée au phénotype caractéristique des cellules normales de la crypte. Ces changements épigénomiques résument donc la perte de différenciation observée dans les cellules cancéreuses. Les enhancers dirigent, via la modulation de l’expression des gènes, le phénotype cellulaire [6–8]. Ainsi, l’acquisition ou la perte de ces éléments devrait se traduire par l’altération du programme transcriptionnel normal. De fait, il a été rapporté que les gènes surexprimés dans des lignées de cellules CCR sont préférentiellement associés aux VEL acquis, alors que les gènes réprimés sont fortement associés aux VEL perdus. De plus, cette relation persiste dans des tumeurs colorectales primaires. Cette relation, qui s’exprime également par des changements épigénétiques des promoteurs associés, valide l’interprétation fonctionnelle de ce nouveau réseau d’enhancers retrouvés systématiquement au cours des différents stades du cancer du côlon. Ce d’autant que cette signature épigénétique se révèle beaucoup plus reproductible comparée à l’hétérogénéité des mutations des gènes driver que détecte le séquençage génomique. |
Les VEL et les variants génétiques Les GWAS (genome wide association study) permettent d’identifier les variants génétiques associés à des phénotypes pathologiques. À ce jour, plus de 20 variants génétiques, présents sous forme de polymorphismes nucléotidiques (SNP), ont été associés au risque de développement du CCR. Les travaux de B. Akhtar-Zaidi et al. [1] révèlent que ces SNP, enrichis dans des régions enhancers marquées par H3K4me1 et spécifiques aux cellules de la crypte, sont détectés dans des régions de VEL acquis ou perdus dans les cellules CCR. Ces résultats démontrent la convergence de mécanismes de dérégulation génétiques et épigénétiques dans des régions clés impliquées dans la carcinogenèse colorectale. Cette association SNP/enhancers a également été retrouvée dans d’autres pathologies [6]. |
Les résultats obtenus par B. Akhtar-Zaidi et al. [1], résumés dans la Figure 2, révèlent l’importance des changements épigénétiques survenus dans des régions fonctionnelles comme fil conducteur du CCR. Plusieurs questions restent toutefois en suspens. Ainsi, ces changements dans l’état de la chromatine surviennent-ils également en réponse aux traitements administrés pour contrer les tumeurs et, si oui, sont-ils impliqués dans l’établissement de la résistance à ces traitements ? Qu’en est-il des bouleversements épigénomiques induits par les carcinogènes environnementaux ? Les modifications de la composition de la chromatine se traduisent-elles par une altération de sa structure ? Comment les SNP influencent-ils l’intégration et la propagation de l’information, provenant de multiples voies de signalisation, aux enhancers ? En définitive, l’identification des mécanismes sous-jacents aux remaniements épigénomiques est nécessaire et ouvrira certainement la voie à de nouveaux outils de diagnostic, mais également de prévision et de suivi de la réponse aux traitements.
 | Figure 2.
Schéma récapitulatif des changements épigénomiques et de leur impact dysfonctionnel dans un modèle de carcinogenèse colorectale. VEL : variant enhancer locus. |
|
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Akhtar-Zaidi
B
,
Cowper-Sal-Lari
R
,
Corradin
O
, et al.
Epigenomic enhancer profiling defines a signature of colon cancer . Science.
2012; ; 336 : :736.–739. 2.
Van Engeland
M
,
Derks
S
,
Smits
KM
, et al.
Colorectal cancer epigenetics: complex simplicity . J Clin Oncol.
2011; ; 29 : :1382.–1391. 3.
Bell
O
,
Tiwari
VK
,
Thomä
NH
,
Schübeler
D.
Determinants and dynamics of genome accessibility . Nat Rev Genet.
2011; ; 12 : :554.–564. 4.
Ong
CT
,
Corces
VG.
Enhancer function: new insights into the regulation of tissue-specific gene expression . Nat Rev Genet.
2011; ; 12 : :283.–293. 5.
Heintzman
ND
,
Stuart
RK
,
Hon
G
, et al.
Distinct and predictive chromatin signatures of transcriptional promoters and enhancers in the human genome . Nat Genet.
2007; ; 39 : :311.–318. 6.
Ernst
J
,
Kheradpour
P
,
Mikkelsen
TS
, et al.
Mapping and analysis of chromatin state dynamics in nine human cell types . Nature.
2011; ; 473 : :43.–49. 7.
Heintzman
ND
,
Hon
GC
,
Hawkins
RD
, et al.
Histone modifications at human enhancers reflect global cell-type-specific gene expression . Nature.
2009; ; 459 : :108.–112. 8.
Lupien
M
,
Eeckhoute
J
,
Meyer
CA
, et al.
FoxA1 translates epigenetic signatures into enhancer-driven lineage-specific transcription . Cell.
2008; ; 132 : :958.–970. 9.
Creyghton
MP
,
Cheng
AW
,
Welstead
GG
, et al.
Histone H3K27ac separates active from poised enhancers and predicts developmental state . Proc Natl Acad Sci USA.
2010; ; 107 : :21931.–21936. 10.
Rada-Iglesias
A
,
Bajpai
R
,
Swigut
T
, et al.
A unique chromatin signature uncovers early developmental enhancers in humans . Nature.
2011; ; 470 : :279.–283. |