| |
| Med Sci (Paris). 2012 November; 28(11): 970–975. Published online 2012 November 12. doi: 10.1051/medsci/20122811016.Rôle de l’ubiquitine ligase c-Cbl dans l’ostéogenèse normale et cancéreuse
Nicolas Sévère1,2 and Pierre Marie1,2* 1Laboratoire de biologie et pathologie de l’ostéoblaste, Inserm UMR 606, Université Paris-Diderot Sorbonne Paris-Cité, Paris, 75475, France 2hôpital Lariboisière, 2, rue Ambroise Paré, 75010, Paris, France |
L’ubiquitination : un signal de mort Le processus d’ubiquitination a été découvert par Aaron Ciechanover, Avram Hershko et Irwin Rose, ce qui leur a valu le prix Nobel de chimie en 2004 (→) [
35]. L’ubiquitination est un signal de mort qui, via le marquage de protéines cibles par une protéine (ubiquitine), permet leur dégradation par les lysosomes ou le protéasome. Plusieurs types d’ubiquitination participent également à l’endocytose, à la réparation de l’ADN ou encore au transport des protéines [
1]. L’ubiquitination conduisant à la dégradation des protéines est une étape importante qui participe au contrôle de l’homéostasie cellulaire. La dégradation des protéines contrôle en effet de nombreux processus cellulaires tels que le cycle cellulaire, la prolifération, la différenciation et l’apoptose. La cascade d’ubiquitination nécessite trois enzymes. La première, E1, recrute l’ubiquitine sur un de ses résidus cystéine grâce à une liaison thioester [
2]. L’ubiquitine est ensuite transférée de l’enzyme E1 sur un résidu cystéine de la seconde enzyme, E2 [
3]. Enfin, la troisième enzyme, E3, est une ubiquitine ligase qui fait le lien entre la protéine cible à dégrader et l’enzyme E2 et, de ce fait, catalyse le transfert de l’ubiquitine sur la protéine cible. Cette réaction est réversible, grâce à l’action de déubiquitinases qui enlèvent l’ubiquitine liée à la protéine cible. Plus de 600 ubiquitine ligases E3 ont été décrites chez l’homme, dont les protéines de la famille Cbl (Casitas B-lineage lymphoma). Ce sont des enzymes clés qui contrôlent la spécificité de la réaction d’ubiquitination [
4].
(→) Voir m/s n° 12, décembre 2004, page 1156
|
Les ubiquitine ligases Cbl : des protéines à multiples domaines d’interaction Le premier membre de la famille Cbl a été isolé à partir d’un rétrovirus Cas-NS1 qui induit un lymphome (Casitas B-lineage lymphoma) issu de cellules préB et proB chez la souris. Cette forme, nommée v-Cbl, est une forme tronquée de la forme sauvage c-Cbl [
5]. Par la suite, trois formes de protéines, c-Cbl, Cbl-b et Cbl-c, ont été identifiées chez les mammifères. Deux protéines Cbl sont présentes chez la drosophile, une isoforme courte d-Cbls et une isoforme longue d-CblL. Une seule protéine Cbl a été identifiée chez le nématode Caenorhabditis elegans, nommée Sli-1. Les protéines Cbl sont des protéines à multiples domaines d’interaction et deux régions sont très conservées : le domaine d’interaction avec les tyrosine kinases (domaine TKB) et le domaine RING (Figure 1).
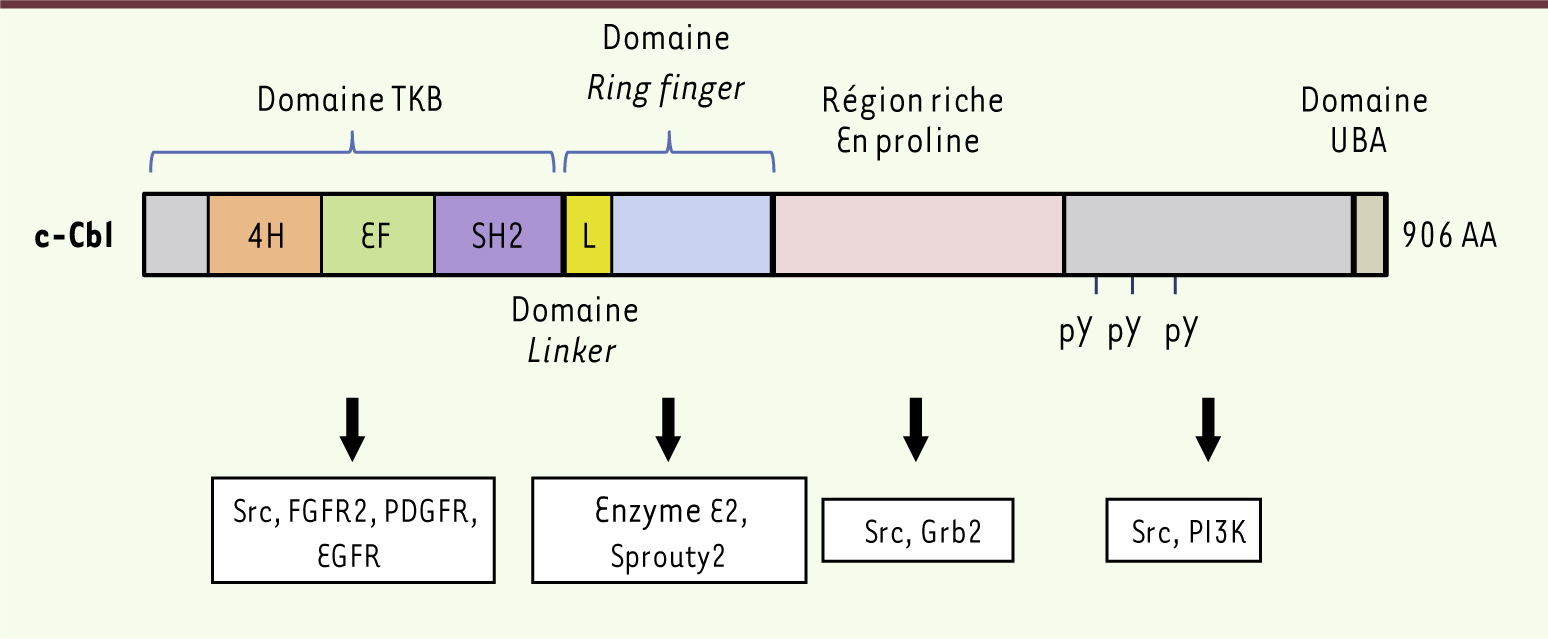
 | Figure 1.
Domaines d’interactions de c-Cbl avec les protéines partenaires. Le domaine TKB permet l’interaction avec les tyrosines phosphorylées des autres protéines. Le domaine RING contrôle l’activité ligase de l’enzyme E3 de Cbl. Le domaine UBA (ubiquitin associated domain) est un domaine d’interaction à l’ubiquitine. Ces domaines sont impliqués dans les interactions avec les protéines indiquées. AA : acides aminés. |
Le domaine TKB permet l’interaction de Cbl avec les tyrosines phosphorylées des autres protéines. Ce domaine est composé de quatre hélices enroulées, d’un domaine d’interaction avec le calcium et d’un domaine SH2 (src homology 2 domain). C’est ce dernier domaine qui dicte la spécificité du substrat avec lequel Cbl va interagir. Le domaine Linker permet de faire le lien entre le domaine TKB et le domaine RING. Il est composé de deux résidus tyrosine essentiels (Y368 et Y371). La délétion d’un des deux résidus entraîne la perte de fonction de Cbl et lui confère un potentiel oncogénique. Ces deux résidus servent de substrat de phosphorylation pour les récepteurs à activité tyrosine kinase (RTK) [
6]. Le domaine RING est un domaine à doigts de zinc (Ring finger) qui contrôle l’activité ligase de l’enzyme E3 et sert de site de fixation à l’enzyme E2. La partie carboxyterminale après le domaine RING est beaucoup moins conservée et sa longueur varie selon les membres de la famille Cbl. Néanmoins, deux éléments sont conservés : la région riche en proline et les résidus tyrosine (trois pour c-Cbl ou deux pour Cbl-b). La région riche en proline est impliquée dans les interactions avec les protéines à domaine SH3 comme PI3K (phosphoinositide 3-kinase), tandis que les tyrosines situées dans la partie en carboxyterminale sont phosphorylées par les Src kinases. Le domaine UBA (ubiquitin associated domain) situé à l’extrémité de c-Cbl et Cbl-b est un domaine d’interaction avec l’ubiquitine. Il interagit de façon non covalente avec l’ubiquitine ou de préférence avec les chaînes de polyubiquitine, et intervient aussi dans les interactions entre les protéines (Figure 1). Des expériences de mutagenèse ont montré que les domaines Linker et RING sont essentiels à l’activité ligase de l’enzyme E3 de Cbl. D’ailleurs, plusieurs mutations dans le domaine RING ont été identifiées dans différents types de cancer. Des mutations dans le domaine TKB inhibent l’interaction de Cbl avec les protéines à activité tyrosine kinase, mais ne semblent pas modifier l’activité ligase de l’enzyme E3 de Cbl. Du fait de leurs multiples domaines d’interactions, les protéines Cbl interagissent avec et contrôlent la dégradation d’un grand nombre de protéines parmi lesquelles on peut citer des récepteurs à activité tyrosine kinase (EGFR [epidermal growth factor receptor], IGF1R [insulin-like growth factor-1 receptor], PDGFR [plateled-derived growth factor receptor], FGFR [fibroblast growth factor receptor], VEGFR [vascular endothelial growth factor receptor], HGFR [hepatocyte growth factor receptor], Kit, etc.), les récepteurs des cytokines IL[interleukin]-2, IL-3, IL-4, CSF-1 (colony stimulating factor-1), les récepteurs à l’antigène sur les lymphocytes T et B, les intégrines (α5β1, αVβ3), les protéines cytosoliques à activité tyrosine kinase (Src kinases) et les protéines cytoplasmiques (PI3K) [
7,
8]. |
Rôle de c-Cbl dans le tissu osseux Pour assurer ses fonctions, le tissu osseux doit être en perpétuel renouvellement. Le remodelage osseux est assuré par deux types de cellules, les ostéoclastes et les ostéoblastes. Les ostéoclastes sont des cellules différenciées issues de précurseurs hématopoïétiques, qui adhèrent à la surface de la matrice osseuse et résorbent l’os minéralisé par des mécanismes enzymatiques complexes [
9]. Les ostéoblastes sont issus de la différenciation de cellules souches du stroma médullaire, et synthétisent les protéines qui vont constituer la matrice osseuse [
10]. Plusieurs études ont montré que les ligases E3 jouent un rôle important dans le remodelage osseux. Rôle de c-Cbl et Cbl-b dans la résorption osseuse par les ostéoclastes c-Cbl joue un rôle dans le contrôle la résorption osseuse par les ostéoclastes. Ces cellules expriment les deux isoformes c-Cbl et Cbl-b, au niveau du podosome. Dans ces cellules, c-Cbl et Cbl-b ont à la fois un rôle spécifique et redondant. En effet, l’extinction de l’expression de c-Cbl ou de Cbl-b entraîne un phénotype osseux modéré chez la souris. Ainsi, chez les souris c-Cbl
-/-
déficientes pour c-Cbl, il y a un défaut d’invasion par les ostéoclastes du cartilage hypertrophique, ce qui réduit l’émergence de la vascularisation nécessaire au remplacement du cartilage par l’os. L’extinction de l’expression de Cbl dans les ostéoclastes diminue leur activité de résorption au niveau du cartilage calcifié, ce qui provoque une persistance du cartilage au niveau des travées osseuses. Cependant, ce phénotype est transitoire et disparaît après la naissance [
11]. In vitro, les ostéoclastes c-Cbl
-/-
ont une adhésion et une mobilité réduites résultant d’anomalies de la fonction des protéines Src et PI3K [
12]. L’expression d’une protéine Cbl mutée dans le domaine RING (C379ACbl) inhibe l’activité d’ubiquitination de c-Cbl et diminue la mobilité des ostéoclastes, ce défaut de mobilité étant corrigé par l’expression de c-Cbl sauvage [
13]. Les ostéoclastes exprimant une forme mutée de c-Cbl (mutation dans le domaine d’interaction avec la PI3K) présentent un défaut de réorganisation du cytosquelette ; de plus, leur survie est augmentée par le M-CSF (macrophage colony stimulating factor) [
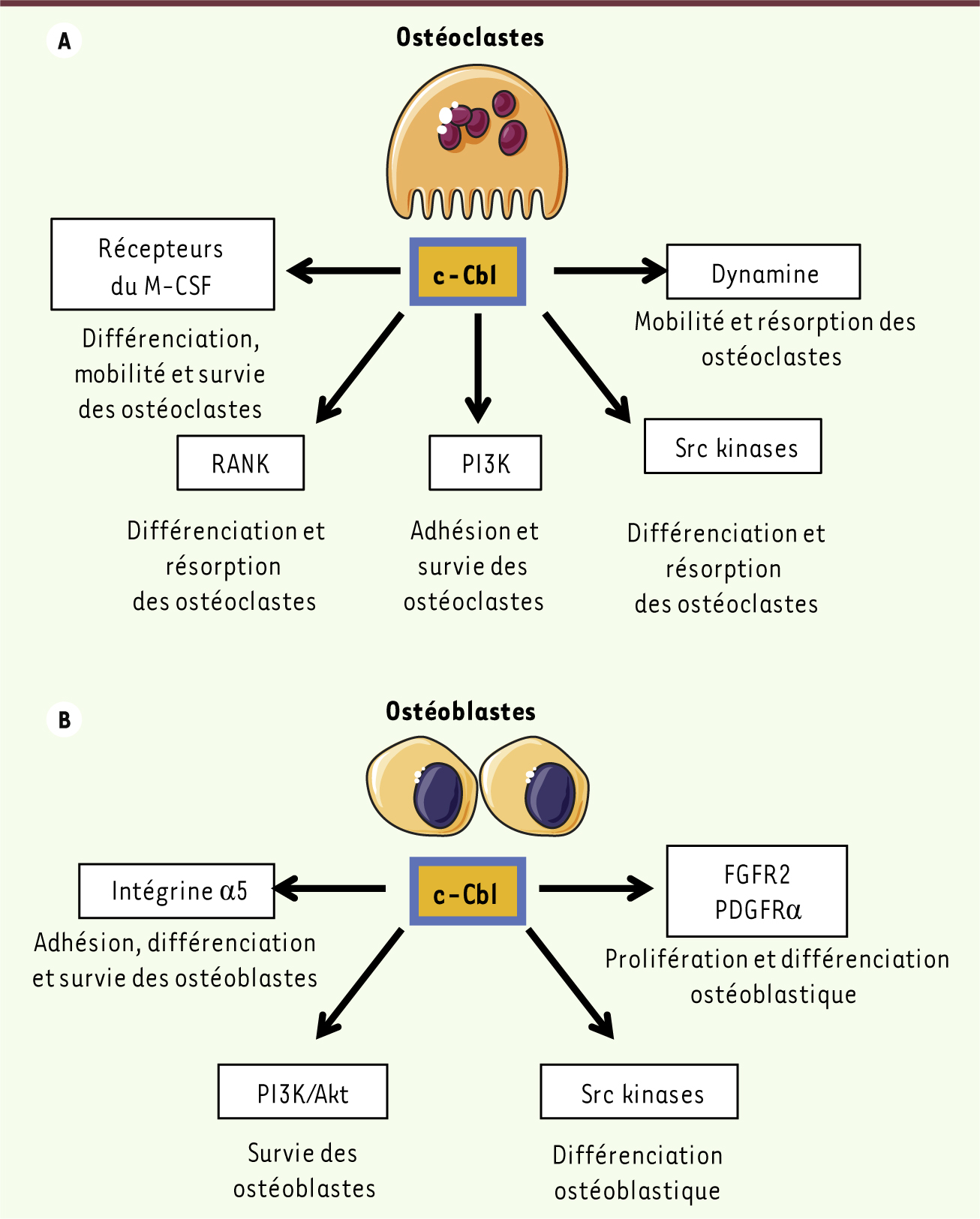
14]. L’interaction Cbl-PI3K est donc importante pour la régulation de la survie et de la réorganisation du cytosquelette dans les ostéoclastes (Figure 2A).
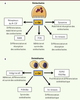 | Figure 2.
Rôle de c-Cbl dans le contrôle des cellules osseuses. Dans le tissu osseux, c-Cbl contrôle l’ubiquitination de nombreuses protéines qui sont impliquées dans l’adhérence, la réplication, la différenciation et la survie (A) des ostéoclastes (cellules résorbant l’os), et (B) des ostéoblastes (cellules formant l’os). |
Le phénotype osseux des souris Cbl-b
-/-
est légèrement différent et plus prononcé que celui des souris c-Cbl
-/-
. L’extinction de l’expression de Cbl-b augmente l’activité des ostéoclastes et induit une perte osseuse [
15]. In vitro, l’activité de résorption des ostéoclastes Cbl-b
-/-
est accrue par rapport à celle des ostéoclastes sauvages ou c-Cbl
-/-
. À l’inverse, la surexpression de la forme sauvage de Cbl-b réduit cette activité de résorption. L’extinction de Cbl-b semble favoriser les voies de signalisation NF-κB, ERK1/2 (extracellular signal-regulated kinase) et la mitogen-activated protein kinase p38. Le phénotype osseux des souris c-Cbl
-/-
et Cbl-b
-/-
n’est pas très prononcé, ce qui suggère que chaque protéine Cbl a son propre rôle dans la régulation des ostéoclastes, probablement en raison de protéines cibles différentes. De plus, une étude a montré que c-Cbl et Cbl-b peuvent se compenser mutuellement pour promouvoir la dégradation de Bcl2/Bim et contrôler l’apoptose des ostéoclastes [
16]. Rôle des protéines Cbl dans l’ostéogenèse Le rôle des protéines Cbl dans l’ostéoformation est moins connu. Les deux isoformes de Cbl sont exprimées dans les ostéoblastes, mais l’étude du phénotype osseux des souris c-Cbl
-/-
et Cbl-b
-/-
n’a pas mis en évidence d’altération importante de la formation osseuse in vivo, ce qui suggère un phénomème de compensation d’une protéine par l’autre [ 11]. Les travaux de notre laboratoire ont cependant montré que c-Cbl régule la formation osseuse en contrôlant la dégradation de plusieurs protéines impliquées dans l’ostéoblastogenèse (Figure 2B). Ainsi, la diminution de l’expression de c-Cbl induite par une mutation génétique du gène Twist provoque une augmentation de l’activité des voies de signalisations PI3K et Akt (protéine kinase B), ce qui entraîne une augmentation de la prolifération des ostéoblastes [
17]. En revanche, le recrutement de c-Cbl induit par l’activation du FGFR2 entraîne une diminution de l’expression des protéines Src, Lyn et Fyn, et d’autres récepteurs à activité tyrosine kinase, ce qui a pour effet d’augmenter l’expression des gènes ostéoblastiques et l’ostéogenèse [
18–
20]. Inversement, l’expression d’un c-Cbl muté dans le domaine RING (7OZ-Cbl) restaure l’expression de Lyn et Fyn et diminue l’expression des gènes ostéoblastiques [ 18]. Enfin, le recrutement de c-Cbl par le FGFR2 activé atténue l’activité de la voie PI3K/Akt, ce qui favorise la mort cellulaire des ostéoblastes [
21]. L’activation de c-Cbl induit aussi une diminution de l’expression de l’intégrine α5, ce qui augmente l’apoptose des ostéoblastes [ 21,
22]. À l’inverse, l’inhibition de l’interaction entre c-Cbl et la PI3K par l’expression d’un Cbl muté (Y 737FCbl) favorise la prolifération des ostéoblastes et augmente la formation osseuse chez la souris [
23]. Il faut noter également que Cbl-b intervient en régulant négativement la formation osseuse. Dans un modèle de perte osseuse par immobilisation chez la souris, l’expression de Cbl-b dans l’os est augmentée. Ceci induit une diminution de l’expression d’IGF1R, rendant les ostéoblastes moins sensibles à la réponse aux facteurs de croissance in vivo, ce qui entraîne une diminution de la formation osseuse [
24]. L’ensemble de ces données montre que c-Cbl joue un rôle dans le contrôle de l’ostéoblastogenèse (Figure 2B). Nos travaux récents ont mis en évidence que c-Cbl contrôle la différenciation en ostéoblastes des cellules souches mésenchymateuses (CSM) en modulant l’activité des RTK. C’est ainsi que lorsqu’on inhibe dans des CSM l’interaction entre c-Cbl et les RTK via un c-Cbl muté (G306ECbl) dans le domaine d’interaction avec les tyrosines phosphorylées (domaine TKB), on diminue l’ubiquitination et la dégradation des récepteurs PDGFRα et FGFR2, et on augmente l’expression des gènes ostéoblastiques, ainsi que l’ostéogenèse in vitro [
25]. Ces données suggèrent que l’on peut cibler l’interaction entre c-Cbl et les RTK pour favoriser la différenciation ostéoblastique des CSM, ce qui ouvre une nouvelle voie thérapeutique potentielle pour favoriser l’ostéogenèse et la réparation osseuse (Figure 3A). L’utilisation de molécules pharmacologiques capables d’inhiber de façon spécifique l’interaction entre c-Cbl et les RTK, molécules actuellement en développement [
26,
27], pourrait ainsi permettre de promouvoir la différenciation ostéogénique des cellules souches mésenchymateuses in vivo.
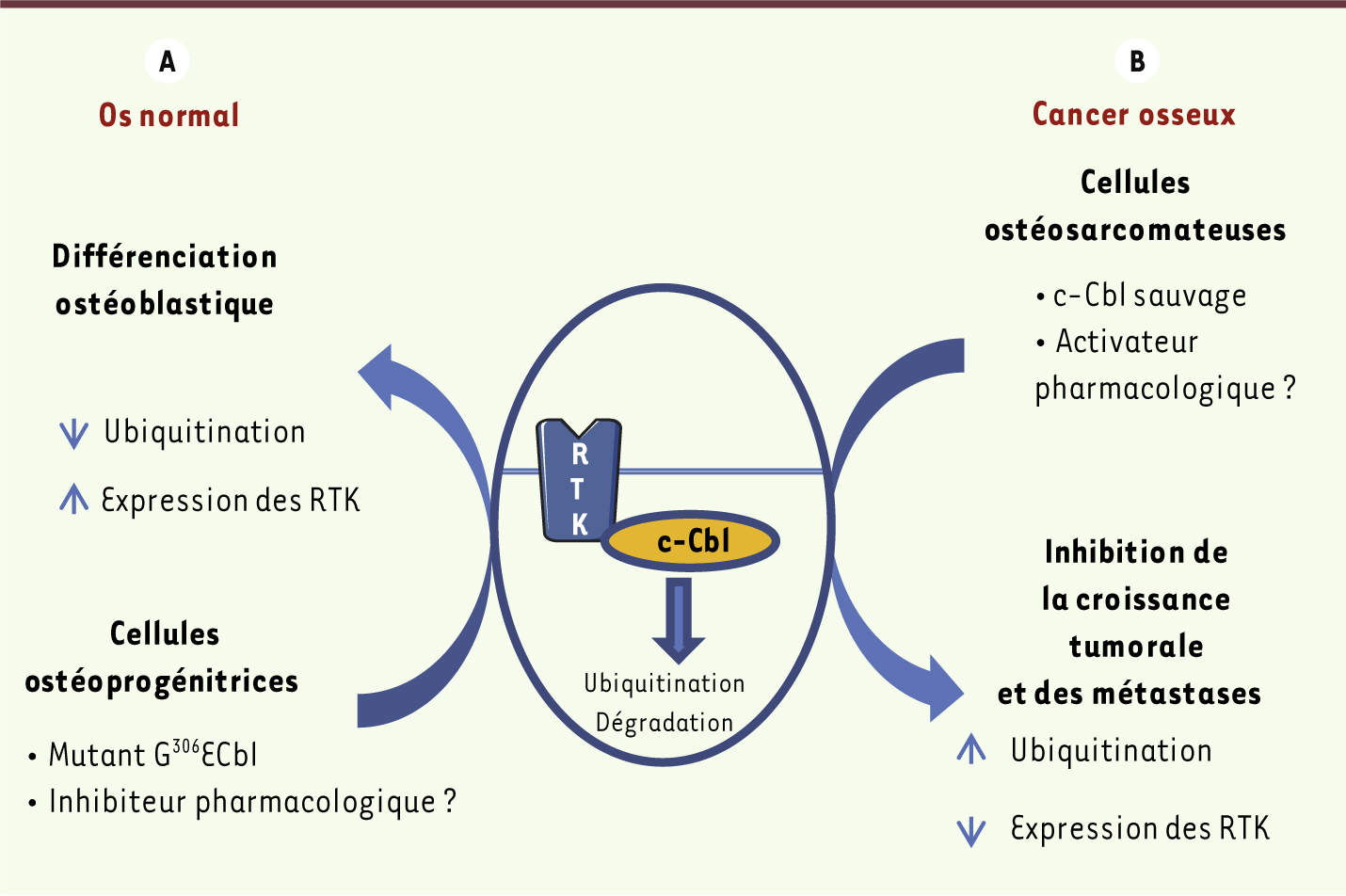
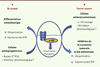 | Figure 3.
Rôle de c-Cbl dans le contrôle de l’ostéogenèse normale et pathologique. A. Dans l’os normal, on peut favoriser la différenciation des cellules ostéoprogénitrices par un c-Cbl muté dans le domaine TKB, ce qui diminue l’ubiquitination et la dégradation des RTK et augmente ainsi l’expression des gènes ostéoblastiques. B. Dans l’os tumoral, la surexpression de c-Cbl induit une dégradation des RTK, ce qui inhibe la croissance et l’invasion des cellules ostéosarcomateuses. L’utilisation d’agents pharmacologiques ciblant c-Cbl pourrait ainsi conduire à de nouvelles voies thérapeutiques pour contrôler l’ostéogenèse normale et pathologique. |
|
c-Cbl : une cible thérapeutique potentielle dans les cancers En contrôlant l’ubiquitination et la dégradation d’un grand nombre de protéines, le système ubiquitine-protéasome est impliqué dans la régulation de processus cellulaires essentiels au développement de l’organisme (prolifération, différenciation, survie, etc.). Il est ainsi compréhensible qu’un dérèglement du système de dégradation protéique puisse induire des aberrations cellulaires conduisant à des pathologies comme les cancers. Ainsi, un défaut de dégradation de protéines oncogéniques peut favoriser le développement de tumeurs mais, à l’inverse, un excès de dégradation de protéines antitumorales peut aussi stimuler les processus de cancérisation [
28]. En particulier, une anomalie de l’activité des protéines Cbl pourrait contribuer au processus tumoral puisqu’elles contrôlent l’ubiquitination et la dégradation de protéines ayant une activité tyrosine kinase. Plusieurs travaux ont en effet montré que des formes mutées de Cbl agissent comme des oncogènes, conférant aux protéines Cbl un statut de suppresseurs de tumeur par leur régulation de la dégradation des protéines [
29]. D’ailleurs, la première forme de Cbl étudiée (v-Cbl) était une forme tronquée du Cbl sauvage dépourvue d’activité ubiquitine ligase. Les travaux les plus convaincants quant au rôle de Cbl dans le développement tumoral ont été menés dans le myélome multiple (MM). Plusieurs mutations de c-Cbl ont en effet été identifiées chez des patients atteints de MM [
30]. La plupart des mutations détectées à ce jour sont dans les domaines Linker ou RING de Cbl, domaines qui contrôlent l’activité ligase de l’enzyme E3. Ces mutations inhibent l’activité ligase de Cbl et participent à l’activité oncogénique de Cbl. C’est ainsi que des mutations de c-Cbl induisent une augmentation de l’activité tyrosine kinase de certains récepteurs impliqués dans les néoplasmes myélofibreux [30]. Des mutations de c-Cbl ont été aussi détectées chez des sujets atteints de cancer du poumon. Ces mutations sont réparties dans tous les domaines de c-Cbl : trois mutations dans le domaine TKB, une mutation dans le domaine RING, deux mutations dans le domaine riche en proline et trois mutations dans la région carboxyterminale. L’expression de formes mutées de c-Cbl dans des cellules de cancer du poumon augmente la viabilité et la migration des cellules in vitro comparées à celles des cellules exprimant la forme sauvage de c-Cbl [
31]. Ce contrôle par les ligases E3 de protéines impliquées dans le développement tumoral suggère que ces enzymes pourraient être des cibles thérapeutiques dans certains types de cancers [
32]. L’ostéosarcome ostéo-condensant est une tumeur résultant de l’activité anormale de cellules tumorales dérivées des ostéoblastes, et la migration de ces cellules conduit au développement de métastases. Nos travaux ont montré que la surexpression de la forme sauvage de c-Cbl dans des lignées d’ostéosarcomes murines et humaines diminue la prolifération, la survie, l’invasion et la migration des cellules tumorales en culture. In vivo, la surexpression de c-Cbl dans un modèle murin d’ostéosarcome diminue la formation tumorale ainsi que le développement de métastases pulmonaires dans un modèle murin d'ostéosarcome. Nous avons montré que cet effet antitumoral de c-Cbl in vivo résulte d’une diminution de la prolifération cellulaire et d’une augmentation de l’apoptose des cellules tumorales, effets relayés par une diminution de l’expression des récepteurs PDGFRα, EGFR et FGFR2 [
33]. Ces données récentes montrent que le rôle antitumoral de c-Cbl passe par l’ubiquitination et la dégradation de ces récepteurs membranaires (Figure 3B). D’un point de vue thérapeutique, ceci suggère une possible stratégie ciblant c-Cbl dans les cancers associés à une activation des RTK. On ne dispose pas encore d’outils pharmacologiques permettant de favoriser spécifiquement l’activité ubiquitine ligase de c-Cbl. Une alternative stratégique potentielle serait de bloquer l’activité des déubiquitinases dont l’activité s’oppose à celle des ubiquitine ligases [
34]. Une troisième possibilité serait de cibler des protéines adaptatrices qui interagissent avec c-Cbl et entrent en compétition avec les RTK (CIN85 [Cbl-interacting protein 85], Abl [Abelson leukemia viral oncogene], Grb2 [growth factor receptor-bound protein 2], FRS2 [fibroblast growth factor receptor substrate 2], etc.). Ainsi, moduler l’ubiquitination et la dégradation des récepteurs impliqués dans le développement tumoral en ciblant c-Cbl pourrait être une stratégie thérapeutique intéressante pour réduire la tumorigenèse dans ces cancers. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Peyron
JF.
Les multiples rôles de l’ubiquitinylation des protéines . Med Sci (Paris).
2001; ; 17 : :1327.–1329. 2.
Schulman
BA
,
Harper
JW.
Ubiquitin-like protein activation by E1 enzymes: the apex for downstream signalling pathways . Nat Rev Mol Cell Biol.
2009; ; 10 : :319.–331. 3.
Ye
Y
,
Rape
M.
Building ubiquitin chains: E2 enzymes at work . Nat Rev Mol Cell Biol.
2009; ; 10 : :755.–764. 4.
Robinson
PA
,
Ardley
HC.
Ubiquitin-protein ligases-novel therapeutic targets?
Curr Protein Pept Sci.
2004; ; 5 : :163.–176. 5.
Langdon
WY
,
Hartley
JW
,
Klinken
SP
, et al.
v-cbl, an oncogene from a dual-recombinant murine retrovirus that induces early B-lineage lymphomas . Proc Natl Acad Sci USA.
1989; ; 86 : :1168.–1172. 6.
Dikic
I
,
Wakatsuki
S
,
Walters
KJ.
Ubiquitin-binding domains: from structures to functions . Nat Rev Mol Cell Biol.
2009; ; 10 : :659.–671. 7.
Thien
CB
,
Langdon
WY.
Cbl: many adaptations to regulate protein tyrosine kinases . Nat Rev Mol Cell Biol.
2001; ; 2 : :294.–307. 8.
Tsygankov
AY
,
Teckchandani
AM
,
Feshchenko
EA
,
Swaminathan
G.
Beyond the RING: CBL proteins as multivalent adapters. , Oncogene.
2001; ; 20 : :6382.–6402. 9.
Baron
R.
L’ostéoclaste et les mécanismes moléculaires de la résorption osseuse . Med Sci (Paris).
2001; ; 17 : :1260.–1269. 10.
Marie
P.
Différenciation, fonction et contrôle de l’ostéoblaste .Med Sci (Paris).
2001; ; 17 : :1252.–1259. 11.
Chiusaroli
R
Sanjay
A
,
Henriksen
K
, et al.
Deletion of the gene encoding c-Cbl alters the ability of osteoclasts to migrate, delaying resorption and ossification of cartilage during the development of long bones . Dev Biol.
2003; ; 261 : :537.–547. 12.
Horne
WC
,
Sanjay
A
,
Bruzzaniti
A
,
Baron
R.
The role(s) of Src kinase and Cbl proteins in the regulation of osteoclast differentiation and function . Immunol Rev.
2005; ; 208 : :106.–125. 13.
Sanjay
A
,
Miyazaki
T
,
Itzstein
C
, et al.
Identification and functional characterization of an Src homology domain 3 domain-binding site on Cbl . FEBS J.
2006; ; 273 : :5442.–5456. 14.
Adapala
NS
,
Barbe
MF
,
Langdon
WY
, et al.
The loss of Cbl-phosphatidylinositol 3-kinase interaction perturbs RANKL-mediated signaling, inhibiting bone resorption and promoting osteoclast survival . J Biol Chem.
2010; ; 285 : :36745.–36758. 15.
Nakajima
A
,
Sanjay
A
,
Chiusaroli
R
, et al.
Loss of Cbl-b increases osteoclast bone-resorbing activity and induces osteopenia . J Bone Miner Res.
2009; ; 24 : :1162.–1172. 16.
Purev
E
,
Neff
L
,
Horne
WC
,
Baron
R.
c-Cbl and Cbl-b act redundantly to protect osteoclasts from apoptosis and to displace HDAC6 from beta-tubulin, stabilizing microtubules and podosomes . Mol Biol Cell.
2009; ; 20 : :4021.–4030. 17.
Guénou
H
,
Kaabeche
K
,
Dufour
C
, et al.
Down-regulation of ubiquitin ligase Cbl induced by twist haploinsufficiency in Saethre-Chotzen syndrome results in increased PI3K/Akt signaling and osteoblast proliferation . Am J Pathol.
2006; ; 169 : :1303.–1311. 18.
Kaabeche
K
,
Lemonnier
J
,
Le Mée
S
, et al.
Cbl-mediated degradation of Lyn and Fyn induced by constitutive FGFR-2 activation supports osteoblast differentiation . J Biol Chem.
2004; ; 279 : :36259.–36267. 19.
Miraoui
H
,
Ringe
J
,
Häupl
T
,
Marie
PJ.
Increased EGF- and PDGFα- receptor signaling by mutant FGF-receptor 2 contributes to osteoblast dysfunction in Apert craniosynostosis . Hum Mol Genet.
2010; ; 19 : :1678.–1689. 20.
Miraoui
H
,
Marie
PJ.
Fibroblast growth factor receptor signaling crosstalk in skeletogenesis . Science Signaling.
2010; ; 3 : :re9.. 21.
Dufour
C
,
Guénou
H
,
Kaabeche
K
, et al.
FGFR2-Cbl interaction in lipid rafts triggers attenuation of PI3K/Akt signaling and osteoblast survival . Bone. , 2008; ; 42 : :1032.–1039. 22.
Kaabeche
K
,
Guénou
H
,
Bouvard
D
, et al.
Cbl-mediated ubiquitination of alpha5 integrin subunit mediates fibronectin-dependent osteoblast detachment and apoptosis induced by FGFR2 activation . J Cell Sci.
2005; ; 118 : :1223.–1232. 23.
Brennan
T
,
Adapala
NS
,
Barbe
MF
, et al.
Abrogation of Cbl-PI3K interaction increases bone formation and osteoblast proliferation . Calcif Tissue Int.
2011; ; 89 : :396.–410. 24.
Suzue
N
,
Nikawa
T
,
Onishi
Y
, et al.
Ubiquitin ligase Cbl-b downregulates bone formation through suppression of IGF-I signaling in osteoblasts during denervation . J Bone Miner Res.
2006; ; 21 : :722.–734. 25.
Sévère
N
,
Miraoui
H
,
Marie
PJ.
The Casitas B lineage lymphoma (Cbl) mutant G306E enhances osteogenic differentiation in human mesenchymal stromal cells in part by decreased Cbl-mediated platelet-derived growth factor receptor alpha and fibroblast growth factor receptor 2 ubiquitination . J Biol Chem.
2011; ; 286 : :24443.–24450. 26.
Kumar
EA
,
Charvet
CD
,
Lokesh
GL
,
Natarajan
A.
High-throughput fluorescence polarization assay to identify inhibitors of Cbl(TKB)-protein tyrosine kinase interactions . Anal Biochem.
2011; ; 411 : :254.–260. 27.
Kumar
EA
,
Yuan
Z
,
Palermo
NY
, et al.
Peptide truncation leads to a twist and an unusual increase in affinity for casitas B-lineage lymphoma tyrosine kinase binding domain . J Med Chem.
2012; ; 55 : :3583.–3587. 28.
Ciechanover
A.
The ubiquitin proteolytic system: from a vague idea, through basic mechanisms, and onto human diseases and drug targeting . Neurology.
2006; ; 66 : :S7.–S19. 29.
Naramura
M
,
Nadeau
S
,
Mohapatra
B
, et al.
Mutant Cbl proteins as oncogenic drivers in myeloproliferative disorders . Oncotarget.
2011; ; 2 : :245.–250. 30.
Kales
SC
,
Ryan
PE
,
Nau
MM
,
Lipkowitz
S.
Cbl and human myeloid neoplasms: the Cbl oncogene comes of age . Cancer Res.
2010; ; 70 : :4789.–4794. 31.
Tan
YH
,
Krishnaswamy
S
,
Nandi
S
, et al.
CBL is frequently altered in lung cancers: its relationship to mutations in MET, EGFR tyrosine kinases . PLoS One.
2010; ; 5 : :e8972.. 32.
Pray
TR
,
Parlati
F
,
Huang
J
, et al.
Cell cycle regulatory E3 ubiquitin ligases as anticancer targets . Drug Resist Updat.
2002; ; 5 : :249.–258. 33.
Sévère
N
,
Dieudonné
F-X
,
Marty
C
, et al.
Targeting the E3 ubiquitin ligase c-Cbl decreases osteosarcoma cell growth and survival and reduces tumorigenesis . J Bone Miner Res.
2012; ; 27 : :2108.–2117. 34.
Shi
D
,
Grossman
SR.
Ubiquitin becomes ubiquitous in cancer: emerging roles of ubiquitin ligases and deubiquitinases in tumorigenesis and as therapeutic targets . Cancer Biol Ther.
2010; ; 10 : :737.–747. 35.
Baldin
V
,
Coux
O.
L’étiquette de la mort . Med Sci (Paris).
2004; ; 20 : :1156.–1157. |