| |
| Med Sci (Paris). 2012 December; 28(12): 1047–1049. Published online 2012 December 21. doi: 10.1051/medsci/20122812009.Rôle crucial des protéines Polycomb d’origine maternelle dans le développement précoce de l’embryon de souris Juliette Salvaing,1,2 Eszter Posfai,3 Antoine H.F.M. Peters,3 and Nathalie Beaujean1,2* 1INRA, UMR 1198 biologie du développement et reproduction, 78350Jouy-en-Josas, France 2École nationale vétérinaire d’Alfort (ENVA), 94700Maisons Alfort, France 3Friedrich Miescher institute for biomedical research (FMI), 4058Basel, Suisse MeSH keywords: Animaux, Chromatine, génétique, physiologie, Malformations, embryologie, métabolisme, Cytoplasme, Embryon de mammifère, Développement embryonnaire, Souris, Modèles biologiques, Ovogenèse, Protéines du groupe Polycomb, ARN messager stocké |
Lors de la fécondation chez les mammifères, la fusion des gamètes paternel et maternel engendre un embryon 1-cellule dans lequel les génomes parentaux forment deux pronoyaux distincts. Ces génomes sont initialement transcriptionnellement inactifs, et le développement précoce est contrôlé par les protéines et les ARN présents dans le cytoplasme ovocytaire. Les expériences de clonage par transfert de noyau ont permis de mettre en évidence la capacité du cytoplasme ovocytaire à reprogrammer les noyaux somatiques afin de reconstituer un embryon capable de se développer à terme. Cependant, l’efficacité de cette reprogrammation apparaît à la fois relativement faible et très variable : les noyaux des cellules germinales ou des cellules embryonnaires souches sont en effet bien plus faciles à reprogrammer que ceux des cellules somatiques [
1], d’où l’idée que la chromatine des cellules germinales est plus compétente pour la reprogrammation, qu’elle est en quelque sorte « préparée » pour le développement embryonnaire. Dans une étude récente que nous avons réalisée avec E. Posfai, A.H.F.M. Peters et leurs collègues, nous nous sommes intéressés au rôle des protéines du groupe Polycomb (PcG) dans la transmission des états chromatiniens de l’ovocyte vers l’embryon [
2]. Les protéines PcG sont impliquées dans la répression de nombreux gènes, notamment des gènes de développement. Elles agissent sous forme de complexes appelés polycomb-group repressive complex 1 (PRC1) et PRC2 capables, respectivement, de mono-ubiquitiner l’histone H2A (H2AK119ub1) et de triméthyler l’histone H3 (H3K27me3) [
3]. Il est important de noter que, dans les cellules de la masse cellulaire interne (ICM) du blastocyste, H3K27me3 est localisée sur des gènes de développement, qui sont alors réprimés [
4]. Or, cette marque est déjà présente aux mêmes locus dans les spermatozoïdes [
5], ce qui suggère que la répression des gènes cibles pourrait être préétablie dans les cellules germinales et transmise ensuite à l’embryon. |
Rôle du complexe PRC1 dans l’ovogenèse et le développement embryonnaire précoce Les protéines Ring1 (ring finger protein 1, Ring1a) et Rnf2 (Ring1b) sont deux paralogues appartenant aux complexes PRC1. Dans l’ovocyte, la composante maternelle de Rnf2 ne semble en effet pas jouer de rôle [
6], suggérant une redondance de fonction entre Ring1 et Rnf2, comme c’est le cas dans les cellules souches embryonnaires [
7]. En revanche, dans l’embryon, la déficience zygotique de Rnf2 est létale au moment de la gastrulation [
8], alors que Ring1 n’apparaît pas essentielle au développement embryonnaire [
9]. Nous avons dans un premier temps confirmé que les gènes Ring1 et Rnf2 sont exprimés pendant l’ovogenèse. En revanche, seul Rnf2 est exprimé dans l’embryon préimplantatoire. Nous avons ensuite réalisé l’inactivation simultanée de Ring1 et Rnf2 dans la lignée germinale femelle, ce qui conduit à une perte de localisation du complexe PRC1 sur la chromatine, alors que la présence d’un seul des deux gènes est suffisante pour sa localisation correcte. Les ovocytes double-mutants ainsi obtenus semblent se développer normalement jusqu’au stade de vésicule germinative, mais la suite de la maturation ovocytaire est retardée, avec notamment des défauts d’alignement des chromosomes sur les plaques métaphasiques lors des deux divisions méiotiques. Les embryons obtenus à partir de ces ovocytes ne se développent pas au-delà du stade 2-cellules, quel que soit le génotype des gamètes mâles utilisés, ce qui souligne l’origine maternelle de cet arrêt. Dans les embryons, la formation des pronoyaux maternel et paternel est retardée, ainsi que la première division mitotique. Un délai dans le début de la phase S est également détecté au cours du premier cycle, et au stade 2-cellules cette phase ne s’achève jamais ; le point de contrôle de la phase S est activé, comme nous l’avons montré par la détection des formes phosphorylées des sérine/thréonine kinases Chk1 et Chk2. L’activation majeure du génome embryonnaire (EGA) a lieu à la fin du stade 2-cellules chez la souris. Afin de voir si elle se déroule normalement, nous avons examiné les ARN néotranscrits dans les embryons double-mutants. Nous avons ainsi mis en évidence une perturbation générale de l’activation du génome embryonnaire, avec une forte réduction du nombre de gènes transcrits de novo. Afin d’essayer de corriger ces défauts, nous avons injecté l’ARN de Rnf2 dans les embryons double-mutants au stade 1-cellule. Si cette injection permet de restaurer la localisation du complexe PRC1, elle ne restaure pas le développement embryonnaire. Il est possible que la reconstitution du complexe PRC1 sur la chromatine soit trop tardive, mais il semble plus probable que la présence de Ring1/Rnf2 soit requise pendant la croissance ovocytaire pour assurer le développement embryonnaire précoce. |
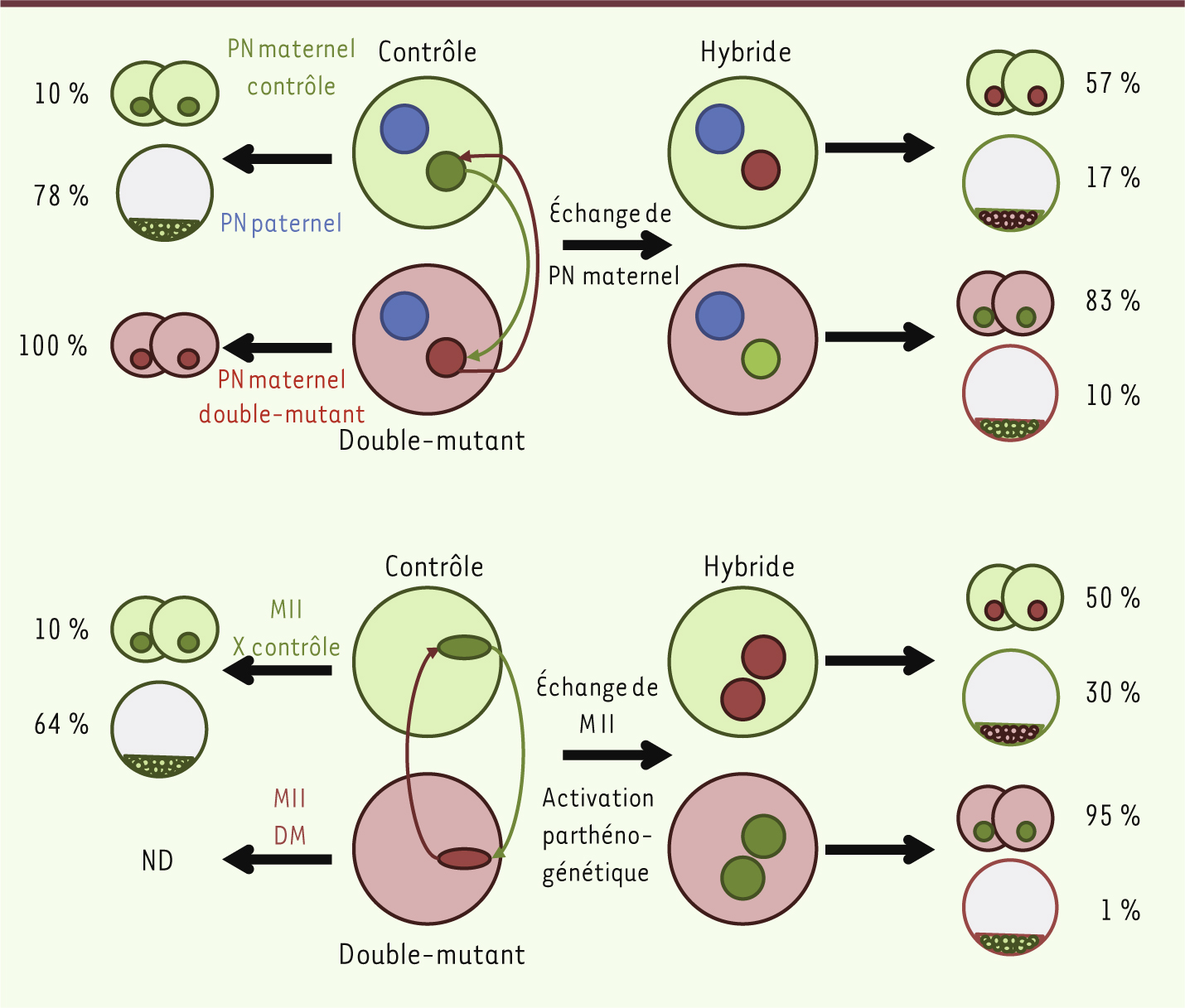
Origine cytoplasmique et chromatinienne des défauts de développement Nous avons ensuite cherché à déterminer si les défauts observés étaient dus à des perturbations de l’expression des transcrits maternels nécessaires au développement (origine cytoplasmique) ou à des défauts de l’organisation chromatinienne affectant la compétence de l’ovocyte pour le développement (origine chromatinienne). Nous avons dans un premier temps examiné le transcriptome total dans les ovocytes et les embryons double-mutants. De nombreux transcrits apparaissent dérégulés aussi bien dans l’ovocyte que dans l’embryon : les transcrits surexprimés dans l’embryon proviennent pour la plupart de l’ovocyte, tandis que les transcrits sous-exprimés résultent pour la plus grande partie de l’échec du processus d’activation majeure du génome embryonnaire. Les gènes de développement sont fortement représentés parmi les gènes surexprimés : nombre de ces gènes sont des cibles connues des protéines PcG dans les cellules souches embryonnaires ou dans les cellules somatiques [
10]. L’étude plus détaillée de certains gènes de différenciation (Eomes [eomesodermin], Pax6 [paired box gene 6] et Gata4) et des protéines correspondantes nous a permis de mettre en évidence un délai entre la surexpression du transcrit (déjà présente dans l’ovocyte) et celle de la protéine (constatée uniquement dans l’embryon), ce qui implique l’existence d’un mécanisme de régulation traductionnelle dans l’ovocyte et pourrait expliquer que les embryons se développent jusqu’au stade 2-cellules. Dans un second temps, et afin de déterminer si la composante chromatinienne est également importante, nous avons réalisé des échanges de pronoyaux maternels entre des embryons issus d’ovocytes contrôles et double-mutants (figure 1A). Le développement de ces embryons hybrides est fortement affecté, avec 10 à 15 % des embryons se développant jusqu’au stade blastocyste et une forte proportion s’arrêtant aux stades 1- ou 2-cellules, ce qui suggère une importance égale des contributions cytoplasmique et chromatinienne. Cependant, l’effet observé aurait pu être expliqué par des défauts de formation des pronoyaux maternels dans les embryons double-mutants. Pour éliminer cette possibilité, nous avons procédé à des échanges de métaphases entre ovocytes bloqués en métaphase II, suivis par une activation parthénogénétique (Figure 1B). La presque totalité (95 %) des embryons avec un cytoplasme double-mutant et une métaphase d’ovocyte contrôle s’arrêtent au stade 1- ou 2-cellules, soulignant à nouveau l’importance de la contribution cytoplasmique. De même, dans la situation inverse, métaphase double-mutante et cytoplasme contrôle, près de 50 % des embryons ne dépassent pas le stade 2-cellules, ce qui démontre que la configuration chromatinienne est également très importante.
 | Figure 1.
Développement d’embryons hybrides obtenus soit par échange du pronoyau maternel (A), soit par échange de métaphases II et activation parthénogénétique (B). Les pourcentages d’embryons dont le développement est stoppé au stade 2-cellules ou plus tôt, et d’embryons qui atteignent le stade blastocyste, sont indiqués pour chaque situation expérimentale. ND : non déterminé. PN : pronoyau ; MII : métaphase II. |
En résumé, nos résultats montrent que les protéines Ring1 et Rnf2 ont une importance capitale pendant l’ovogenèse, tant pour la régulation transcriptionnelle des ARN maternels que pour la structuration de la chromatine maternelle. Leur absence entraîne un arrêt de développement au stade 2-cellules, caractérisé notamment par l’activation du point de contrôle de phase S et par de graves défauts dans la mise en route du génome embryonnaire. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Beaujean
N
,
Martin
C
,
Debey
P
, et al.
Reprogrammation et épigenèse . Med Sci (Paris).
2005; ; 21 : :412.–411. 2.
Posfai
E
,
Kunzmann
R
,
Brochard
V
, et al.
Polycomb function during oogenesis is required for mouse embryonic development . Genes Dev.
2012; ; 26 : :920.–932. 3.
Simon
JA
,
Kingston
RE.
Mechanisms of polycomb gene silencing: knowns and unknowns . Nat Rev Mol Cell Biol.
2009; ; 10 : :697.–708. 4.
Dahl
JA
,
Reiner
AH
,
Klungland
A
, et al.
Histone H3 lysine 27 methylation asymmetry on developmentally-regulated promoters distinguish the first two lineages in mouse preimplantation embryos . PLoS One.
2010; ; 5 : :11.. 5.
Brykczynska
U
,
Hisano
M
,
Erkek
S
, et al.
Repressive and active histone methylation mark distinct promoters in human and mouse spermatozoa . Nat Struct Mol Biol.
2010; ; 17 : :679.–687. 6.
Terranova
R
,
Yokobayashi
S
,
Stadler
MB
, et al.
Polycomb group proteins Ezh2 and Rnf2 direct genomic contraction and imprinted repression in early mouse embryos . Dev Cell.
2008; ; 15 : :668.–679. 7.
Endoh
M
,
Endo
TA
,
Endoh
T
, et al.
Polycomb group proteins Ring1A/B are functionally linked to the core transcriptional regulatory circuitry to maintain ES cell identity . Development.
2008; ; 135 : :1513.–1524. 8.
Valk-Lingbeek
ME
,
Bruggeman
SWM
,
Van Lohuizen
M.
Stem cells and cancer; the polycomb connection . Cell.
2004; ; 118 : :409.–418. 9.
Del Mar Lorente
M
,
Marcos-Gutiérrez
C
,
Pérez
C
, et al.
Loss- and gain-of-function mutations show a polycomb group function for Ring1A in mice . Development.
2000; ; 127 : :5093.–5100. 10.
Boyer
LA
,
Plath
K
,
Zeitlinger
J
, et al.
Polycomb complexes repress developmental regulators in murine embryonic stem cells . Nature.
2006; ; 441 : :349.–353. |