| |
| Med Sci (Paris). 2013 April; 29(4): 405–410. Published online 2013 April 26. doi: 10.1051/medsci/2013294014.La reprogrammation, un jeu de hasard ? Laurent David1,2* and John De Vos3 1Inserm UMR 1064, faculté de médecine, université de Nantes, 44093Nantes Cedex 1, France 2Plate-forme iPS Nantes, SFR F. Bonamy, faculté de médecine, université de Nantes, 44007Nantes Cedex 1, France 3Inserm U1040, hôpital Saint-Eloi, 80, avenue Augustin Fliche, 34295Montpellier Cedex 5, France |
L’énigme de la reprogrammation : stochastique, déterministe ou mixte ?
Vignette (Photo © Inserm - Lionel Simonneau). La possibilité qu’ont les cellules pluripotentes de se différencier en tout type cellulaire en font un outil de choix pour la médecine régénérative et le criblage de nouveaux médicaments [
1]. La possibilité de générer des cellules pluripotentes induites (iPSC, induced pluripotent stem cells) à partir de cellules somatiques vient ainsi d’être reconnue comme une contribution majeure à la recherche médicale et a été récompensée par le prix Nobel de Médecine 2012 [
2]. Si nos connaissances des mécanismes moléculaires contrôlant la reprogrammation ont grandement progressé depuis la découverte des iPSC, plusieurs zones d’ombre demeurent [
3]. Une des plus grandes énigmes est sans doute la très faible fréquence avec laquelle une cellule peut être reprogrammée avec succès, puisque moins de une cellule sur 2 000 est reprogrammée lorsqu’on utilise le protocole canonique publié initialement par K. Takahashi et S. Yamanaka, fondé sur l’expression forcée des quatre facteurs Myc, Klf4 (Krüppel-like factor 4), Sox2 ([sex determining region Y]-box2 et Oct4 [
4]. L’hypothèse selon laquelle seules de rares cellules, immatures et peut-être déjà très proches des cellules souches pluripotentes, peuvent se reprogrammer a été rapidement récusée. Pour le vérifier, les auteurs ont utilisé des cellules différenciées qui exprimaient des marques génétiques caractéristiques : des hépatocytes dans lesquels le promoteur de l’albumine est activé, ou des lymphocytes B dans lesquels le locus des immunoglobulines est recombiné [
5,
6]. Plus récemment, le groupe de R. Jaenisch a tiré parti d’un système de reprogrammation « secondaire » (Figure 1) dans lequel l’expression des quatre transgènes Myc, Klf4 (Krüppel-like factor 4), Sox2 (sex determining region Y)-box2 et Oct4 - que nous désignerons par OKMS - est sous le contrôle d’un promoteur inductible par la doxycycline (Dox), pour montrer que quasiment toutes les cellules finissaient par être reprogrammées si l’on poursuit l’expérience suffisamment longtemps (plus de quatre mois !) [
7]. La modélisation mathématique de ces observations suggérait que la reprogrammation était un processus stochastique, c’est-à-dire que l’expression des quatre transgènes n’induisait pas le même effet dans chaque cellule d’une population : certaines cellules se reprogrammaient rapidement, d’autres plus tardivement, certaines jamais. Cette vue s’opposait à un modèle déterministe dans lequel la durée de la reprogrammation aurait été fixe et toujours la même. Mais la faible fréquence de la reprogrammation et son caractère stochastique (les cellules ne se reprogramment pas toutes simultanément) rendaient l’analyse moléculaire de ce processus particulièrement délicate. Par l’utilisation de techniques d’analyse de cellules isolées ou de populations cellulaires triées, plusieurs groupes ont montré récemment que la reprogrammation comportait également une part importante de déterminisme, c’est-à-dire d’événements séquentiels reproductibles d’une reprogrammation à une autre.
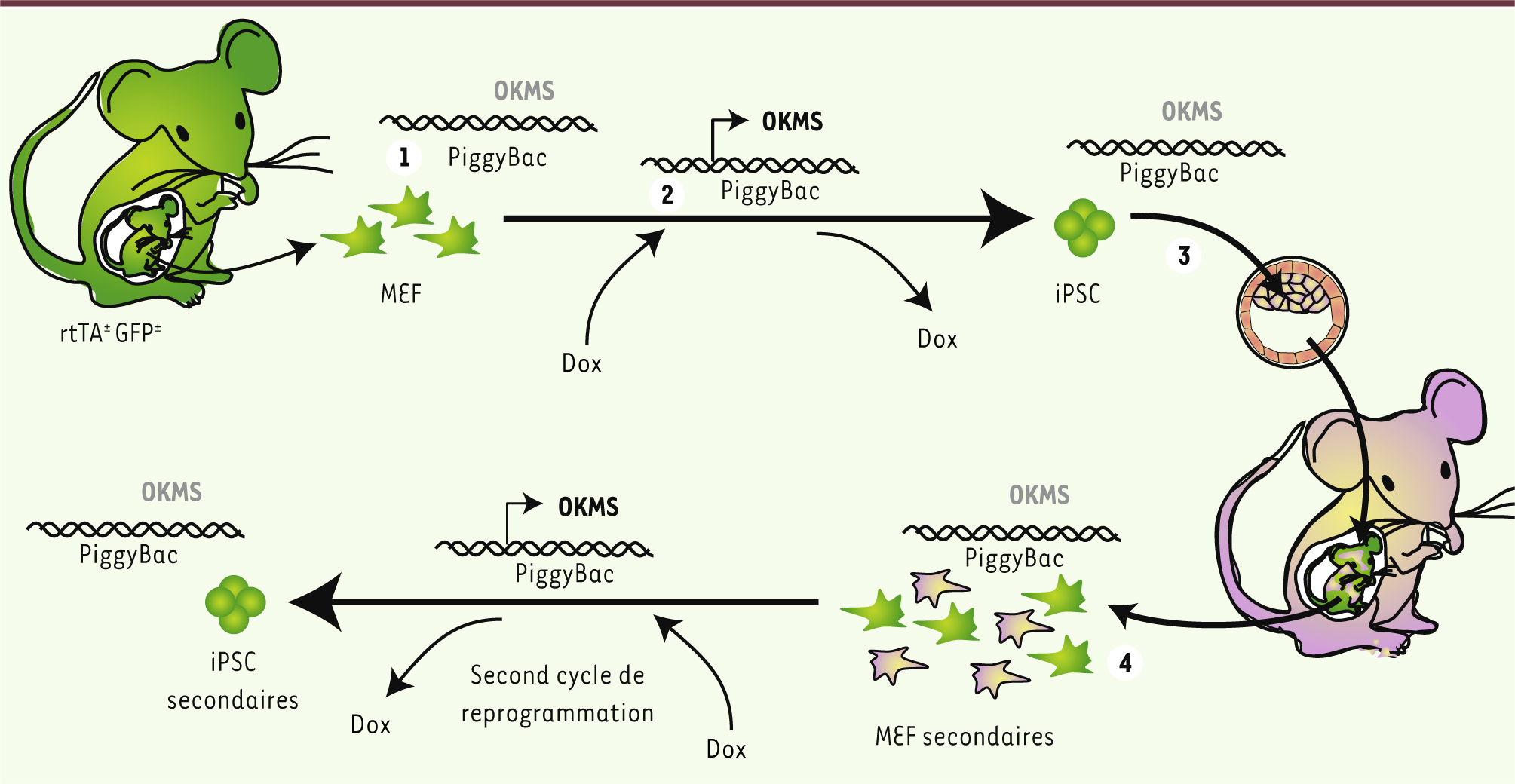
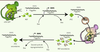 | Figure 1.
Système de reprogrammation secondaire. Exemple du système piggyBac. (1) Des cellules MEF exprimant la rtTA et la GFP (rtTA+/- GFP+/-) sont reprogrammés par un système piggyBac qui s’intègre dans le génome et (2) qui exprime les séquences codant pour quatre facteurs Oct4, Klf4, Myc et Sox2 (OKMS) sous le contrôle de la doxycycline (Dox). (3) Les iPS Dox indépendantes servent à générer des souris chimères par injection dans un blastocyste de souris. (4) Les MEF obtenus de ces souris chimères contiennent des cellules génétiquement modifiées qui expriment les quatre facteurs OKMS en présence de Dox. Elles offrent 2 avantages : il y a un très grand nombre de cellules reprogrammables et la reprogrammation de ces cellules est très efficace. MEF : mouse embryonic fibroblasts ; OKMS : Oct 4, Klf4, Myc, Sox2 ; rtTA : reverse tetracycline transcriptional activator ; GFP : green fluorescent protein ; iPSC : induced pluripotent stem cells. |
Dès 2010, plusieurs travaux ont défendu l’hypothèse du caractère déterministe de la reprogrammation cellulaire. Celle-ci résulterait d’une succession d’événements définis. Ce fut le cas notamment d’expériences de suivi en temps réel par vidéomicroscopie de cellules en cours de reprogrammation, ainsi que de notre travail au sein du groupe de J. Wrana [
8]. Dans un modèle dans lequel la reprogrammation était massive et rapide, 10 à 20 % des cellules exprimant les transgènes se reprogrammaient en iPSC en trois semaines, nous avions pu distinguer trois phases distinctes lors de la reprogrammation de fibroblastes en iPSC : l’initiation, la maturation et la stabilisation. L’initiation comprend une transition mésenchymo-épithéliale qui se traduit par un changement phénotypique et une modification profonde de la signature transcriptomique [
9,
10]. Ces résultats nous ont permis d’améliorer la reprogrammation des cellules humaines, par exemple en utilisant des microARN de la famille miR-200 pour stimuler cette transition mésenchymo-épithéliale et donc augmenter l’efficacité de reprogrammation [
11]. La phase de maturation est caractérisée par l’expression de quelques marqueurs de pluripotence (Nanog, Sall4 [sal-like 4 (Drosophila)], Oct4 endogène), et elle est suivie d’une phase de stabilisation au cours de laquelle sont induits un certain nombre de marqueurs de la pluripotence qui ne s’exprimaient pas encore, tels que Sox2 endogène, Pecam (plateled endothelial cell adhesion molecule), DppaIV (developmental pluripotency associated 4) [10]. Malgré ces avancées, de nombreuses questions restaient en suspens concernant notamment la nature des événements tardifs lors de la reprogrammation, le rôle des transgènes au cours des différentes phases de la reprogrammation ou encore la validation de la signature transcriptomique obtenue sur des populations de cellules analysées individuellement. |
Événements séquentiels et hiérarchiques caractérisant la reprogrammation des fibroblastes Fin 2012, plusieurs groupes ont reconsidéré la question des modifications transcriptionnelles successives au cours de la reprogrammation de cellules différenciées en iPSC par des approches utilisant des cellules triées ou isolées et/ou des systèmes de reprogrammation secondaire (Figure 2). Il est intéressant de noter qu’alors que les facteurs de reprogrammation utilisés par les trois équipes étaient les mêmes (OKMS), chaque équipe a utilisé un système secondaire qui lui était propre [
12–
14] (Figure 2).
-
Le groupe de R. Jaenisch (Figure 2B) a utilisé une approche clonale (reprogrammation de cellules individuellessuivies au cours du temps) couplée à une analyse par PCR (polymerase chain reaction) de cellules uniques (tri cellulaire et PCR digitale), analysant l’expression de 48 gènes. Cette nouvelle approche a confirmé l’acquisition séquentielle de marqueurs précédemment identifiés par notre analyse temporelle d’une population de cellules en cours de reprogrammation, et a proposé de diviser le processus de reprogrammation en une première partie stochastique et une deuxième partie déterministe [
15].
-
Parallèlement, le groupe de K. Hochedlinger (Figure 2C) a utilisé des marqueurs apparaissant successivement au cours de la reprogrammation (d’abord Thy1, puis SSEA1 [stage-specific embryonic antigen 1], puis Oct4) pour trier les fractions cellulaires engagées avec succès vers le stade iPSC. Ce tri cellulaire diminue le bruit de fond de l’analyse transcriptomique et montre qu’à chaque acquisition d’un des marqueurs choisis pour le tri cellulaire correspond une signature transcriptomique distincte. Là aussi, ces résultats mettent en évidence une signature équivalente à celle que nous avions publiée auparavant. K. Hochedlinger décrit notamment deux phases au cours desquelles se produisent des modifications majeures du transcriptome : la première sous le contrôle principal de Myc et Klf4 (phase d’initiation), et la seconde sous le contrôle de Oct4, Sox2 et Klf4 (équivalente à la phase de maturation). Cette dernière prépare l’entrée des cellules dans la phase de stabilisation, qui marque l’acquisition par les cellules du caractère pluripotent [
16]. Ce groupe a couplé la signature transcriptomique de reprogrammation avec les données de ChIP-Seq (chromatin immunoprecipitation with parallel DNA sequencing) obtenues précédemment par H. Ng dans des cellules souches embryonnaires et avec l’analyse de la contribution relative d’OKMS lors de l’initiation de la reprogrammation dans des fibroblastes murins réalisée par le groupe de K. Plath [
17,
18]. La conclusion de cette analyse est que les changements profonds de l’épigénome sont différemment répartis au cours du temps, les modifications des histones étant précoces tandis que les modifications de la méthylation de l’ADN sont tardives. Ces observations suggèrent que le changement de profil de méthylation de l’ADN est un des phénomènes terminaux qui permettent l’acquisition de la pluripotence par les cellules en cours de reprogrammation. De manière intéressante, les cellules utilisées par K. Hochedlinger éteignent les transgènes utilisés pour forcer l’expression initiale de OKMS de manière spontanée, peu après l’expression d’Oct4 endogène, ce qui entraîne un bouleversement massif du transcriptome.
-
Notre système (Figure 2A) nous permet de contrôler l’extinction des transgènes. Ceci a pour conséquence de nous donner une meilleure résolution des événements de reprogrammation, et montre que les cellules doivent acquérir un programme particulier pour survivre à l’extinction des transgènes [
19]. Les données de K. Hochedlinger suggèrent qu’il s’agit de modifications du profil de méthylation du génome, spécifiquement pour une partie des facteurs régulant la pluripotence (marqueurs de la phase de stabilisation).
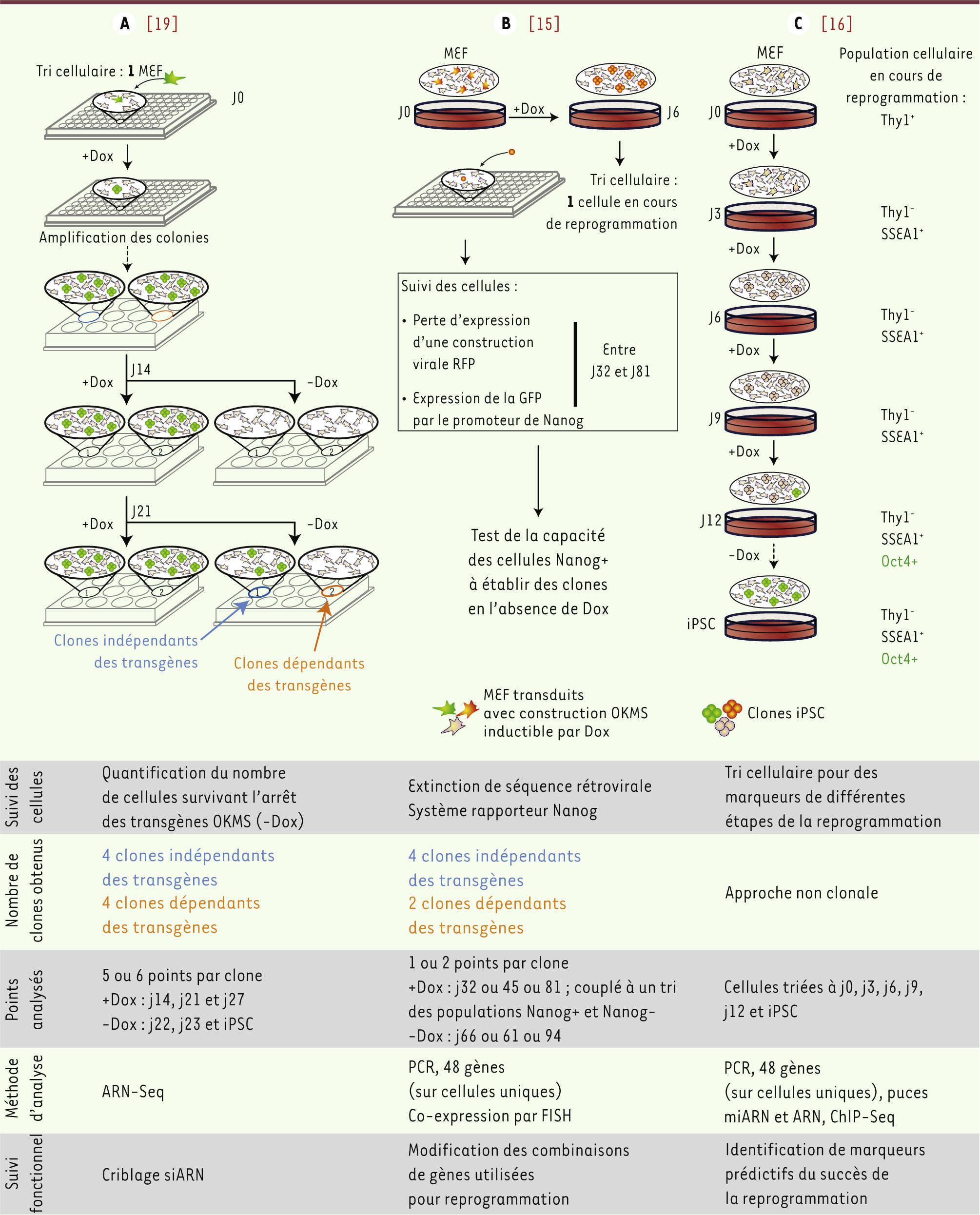
 | Figure 2.
Résumé des stratégies expérimentales utilisées. Les stratégies utilisées à partir de MEF secondaires par les 3 équipes [ 15, 16, 19] sont décrites. L’originalité de ces études est que l’expérience a été conduite de manière clonale et/ou que l’analyse a été réalisée sur cellules uniques. Thy1 - : cellules n’exprimant pas Thy1, SSEA1 + : cellules ayant une modification glucidique au niveau de leur membrane reconnue comme l’antigène SSEA1, Nanog + ou Oct4 + : cellules exprimant Nanog ou Oct4. |
Toutes ces données nous offrent maintenant une vision assez précise de la séquence d’événements qui s’enchaînent lors de la reprogrammation de fibroblastes murins en iPSC (Figure 3). Une partie de ces événements ont été validés récemment lors de la reprogrammation de cellules humaines, montrant une fois de plus l’utilité de systèmes murins pour faire avancer les connaissances des mécanismes de reprogrammation [
20].
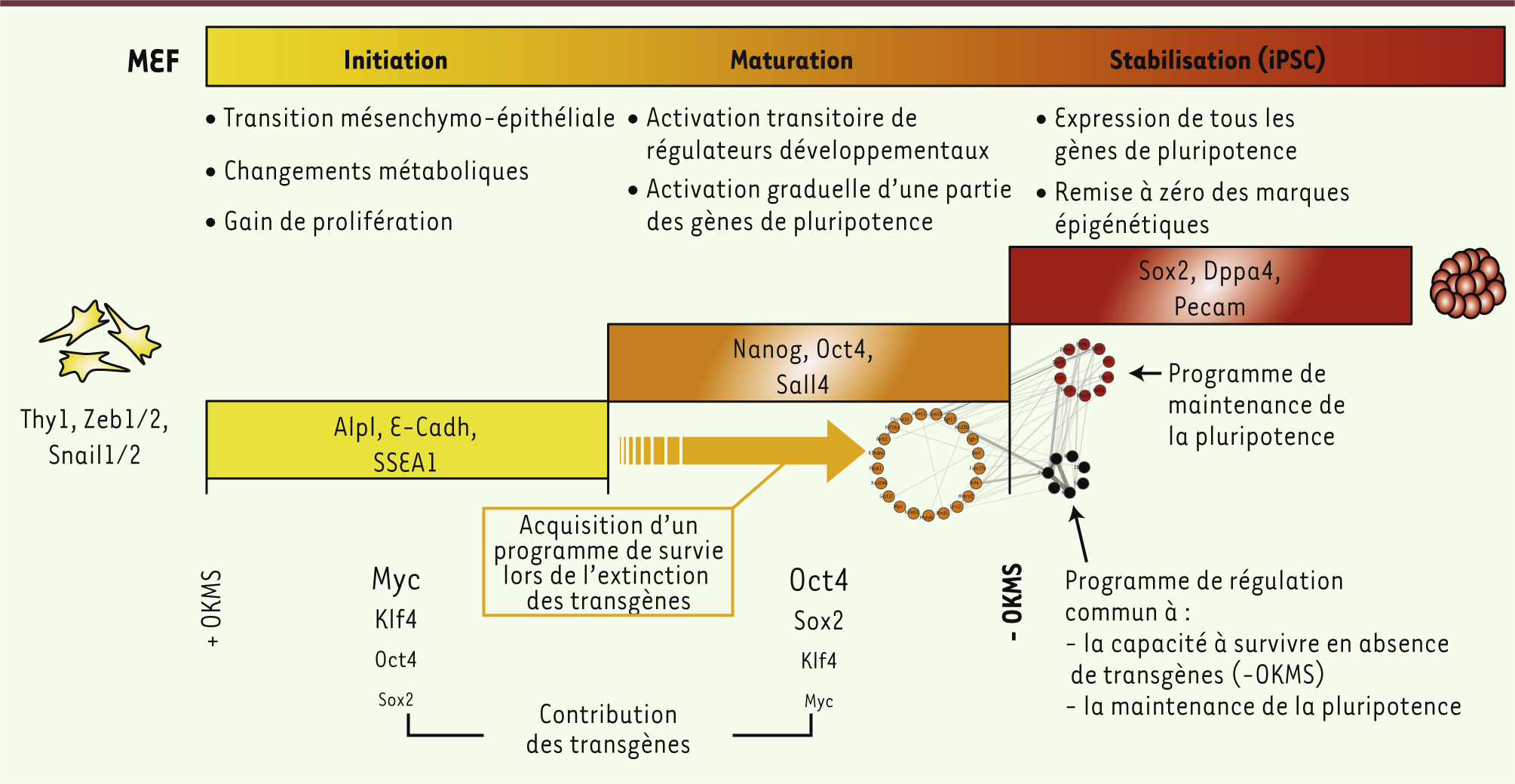
 | Figure 3.
Événements séquentiels rythmant la reprogrammation et contribution des différents transgènes. La phase d’initiation se caractérise par une transition mésenchymo-épithéliale (MET) sans laquelle les fibroblastes sont incapables d’être reprogrammés. Durant la MET, les marqueurs mésenchymateux (par exemple Snail) sont perdus au profit de marqueurs épithéliaux (par exemple la E-cadhérine). Après la phase d’initiation, les marqueurs de pluripotence sont exprimés (Nanog, Oct4 endogène, Sall4, etc.), définissant la phase de maturation. À partir de ce moment, les cellules sont dépendantes de l’expression des transgènes, et doivent acquérir un programme de régulation spécifique pour survivre à l’extinction de ces derniers. Une fois les transgènes éteints, les cellules entreront dans la phase de stabilisation. La taille des lettres reflète l’importance respective des facteurs. MEF : mouse embryonic fibroblasts ; E-Cadh : E-cadhérine. |
|
Les transgènes inhibent la progression vers la phase de stabilisation Les études de K. Hochedlinger et R. Jaenisch ont confirmé l’existence d’événements tardifs de reprogrammation dans les cellules qui vont devenir des iPSC. Nous nous sommes intéressés à la régulation de ces événements tardifs [19]. Notre première découverte a été que l’expression des transgènes inhibe le passage de la phase de maturation à la phase de stabilisation. Néanmoins, nous avons observé que les cellules devaient acquérir des mécanismes leur permettant de survivre en l’absence des transgènes, un processus qui imprime une composante temporelle forte. Ceci signifie que les cellules en cours de reprogrammation ont besoin de plus de 10 jours pour acquérir la capacité de s’autorenouveler en l’absence de la surexpression des facteurs OKMS exogènes. Nous nous sommes donc orientés vers une approche clonale. Le fait d’utiliser des cellules uniques lors des études de reprogrammation d’une population cellulaire permet d’analyser des clones représentatifs des différentes sous-populations de cellules. Ainsi, nous avons obtenu deux types de clones : certains avaient un fort potentiel de survie après extinction des transgènes, d’autres étaient composés presque exclusivement de cellules dépendantes des transgènes pour leur survie. Il faut noter que - à des temps précoces (milieu de la phase de maturation - la survie de tous les clones était dépendante de l’expression des facteurs exogènes - donc de l’expression des transgènes -, malgré une expression déjà marquée de Nanog et Oct4 endogènes, ou encore de Sall4. En analysant les profils d’expression génique en fonction du phénotype des clones, nous avons identifié une signature correspondant à la capacité des cellules à survivre à l’extinction des transgènes. Nous avons validé cette signature d’un point de vue fonctionnel par un criblage de siARN correspondant à une librairie composée d’ARN présents dans la signature, mais aussi d’autres connus pour réguler la pluripotence. Ce criblage a été effectué à deux étapes de la reprogrammation : dans des cellules transitant de la phase de maturation vers la phase de stabilisation, et dans des cellules déjà entrées dans la phase de stabilisation (que l’on peut qualifier d’iPSC). La comparaison des résultats de ces deux criblages a montré que les cellules abordant la phase de stabilisation étaient dépendantes d’un réseau moléculaire de régulation propre, distinct du réseau régulant la stabilisation de la pluripotence (Figure 3). Ceci montre que les cellules en cours de reprogrammation doivent acquérir un programme spécifique qui leur permet de survivre à l’extinction des transgènes afin de progresser vers le statut d’iPSC. Ceci est cohérent avec l’acquisition séquentielle de la pluripotence et l’observation de cellules qui restent « bloquées » à différentes étapes sans parvenir au stade d’iPS. |
Vers une reprogrammation plus rapide et des iPS plus sûres ? La hiérarchie des marqueurs de pluripotence qui sont acquis séquentiellement lors de la reprogrammation commence donc à être établie, mais beaucoup reste à faire. Tout d’abord, ces résultats, qui ont été obtenus chez la souris, devront maintenant être validés dans les cellules humaines. Une autre question importante est celle de la régulation du passage d’une étape à une autre. Pourquoi les marqueurs de pluripotence sont-ils activés en deux phases distinctes ? Quel(s) transgène(s) inhibe(nt) la transition de la phase de maturation à la phase de stabilisation ? Pour répondre à ces questions, il sera essentiel d’approfondir les observations effectuées sur cellules uniques. Cela permettra de comprendre les étapes limitant la reprogrammation par des facteurs définis. Cela permettra également d’expliquer pourquoi cette reprogrammation semble au moins partiellement stochastique, alors que les autres méthodes de reprogrammation (transfert nucléaire et fusion cellulaire) sont déterministes. Peut-être la force motrice de reprogrammation est-elle moindre dans le cas d’une reprogrammation par des facteurs définis - OKMS ici - que lorsque tous les déterminants de la pluripotence sont présents, ce qui est le cas lors d’un transfert nucléaire ou d’une fusion cellulaire. Cette perte de force motrice entraîne un ralentissement de la reprogrammation qui permet à plusieurs événements « parasites » de s’exprimer : apparition de cellules intermédiaires (cellules qui n’ont effectué qu’une partie de la reprogrammation) et de cellules qui s’apparentent à des progéniteurs de voies de différenciation spécifiques (pouvant n’être que des cellules partiellement reprogrammées et qui ont commencé à se différencier de manière incontrôlée). Un des enjeux principaux dans le domaine des iPSC reste le perfectionnement des techniques de reprogrammation pour obtenir des iPSC normales sur le plan génétique et exprimant les attributs chromatiniens et transcriptomiques complets d’un programme pluripotent. Les étapes qui apparaissent comme stochastiques, car peu fréquentes, incarnent des goulots d’étranglement de la reprogrammation et sont responsables des ralentissements, échecs et anomalies liés à la reprogrammation. Ceci revient à dire que si nous modifions le « cocktail » (OKMS) de reprogrammation de S. Yamanaka, en particulier en le complétant par des facteurs clefs des étapes intermédiaires de la reprogrammation, la reprogrammation pourrait être plus rapide et plus efficace. Ceci a été observé par le groupe de K. Hochedlinger, qui a utilisé ERas, un marqueur de la phase de stabilisation, pour accélérer la reprogrammation. Le groupe de R. Jaenisch, quant à lui, a utilisé Esrrb (estrogen related receptor, beta), un marqueur de la phase de maturation exprimé plus fortement dans les cellules ayant la capacité de survivre à l’extinction des transgènes. Ce groupe a montré qu’Esrrb peut remplacer de nombreux facteurs, en accord avec des publications récentes qui le placent parmi les facteurs les plus importants régulant la pluripotence [
21,
22]. De plus, ces facteurs, ainsi que ceux que nous avons identifiés comme étant fonctionnellement importants pour la reprogrammation, seront d’excellents marqueurs prédictifs de la reprogrammation et permettront de détecter plus précocement et avec plus de fiabilité les cellules qui vont être correctement reprogrammée, facilitant ainsi le travail des chercheurs. Enfin, comme la fréquence des anomalies épigénétiques augmente avec l’utilisation de protocoles de reprogrammation « imparfaits » (comme cela a été démontré pour les anomalies épigénétiques [
23]), il est très probable qu’en améliorant l’efficacité de la reprogrammation, on limitera l’incidence des aberrations génomiques et épigénomiques parfois observées dans les iPSC. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Bai
Q
,
Desprat
R
,
Klein
B
, et al.
Embryonic stem cells or induced pluripotent stem cells? A DNA integrity perspective . Curr Gene Ther.
2013 ; Jan;
:7.. [Epub ahead of print].
2.
Vogel
G
,
Normile
D
. Nobel Prize in physiology or medicine. Reprogrammed cells earn biologists top honor . Science.
2012; ; 338 : :178.–179. 3.
David
L
,
Samavarchi-Tehrani
P
,
Golipour
A
,
Wrana
JL
. Looking into the black box: insights into the mechanisms of somatic cell reprogramming . Genes.
2011; ; 2 : :81.–106. 4.
Takahashi
K
,
Yamanaka
S
. Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors . Cell.
2006; ; 126 : :663.–676. 5.
Aoi
T
,
Yae
K
,
Nakagawa
M
, et al.
Generation of pluripotent stem cells from adult mouse liver and stomach cells . Science.
2008; ; 321 : :699.–702. 6.
Hanna
J
,
Markoulaki
S
,
Schorderet
P
, et al.
Direct reprogramming of terminally differentiated mature B lymphocytes to pluripotency . Cell.
2008; ; 133 : :250.–264. 7.
Hanna
J
,
Saha
K
,
Pando
B
, et al.
Direct cell reprogramming is a stochastic process amenable to acceleration . Nature.
2009; ; 462 : :595.–601. 8.
David
L
. La transition mésenchymato-épithéliale, passage obligatoire pour la reprogrammation de fibroblastes en iPSC . Med Sci(Paris).
2010; ; 26 : :1030.–1032. 9.
Smith
ZD
,
Nachman
I
,
Regev
A
,
Meissner
A
. Dynamic single-cell imaging of direct reprogramming reveals an early specifying event . Nat Biotech.
2010; ; 28 : :521.–526. 10.
Samavarchi-Tehrani
P
,
Golipour
A
,
David
L
, et al.
Functional genomics reveals a BMP-driven mesenchymal-to-epithelial transition in the initiation of somatic cell reprogramming . Cell Stem Cell.
2010; ; 7 : :64.–77. 11.
Subramanyam
D
,
Lamouille
S
,
Judson
RL
, et al.
Multiple targets of miR-302 and miR-372 promote reprogramming of human fibroblasts to induced pluripotent stem cells . Nat Biotech.
2011; ; 29 : :443.–448. 12.
Woltjen
K
,
Michael
IP
,
Mohseni
P
, et al.
piggyBac transposition reprograms fibroblasts to induced pluripotent stem cells . Nature.
2009; ; 458 : :766.–770. 13.
Stadtfeld
M
,
Maherali
N
,
Borkent
M
,
Hochedlinger
K
. A reprogrammable mouse strain from gene-targeted embryonic stem cells . Nat Methods.
2010; ; 7 : :53.–55. 14.
Carey
BW
,
Markoulaki
S
,
Beard
C
, et al.
Single-gene transgenic mouse strains for reprogramming adult somatic cells . Nat Methods.
2010; ; 7 : :56.–59. 15.
Buganim
Y
,
Faddah
DA
,
Cheng
AW
, et al.
Single-cell expression analyses during cellular reprogramming reveal an early stochastic and a late hierarchic phase . Cell.
2012; ; 150 : :1209.–1222. 16.
Polo
JM
,
Anderssen
E
,
Walsh
RM
, et al.
A Molecular roadmap of reprogramming somatic cells into iPS cells . Cell.
2012; ; 151 : :1617.–1632. 17.
Sridharan
R
,
Tchieu
J
,
Mason
MJ
, et al.
Role of the murine reprogramming factors in the induction of pluripotency . Cell.
2009; ; 136 : :364.–377. 18.
Chen
X
,
Xu
H
,
Yuan
P
, et al.
Integration of external signaling pathways with the core transcriptional network in embryonic stem cells . Cell.
2008; ; 133 : :1106.–1117. 19.
Golipour
A
,
David
L
,
Liu
Y
, et al.
A late transition in somatic cell reprogramming requires regulators distinct from the pluripotency network . Cell Stem Cell.
2012; ; 11 : :769.–782. 20.
Soufi
A
,
Donahue
G
,
Zaret
KS
. Facilitators and impediments of the pluripotency reprogramming factors’ initial engagement with the genome . Cell.
2012; ; 151 : :994.–1004. 21.
Martello
G
,
Sugimoto
T
,
Diamanti
E
, et al.
Esrrb is a pivotal target of the gsk3/tcf3 axis regulating embryonic stem cell self-renewal . Cell Stem Cell.
2012; ; 11 : :491.–504. 22.
Festuccia
N
,
Osorno
R
,
Halbritter
F
, et al.
Esrrb Is a direct Nanog target gene that can substitute for Nanog function in pluripotent cells . Cell Stem Cell.
2012; ; 11 : :477.–490. 23.
Carey
BW
,
Markoulaki
S
,
Hanna
JH
, et al.
Reprogramming factor stoichiometry influences the epigenetic state and biological properties of induced pluripotent stem cells . Cell Stem Cell.
2011; ; 9 : :588.–598. |