| |
| Med Sci (Paris). 2013 April; 29(4): 434–436. Published online 2013 April 26. doi: 10.1051/medsci/2013294019.Chroniques génomiques La génétique du trou de souris Bertrand Jordan1* 1CoReBio PACA, case 901, parc scientifique de Luminy, 13288Marseille Cedex 9, France MeSH keywords: Animaux, Comportement animal, Cartographie chromosomique, Projet génome humain, Peromyscus, génétique |

Ces chroniques sont souvent consacrées à dissiper les illusions produites par les annonces des tenants du « tout génétique ». Pour changer, voici cette fois un véritable triomphe de la génétique du comportement, aidée par les nouvelles techniques d’analyse du génome et, singulièrement, par le séquençage de nouvelle génération. |
Il y a terrier et terrier L’étude qui fait l’objet de cette chronique, publiée [
1] et commentée [
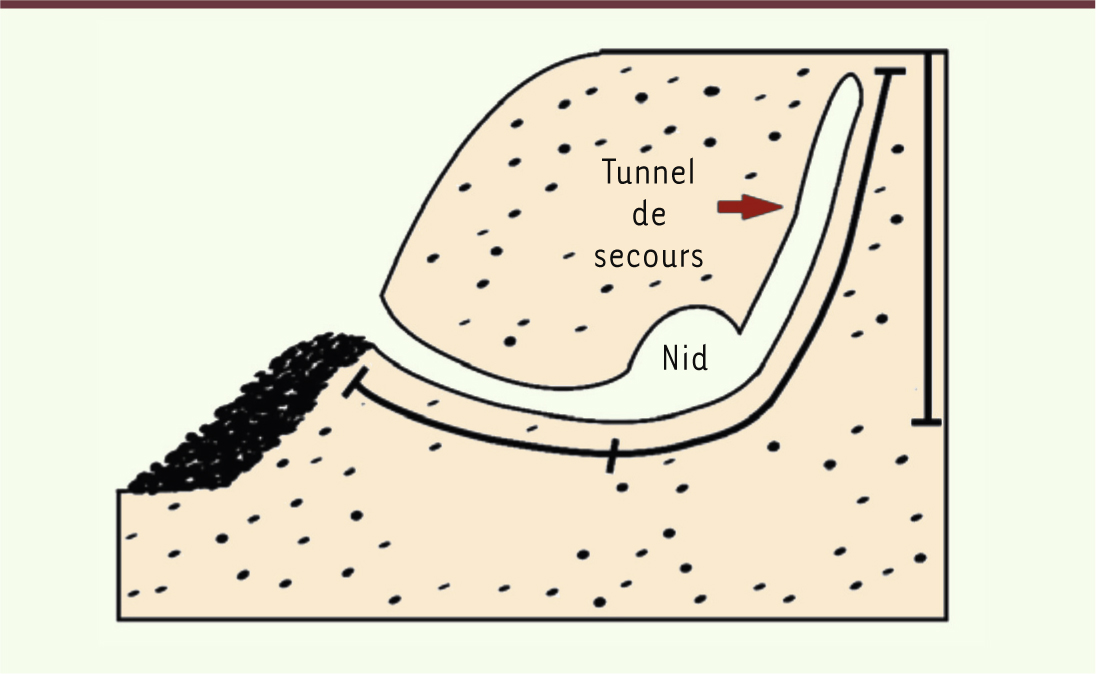
2] dans Nature, porte sur deux espèces apparentées, la « Souris des sables » (Oldfield mouse, Peromyscus polionotus) et la « Souris sylvestre » (Deer mouse, Peromyscus maniculatus). Ces deux « espèces-sœurs » sont séparées dans la nature mais restent fertiles dans leurs croisements au laboratoire. Elles construisent des terriers très différents : ceux de la souris des sables sont assez élaborés, avec un tunnel de secours (Figure 1), alors que sa cousine sylvestre creuse un terrier très sommaire comportant uniquement un court tunnel d’accès au nid.
 | Figure 1.
Aspect des terriers construits par la souris des sables (Peromyscus polionotus). Noter le tunnel de secours, utilisé notamment en cas d’invasion du nid par un serpent (Figure redessinée à partir de [ 1]). |
Ce comportement est inné : dès qu’on les introduit dans une enceinte ad hoc contenant du sable bien tassé, des souris élevées au laboratoire depuis des générations creusent spontanément un terrier dont l’architecture correspond dans tous les cas au modèle caractéristique de la sous-espèce considérée. Il s’agit donc bien d’une conduite génétiquement déterminée, qui plus est facilement quantifiable : on récupère la souris, on insuffle dans le terrier de la mousse de polyuréthane et, après solidification, on mesure les différentes longueurs (tunnel d’entrée, tunnel de secours, profondeur) sur ce moulage (Figure 1). |
Le tunnel de secours est dominant En bonne généticienne, la responsable de l’étude, Hopi Hoekstra (Cambridge, États-Unis), a commencé par croiser les deux souches. Elle a constaté que les souris de la génération F1 construisaient toutes des terriers de type polionotus, avec issue de secours : ce comportement est donc dominant. Elle a ensuite procédé au classique backcross, dans lequel on accouple des animaux F1 avec l’un des parents, la souche maniculatus en l’occurrence. Parmi les 272 animaux issus de ce croisement, la moitié (46 % exactement) creusent un tunnel de secours. Tout se passe donc comme si ce comportement complexe (qui implique toute une série d’actions coordonnées) était déterminé par un seul gène à effet dominant. Reste à aller plus loin sur le plan génétique, à voir si réellement un seul gène est impliqué et, si possible, à déterminer sa localisation. Et ici on se heurte à une difficulté de taille : il n’existe ni carte génétique, ni carte physique de Peromyscus, aucun snip caractérisé et encore moins de microarray permettant de caractériser rapidement un individu du point de vue génétique ! Il s’agit certes d’une « souris », d’un membre de l’ordre des rongeurs, mais on ne peut pas lui appliquer les données connues pour la souris de laboratoire. D’ailleurs, Peromyscus a 24 paires de chromosomes, et non 20 comme notre Mus musculus favorite. |
Le séquençage à la rescousse C’est là qu’intervient une nouvelle technique appelée Double Digest RADseq [
3], qui permet de bâtir rapidement et à peu de frais une carte génétique pour n’importe quelle espèce animale, grâce au séquençage à haut débit. J’en présente d’abord le principe dans le cas de la recherche de snip. L’ADN de chacune des deux souches de souris est digéré par deux enzymes de restriction (EcoR1 et Msp1) et la fraction de taille proche de 300 paires de bases est isolée. Après ligation d’adaptateurs différents pour chaque souche, les ADN sont mélangés, amplifiés par PCR (polymerase chain reaction) puis séquencés. L’analyse distingue ce qui vient de chaque souche grâce aux adaptateurs, et permet de repérer les snip qui sont fixes dans chaque souche et différents entre les deux - les auteurs en ont initialement trouvé 1 700 et en ont conservé 526 après différents contrôles de qualité. On peut alors passer à l’analyse des ADN des souris provenant du backcross, qui contiennent donc les chromosomes parentaux avec un certain nombre de recombinaisons. Chaque ADN provenant d’un individu est marqué par un adaptateur spécifique, puis les 272 ADN sont mélangés et séquencés ensemble en un seul cycle d’une machine Illumina. L’analyse des résultats permet de construire une carte génétique comportant 24 groupes de liaison correspondant aux 24 paires de chromosomes. On a ainsi, en deux expériences, construit une carte génétique portant 526 marqueurs, pour une espèce dont on ignorait tout au départ1 ! L’apport du séquençage ne s’arrête pas là : puisqu’on connaît maintenant l’assortiment d’allèles présent dans chacun des 272 animaux issus du backcross, on peut étudier l’association des différents segments chromosomiques ainsi définis avec le phénotype, en l’espèce la construction (ou non) d’un tunnel de secours. Et le résultat est sans ambiguïté : le creusement d’une issue de secours est lié à une seule région du groupe de liaison (chromosome) 5. Cette zone contient donc un (ou peut-être plusieurs ?) gène(s) dont un allèle induit, de manière dominante, le comportement « tunnel de secours ». On attend avec intérêt l’identification et l’étude de ce gène, qui est sûrement l’objet des travaux en cours dans le laboratoire de Hopi Hoekstra. |
Des conclusions généralisables et importantes Mais tels qu’ils sont déjà, ces travaux apportent des éléments importants. Je n’ai pas rapporté la partie de l’étude qui porte sur la longueur du tunnel d’entrée (long chez polionotus, court chez maniculatus), mais là aussi les conclusions sont nettes et identifient trois locus gouvernant de manière additive ce paramètre. On voit que ces aspects du comportement sont, du point de vue génétique, modulaires : chaque locus intervient de manière indépendante et additive, ce qui a des conséquences immédiates au niveau de l’évolution. Un comportement complexe (entrée longue, issue de secours) peut ainsi être construit pas à pas, et privilégié par la sélection à chacune de ses étapes : cette démonstration sans appel est très importante pour la compréhension des mécanismes évolutifs. Sur un plan plus général, ce très beau travail illustre la pertinence du choix fait par ses auteurs, celui d’étudier le comportement par le biais d’une architecture animale (dont les ruches, les nids d’oiseau ou les toiles d’araignée (Figure 2) donnent de multiples exemples). Ces édifices sont caractéristiques et facilement quantifiables et, dans la mesure où ils sont génétiquement déterminés, ils permettent une mise en relation directe des gènes et du comportement.
 | Figure 2.
Une autre architecture animale à étudier ? Toiles d’araignée en spirale (Australie de l’Ouest) (tirée de © Wikipedia Commons). |
D’autres aspects du comportement animal sont certes accessibles à l’étude génétique, comme le montre notamment une étude récente sur l’acceptation, ou non, de plusieurs reines au sein d’une colonie de termites, un comportement social qui est contrôlé par un cluster associé à un gène appelé Gp-9 [
5]. Notons aussi que l’utilisation astucieuse du séquençage de nouvelle génération pour bâtir rapidement et à peu de frais la carte génétique de n’importe quelle espèce a été un élément essentiel de cette étude (l’article cité [3] émane d’ailleurs de la même équipe et constitue une amélioration d’une technique publiée il y a quelques années [
6]). Ce bel exemple montre enfin la futilité de toute doctrine prônant le « tout environnemental » ou le « tout génétique ». Le rôle de l’environnement est généralement important, et doit être rappelé, à une époque où les média ont un peu trop tendance à tout attribuer à l’ADN. Mais la performance de ces souris qui n’ont jamais vu un tas de sable et qui, sitôt lâchées, s’empressent de creuser le terrier que leur dictent leurs gènes, nous démontre que certains comportements sont bel et bien innés, et que toute généralisation dans un sens ou dans l’autre est abusive. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Weber
JN
,
Peterson
BK
,
Hoekstra
HE
. Discrete genetic modules are responsible for complex burrow evolution in Peromyscus mice . Nature.
2013; ; 493 : :402.–405. 2.
Callaway
E
. Behaviour genes unearthed . Nature . , 2013; ; 493 : :284.. 3.
Peterson
BK
,
Weber
JN
,
Kay
EH
, et al.
Double digest RADseq: an inexpensive method for de novo SNP discovery and genotyping in model and non-model species . PLoS One.
2012; ;7 : :e37135.. 4.
Donis-Keller
H
,
Green
P
,
Helms
C
, et al.
A genetic linkage map of the human genome . Cell.
1987; ; 51 : :319.–337. 5.
Bourke
AFG
,
Mank
JE
. Genetics: A social rearrangement . Nature.
2012; ; 493 : :612.–613. 6.
Baird
NA
,
Etter
PD
,
Atwood
TS
, et al.
Rapid SNP discovery, genetic mapping using sequenced RAD markers . PLoS One . 2008; ; 3 : :e3376.. |