| |
| Med Sci (Paris). 2013 May; 29(5): 545–547. Published online 2013 May 28. doi: 10.1051/medsci/2013295020.Chroniques génomiques Un hasard dirigé Bertrand Jordan1*** 1CoReBio PACA, case901, parc scientifique de Luminy, 13288Marseille Cedex 9, France MeSH keywords: Trouble autistique, génétique, Humains, Mutation |

Le séquençage d’ADN à haut débit continue à nous apporter de multiples informations. Il établit notamment sur des bases solides la valeur de paramètres aussi fondamentaux que le taux de mutation dans notre génome - chiffre essentiel pour tout travail sur l’évolution mais jusqu’ici estimé de manière assez indirecte et pour tout dire un peu pifométrique. C’est à mon point de vue l’apport essentiel d’un important article récemment paru dans Cell [
1], même si son titre (Whole-genome sequencing in autism) laisse supposer qu’il est centré sur le déterminisme génétique de l’autisme. |
De l’importance des mutations de novo
On a pu, durant quelques années, négliger un peu les « nouvelles mutations », celles qui sont présentes chez l’individu atteint mais pas chez ses parents, pour s’intéresser surtout aux altérations transmises au fil des générations et qui permettent, par les approches du clonage positionnel, de remonter de la maladie au gène impliqué. Néanmoins, la persistance dans la population d’affections gravissimes comme la myopathie de Duchenne, bien qu’elle élimine ses porteurs du processus reproductif, indiquait dès le début que les mutations de novo (rendues plus probables dans ce cas par la très grande taille du gène concerné) devaient jouer un rôle. Et, de fait, un tiers environ des cas de cette myopathie correspondent à de nouvelles mutations [
2]. Plus récemment, l’étiologie génétique déjà fort complexe (bien qu’indubitable) de l’autisme [
3] s’est encore « enrichie » de la découverte de nombreuses mutations et CNV (copy number variations, regroupant insertions, délétions, inversions) apparues de novo chez ces patients et dont certaines semblent jouer un rôle causal [
4]. Mais ces données restaient relativement anecdotiques, reposant sur un examen partiel du génome des malades et ne pouvant prétendre à l’exhaustivité. Ici encore, les nouvelles techniques de séquençage permettent de se livrer à un examen réellement complet et fournissent des résultats solides. |
En avant pour le quadrille ! Les auteurs ont choisi de séquencer intégralement l’ADN de dix couples de jumeaux homozygotes ainsi que celui de leurs parents. Disposer de ces données pour de tels quads (quadrilles) présente de nombreux avantages : les mutations retrouvées chez les deux jumeaux mais absentes chez les parents sont presque à coup sûr des altérations germinales de novo, apparues dans un ovule ou un spermatozoïde. De plus, les couples de jumeaux choisis pour cette étude sont concordants pour l’autisme, ce qui justifie le titre de l’article même si, nous le verrons, ce dernier n’apporte pas d’éléments très nouveaux sur les déterminants génétiques de cette affection. Du point de vue technique, le séquençage - sous-traité au centre chinois BGI1, - a été effectué à une redondance respectable (40 fois) sur une machine Illumina HiSeq. Reste que des erreurs de séquençage sont toujours possibles2, et qu’une telle somme de données (quarante séquences humaines complètes !) ne peut pas être validée en détail « à la main ». Les auteurs ont donc mis au point un classificateur appelé Forest DeNovoMutation (ForestDNM) censé repérer les « vraies » mutations et éliminer les faux positifs. Ce logiciel utilise les informations fournies par le système d’acquisition des données sur la qualité des séquences : nombre de passages effectifs en un point donné, et existence ou non de conflits entre passages successifs. Testé sur une famille témoin (validée, elle, de manière manuelle et exhaustive), il a donné d’assez bons résultats (sensibilité et spécificité d’environ 90 %) et a donc été employé pour le reste de l’échantillon. |
Finalement, on détecte ainsi, grâce à cette approche qui peut prétendre être exhaustive, 581 DNM (de novo mutations, terme regroupant mutations ponctuelles et CNV) au sein des dix paires de jumeaux. Ces mutations, par définition, sont présentes chez les deux jumeaux et absentes chez leurs parents. Cela fait donc une soixantaine de DNM par embryon et, comme on a analysé l’ensemble du génome diploïde (six gigabases), le taux de mutation par génération s’établit ainsi à 10-8 - une valeur un peu plus basse (mais considérablement plus solide) que les estimations précédentes. Les trois quarts de ces DNM proviennent du père3 ; leur nombre exact varie de 42 à 75 et est nettement corrélé avec l’âge paternel : il passe d’environ 45 pour les pères âgés de moins de 30 ans à plus de 65 pour ceux qui ont dépassé la quarantaine. Le résultat le plus important de cet article concerne la distribution de ces DNM le long du génome. Celle-ci dévie de manière marquée d’une répartition au hasard, comme le montre la
Figure 1.
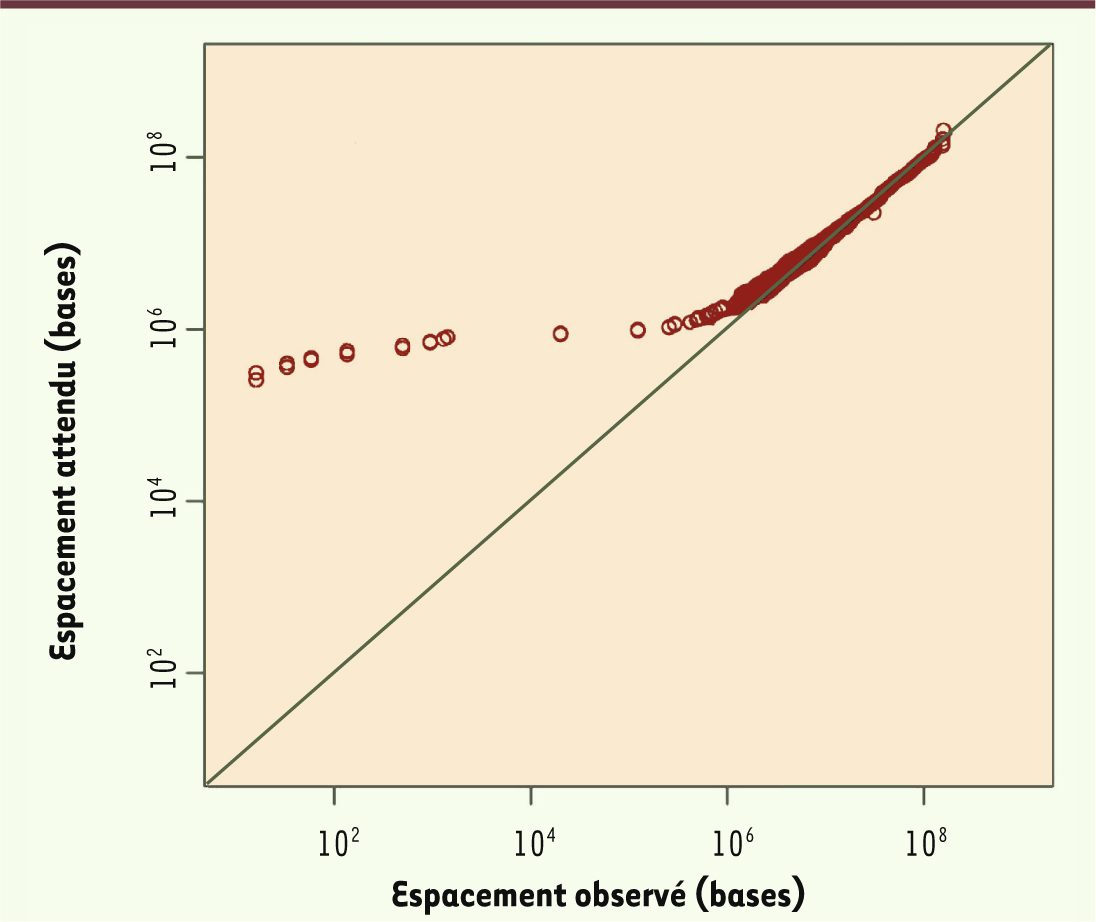
 | Figure 1.
Distribution attendue (ordonnée) et observée (abscisse) des distances entre DNM au sein de chaque génome séquencé (nombre de bases, en échelle logarithmique) (extrait redessiné de la figure 2 de [ 1]). |
On voit que si la grande majorité des distances entre DNM se répartit de la manière attendue (diagonale), on observe un excès significatif de courtes distances signalant des zones dans lesquelles le taux de mutation est plus important. À partir de ces données, les auteurs définissent alors un « index de mutabilité » (MI, mutability index), qui est la valeur du taux de mutation par nucléotide, afin de pouvoir décrire en détail le « paysage de mutabilité » (landscape of mutability) tout le long du génome. L’analyse révèle alors de nombreuses régions, longues de dizaines ou centaines de kilobases, au sein desquelles le taux de mutation est significativement différent de la moyenne. Par exemple, les zones de très forte mutabilité (plus de 7 fois la valeur moyenne) occupent 0,02 % du génome, celles de forte mutabilité (2 à 3 fois la valeur moyenne) s’étendent sur 9 % de notre ADN. |
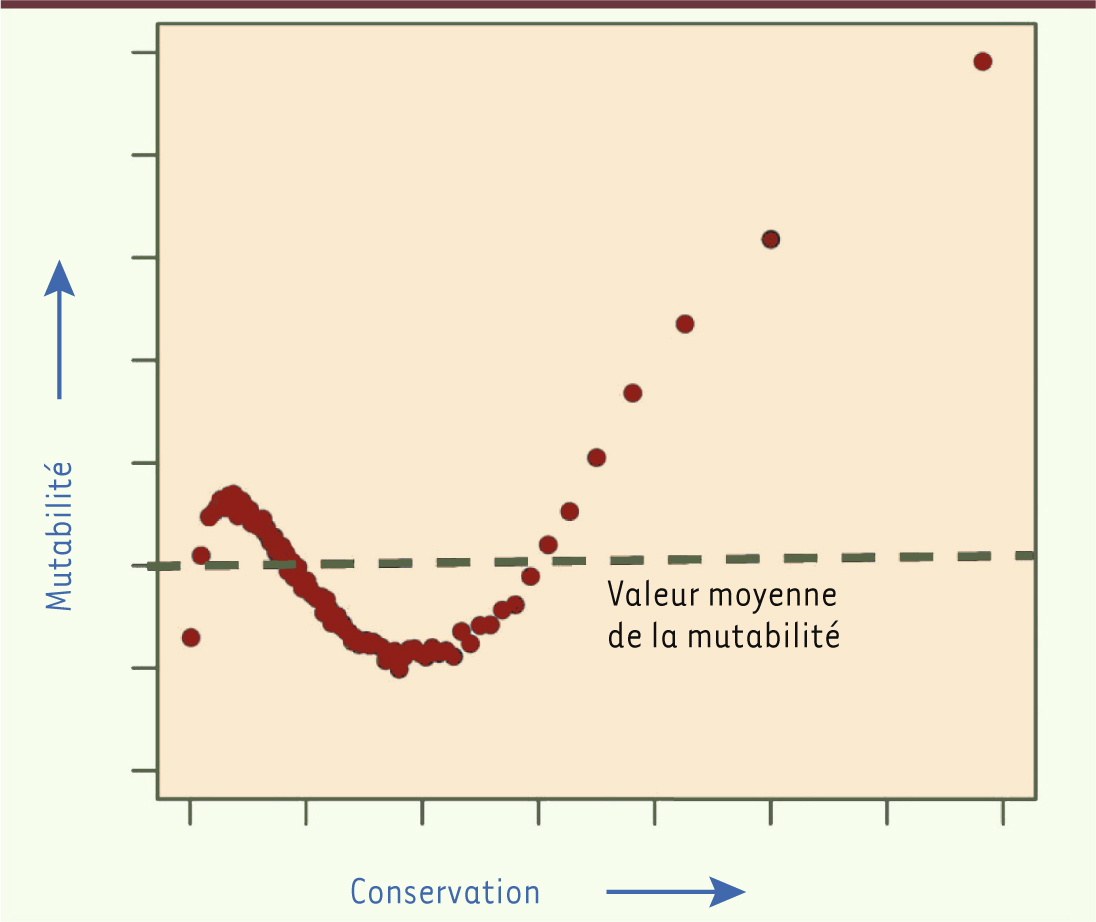
Mutabilité et conservation : une relation paradoxale Mais le résultat le plus surprenant de cette étude apparaît lorsque la mutabilité (l’index MI) est comparée au degré de conservation des séquences. Pour la majeure partie du génome, relativement peu conservée au cours de l’évolution, on observe bien la relation attendue : la mutabilité est élevée dans les zones les moins conservées et décroît lorsque la conservation augmente (Figure 2, partie gauche de la courbe).
 | Figure 2.
Corrélation entre mutabilité et conservation au sein du génome (unités arbitraires, échelle linéaire) (extrait partiel et redessiné de la figure 6 de [ 1]). |
Mais pour les régions les plus conservées et notamment les exons, la relation s’inverse, et la mutabilité augmente en même temps que la conservation (Figure 2, partie droite de la courbe) ! Ceci est confirmé par une étude portant uniquement sur les exons, qui montre là aussi une corrélation positive très nette entre conservation et mutabilité. En d’autres termes, certaines des séquences les plus « mutables » dans notre génome sont aussi les mieux conservées. Les auteurs avancent plusieurs hypothèses pour expliquer ce résultat étonnant, la plus séduisante peut-être étant celle de la « mutation adaptative » [
5] selon laquelle cette mutabilité pourrait être un trait positivement sélectionné car favorisant la diversité à un locus donné. On peut aussi imaginer un couplage entre réparation et transcription aboutissant à ce que les zones les plus activement transcrites soient aussi les plus susceptibles de muter. Quoi qu’il en soit, ce résultat est très important puisqu’il indique que les patrons de mutation de notre génome semblent favoriser les changements génétiques qui influent sur des fonctions biologiques. |
Nous avons un peu perdu de vue ce qui était sans doute l’objectif initial de ce travail, la recherche de mutations de novo impliquées dans l’étiologie de l’autisme. En fait, comme je l’ai déjà indiqué, la moisson à ce niveau est relativement limitée. Le taux de mutation n’apparaît pas plus élevé chez les patients, du moins d’après une comparaison effectuée sur les exons. La mutabilité est particulièrement élevée pour les gènes exprimés dans le cerveau, qui comprennent naturellement un certain nombre de gènes candidats pour la vulnérabilité à l’autisme. Et parmi ceux d’entre eux qui présentent une DNM dans cette étude, plusieurs s’avèrent corrélés avec l’autisme dans une autre étude brièvement mentionnée dans la discussion. Des indications donc, mais rien de vraiment nouveau ou de réellement concluant. |
Je n’ai décrit ici que les deux aspects qui m’ont semblé les plus importants de ce travail considérable, rapporté dans un article touffu dont on pourrait sans doute extraire bien d’autres informations. Il illustre la richesse des aperçus qu’offre aujourd’hui la possibilité de séquencer des génomes entiers, l’intérêt de concevoir les schémas expérimentaux les plus à même de fournir des données interprétables - et aussi le travail considérable d’analyse inhérent à de telles études. Cette étude apporte vraiment du nouveau sur le taux et la répartition des mutations de novo, même si elle ne s’avère pas particulièrement éclairante en ce qui concerne l’autisme. Reste, malgré l’abondance des résultats rapportés, un point qui n’est pratiquement pas évoqué par Michaelson et al. [1], celui de la nature des DNM observées : on aimerait savoir comment elles se répartissent entre mutations ponctuelles, insertions/délétions et autres anomalies. Ce sera sans doute le sujet d’une prochaine publication. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Michaelson
JJ
,
Shi
Y
,
Gujral
M
, et al.
Whole-genome sequencing in autism identifies hot spots for de novo germline mutation . Cell.
2012; ; 151 : :1431.–1442. 2.
Emery
AEH
. Principles and practice of medical genetics . In :
Emery
AEH
,
Rimoin
DL
, eds.
Edinburgh: : Churchill Livingstone; , 1983. 3.
Jordan
B.
, Autisme le gène introuvable . Paris: : Seuil;
2012. 4.
Sanders
SJ
,
Ercan-Sencicek
AG
,
Hus
V
, et al.
Multiple recurrent de novo CNVs, including duplications of the 7q11.23 Williams syndrome region, are strongly associated with autism . Neuron.
2011; ; 70 : :863.–885. 5.
Rosenberg
SM
. Evolving responsively: adaptive mutation . Nat Rev Genet.
2001; ; 2 : :504.–515. |