| |
| Med Sci (Paris). 2013 June; 29(6-7): 582–585. Published online 2013 July 12. doi: 10.1051/medsci/2013296010.Microbiome Un nouveau facteur de risque de cancer colorectal ? Christian Jobin1* 1Associate Professor, Department of medicine, pharmacology, immunology/microbiology, University of North Carolina, 111 Mason Farm rd, 7341B Medical Biomolecular Research Bldg, CB # 7032, NC 27599Chapel Hill, États-Unis MeSH keywords: Infections bactériennes, complications, Tumeurs colorectales, microbiologie, Infections à Escherichia coli, Humains, Métagénome, physiologie, Facteurs de risque |
Le cancer colorectal représente la troisième cause la plus fréquente de cancer, avec un million de nouveaux cas dans le monde chaque année, et sa mortalité avoisine 30%. Le développement des techniques « -omiques » , génomique, protéomique, métabolomique, nous permet aujourd’hui d’appréhender de façon assez précise les mécanismes moléculaires à l’œuvre dans l’émergence et la progression de cette forme de cancer. Malgré l’impact que ces connaissances ont eu sur la prise en charge diagnostique, et thérapeutique, mais aussi sur la prévention de ce cancer, celui-ci est loin d’être vaincu, et la poursuite des recherches est indispensable. Parmi les domaines les plus étudiés, celui qui analyse les relations entre environnement et développement tumoral est particulièrement actif. Déjà au xix
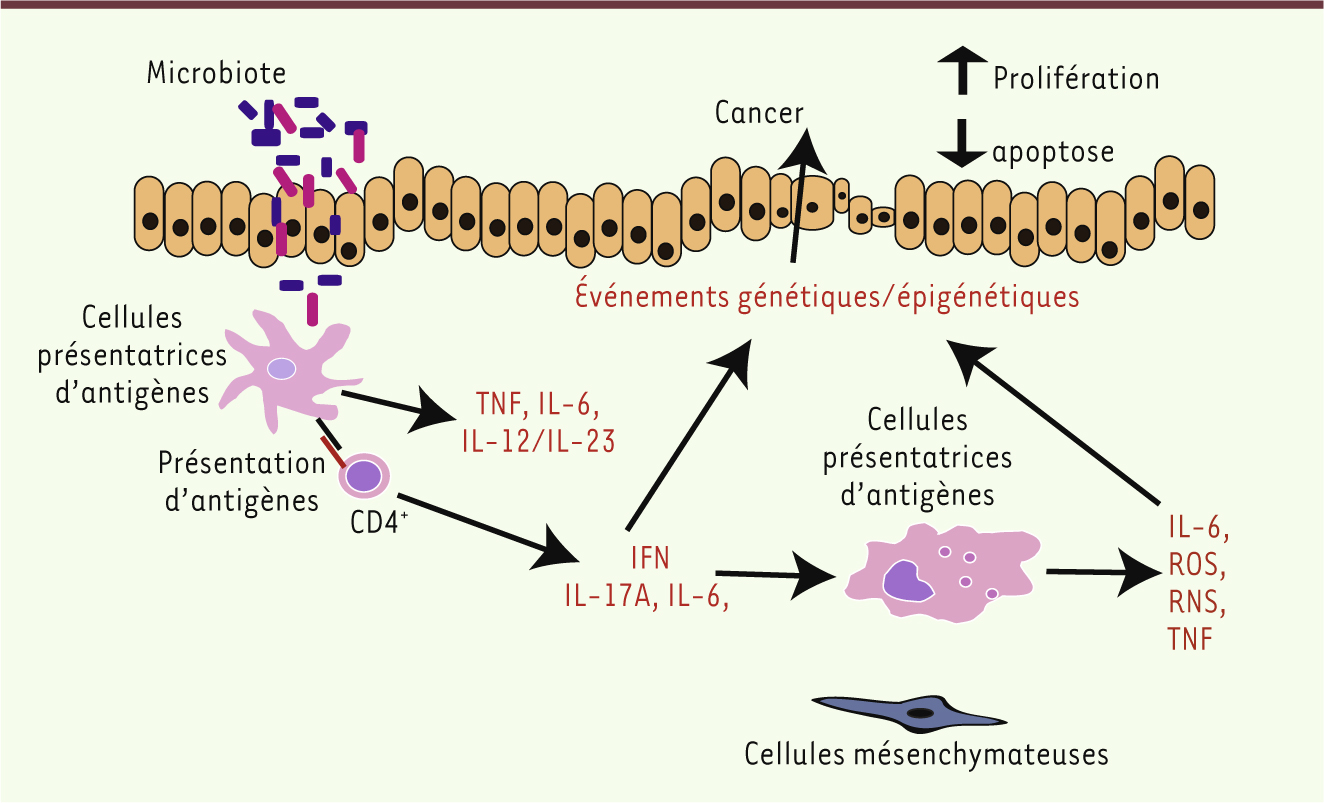
e siècle, le célèbre anatomopathologiste Rodolf Virchow notait la présence de cellules inflammatoires (leucocytes) dans les lésions tumorales de différents patients. Cette observation toute simple a probablement contribué au célèbre concept selon lequel l’inflammation fait le lit du cancer. Concept largement vérifié depuis, en particulier dans les localisations digestives estomac, côlon et foie. Dans l’exemple du cancer colorectal se développant dans un contexte de colite inflammatoire, les protagonistes sont connus : médiateurs de l’inflammation (TNF [tumor necrosis factor], IL-17A, IL-23), molécules génotoxiques (reactive oxygen and nitrogen oxygen species [ROS, RNS]) produites par des cellules variées (cellules de l’immunité, cellules mésenchymateuses), inductrices de modifications génétiques et épigénétiques à l’origine de lésions prénéoplasiques et finalement de tumeurs (Figure 1). Or, dans les maladies inflammatoires chroniques de l’intestin, le trio microorganisme-inflammation-cancer colorectal a été scruté de près par beaucoup d’équipes de recherche. Mais, si le lien entre microorganismes et inflammation est familier, il est intéressant de s’interroger sur les cibles de cette inflammation survenant chez l’hôte ? Concernent-elles exclusivement les cellules de l’hôte, des cellules mammifères; ne pourraient-elles pas modifier aussi l’équilibre des populations bactériennes colonisant l’intestin ?
 | Figure 1 Mécanismes pro-inflammatoires conduisant à l’induction d’événements génétiques et épigénétiques facilitant la cancérogenèse. ROS : reactive oxygen species; RNS : reactive nitrogen species.
|
|
Microbiome, inflammation et cancer colorectal Certains microorganismes pathogènes ont été impliqués dans des tumeurs : Helicobacter pylori dans le développement d’un cancer gastrique, le virus de l’hépatite C dans celui d’un carcinome hépatique. L’implication de microorganismes pathogènes dans le développement du cancer colorectal chez l’humain n’est pas établie. Encore plus important, la possible implication du microbiote endogène comme facteur de risque environnemental du développement d’un cancer colorectal n’a guère été étudiée. Ce qu’on pourrait interpréter comme un manque d’intérêt de la part de la communauté est d’autant plus curieux que la population bactérienne qui réside dans notre intestin – site d’origine de ces tumeurs – est énorme puisqu’elle est forte de 1013 à 1014 bactéries dont le génome ne comprend pas moins de 3 x 106 gènes. Cet écosystème microbien qui vit a proximité de l’épithélium intestinal exerce des fonctions essentielles pour l’équilibre homéostatique de l’hôte, qu’il s’agisse de la synthèse de vitamines essentielles, de la digestion et de l’hydrolyse de sucres complexes issus de l’alimentation, de l’éducation du système immunitaire muqueux, voire de la protection qu’assure le microbiote commensal vis-à-vis de pathogènes avec lesquels il entre en compétition pour les niches écologiques. Le revers de la médaille est que ces populations bactériennes inoffensives peuvent aussi être source de stimulations antigéniques néfastes chez un hôte sensibilisé, un processus démontré dans les maladies inflammatoires chroniques de l’intestin (MICI). |
Analyse métagénomique bactérienne et cancer colorectal On sait qu’une dérégulation de la réponse immune vis-à-vis du microbiote endogène de l’hôte est un événement clé de la survenue des MICI; or ces maladies représentent un facteur de risque avéré de cancer colorectal. L’avènement récent de la métagénomique, qui permet de s’affranchir de la culture des microorganismes [
12] notamment via les techniques de next-generation sequencing (NGS) basées sur l’analyse des gènes ribosomiaux bactériens 16S, offre aujourd’hui la possibilité d’analyser ce vaste écosystème de plus de 100 trillions de microorganismes, dont beaucoup ne peuvent pas être cultivés in vitro. L’identification des changements associés à diverses situations pathologiques, dont les MICI et les cancers colorectaux, est donc à notre portée. La comparaison des résultats obtenus à partir d’échantillons tissulaires ou de matières fécales chez des sujets contrôles et des patients cancéreux a permis de mettre en évidence chez ces derniers des modifications de la composition de la flore microbienne, un phénomène désigné par le terme de « dysbiose » . La divergence des microbiotes entre sujets sains et patients cancéreux était constante, et plusieurs groupes de microorganismes ont été identifiés, sans toutefois que l’on puisse assigner une catégorie particulière de microorganismes à la présence d’un cancer chez l’hôte. Ainsi, les selles de patients cancéreux étaient enrichies en bactéries anaérobies du groupe Bacteroides-Prevotella en comparaison de selles de sujets sains [
1]. Dans une autre étude, des microorganismes des genres Enterococcus, Escherichia/Shigella, Klebsiella, Streptococcus et Peptostreptococcus prédominaient dans le compartiment luminal des patients cancéreux comparés aux sujets contrôles, alors que la famille Lachnospiraceae, qui comprend des bactéries productrices de butyrate, une substance bénéfique pour l’intestin, y étaient moins abondantes [
2]. Firmicutes, Bacteroidetes et Proteobacteria sont observées en abondance à la surface de la muqueuse intestinale chez les patients porteurs d’adénomes comparés aux sujets indemnes [
3]. Chez un même patient, l’analyse comparée d’échantillons provenant de résections tumorales et du tissu sain adjacent a révélé une augmentation des microorganismes des phylums Bacteroidetes, et la diminution des phylums Firmicutes sélectivement dans les fragments tumoraux [
4]. Si l’on analyse les genres, Coriobacteridae, Roseburia, Fusobacterium et Faecalibacterium étaient sur-représentés et Enterobacteriaceae sous-représentés [4]. Cette augmentation de la quantité relative des Fusobacterium chez les patients atteints de cancer colorectal a également été observée par d’autres équipes travaillant avec des échantillons de tissu colique ou des écouvillons rectaux [
5–
7]. Or la présence de Fusobacterium nucleatum a été corrélée au développement de MICI [
8]. Mais, toutes ces données, qui traduisent un effort certain d’analyse comparative entre sujets sains et patients atteints de cancer, restent très descriptives et se fondent sur des corrélations, incitant à une interprétation prudente. Il se peut que ces modifications du microbiome traduisent simplement des changements dans l’environnement secondaires au développement de la tumeur. Prouver un lien causal entre altérations du microbiome et cancer requiert des analyses longitudinales et la réalisation d’expériences fonctionnelles dans des modèles expérimentaux où la composition du microbiote peut être modifiée, et les conséquences suivies et analysées. Il n’en reste pas moins vrai que les différences récurrentes de la composition des communautés bactériennes intestinales entre patients et sujets sains incitent à en décrypter l’origine et les conséquences. |
E.coli, maladies inflammatoires de l’intestin et cancer colorectal Des résultats in vitro ont précédemment démontré qu’un nombre élevé de souches pathogènes de Escherichia coli appartenant au pathovar AIEC pour adherent-invasive E. coli, adhéraient à la surface d’échantillons de muqueuse iléale et colique de patients atteints de MICI ou de cancer colorectal, par comparaison avec les muqueuses saines. Chez les patients atteints de MICI, l’étude métagénomique a identifié une augmentation des Proteobacteria, en particulier des Enterobacteriaceae, genre auquel appartient E. coli [
9]. Mais, comme précédemment, cette altération du profil des bactéries identifiées peut être une conséquence, et non la cause, de l’inflammation intestinale. Nous avons récemment montré [
10] dans un modèle de souris invalidées pour le gène codant l’interleukine 10 (Il10
−/-
) – qui développent des colites inflammatoires et représentent un modèle de MICI –, une augmentation considérable des bactéries de type Proteobacteria, en particulier E. coli, dans la lumière intestinale des souris qui développent un cancer colique. Pour analyser plus finement le lien entre cette prédominance d’E. coli et le développement d’un cancer colorectal dans ce contexte inflammatoire, nous avons inoculé à des souris Il10
−/-
axéniques soit une souche d’E. coli de type AIEC spécifique des souris (NC101), soit une souche Enterococcus faecalis humaine. Toutes deux induisent une inflammation intestinale chronique sévère chez ces hôtes, mais seules les souris ayant reçu la souche NC101 développent une tumeur colique. Cette sélectivité va à l’encontre de l’idée consensuelle que toute inflammation fait le lit du cancer, comme nous le mentionnions en introduction, et révèle que la composition microbienne est un paramètre important pour le développement tumoral. Afin d’analyser le mécanisme à l’œuvre, nous avons recherché dans le génome de ces E. Coli-NC101 des gènes codant pour des facteurs intervenant dans la réponse génotoxique de l’hôte. Un ilot génomique pks (polyketide synthase qui code pour des synthases de polycétides et de peptides non ribosomaux) a été précédemment identifié dans le génome de bactéries E. coli extra-intestinales [
11]. Le produit de cet ilot pks est la colibactine, complexe qui, même s’il n’a été ni purifié ni caractérisé jusqu’à présent, semble induire les voies de la réponse cellulaire aux dommages de l’ADN [11]. Or la souche E. coli NC101 contient cet ilot pks. Pour en comprendre l’influence dans le développement des cancers coliques chez les souris il10
−/-
infectées avec NC101, nous avons répété ces expéreinces en inoculant des souris axéniques il10
−/-
avec une souche NC101 dépourvue de cet ilot pks. Alors que les deux populations de souris, pks+ ou pks-, ayant reçu E. Coli développent une inflammation intestinale, seules les premières (pks+) développent une tumeur. Nous pouvons donc conclure que certes l’inflammation intestinale est un facteur de risque de développement d’un cancer, mais, à elle seule, elle n’est pas suffisante pour induire l’émergence tumorale dans le modèle murin utilisé. Il est donc probable que le microbiome, et les produits des gènes qu’ils portent (comme ici pks/colibactine) sont un déterminant essentiel pour le développement d’une pathologie tumorale. |
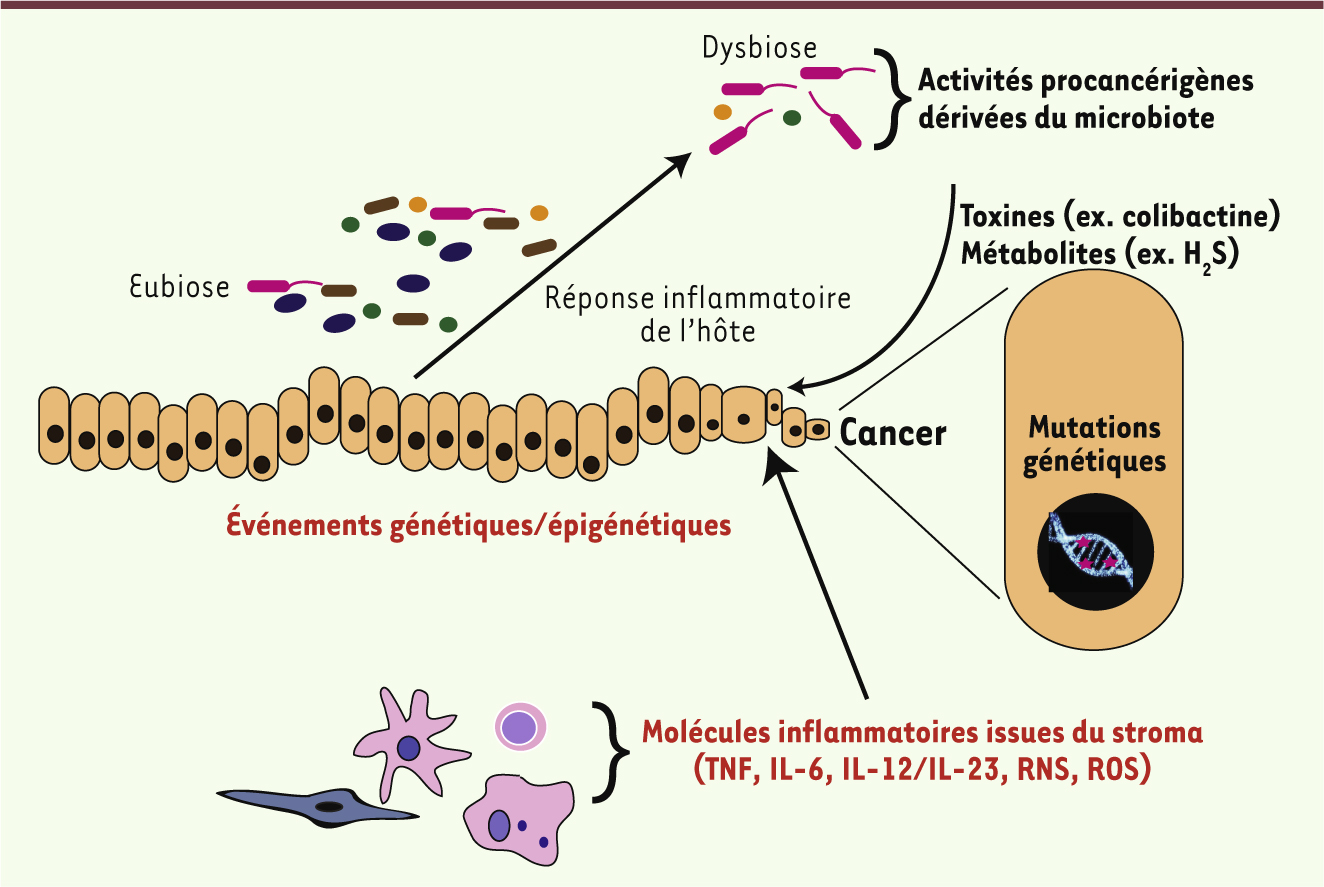
L’implication potentielle de symbiotes (organisme vivant dans un autre organisme et dont l’association est à bénéfices réciproques), bactéries non pathogènes, dans le développement de cancers, est un concept relativement neuf. Les récentes études du microbiome chez les patients atteints de cancer colorectal à différents stades de progression ont apporté des informations nouvelles sur les communautés bactériennes vivant dans les différentes niches écologiques de notre tube digestif. Elles ont ainsi révélé un lien potentiel entre les souches présentes et l’émergence de cellules tumorales, dont la causalité a été obtenue dans des modèles précliniques. Mais tout aussi important pour notre connaissance de la pathogenèse des cancers colorectaux est la reconnaissance de l’impact délètère de l’inflammation sur la composition microbienne. Il n’y a pas si longtemps, on pensait que la seule conséquence néfaste de l’inflammation sur l’environnement opérait via la sécrétion de molécules génotoxiques et de médiateurs de l’inflammation, tous deux compromettant la stabilité génétique et la physiologie des cellules épithéliales (Figure 1). Il est maintenant clair que, outre son retentissement cellulaire, cette inflammation influence également, et de façon probablement déterminante, la composition du microbiote intestinal. Une hypothèse très plausible aujourd’hui est que le microbiote, en réponse aux influences endogènes/exogènes (liées par exemple à l’alimentation, au stress, à l’inflammation) influencerait l’émergence/progression des cancers via la production de composés carcinogènes (par exemple la colibactine comme dans notre modèle, H2S, etc.) (Figure 2). Dans cette nouvelle vision des interactions microbiote-hôte, le microbiote joue un double jeu : il est certes à l’origine de la réponse inflammatoire de l’hôte (illustrée sur la
Figure 1
), mais celle-ci, via ses médiateurs, modifie considérablement la composition microbienne, conduisant à une dysbiose (par opposition à eubiose). En réponse à ces modifications de leur environnement, les microorganismes induisent des changements complexes de leurs réponses transcriptionnelles et métaboliques et produisent des molécules (toxines, métabolites, enzymes, etc.) qui peuvent influencer le développement d’une tumeur. Il est donc logique d’inclure le microbiome comme un des protagonistes important de la chaîne des évènements dont l’aboutissement est la transformation cellulaire maligne des cellules épithéliales coliques.
 | Figure 2
Changements du microbiote induits par l’inflammation qui facilitent l’émergence d’une tumeur. |
Dans le futur, il faudra corréler les réponses de l’hôte avec les caractéristiques du microbiome explorées par les approches métagénomique, métabolomique, métatranscriptomique. Dans ce cadre, l’analyse de l’influence du mode de vie (alimentation, stress, etc.) et du statut génétique sur le microbiome et la susceptibilité de l’individu au risque de cancer, conduira à une approche intégrative qui pourrait révéler de nouvelles voies de développement d’un cancer. Il est possible dans ce cadre que de nouveaux biomarqueurs, issus de l’analyse du microbiome, fassent leur apparition ainsi que des approches thérapeutiques ou préventives visant à moduler ce microbiote, dont l’objectif final sera d’améliorer la prise en charge des patients cancéreux. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article. |
Footnotes |
1.
Sobhani
I
,
Tap
J
,
Roudot-Thoraval
F
, et al. Microbial dysbiosis in colorectal cancer (CRC) patients . PLoS One.
2011; ; 6 : :e16393.. 2.
Wang
T
,
Cai
G
,
Qiu
Y
, et al. Structural segregation of gut microbiota between colorectal cancer patients and healthy volunteers . ISME J.
2011; ; 6 : :320.–329. 3.
Sanapareddy
N
,
Legge
RM
,
Jovov
B
, et al. Increased rectal microbial richness is associated with the presence of colorectal adenomas in humans . ISME J.
2012; ; 6 : :1858.–1868. 4.
Marchesi
JR
,
Dutilh
BE
,
Hall
N
, et al. Towards the human colorectal cancer microbiome . In
Ahmed
N
, ed.
PLoS One.
2011; ; 6 : :e20447.. 5.
Kostic
AD
,
Gevers
D
,
Pedamallu
CS
, et al. Genomic analysis identifies association of Fusobacterium with colorectal carcinoma . Genome Res.
2012; ; 22 : :292.–298. 6.
Castellarin
M
,
Warren
RL
,
Freeman
JD
, et al.
Fusobacterium nucleatum infection is prevalent in human colorectal carcinoma . Genome Res.
2012; ; 22 : :299.–306. 7.
Chen
W
,
Liu
F
,
Ling
Z
,
Tong
X
,
Xiang
C.
. Human intestinal lumen and mucosa-associated microbiota in patients with colorectal cancer . PLoS One.
2012; ; 7 : :e39743.. 8.
Strauss
J
,
Kaplan
GG
,
Beck
PL
, et al. Invasive potential of gut mucosa-derived Fusobacterium nucleatum positively correlates with IBD status of the host . Inflamm Bowel Dis.
2011; ; 17 : :1971.–1978. 9.
Mukhopadhya
I
,
Hansen
R
,
El-Omar
EM
,
Hold
GL
. IBD: what role do Proteobacteria play? . Nat Rev Gastroenterol Hepatol.
2012; ; 9 : :219.–230. 10.
Arthur
JC
,
Perez-Chanona
E
,
Mühlbauer
M
, et al. Intestinal inflammation targets cancer-inducing activity of the microbiota . Science.
2012; ; 338 : :120.–123. 11.
Nougayrede
JP
,
Homburg
S
,
Taieb
F
, et al.
Escherichia coli induces DNA double-strand breaks in eukaryotic cells . Science.
2006; ; 313 : :848.–851. 12.
Bernardo
P
,
Albina
E
,
Eloit
M
,
Roumagnac
P.
. Métagénomique virale et pathologie, une histoire récente . Med Sci (Paris).
2013; ; 29 : :501.–508. |