| |
| Med Sci (Paris). 2013 October; 29(10): 843–845. Published online 2013 October 18. doi: 10.1051/medsci/20132910010.Reprogrammation épigénétique des cellules hôtes par Legionella pneumophila
Monica Rolando,1 Christophe Rusniok,1 Raphael Margueron,2 and Carmen Buchrieser1* 1Institut Pasteur, unité biologie des bactéries intracellulaires, CNRS UMR 3525, 25-28, rue du Docteur Roux, 75015Paris, France 2Institut Curie, unité génétique et biologie du développement, CNRS UMR3215, Inserm U934, 26, rue d’Ulm, 75248Paris Cedex 05, France MeSH keywords: Protéines bactériennes, métabolisme, physiologie, Assemblage et désassemblage de la chromatine, génétique, Épigenèse génétique, Histone-lysine N-methyltransferase, Histone, Interactions hôte-pathogène, Humains, Legionella pneumophila, Maladie des légionnaires, immunologie, Macrophages, microbiologie, Méthylation |
Legionella pneumophila, une bactérie qui exploite l’hôte à son avantage
Legionella pneumophila est un bacille à Gram négatif à l’origine d’une infection pulmonaire grave appelée légionellose (ou maladie du légionnaire) [
11].
(→) Voir l’article de Y. Jamilloux et al., m/s n° 6-7, juin-juillet 2012, page 639
Les légionelles sont des bactéries vivant en milieu aquatique et affectionnant particulièrement les eaux tièdes. Ce sont des bactéries intracellulaires facultatives qui colonisent habituellement les amibes, protozoaires qui prolifèrent dans l’eau, mais elles sont également capables d’infecter l’homme par le biais d’aérosols. Une fois phagocytées, les légionelles survivent et se multiplient au détriment de la cellule hôte : cette réplication intracellulaire semble constituer le moyen prédominant de multiplication de ces bactéries dans l’environnement. La capacité de Legionella à infecter les cellules eucaryotes est intimement liée à la faculté de manipuler les mécanismes de la cellule afin de créer une niche intracellulaire pour sa réplication, et ceci grâce à un arsenal de protéines sécrétées dans la cellule hôte (effecteurs) qui agissent tout au long de l’infection. Plus particulièrement, un système de sécrétion de type IV (appelé Dot/Icm) entre en jeu pour transporter ces effecteurs dans la cellule cible [
12]. Ces effecteurs redirigent alors le trafic du phagosome de L. pneumophila, permettant ainsi sa transformation en un organite dérivé du réticulum endoplasmique (RE) [
1]. Le séquençage complet du génome de L. pneumophila en 2004 [
2,
3] a constitué une avancée importante dans la recherche sur les légionelles. Sa particularité la plus étonnante est la présence d’un grand nombre et d’une grande variété de gènes codant pour des protéines similaires à des protéines eucaryotes, dites eukaryotic-like proteins (ELP), ainsi qu’à des protéines contenant des domaines eucaryotes (EDP). Ces protéines sont les premières candidates à être impliquées dans le détournement des fonctions cellulaires [
4]. |
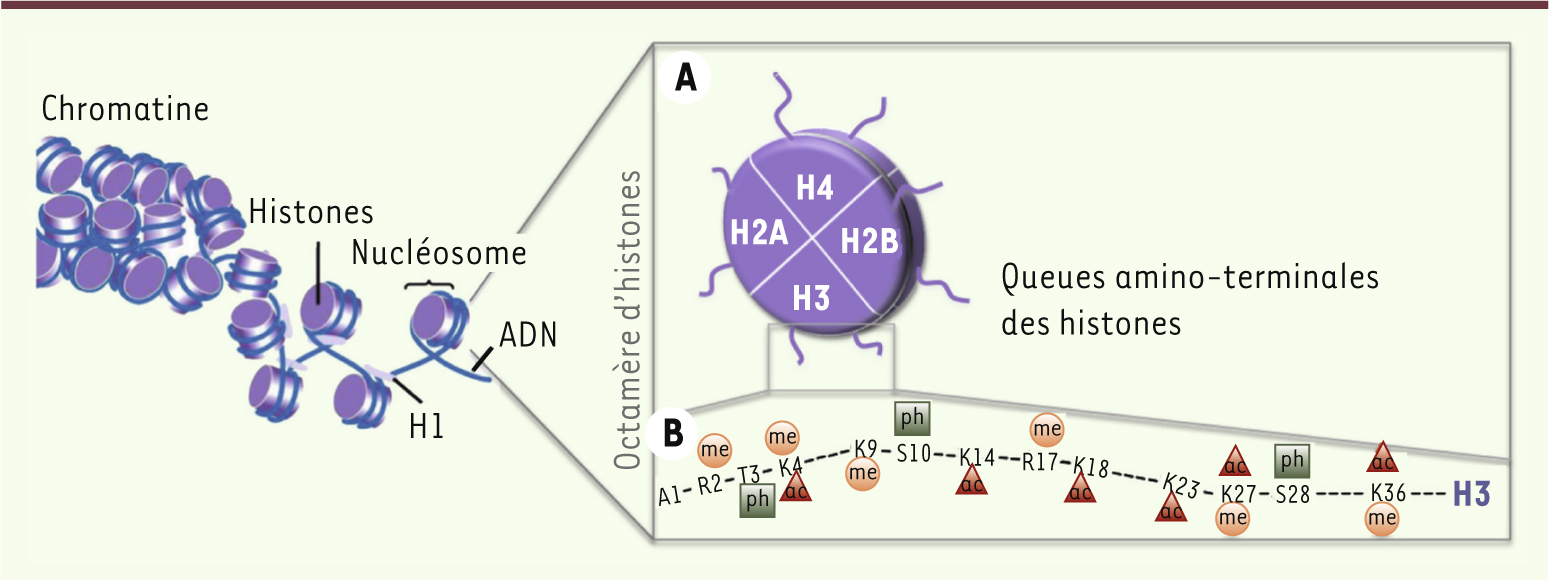
Organisation chromatinienne dans les cellules eucaryotes Le stockage de l’ADN dans le noyau des cellules eucaryotes requiert un fort degré de compaction. L’ADN est enroulé autour d’un complexe protéique, l’octamère d’histones, pour former le nucléosome. L’enchaînement des nucléosomes constitue la chromatine, le premier degré de compaction de l’ADN. L’octamère est formé d’un tétramère d’histones H3 et H4 et de deux dimères d’histones H2A et H2B (Figure 1). Chaque histone est composée, d’une part d’un domaine globulaire très structuré et nécessaire à la formation de l’octamère et, d’autre part, d’une queue amino-terminale se projetant en dehors du nucléosome et plus flexible. Cette dernière est chargée positivement grâce aux nombreux résidus basiques (Lys et Arg) qui la constituent. Les queues des histones font l’objet de diverses modifications covalentes post-traductionnelles dont les plus connues sont l’acétylation, la méthylation et l’ubiquitination des lysines, la méthylation des arginines, ainsi que la phosphorylation des sérines et thréonines (Figure 1). Ces modifications peuvent changer la charge des histones et induire ainsi une forme plus ouverte de la chromatine, propice, par exemple, au fonctionnement de la machinerie transcriptionnelle. Les modifications des histones peuvent également stabiliser la liaison de protéines à la chromatine sans nécessairement altérer sa structure. Dans le cas où des modifications régulant la fonction de l’ADN sont transmises en l’absence du stimulus qui les a induites, elles sont qualifiées d’épigénétiques [
5].
 | Figure 1.
Représentation schématique de l’organisation de la chromatine dans la cellule eucaryote. La chromatine provient de l’empaquetage de plusieurs nucléosomes. Ceux-ci sont formés d’octamères d’histones enroulés d’ADN à l’aide d’une molécule d’histone H1. A. L’octamère est formé par un tétramère d’histones H3 et H4, et de deux dimères d’histones H2A et H2B qui ont une forme globulaire et à partir desquelles les queues amino-terminales se projettent à l’extérieur. B. Détail de la queue amino-terminale de l’histone H3 sur laquelle différentes modifications post-traductionnelles connues sont indiquées (me : méthylation ; ph : phosphorylation ; ac : acétylation). |
De plus en plus de pathologies sont associées à un dysfonctionnement de ces régulations épigénétiques [
6]. |
RomA cible spécifiquement la chromatine de la cellule hôte En cherchant à mieux caractériser les protéines sécrétées dans la cellule infectée par L. pneumophila, nous nous sommes focalisés sur l’une d’entre elles, qui est codée par le gène lpp1683 et possède un domaine eucaryote SET. Le domaine SET (pour supressor of variegation-enhancer of zeste-trithorax) est un domaine très conservé d’environ 130 acides aminés, caractéristique de la plupart des « histone lysine méthyl-transférases » (HKMTases). Ces enzymes eucaryotes catalysent le transfert d’un groupe méthyle sur les résidus lysine (K) de différentes histones [
7]. Par des approches biochimiques, nous avons étudié la capacité de la protéine bactérienne codée par le gène lpp1683 à méthyler des histones. Nous avons ainsi mis en évidence que cette protéine est spécifique de l’histone H3, un substrat absent chez les procaryotes. Pour cette raison, nous avons nommé cette protéine RomA (pour regulator of methylation-A). Dans un article récemment publié dans Cell Host and Microbe [
8], nous rapportons l’importance de cette protéine au cours de la réplication bactérienne dans la cellule hôte. En effet, une bactérie mutante pour le gène lpp1683, construite au laboratoire, possède un défaut de virulence vis-à-vis des cellules eucaryotes qu’elle cible (soit des amibes, soit des lignées cellulaires humaines). RomA semble donc clairement impliquée dans la modulation des activités cellulaires de l’hôte, et plus particulièrement par son activité enzymatique de HKMTase. Ainsi RomA, tout comme les autres ELP/EDP, pourrait avoir été acquise au cours de la coévolution étroite des légionelles avec l’hôte eucaryote, afin d’exploiter au mieux les fonctions de la cellule hôte à l’avantage de la bactérie. Notre étude phylogénétique de RomA montre que le domaine SET porté par cette protéine est d’origine eucaryote, ce qui suggère que Legionella aurait acquis ce gène par une intégration dans son génome par transfert horizontal. Ce dernier se réfère à un processus dans lequel un organisme intègre du matériel génétique provenant d’un autre organisme sans en être le descendant [
13]. Nous avons en effet déjà montré que des transferts horizontaux de gènes eucaryotes vers Legionella et l’adaptation graduelle de ces protéines eucaryotes à Legionella sont un facteur important dans l’évolution de L. pneumophila [
9]. De façon intéressante, nous avons mis en évidence une spécificité particulière en termes de résidu ciblé par le domaine SET de L. pneumophila. En effet, nous avons identifié, par des techniques de spectrométrie de masse, que le résidu cible de RomA est la lysine 14 de l’histone H3 (noté H3K14) (Figure 2).
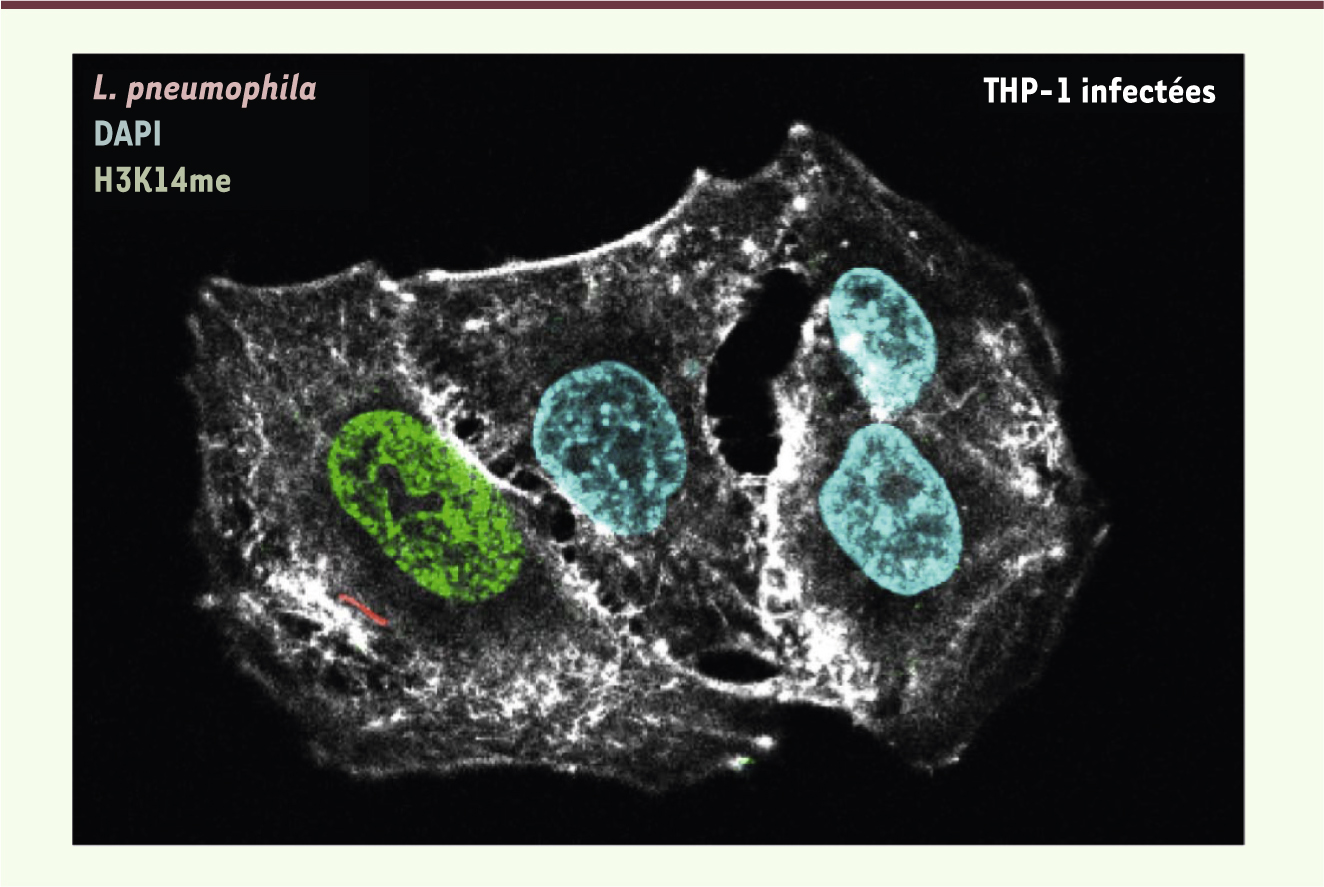
 | Figure 2.
Méthylation de la lysine 14 de l’histone H3 dans des cellules humaines infectées par Legionella pneumophila. Une lignée cellulaire de monocytes humains différenciés en macrophages (THP-1) est infectée avec une souche sauvage de L. pneumophila exprimant la protéine fluorescente DsRed (visible en rouge). Les noyaux sont marqués au DAPI, une molécule fluorescente capable de se lier fortement à l’ADN (cyan) et la méthylation de la lysine 14 de l’histone H3 est marquée avec un anticorps spécifique révélé en vert (H3K14me). Seule la cellule infectée possède une méthylation de la lysine 14 de l’histone H3. |
Cette découverte est très surprenante, car, à ce jour, seule l’acétylation de H3K14 a été observée. L’état d’acétylation des lysines au niveau des histones est régulé de manière très dynamique lors de la transcription. Ainsi, l’acétylation du résidu H3K14 corrèle fortement avec l’activation de la transcription. La méthylation de ce même résidu, en bloquant son acétylation, pourrait donc interférer avec la transcription. Par conséquent, notre hypothèse de travail était que L. pneumophila aurait acquis un gène codant pour une HKMTase afin de diminuer la réponse transcriptionnelle de la cellule hôte au cours de sa réplication. Ceci a tout d’abord été confirmé en observant que la méthylation de l’H3K14 (notée H3K14me) est enrichie au sein de l’hétérochromatine, une forme particulièrement condensée de la chromatine et transcriptionnellement peu active. En utilisant un système artificiel permettant de recruter la protéine RomA sur un gène rapporteur, nous avons confirmé que les modifications de la chromatine induites par RomA entraînaient une baisse de l’expression du gène rapporteur. Ce résultat a été reproduit au niveau de cibles endogènes de la cellule hôte. Nous avons ensuite entrepris une cartographie des régions de la chromatine ciblées par RomA à l’échelle du génome entier (ChIPseq : séquençage de chromatine immunoprécipitée). L’analyse de la nature des gènes cibles (Gene Ontology) nous a révélé qu’ils sont impliqués dans diverses voies de signalisation de la cellule, dont la régulation de la mort cellulaire ou des processus immunitaires. |
Conclusions et perspectives Ce travail nous a permis de mettre en évidence un nouveau mécanisme épigénétique de régulation du fonctionnement de la cellule eucaryote instauré par la bactérie à son profit. Même si l’on savait que des agents infectieux (bactéries ou virus) pouvaient recruter des HKMTases afin de réguler la transcription chez l’hôte, peu d’entre eux étaient connus pour cibler directement les histones in vivo [
10]. De plus, ce mécanisme inédit nous a aidés à mieux déchiffrer la régulation épigénétique chez les eucaryotes et ouvre de nouvelles perspectives de recherche sur la façon dont les bactéries intracellulaires ciblent la chromatine pour favoriser leur prolifération. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Nous remercions nos collègues qui ont participé à cette étude et présentons nos excuses à ceux dont les travaux n’ont pu être cités faute d’espace. Ces travaux étaient financés par l’Institut Pasteur, le Centre national de la recherche scientifique (CNRS), l’Institut Carnot-Pasteur MI, la Fondation pour la recherche médicale (FRM ; grant n° DEQ20120323697), l’île-de-France (DIM Malinf), l’ANR (n°ANR-10-LABX-62-IBEID), le programme ATIP-Avenir et l’Institut Curie. Monica Rolando est financée par une bourse Roux de l’Institut Pasteur.
|
1.
Hubber
A
,
Roy
CR
. Modulation of host cell function by Legionella pneumophila type IV effectors . Annu Rev Cell Dev Biol.
2010; ; 26 : :261.–283.
2.
Cazalet
C
,
Rusniok
C
,
Brüggemann
H
, et al.
Evidence in the Legionella pneumophila genome for exploitation of host cell functions and high genome plasticity . Nat Genet.
2004; ; 36 : :1165.–1173.
3.
Cazalet
C
,
Buchrieser
C
. Qu’apprend-on du génome de Legionella pneumophila?
Med Sci (Paris).
2005; ; 21 : :455.–457. 4.
Gomez-Valero
L
,
Rusniok
C
,
Cazalet
C
,
Buchrieser
C.
, Comparative and functional genomics of Legionella identified eukaryotic like proteins as key players in host-pathogen interactions . Front Microbiol.
2011; ; 2 : :208.. 5.
Margueron
R
,
Reinberg
D
. Chromatin structure and the inheritance of epigenetic information . Nat Rev Genet.
2010; ; 11 : :285.–296. 6.
Paschos
K
,
Allday
MJ
. Epigenetic reprogramming of host genes in viral and microbial pathogenesis . Trends Microbiol.
2010; ; 18 : :439.–447. 7.
Greer
EL
,
Shi
Y
. Histone methylation: a dynamic mark in health, disease and inheritance . Nat Rev Genet.
2012; ; 13 : :343.–357.
8.
Rolando
M
,
Sanulli
S
,
Rusniok
C
, et al.
Legionella pneumophila effector RomA uniquely modifies host chromatin to repress gene expression and promote intracellular bacterial replication . Cell Host Microbe.
2013; ; 13 : :395.–405.
9.
Lurie-Weinberger
MN
,
Gomez-Valero
L
,
Merault
N
, et al.
The origins of eukaryotic-like proteins in Legionella pneumophila . Int J Med Microbiol.
2010; ; 300 : :470.–481. 10.
Bierne
H
,
Hamon
M
,
Cossart
P.
, Epigenetics and bacterial infections . Cold Spring Harb Perspect Med.
2012; ; 2 : :a010272.. 11.
Jamilloux
Y
,
Jarraud
S
,
Lina
G
,
Etienne
J
,
Ader
F
. Legionella, légionellose . Med Sci (Paris).
2012; ; 28 : :639.–645. 12.
Foulongne
V
,
Michaux-Charachon
S
,
O’Callaghan
D
,
Ramuz
M
. Systèmes de sécrétion des protéines de type IV et virulence bactérienne . Med Sci (Paris).
2002; ; 18 : :439.–447.
13.
Daubin
V
,
Abby
S
. Les transferts horizontaux de gènes et l’arbre de la vie . Med Sci (Paris).
2012; ; 28 : :695.–698. |