| |
| Med Sci (Paris). 2013 October; 29(10): 846–848. Published online 2013 October 18. doi: 10.1051/medsci/20132910011.Xénobiotiques et microbiome intestinal actif Des effets insoupçonnés Corinne F. Maurice1* 1FAS Center for systems biology, Harvard university, 52 Oxford street, Cambridge, MA02138, États-Unis MeSH keywords: Femelle, Tube digestif, effets des médicaments et des substances chimiques, métabolisme, microbiologie, Régulation de l'expression des gènes bactériens, Interaction entre gènes et environnement, Humains, Métagénome, physiologie, Viabilité microbienne, génétique, Microbiote, Modèles biologiques, Xénobiotique, effets indésirables, pharmacologie |
Le corps humain contient des milliards de bactéries distribuées au niveau de la peau, de la bouche, du vagin, ou encore du tube digestif, qui correspondent collectivement au microbiote humain [
1,
2]. Le tube digestif est l’habitat le plus densément colonisé par ces bactéries (formant le microbiote intestinal), qui assurent des fonctions métaboliques essentielles pour l’homme. En effet, l’activité de l’ensemble des gènes bactériens dans l’intestin - ou microbiome intestinal - permet par exemple la dégradation de nombreux composés alimentaires, la mise en place d’un système immunitaire adapté, et peut influencer l’équilibre énergétique alimentaire, jouant ainsi un rôle déterminant dans l’obésité [1,
3]. Cependant, l’identification des bactéries du microbiote intestinal par séquençage de l’ADN ribosomal 16S ou du génome complet/partiel bactérien ne fournit pas ou peu d’indication sur leur activité. Malgré le rôle crucial du microbiome intestinal pour la santé, de nombreuses zones d’ombres reliant activité et diversité bactérienne persistent. En effet, bien que les membres du microbiote intestinal soient impliqués dans la biodisponibilité, la toxicité, et l’activité de plus de 40 composés pharmaceutiques [
4,
5], leurs réponses face à ces xénobiotiques (substances étrangères à notre corps) restent largement inconnues. En combinant des techniques de cytométrie en flux, de métagénomique et de métatranscriptomique, nous avons exploré, chez trois individus sains, l’impact de xénobiotiques (huit antibiotiques et six médicaments ayant une activité analgésique, cardiaque, antihelminthique, anti-inflammatoire ou antihistaminique) sur la physiologie et la diversité du microbiote intestinal, ainsi que sur les principales voies métaboliques du microbiome intestinal [
6]. |
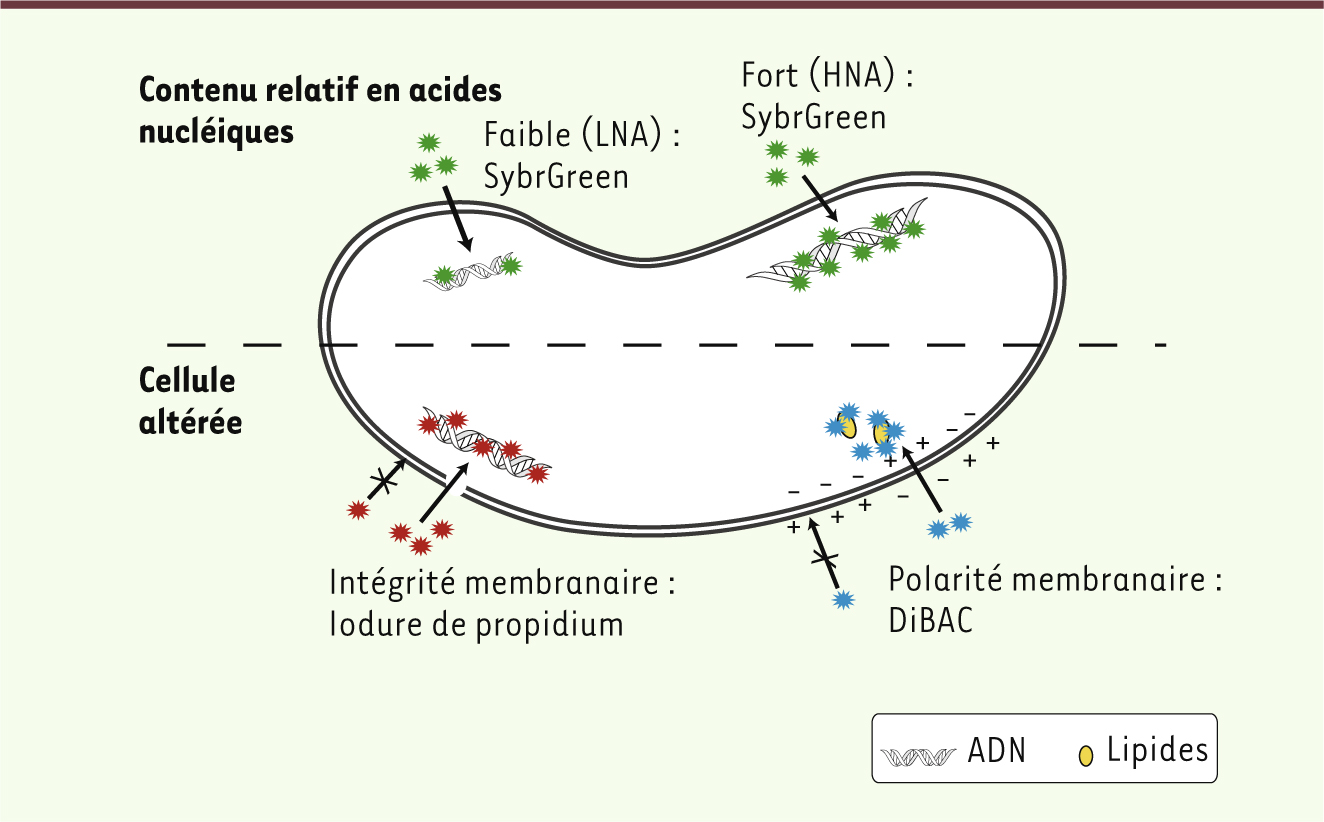
Les Firmicutes, les membres les plus actifs du microbiote intestinal Grâce à la cytométrie en flux et des marqueurs fluorescents de la physiologie bactérienne (Figure 1), nous avons déterminé dans 21 échantillons fécaux de trois individus les proportions relatives de bactéries ayant des contenus en acides nucléiques distincts (fort contenu : HNA ; faible contenu : LNA), et des membranes altérées. La perte de polarité membranaire a été détectée par l’indicateur DiBAC+ (bis-[1,3-dibutylbarbituric acid] trimethime oxonol) et la perte d’intégrité membranaire avec de l’iodure de propidium intracellulaire. Les cellules ayant un fort contenu en acides nucléiques sont métaboliquement très actives et/ou en division [
7], et représentent plus de 55 % du microbiote intestinal. Les cellules altérées avec perte de polarité ou d’intégrité membranaire représentent tout juste un tiers de la communauté [6]. Ces proportions, comparables d’un individu à l’autre, sont néanmoins variables chez un même individu au cours du temps, suggérant que le microbiote intestinal est dynamique et capable de réagir à des variations de signaux extérieurs qui restent à identifier (régime alimentaire, statut immunitaire, etc.).
 | Figure 1.
Fluorochromes et cibles intracellulaires. Le SybrGreen pénètre toutes les bactéries indépendamment de leur intégrité membranaire, se fixe aux acides nucléiques, et permet de distinguer les cellules HNA (fort contenu en acides nucléiques) des cellules LNA (faible contenu en acides nucléiques). L’iodure de propidium (Pi) se fixe aussi aux acides nucléiques, mais ne peut traverser une membrane intacte. Le DiBAC (bis-[1,3-dibutylbarbituric acid] trimethime oxonol) est un anion qui se fixe aux lipides intracellulaires, mais ne peut pas pénétrer dans une cellule qui maintient sa polarité membranaire négative. |
En combinant cytométrie en flux et séquençage (FACS-Seq) de la région variable V4 de l’ADNr 16S, nous avons trié et identifié les bactéries HNA, LNA, DiBAC+, et Pi+
(Figure 1). Les bactéries de l’ordre des Clostridiales (phylum des Firmicutes) prédominent parmi les cellules très actives HNA et les cellules altérées DiBAC+ et Pi+, alors que le phylum des Bifidobacteriaceae est majoritaire au sein du groupe des cellules moins actives LNA. Ces résultats suggèrent une différence fonctionnelle entre les deux principaux phylums du système digestif : les Firmicutes seraient métaboliquement actives et fréquemment renouvelées, contrairement aux Bacteroidetes. De plus, en comparant les niveaux d’expression de gènes transcrits par métatranscriptomique (RNASeq), nous avons observé que le phylum des Firmicutes contenait significativement plus de gènes transcrits que celui des Bacteroidetes, et les niveaux d’expression de 15 taxons parmi les Firmicutes étaient particulièrement élevés [6]. Collectivement, ces données suggèrent que les membres du microbiote intestinal seraient regroupés en niches écologiques définies, non seulement par leur emplacement physique et les ressources disponibles dans le système digestif, mais aussi par leur niveau d’activité. Par la suite, il serait intéressant d’explorer les patrons d’activité de ces taxons bactériens et leurs interactions. |
Modifications du microbiote intestinal induites par les xénobiotiques Le microbiote intestinal est souvent exposé à des perturbations de nature biotique et abiotique, comme une exposition à des xénobiotiques, généralement dans un cadre thérapeutique. Identifier les réponses du microbiome intestinal à une exposition de courte durée à des composés, qu’il s’agisse ou non d’antibiotiques, est nécessaire pour l’élaboration de traitements plus adaptés qui tiendraient également compte des modifications subies par le microbiome intestinal. Une incubation de 4 h de communautés fécales de trois individus, avec des antibiotiques, a significativement modifié la physiologie, l’expression des gènes et la structure des communautés microbiennes [6,
8]. La proportion des cellules altérées DiBAC+ a augmenté de plus de 10 %, et celle des cellules fortement altérées Pi+ a, quant à elle, doublé. Il est intéressant de noter que la réponse aux antibiotiques de chaque individu était très variable, sans que cela puisse être expliqué par les informations recueillies auprès des donneurs. En accord avec de précédentes études, les antibiotiques ont modifié la structure du microbiote intestinal [8], et 90 % des taxons négativement modifiés étaient des Firmicutes. De plus, les antibiotiques ont induit l’expression de nombreux gènes impliqués dans le transport de phosphate, la résistance aux antibiotiques, l’induction de prophages, la biosynthèse de vitamines et d’ARNt, ou la traduction [6]. Les autres composés, qui n’étaient pas des antibiotiques, ont peu modifié la physiologie et la diversité du microbiote intestinal. Cependant, nos données de métatranscriptomique ont mis en évidence d’importants changements dans l’expression de gènes impliqués dans le métabolisme et la dégradation de xénobiotiques, le transport membranaire, les voies métaboliques des polysaccharides et dans les réponses générales au stress. Plus de 300 familles de gènes dont les niveaux d’expression étaient fortement modifiés ont ainsi été identifiées, sans modification de la structure ni de la physiologie du microbiote. Ces changements d’expression génique suggèrent que de nombreuses interactions jusqu’à présent inconnues existent entre le microbiome intestinal et des xénobiotiques non bactéricides ou bactériostatiques. |
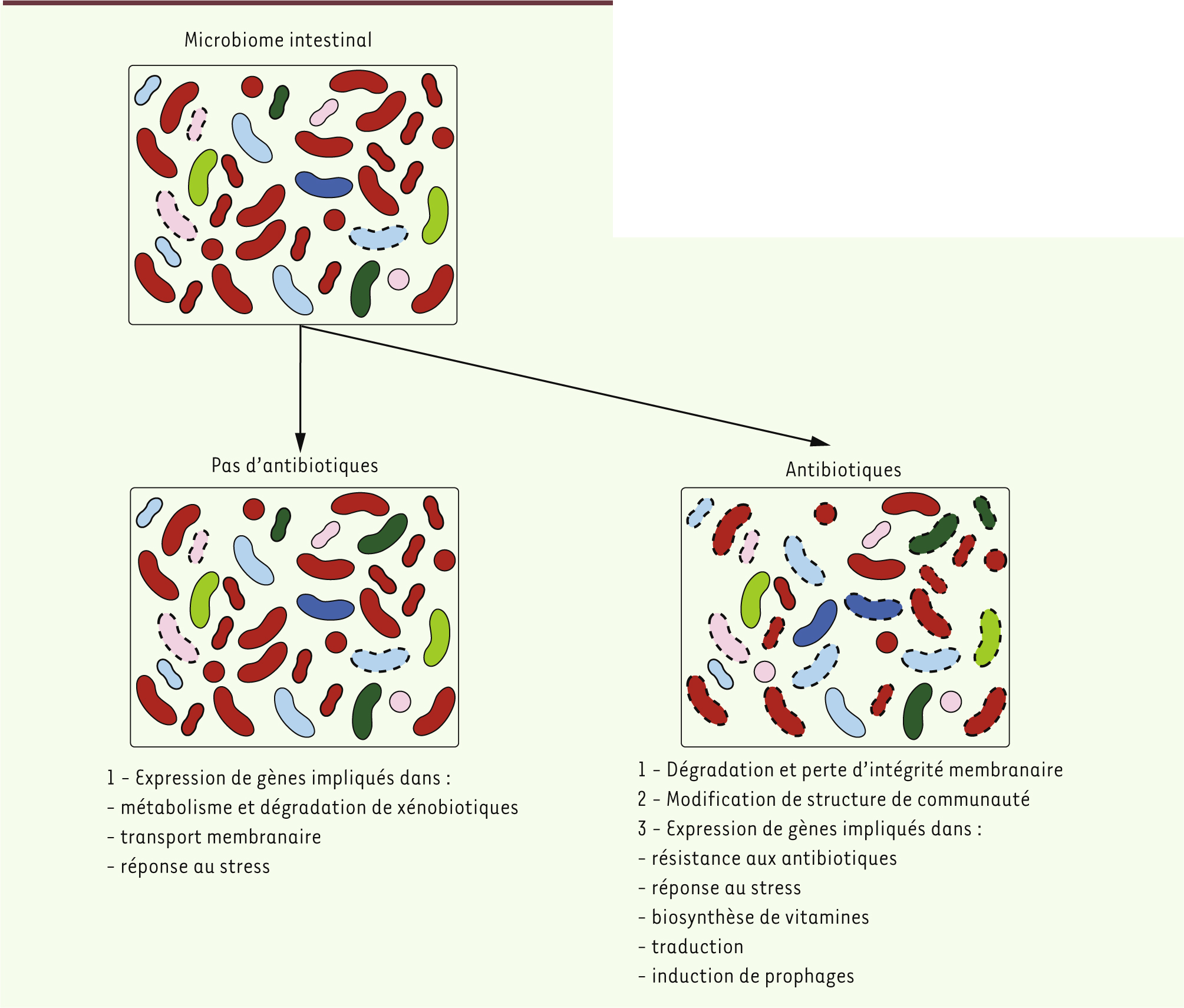
Conclusions et perspectives De nombreux composés pharmaceutiques peuvent être métabolisés par le microbiome intestinal, ce qui a d’importantes implications pour la santé humaine [5]. Identifier les bactéries qui métabolisent des xénobiotiques est crucial pour comprendre l’efficacité et la toxicité de certains composés. En déterminant que des xénobiotiques peuvent modifier de façon significative l’expression de gènes sans altérer la physiologie ou la structure des communautés bactériennes (Figure 2), notre étude souligne l’importance d’utiliser des approches expérimentales complémentaires reliant diversité, physiologie et activité bactériennes. L’application de ces dernières à un plus grand nombre d’individus permettra d’affiner ces patrons d’activité, ainsi que d’explorer les effets d’autres classes de composés pharmaceutiques. Les implications de telles études sont nombreuses : explorer les interactions entre les différents membres du microbiote intestinal, ou entre le microbiote et les cellules humaines, mener des études de β-diversité1 entre populations humaines et, à plus long terme, développer des traitements pharmaceutiques personnalisés pour limiter les effets secondaires. Nous commençons tout juste à mesurer l’importance du microbiome humain pour la santé, mais une chose est sûre : il est temps d’aller au-delà des études descriptives et de passer à la « vitesse fonctionnelle », pour révéler toute la complexité des interactions entre les xénobiotiques et le microbiome intestinal.
 | Figure 2.
Principales modifications subies par le microbiome intestinal après une exposition à des xénobiotiques. Schéma du microbiome intestinal composé de Firmicutes (rouge), Bacteroidetes (bleu) et Actinobactéries (vert). Les cellules actives sont représentées par des couleurs vives, alors que les moins actives sont en pastel. La membrane bactérienne intacte est représentée par un trait plein, et est hachurée lorsque la membrane est altérée. |
|
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiés dans cet article.
|
Footnotes |
1.
Consortium
HMP
. Structure, function and diversity of the healthy human microbiome . Nature.
2012; ; 486 : :207.–214.
2.
Costello
EK
,
Lauber
CL
,
Hamady
M
, et al.
Bacterial community variation in human body habitats across space and time . Science.
2009; ; 326 : :1694.–1697. 3.
Turnbaugh
PJ
,
Ridaura
VK
,
Faith
JJ
, et al.
The effect of diet on the human gut microbiome: a metagenomic analysis in humanized gnotobiotic mice . Sci Transl Med.
2009; ; 1 : :6.ra14.
4.
Sousa
T
,
Paterson
R
,
Moore
V
, et al.
The gastrointestinal microbiota as a site for the biotransformation of drugs . Int J Pharm.
2008; ; 363 : :1.–25. 5.
Haiser
HJ
,
Turnbaugh
PJ
. Is it time for a metagenomic basis of therapeutics?
Science.
2012; ; 336 : :1253.–1255. 6.
Maurice
CF
,
Haiser
HJ
,
Turnbaugh
PJ
. Xenobiotics shape the physiology and gene expression of the active human gut microbiome . Cell.
2013; ; 152 : :39.–50. 7.
Bouvier
T
,
del Giorgio
PA
,
Gasol
JM
. A comparative study of the cytometric characteristics of high and low nucleic-acid bacterioplankton cells from different aquatic ecosystems . Environ Microbiol.
2007; ; 9 : :2050.–2066. 8.
Dethlefsen
L
,
Relman
DA
. Incomplete recovery and individualized responses of the human distal gut microbiota to repeated antibiotic perturbation . Proc Natl Acad Sci USA.
2011; ; 108 : :4554.–4561. |