| |
| Med Sci (Paris). 2013 December; 29(12): 1077–1079. Published online 2013 December 20. doi: 10.1051/medsci/20132912005.Complexité du transcriptome dans les cellules germinales Magali Soumillon1,2* 1Harvard department of stem cell and regenerative biology, Harvard University, MA02138, États-Unis 2Broad Institute, Cambridge, MA02142, États-Unis MeSH keywords: Animaux, Cellules germinales, physiologie, Humains, Mâle, Spermatogenèse, génétique, Testicule, Transcriptome |
La complexité des transcriptomes Alors que l’information génomique est globalement identique dans toutes les cellules d’un individu, l’activation spatio-temporelle des différents gènes caractérise les différents organes et les cellules qui les constituent. Ainsi, le transcriptome (c’est-à-dire l’ensemble des molécules d’ARN résultant de l’activité des gènes exprimés) constitue une véritable signature et un lien clé entre génome et phénotype. L’avènement récent des nouvelles technologies de séquençage à haut-débit rend désormais accessible l’étude du transcriptome à l’échelle du génome dans son intégralité en surpassant les principales limitations des techniques jusqu’alors utilisées. Une étude récente utilisant le séquençage d’ARN (RNA-seq) a ainsi pu explorer l’étendue du transcriptome dans les tissus des organismes modèles que sont l’homme et la souris [
1]. Muscle et foie présentent des transcriptomes relativement peu complexes car la majorité des transcrits proviennent de quelques gènes fortement exprimés. Au contraire, rein, cerveau, et en particulier testicule, expriment un grand nombre de gènes, mais à un niveau d’expression plus homogène. Tirant parti d’un large jeu de données publiées récemment par notre groupe [
2], nous avons confirmé la complexité du transcriptome dans le testicule chez de nombreuses espèces couvrant toutes les principales lignées des mammifères et des oiseaux (homme, macaque, souris, opossum, ornithorynque et poulet) [
3]. En utilisant ces données de RNA-seq à large échelle, nous avons pu observer que les gènes codant pour des protéines ne sont pas les seuls éléments les plus abondamment transcrits dans ces tissus dits « complexes ». En effet, nous avons aussi pu identifier un grand nombre de transcrits multiexoniques jusqu’alors non annotés et ayant les caractéristiques des larges ARN non codants intergéniques (lincARN, potentiels régulateurs de l’expression des gènes [
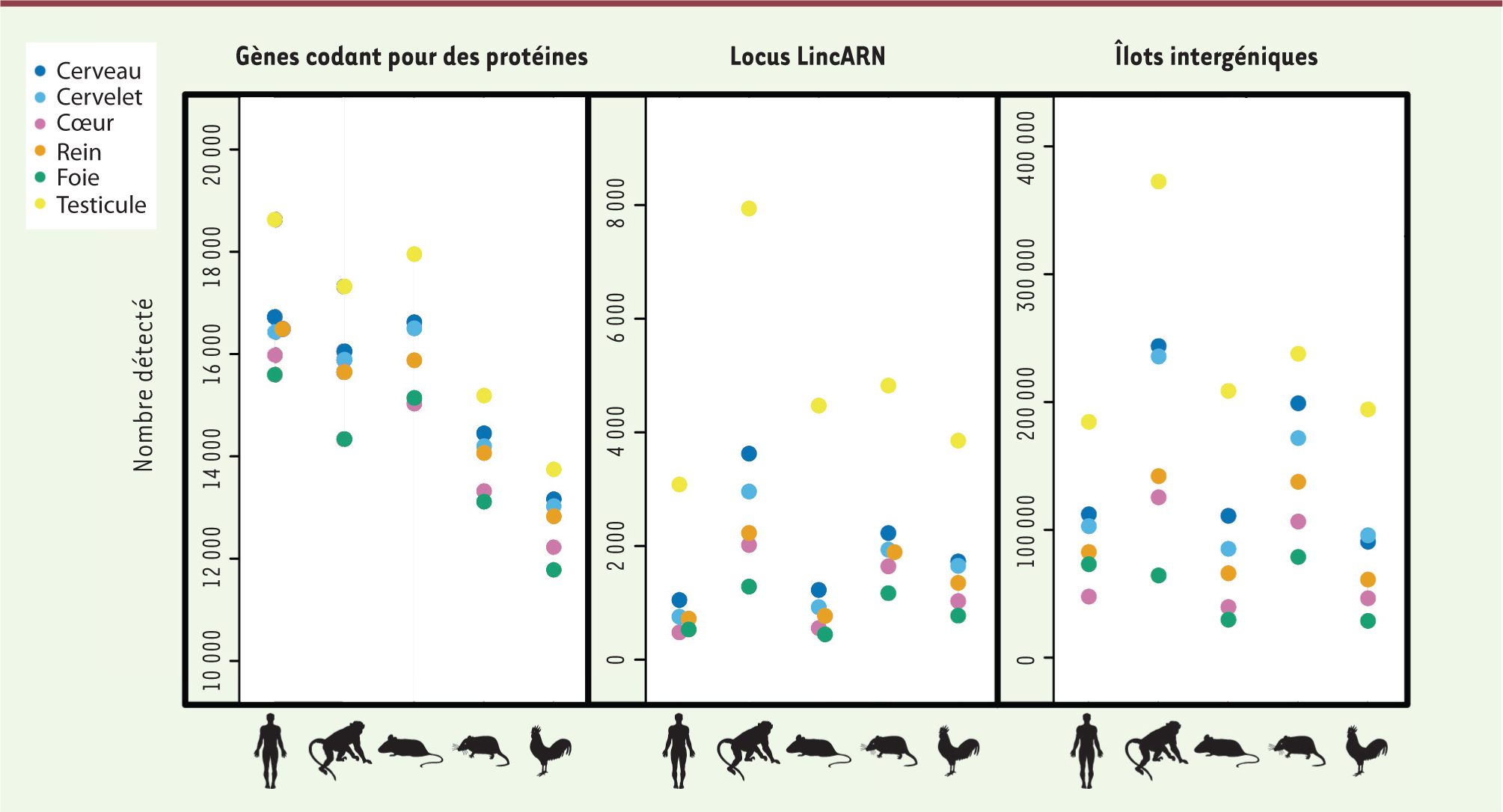
4]), ainsi qu’un grand nombre d’éléments intergéniques aussi préférentiellement transcrits dans ces tissus (Figure 1).
 | Figure 1.
Complexité du transcriptome dans le testicule chez les mammifères et les oiseaux. Nombre de gènes codant pour des protéines, lincARN prédits et éléments intergéniques autosomiques transcrits dans six organes pour cinq espèces (de gauche à droite : homme, macaque, souris, opossum et poulet). |
|
L’origine de la complexité du transcriptome dans le testicule Déterminer l’origine de la complexité du transcriptome nécessite de prendre en considération la composition de chacun des différents tissus. Afin d’examiner les particularités transcriptionnelles du testicule, nous nous sommes concentrés sur la souris en tant qu’organisme modèle, et avons isolé les cinq types cellulaires majeurs impliqués dans la spermatogenèse [3] : les cellules de Sertoli, cellules somatiques qui nourrissent les cellules germinales au cours de leur maturation ; les spermatogonies de type A, précurseurs mitotiques des cellules germinales comprenant des cellules souches de la spermatogenèse et des spermatogonies différenciées ; les spermatocytes en phase pachytène, résultant de la différenciation des spermatogonies et au sein desquels a lieu la méiose et son brassage génétique ; les spermatides rondes, cellules germinales haploïdes dérivant des spermatocytes ; et les spermatozoïdes. En générant une quantité significative de données pour ces différents types cellulaires et en les comparant à celles obtenues de tissus ayant différents degrés de complexité, nous avons pu identifier les spermatocytes et les spermatides comme étant les principaux contributeurs de la complexité du testicule dans son ensemble. Ces cellules contribuent à la fois par la variété des éléments qui y sont transcrits mais aussi par la variété des motifs d’épissage alternatif et par la quantité totale d’ARN produite par ces cellules. Un large panel de gènes codant pour des protéines, de potentiels lincARN nouvellement prédits, mais aussi un nombre important d’éléments intergéniques tels que des rétrocopies (copies de gènes se réintégrant dans l’ADN par transcriptase inverse et caractérisées par l’absence d’introns), des éléments transposables et des îlots intergéniques non annotés sont préférentiellement transcrits dans ces cellules. |
Les cellules germinales comme catalyseurs de la naissance de nouveaux gènes ? De nombreuses études évolutives ont présenté le testicule comme un organe à part qui évolue très rapidement et constitue un potentiel catalyseur de la naissance et de l’évolution de nouveaux gènes par les pressions sélectives auxquelles il est soumis [
5]. Les gènes nouvellement formés, qu’ils s’agissent de rétrogènes, de gènes dupliqués ou de gènes codant pour des protéines, ont une tendance à être spécifiquement ou préférentiellement exprimés dans le testicule avant d’éventuellement acquérir d’autres fonctions dans d’autres tissus somatiques [
6,
7]. |
La transcription globale observée reflète-t-elle nécessairement une fonctionnalité pour chacun des éléments transcrits ? L’analyse du transcriptome des différents types cellulaires intervenant au cours de la spermatogenèse indique que la diversité du transcriptome au sein des seuls spermatides surpasse étonnamment celle d’organes complets. La question se pose de savoir si tous les transcrits détectés au cours de la spermatogenèse, et plus spécifiquement durant la spermiogenèse (phase finale de la spermatogenèse), jouent un rôle fonctionnel. Un grand nombre de gènes codant pour des protéines, notamment ceux qui sont activés dans les spermatocytes et les spermatides, exercent une fonction indéniable au cours du processus complexe qu’est la spermatogenèse. Parmi ces gènes, on trouve par exemple un excès de gènes codant pour des protéines localisées dans les flagelles et dans les mitochondries, tous deux essentiels à la motilité des spermatozoïdes. Au contraire, la faible conservation au cours de l’évolution des îlots intergéniques, également transcrits abondamment dans les spermatocytes et les spermatides, reflète le caractère globalement non fonctionnel de tels éléments. Néanmoins, il semble qu’une fraction de ces régions transcrites ait été soumise à des contraintes sélectives récentes pouvant préfigurer un éventuel gain de fonction [3]. |
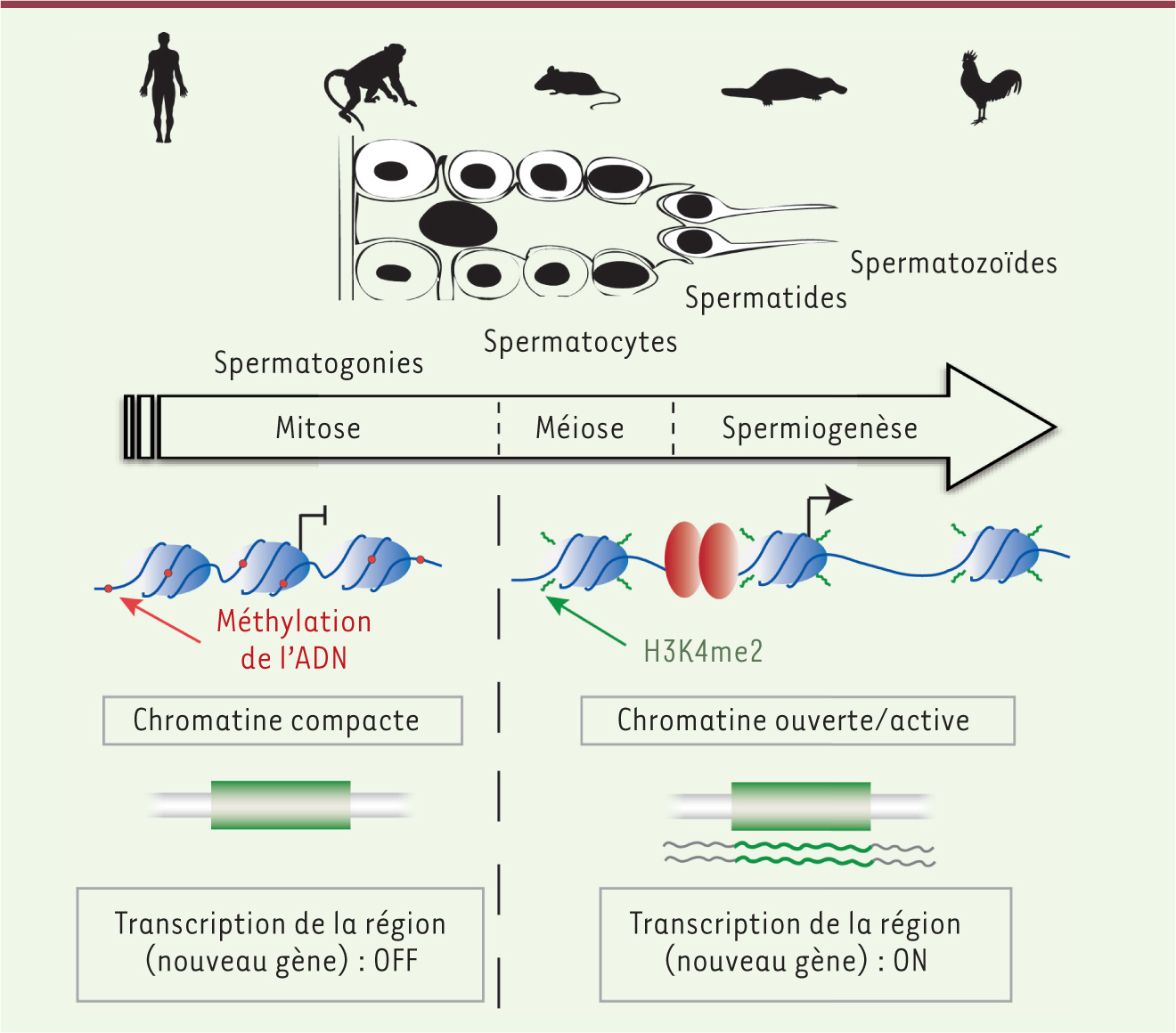
Quels sont les mécanismes potentiels à l’origine de cette transcription globale ? La chromatine est remaniée de manière intensive durant la spermatogenèse et plus particulièrement pendant la spermiogenèse, phase durant laquelle le matériel génétique est condensé de façon très intense [
8,
9]. Cette étape de condensation implique notamment le remplacement des histones standard par d’autres variantes d’histones puis plus tard par les protamines, petites protéines nucléaires [
10]. Cette phase de réarrangement de la chromatine s’avère être potentiellement propice à une transcription extensive due à une ouverture globale de la chromatine. Notre étude de la composante épigénétique dans les cellules méiotiques et post-méiotiques indique que la déméthylation des dinucléotides CpG, associée à un état ouvert/actif de la chromatine des régions promotrices des divers éléments, joue généralement un rôle favorable à la transcription globale observée. De plus, nos données concernant le niveau de diméthylation de la lysine 4 de l’histone 3 (H3K4me2), reflétant potentiellement les échanges d’histones au cours de la compaction du matériel génétique, confirme l’état général d’ouverture de la chromatine, corrélé à l’expression des divers éléments détectés lors de l’analyse du transcriptome (Figure 2).
 | Figure 2.
Schéma synthétisant les modifications épigénétiques au cours de la spermatogenèse et leur impact sur la transcription.
|
|
L’étendue de la transcription dans les cellules germinales mâles, en particulier dans les spermatocytes et les spermatides, semble facilitée par l’état globalement ouvert de la structure de l’ADN en conséquence du réarrangement de la chromatine en vue de la compaction finale du matériel génétique (Figure 2). Même si cette transcription globale semble n’être qu’une conséquence secondaire de la réorganisation de la chromatine, elle implique pourtant des conséquences évolutives importantes. En effet, elle apparaît comme un terrain propice à la naissance de nouveaux gènes, à travers la transcription initiale de gènes dupliqués ou l’émergence de potentiels nouveaux lincARN. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Ramsköld
D
,
Wang
ET
,
Burge
CB
,
An
Sandberg R.
, abundance of ubiquitously expressed genes revealed by tissue transcriptome sequence data . PLoS Comput Biol.
2009; ; 5 : :e1000598.. 2.
Brawand
D
,
Soumillon
M
,
Necsulea
A
, et al.
The evolution of gene expression levels in mammalian organs . Nature.
2011; ; 478 : :343.–348. 3.
Soumillon
M
,
Necsulea
A
,
Weier
M
, et al.
Cellular source and mechanisms of high transcriptome complexity in the mammalian testis . Cell Rep.
2013; ; 3 : :2179.–2190. 4.
Derrien
T
,
Guigo
R
. De longs ARN non codants activateurs de la transcription des gènes . Med Sci (Paris).
2011; ; 27 : :359.–361. 5.
Nielsen
R
,
Bustamante
C
,
Clark
AG
, et al.
A scan for positively selected genes in the genomes of humans, chimpanzees . PLoS Biol. , 2005; ; 3 : :e170.. 6.
Marques
A
,
Dupanloup
I
,
Vinckenbosch
N
,
Reymond
A
,
Kaessmann
H.
, Emergence of young human genes after a burst of retroposition in primates . PLoS Biol.
2005; ; 3 : :e357.. 7.
Kaessmann
H
. Origins, evolution, and phenotypic impact of new genes . Genome Res.
2010; ; 20 : :1313.–1326. 8.
Kimmins
S
,
Sassone-Corsi
P
. Chromatin remodelling and epige- netic features of germ cells . Nature.
2005; ; 434 : :583.–589. 9.
Sassone-Corsi
P.
2002. Unique chromatin remodeling and transcriptional regulation in spermatogenesis . Science.
2002; ; 296 : :2176.–2178. 10.
Montellier
E
,
Rousseaux
S
,
Khochbin
S
. Feux croisés sur le nucléosome : bases moléculaires de la compaction du génome mâle haploïde . Med Sci (Paris).
2012; ; 28 : :485.–489. |