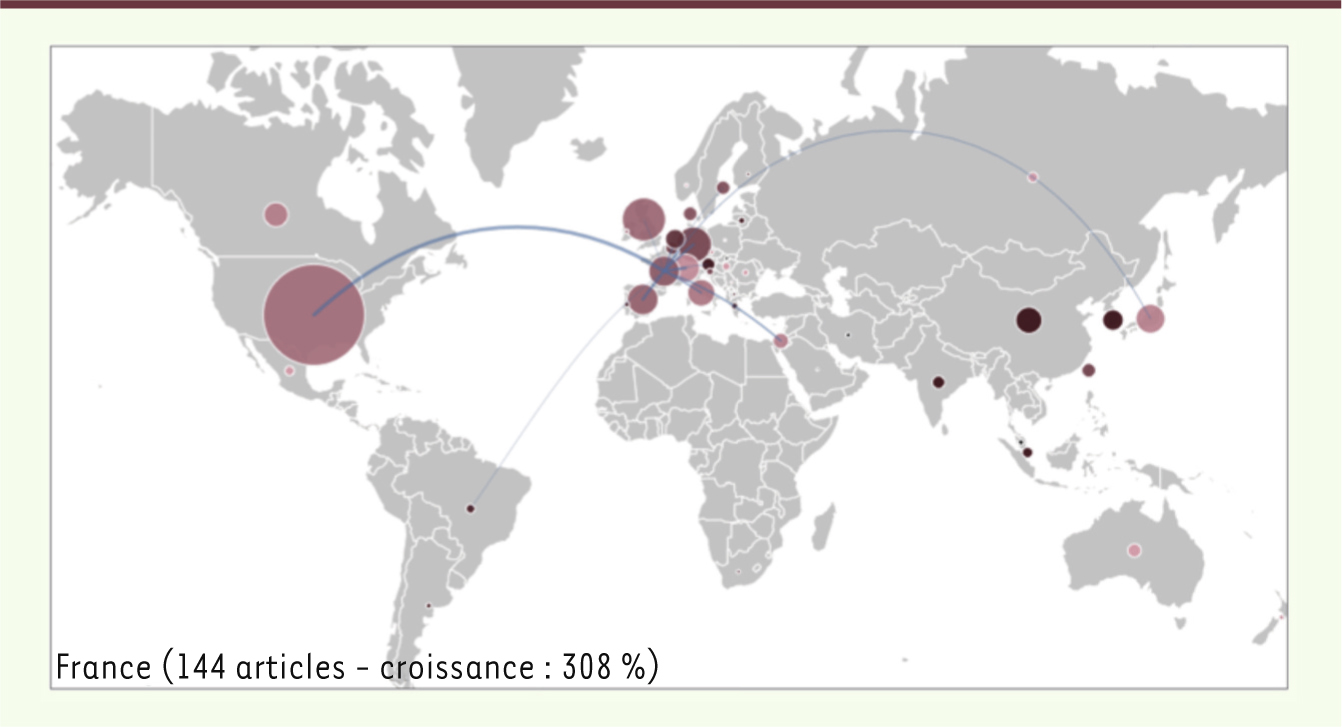
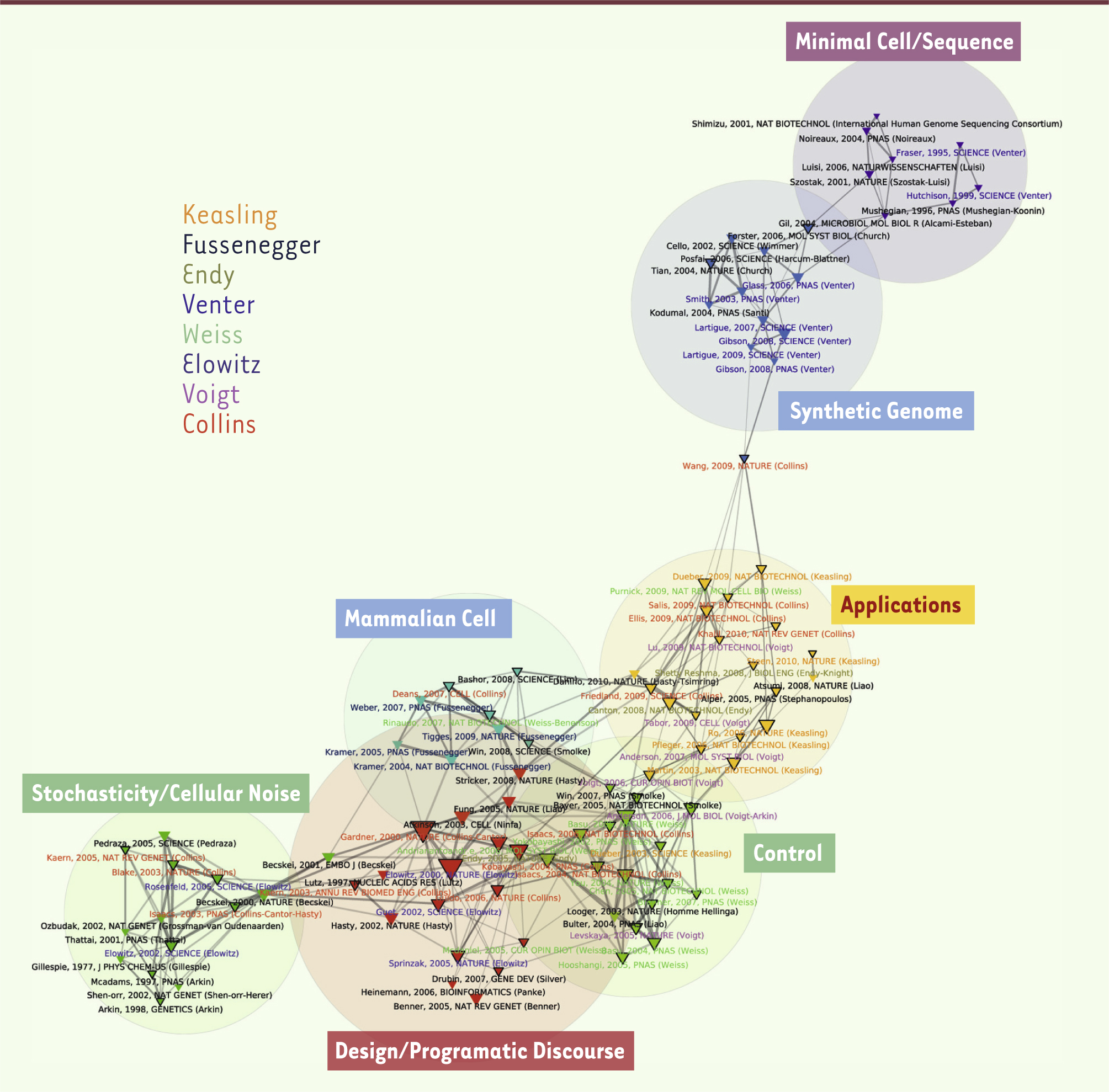
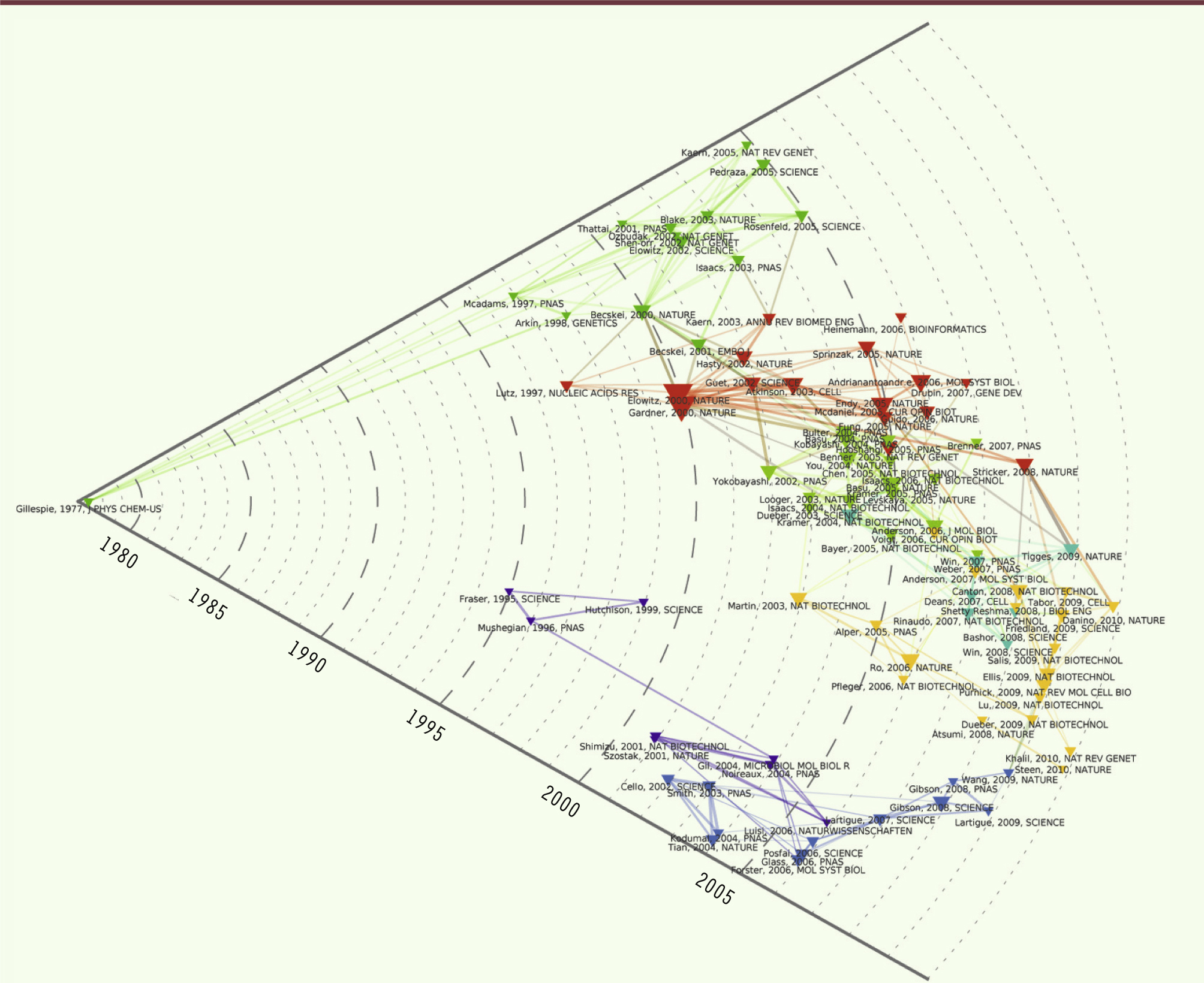
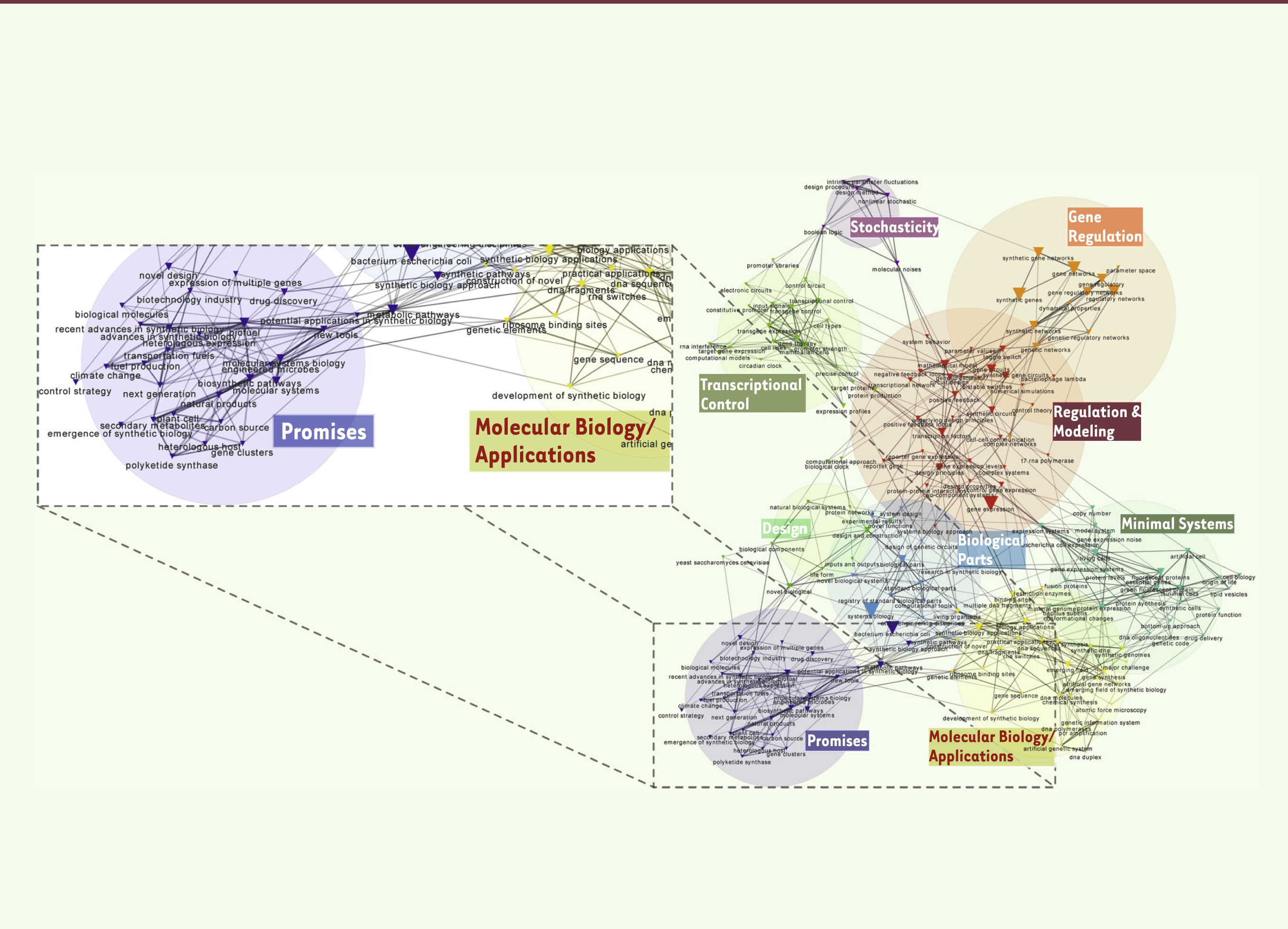
Paul de Brem Je propose à Pierre Monsan et Jacques Haiech de nous rejoindre pour cette discussion. Ces travaux de scientométrie sont fascinants. Ils montrent que sur les 100 articles les plus cités, 60 sont liés directement ou indirectement à Drew Endy, et qu’il existe également un véritable pôle autour de Craig Venter.
Pierre Monsan Je suis fasciné par la puissance de cette analyse. Je serai tout à fait preneur de cet article lorsqu’il sera publié.
Morgan Meyer Vous avez évoqué l’apparition de frontières. Pour Thomas Gieryn
2, la frontière renvoie à l’expulsion, à l’expansion et à la protection de l’autonomie. Vous montrez plutôt l’apparition de nouvelles frontières. Mais quelle est la matérialité de ces frontières ?
Ma deuxième question renvoie aux propos de Thomas Heams ce matin sur la notion d’émergence. Vos cartes sont explicites, mais je m’interroge sur le contexte politique, économique, financier et culturel qui fait émerger une science. Cette notion d’émergence peut également être critiquée car il n’est pas toujours facile de la caractériser.
Pierre-Benoît Joly Notre référence aux travaux de Thomas Gieryn est assez indirecte et nous nous inspirons principalement de son concept de
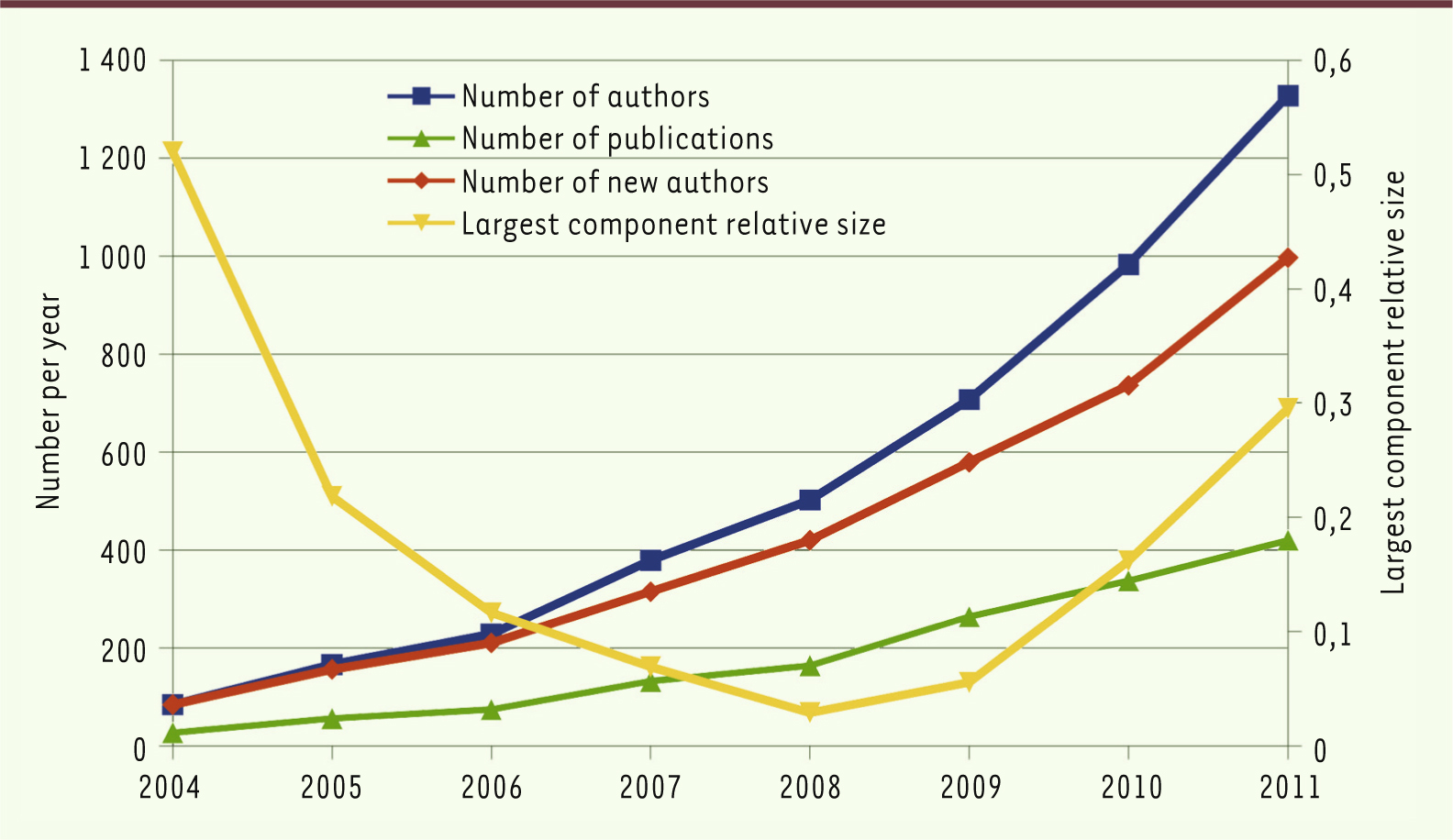
boundary work. L’idée est que les frontières entre les disciplines ne sont pas des données, mais qu’elles doivent être expliquées, particulièrement dans le cadre d’un processus d’émergence. La théorie que nous développons dans l’article concerne la qualité d’une frontière dans le contexte de l’émergence, car même en fin de période, il apparaît 75 % de nouveaux auteurs chaque année. La constitution d’un nouveau champ se fait par attraction d’auteurs qui s’intéressent à un domaine. Mais le champ doit également posséder une consistance, laquelle passe par l’élaboration de normes permettant de distinguer ce qui relève de la biologie de synthèse et ce qui lui est étranger. Il nous a semblé intéressant de pointer ce caractère contradictoire des frontières qui doivent être à la fois perméables et bien marquées. Cela passe par des définitions scientifiques, mais aussi par des normes de production dans lesquelles les membres de la communauté se retrouvent. Les entrepreneurs institutionnels jouent à ce titre un rôle très important.
Concernant la seconde question, nous n’avons pas mené d’analyse systématique du lien entre la publication scientifique et les instruments institutionnels, les programmes qui soutiennent les recherches, ou bien du lien avec les utilisateurs potentiels, notamment avec les industriels. Il est possible d’approfondir le sujet avec les outils scientométriques, mais il s’agit de techniques particulières que nous n’avons pas encore utilisées et qui s’inscrivent dans le prolongement de nos travaux.
Pierre Monsan Assiste-t-on réellement à l’émergence d’une nouvelle science ou bien constate-t-on l’arrivée à maturité d’une science qui débouche sur une technologie ?
Jacques Haiech Je sais ce qu’est une discipline mais j’ignore ce qu’est une science. La science se définit selon moi par une question générale qui est « comment ça marche ? ». La discipline se définit par la création d’un club, l’apparition d’un réseau. Il s’agit d’un réseau hiérarchique, sans échelle, semblable aux réseaux sociaux, avec des personnalités plus connectées que d’autres.
De nouvelles façons d’analyser le vivant apparaissent. Je ne suis pas certain qu’elles soient issues simplement d’entrepreneurs institutionnels. Si l’on avait posé la même question à l’époque d’Apple 2 contre IBM, et si l’on s’était fondé sur les seules publications, Steve Jobs aurait été totalement invisible. Dans le domaine de la biologie de synthèse, le DIY bio, iGEM, et un certain nombre de philosophies open source sont invisibles au niveau des publications. Il serait intéressant d’utiliser l’outil scientométrique à partir des rapports iGEM pour déterminer si ce n’est pas à ce niveau qu’apparaît l’émergence ou s’il existe des idées différentes de celles des entrepreneurs institutionnels.
Benjamin Raimbault Le concours iGEM donne lieu à de la biologie de synthèse, et je rappelle que son fondateur est Drew Endy. En outre, DIY bio se revendique de l’open source tout comme Drew Endy le revendique également. Dans les publications scientifiques, on retrouve cette littérature grise qui naît en marge des institutions. Il existe certainement des biais dans la constitution du corpus, mais l’influence de l’iGEM est bel est bien présente.
Pierre-Benoît Joly Nous nous basons sur la littérature concernant l’émergence des champs scientifiques. La question est moins de savoir s’il s’agit d’une nouvelle science que d’un nouveau champ. La différence porte sur des éléments qui permettent de distinguer un nouveau champ. Ces éléments relèvent effectivement de la densité des relations entre chercheurs, mais également des structurations de la communauté du champ (écoles, revues, associations spécialisées, etc.). Un article de Mullins sur la constitution de la biologie moléculaire a montré en 1972 [
1] comment, à partir d’un certain stade, on pouvait parler d’un champ possédant une relative autonomie.
De la salle Ma question est méthodologique et concerne l’apparition de nouveaux auteurs, qui contraste avec le nombre relativement faible de PI (principal investigator) restant dans le champ. S’agit-il d’un artéfact du fonctionnement des laboratoires ? Ne peut-on pas le contrôler par comparaison avec un champ qui ne serait pas caractérisé comme en émergence ?
Jean-Philippe Cointet Nous nous sommes posé la même question. Nous avons effectivement fait la comparaison. Nous avons calculé exactement les mêmes statistiques − taux de nouveaux entrants, taille du cœur structurel dans lequel l’ensemble des auteurs sont connectés les uns aux autres - sur un corpus définissant la communauté des biologistes du développement spécialisés dans le poissson zébré, un animal modèle, et nous avons constaté qu’à taux de maturité égal, c’est-à-dire à nombre de publications égal, le taux de nouveaux entrants était de 60 % chez les biologistes du développement, contre 75 % pour la biologie de synthèse. Actuellement, la part de chercheurs appartenant à la plus grande composante connexe dans la communauté du poisson zébré dépasse 80 %, nombre à mettre en perspective avec la communauté de la biologie de synthèse encore en cours de maturation.
Jean Weissenbach Le génie génétique est apparu il y a 30 ou 40 ans. Le phénomène était à peu près similaire : il tournait autour de l’ingénierie. Il a donné lieu aux mêmes discussions sur l’apparition d’un nouveau domaine. Or finalement, le génie génétique n’est jamais devenu un champ. Il s’agit d’une technologie appliquée à la fois à des projets d’intérêt fondamental en tant qu’outil d’exploration, et à un certain nombre d’applications. Il serait peut-être intéressant d’établir une comparaison avec les premières expériences de recombinaison in vitro dans les années 1970.
Pierre-Benoît Joly La biologie synthétique est-elle un champ scientifique ou bien relève-t-elle plutôt de la technologie et des outils ? Nous n’avons pas les moyens de répondre à cette question, mais il est clair qu’il s’agit d’une hypothèse à prendre en compte. Un champ se définit autour d’un certain nombre de questions fondamentales en biologie. Dans le même temps, l’applicatif joue un rôle très important dans la structuration du champ. Nous avons tendance à penser que ce champ se caractérise par son caractère hybride, portant à la fois sur des questions fondamentales, y compris l’origine de la vie, le génome minimal, etc., et sur des aspects applicatifs. J’ignore si nous pouvons trancher cette question à notre niveau. Certains chercheurs identifiés par notre analyse relèvent sans aucun doute de la recherche fondamentale.
Jean Weissenbach Je ne veux pas mettre en avant l’opposition entre fondamental et appliqué mais l’analogie avec l’émergence du génie génétique, qui n’est pas devenu une discipline.
Jacques Haiech Il se peut que le génie génétique ne soit pas devenu une discipline parce qu’il ne s’est pas figé. Si l’on définit un champ comme un domaine prenant une place dans l’histoire de la science et continuant à se reproduire, le génie génétique ne correspond pas à cette définition. Mais sans le génie génétique, il n’y aurait pas eu les « -omiques » voire la biologie de synthèse. Il faudrait définir quelque chose relevant davantage d’un flux que de la création d’un « lac ».
Pierre Monsan Ne s’agit-il pas justement de la convergence de nombreux ruisseaux qui se rejoignent pour former un fleuve ?
Morgan Meyer On peut tout de même parler de discipline dans la mesure où elle donne lieu à de nombreux Masters, à des revues contenant « synthetic biology » dans leur titre, et il existe une conférence sur la biologie de synthèse depuis 2004. La biologie de synthèse est en cours d’institutionnalisation et on peut parler d’un champ en cours de création.
Paul de Brem Vous m’avez indiqué que certains résultats de vos travaux étaient en contradiction avec des résultats préalablement publiés.
Pierre-Benoît Joly J’ai effectivement indiqué qu’en partant à peu près des mêmes données, nous observions des phénomènes que les auteurs qui avaient travaillé sur ces cartes n’avaient pas du tout perçus, parce que nous disposons d’algorithmes qu’ils n’ont pas. En dépit d’un champ très fluide, avec un grand nombre de nouveaux entrants et une très forte hétérogénéité, les analyses de dépendance montrent qu’il existe en réalité deux pôles extrêmement importants dans la structuration du champ : le pôle Craig Venter et le pôle constitué autour de Drew Endy, fonctionnant sur des bases assez différentes. Ce résultat est relativement robuste. Un article récent montre très clairement qu’il existe deux façons de penser la propriété intellectuelle en matière de biologie de synthèse, l’une assez traditionnelle qui est celle de Craig Venter, et l’autre plutôt basée sur la métaphore open source, qui est celle de Drew Endy, avec un fonctionnement communautaire et une structuration très différente. Ce caractère quelque peu bipolaire est original.
Paul de Brem Pouvez-vous rappeler en quoi consiste la plate-forme CorText ?
Jean-Philippe Cointet CorText est une plate-forme de l’IFRIS, à la fois infrastructure technique, collectif de chercheurs en linguistique, sciences des systèmes complexes et modélisation et d’ingénieurs web. Son objectif consiste à développer des outils et des méthodologies d’analyse de grands corpus textuels, qui ne se limitent pas nécessairement à des corpus scientométriques. L’enjeu est d’accompagner des projets en sciences sociales au sein de l’IFRIS pour produire des connaissances sur les dynamiques sociosémantiques à partir de grands corpus de données définis par des chercheurs de l’IFRIS. L’ambition consiste à mêler cette approche méthodologique provenant des sciences dures à des questionnements venus, eux, des sciences sociales. C’est ce qui constitue l’originalité de la démarche.