| |
| Med Sci (Paris). 2014 January; 30(1): 12–15. Published online 2014 January 24. doi: 10.1051/medsci/20143001003.Contrôle de la mitophagie par les microARN Une étape clé de l’érythropoïèse Isabelle Barde,1 Benjamin Rauwel,1 Ray Marcel Marin-Florez,2 Andrea Corsinotti,1 Elisa Laurenti,1,3 Sonia Verp,1 Sandra Offner,1 Julien Marquis,1 Adamandia Kapopoulou,1 Jiri Vanicek,2 and Didier Trono1* 1School of Life Sciences and Frontiers in Genetics Program 2School of Basic Sciences, École Polytechnique Fédérale de Lausanne (EPFL), 1015Lausanne, Suisse 3Adresse actuelle : Campbell Family Institute for Cancer Research, Ontario Cancer Institute, Princess Margaret Cancer Centre, University Health Network and Department of Molecular Genetics, University of Toronto, Toronto, Ontario, Canada MeSH keywords: Animaux, Protéines de transport, physiologie, Différenciation cellulaire, génétique, Érythropoïèse, Humains, Souris, Souris knockout, microARN, Dégradation des mitochondries, Protéines nucléaires, Protéines de répression, Transduction du signal |
Un adulte en bonne santé produit jusqu’à cent milliards de nouveaux globules rouges chaque jour, afin d’en maintenir les stocks sanguins. Le globule rouge, ou érythrocyte, contient l’hémoglobine responsable de transporter l’oxygène des poumons vers l’ensemble du corps. L’érythrocyte a son origine dans la moelle osseuse, dérivant de la cellule souche hématopoïétique via un processus complexe de prolifération et de différenciation [
1,
2]. Il reste encore beaucoup à apprendre sur les mécanismes moléculaires de ces événements, non seulement pour comprendre la cause de certains troubles hématologiques, mais aussi pour envisager un jour la production de globules rouges en laboratoire – un enjeu important pour la transfusion. |
Les génomes des vertébrés supérieurs codent pour des centaines de protéines à doigt de zinc dotées d’un domaine KRAB (KRAB-ZFP). Ces protéines reconnaissent des séquences spécifiques de l’ADN via leurs doigts de zinc, et y recrutent leur cofacteur universel KAP1 via le motif KRAB (Krueppel-associated box). KAP1, également connu sous les noms de TRIM28 (protéine à motif tripartite 28), TIF1β (facteur de transcription intermédiaire 1β), ou Krip-1 (KRAB-interacting protein 1), agit ensuite comme une plate-forme pour un complexe multimoléculaire qui réprime la transcription par l’induction d’hétérochromatine [
3,
4]. Le système KRAB/KAP1 a probablement d’abord évolué pour minimiser les perturbations du génome induites par l’invasion des rétroéléments [
5], mais des données récentes indiquent qu’il contrôle également de multiples aspects de la physiologie des mammifères [
6,
7]. |
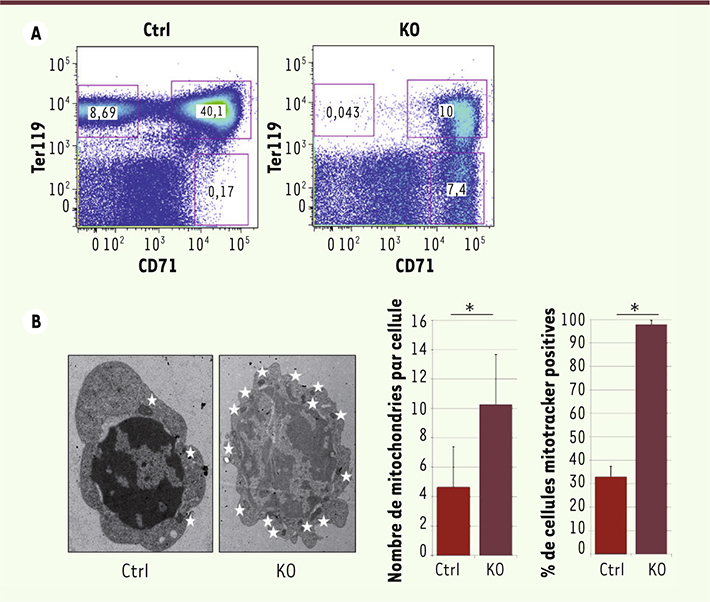
Défaut de mitophagie en l’absence de KAP1 Afin d’étudier le rôle potentiel de ce système dans l’hématopoïèse, nous avons construit une souris adulte dont le gène codant pour KAP1 est invalidé sélectivement (knock-out, KO) dans les lignées hématopoïétiques. Le phénotype de ces souris se caractérise par une série d’anomalies hématologiques dont la plus remarquable est une anémie due à un arrêt de différenciation des précurseurs des érythrocytes - les érythroblastes - au stade où ils expriment fortement les marqueurs de surface CD71 (le récepteur de la transferrine) et Ter119 (marqueur associé à la glycophorine A chez la souris) (Figure 1A).
 | Figure 1.
Phénotype des érythroblastes KAP1 KO : arrêt de maturation et accumulation des mitochondries. A. Analyse par FACS (fluorescence-activated cell sorting) des cellules exprimant les marqueurs de surface CD71 et Ter119, issues de la moelle osseuse des souris contrôles (Ctrl) ou de souris dont le gène KAP1 a été invalidé dans les lignées hématopoïétiques (KO). Le pourcentage de chaque population dans la moelle totale est indiqué sur le graphique. B. Microscopie électronique des cellules CD71+Ter119+ contrôles et KAP1 KO. Panneau de gauche : les étoiles indiquent les mitochondries observées ; panneau du milieu : nombre moyen de mitochondries mesuré par cellule (n = 10, *p < 0,05) ; panneau de droite : résultats obtenus avec la coloration MitoTracker (n = 4, *p < 0,05). |
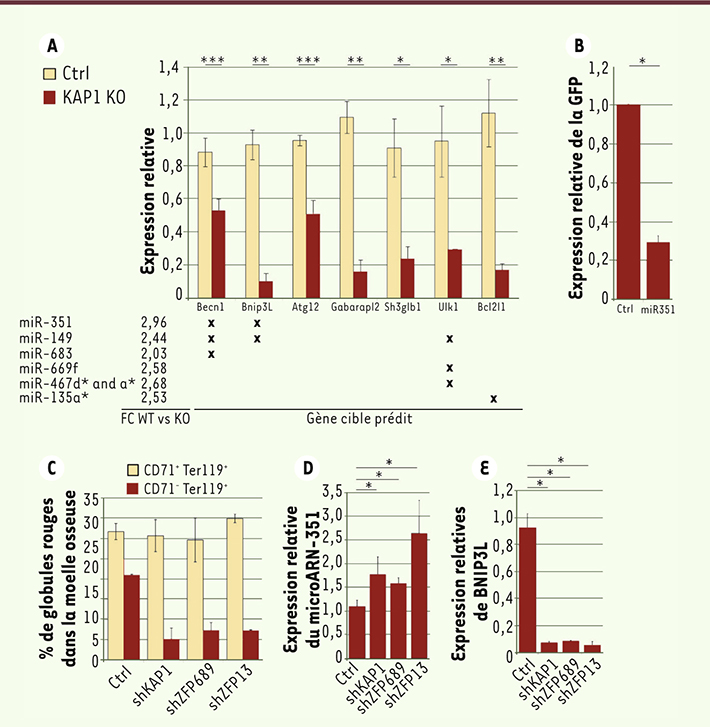
L’examen de ces érythroblastes dépourvus de KAP1 en microscopie électronique et par une technique de coloration spécifique (MitoTracker) a révélé qu’ils contenaient plus de mitochondries que les contrôles (Figure 1B). En corollaire de cette observation, l’analyse de l’expression des gènes a montré dans ces cellules une diminution des transcrits (comme ceux de BNIP3L, ULK1 [unc-51 like autophagy activating kinase 1], Gabarapl2 [GABA(A) receptor-associated protein-like 2], Sh3glb1 [SH3-domain GRB2-like endophilin B1], Atg12 [autophagy related 12], Becn1 [beclin 1] et BCL2L1) impliqués dans la mitophagie, mécanisme d’élimination des mitochondries (Figure 2A). Cette dernière représente une étape importante dans le processus de différenciation des globules rouges.
 | Figure 2.
Une cascade impliquant des KRAB-ZFP spécifiques des érythroblastes, le microARN-351 et le gène BNIP3L contrôle la mitophagie dans les globules rouges. A. Expression des gènes impliqués dans la mitophagie dans les érythroblastes contrôles (Ctrl) et KAP1 KO (n = 4, *p < 0,05, **p < 0,01, ***p < 0,001). En dessous du graphique est indiquée l’expression des microARN dans les mêmes échantillons, la croix (X) désignant leurs cibles potentielles prédites. B. Le microARN351 cible directement la région terminale non codante du gène BNIP3L. C. Analyse par FACS des globules rouges de la moelle osseuse de souris greffées avec des cellules souches hématopoïétiques modifiées génétiquement par un vecteur lentiviral permettant l’extinction de l’expression de ZFP689 ou 13. Le contrôle (Ctrl) correspond à l’utilisation d’un vecteur vide ou ne ciblant aucun gène. Pour chaque condition, l’expression du microARN351 (D) et du gène BNIP3L
(E) dans les cellules CD71+Ter119+ a été mesurée (n = 6, *p < 0,05). |
|
Comment l’absence de KAP1 entraîne-t-elle un défaut de mitophagie ? KAP1 régule négativement plusieurs microARN contrôlant un gène clé de la mitophagie dans les érythroblastes Cependant, comme le système KRAB/KAP1 est surtout connu pour induire la répression de la transcription [
8], il semblait peu probable que la diminution de l’expression de ces gènes de la mitophagie puisse être due à un effet direct du KO de KAP1. L’analyse des petits ARN a révélé une augmentation de 11 des 455 microARN testés dans les érythroblastes KAP1-KO. Les microARN sont des régulateurs post-transcriptionnels capables d’atténuer l’expression d’un gène. Grâce à un système de prédiction informatique récemment développé au sein d’un laboratoire de l’EPFL [
9], nous avons pu établir une relation entre l’augmentation de l’expression de certains microARN et la diminution de transcrits spécifiques de la mitophagie dans les érythroblastes KAP1 KO. En particulier, une des prédictions concernait le microARN-351, susceptible d’agir sur la région terminale non codante du gène BNIP3L ( BCL2/adenovirus E1B 19kDa interacting protein 3-like), gène clé de la mitophagie dans les érythrocytes. Or, l’analyse de l’expression naturelle de ce microARN-351 a révélé qu’elle était diminuée spécifiquement dans les érythroblastes CD71 +Ter119 + tandis que l’expression de sa cible, BNIP3L, elle, augmentait au cours de la différenciation des globules rouges. La preuve de l’interaction directe entre miARN-351 et BNIP3L a été obtenue en surexprimant dans la lignée cellulaire MEL (une lignée établie de cellules murines issues d’une érythroleucémie) microARN351 et sa séquence cible insérée dans la région terminale non codante du gène codant pour la GFP ( green fluorescent protein). La surexpression du microARN a induit dans les cellules MEL l’extinction spécifique de l’expression de la eGFP qui portait la cible prédite (Figure 2B). KAP1 contrôle les miARN par l’intermédiaire de protéines KRAB Deux autres microARN (503 et 322), proches du microARN-351 et formant avec ce dernier un petit cluster sur le chromosome X, étaient également exprimés plus fortement dans les érythroblastes KAP1 KO. Or, un site de liaison de KAP1 a été mis en évidence par immunoprécipitation de la chromatine couplée à un séquençage d’ADN à moins de 4kb de ce groupe de gènes codant pour les trois microARN, suggérant fortement une possible régulation de ce locus génomique par le système KRAB/KAP1. Comme KAP1 n’est pas une protéine de liaison directe à l’ADN, nous avons cherché à identifier les protéines KRAB-ZFP responsables de son recrutement dans cette région. Neuf protéines KRAB-ZFP ont été identifiées qui sont exprimées exclusivement dans les érythroblastes et ont un orthologue humain. Deux d’entre elles, ZFP689 et ZFP13, sont exprimées spécifiquement à deux étapes de différenciation successives de l’érythropoïèse (CD71 +Ter119 + et CD71 +Ter119 -, respectivement), et se sont révélées être – dans le système cellulaire des MEL - des régulateurs potentiels de l’expression du microARN351 et du gène BNIP3L. Nous avons ensuite validé la fonction de ces deux candidats in vivo dans un modèle animal permettant d’éteindre leur expression dans le système hématopoïétique : des cellules hématopoïétiques ont été modifiées génétiquement par un vecteur lentiviral permettant l’expression d’un petit ARN interférent (shARN) ciblant spécifiquement ZFP689 ou 13, puis greffées à un animal receveur. L’analyse de la lignée érythroblastique appauvrie en ZFP689 ou 13 a révélé une diminution du nombre de cellules CD71 +Ter119 + de même ampleur que celle qu’entraîne la diminution de l’expression de KAP1 (Figure 2C). De plus, les analyses moléculaires ont confirmé l’expression accrue du microARN351 et celle diminuée de BNIP3L dans les cellules appauvries en ZFP689 ou 13, un profil identique à celui des cellules KAP1 KO (Figure 2D–E). Finalement, dans une dernière série d’expériences, nous avons pu montrer que la neutralisation du système KAP1 avait un effet similaire sur des globules rouges humains, l’orthologue du microARN351 semblant être le microARN-125a-5p. |
Différenciation érythroblastique : une subtile combinaison de protéines et ARN Ces résultats révèlent l’existence d’un système de régulation de la transcription où protéines et ARN se superposent en une combinatoire subtile pour que soit déclenchée au moment opportun une étape clé de l’érythropoïèse. Dans ce processus, plusieurs protéines KRAB-ZFP sont impliquées dans le contrôle de plusieurs microARN, eux-mêmes ayant pour cibles plusieurs transcrits de gènes associés avec la mitophagie ou d’autres événements intervenant lors de la maturation érythrocytaire. Cette apparente redondance - ou plutôt addition - d’effets parallèles ciblant le même processus physiologique a déjà été relevée dans le domaine de l’interférence ARN [
10]. La découverte que ce dernier puisse lui-même être modulé par le système des KRAB-ZFP, qui agissent également de façon multifactorielle, ajoute un remarquable degré de modularité et de robustesse à ce type de régulation. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
L'auteur remercie Franck Aguila pour ses conseils artistiques sur les figures, Thomas Lorivel et Joëlle Chabry pour leur relecture attentive, Audrey Recouly, Fabienne Chevallier et Florence Servent de la Société CISBIO Bioassays, Codolet, France pour les expériences en FRET utilisant la technologie Tag-lite et Richard Miller (Chicago) pour nous avoir fourni le clone de CXCR4.
|
1.
Hattangadi
SM
,
Wong
P
,
Zhang
L
,
Flygare
J
,
Lodish
HF
. From stem cell to red cell: regulation of erythropoiesis at multiple levels by multiple proteins, RNAs, and chromatin modifications . Blood.
2011; ; 118 : :6258.–6268. 2.
Xu
J
,
Shao
Z
,
Glass
K
, et al.
Combinatorial assembly of developmental stage-specific enhancers controls gene expression programs during human erythropoiesis . Dev Cell.
2012; ; 23 : :796.–811. 3.
Nielsen
AL
,
Ortiz
JA
,
You
J
, et al.
Interaction with members of the heterochromatin protein 1 (HP1) family and histone deacetylation are differentially involved in transcriptional silencing by members of the TIF1 family . EMBO J.
1999; ; 18 : :6385.–6395. 4.
Schultz
DC
,
Ayyanathan
K
,
Negorev
D
,
Maul
GG
,
Rauscher
FJ
3rd
. SETDB1: a novel KAP-1-associated histone H3, lysine 9-specific methyltransferase that contributes to HP1-mediated silencing of euchromatic genes by KRAB zinc-finger proteins . Genes Dev.
2002; ; 16 : :919.–932. 5.
Rowe
HM
,
Jakobsson
J
,
Mesnard
D
, et al.
KAP1 controls endogenous retroviruses in embryonic stem cells . Nature.
2010; ; 463 : :237.–240. 6.
Jakobsson
J
,
Cordero
MI
,
Bisaz
R
, et al.
KAP1-mediated epigenetic repression in the forebrain modulates behavioral vulnerability to stress . Neuron.
2008; ; 60 : :818.–831. 7.
Santoni de Sio
FR
,
Massacand
J
,
Barde
I
, et al.
KAP1 regulates gene networks controlling mouse B-lymphoid cell differentiation and function . Blood.
2012; ; 119 : :4675.–4685. 8.
Schultz
DC
,
Friedman
JR
,
Rauscher
FJ
3rd
. Targeting histone deacetylase complexes via KRAB-zinc finger proteins: the PHD and bromodomains of KAP-1 form a cooperative unit that recruits a novel isoform of the Mi-2alpha subunit of NuRD . Genes Dev.
2001; ; 15 : :428.–443. 9.
Marin
RM
,
Vanicek
J
. Efficient use of accessibility in microRNA target prediction . Nucleic Acids Res.
2011; ; 39 : :19.–29. 10.
Ebert
MS
,
Sharp
PA
. Roles for microRNAs in conferring robustness to biological processes . Cell.
2012; ; 149 : :515.–524. |