| |
| Med Sci (Paris). 2014 January; 30(1): 55–63. Published online 2014 January 24. doi: 10.1051/medsci/20143001014.Les remaniements chromosomiques complexes Un paradigme pour l’étude de l’instabilité chromosomique Vincent Gatinois,1 Jacques Puechberty,1 Geneviève Lefort,1 David Geneviève,1 and Franck Pellestor1* 1Laboratoire de génétique chromosomique, hôpital Arnaud de Villeneuve, CHRU de Montpellier, 371, avenue du doyen Gaston Giraud, 34295Montpellier, France |
Vignette (Photo © Franck Pellestor). Le terme de remaniement chromosomique désigne les anomalies de la structure des chromosomes, qu’elles soient équilibrées ou non : translocations, délétions, duplications, inversions, insertions, anneaux, etc. Pour caractériser ces anomalies, on utilise la notion de point de cassure. Ainsi définit-on un réarrangement chromosomique comme simple lorsqu’il ne fait intervenir que deux points de cassure, ou complexe (CCR pour complex chromosomal rearrangements) s’il nécessite trois points de cassure au minimum pour se former. Il en existe une extrême variété. Toutefois, en dehors du contexte onco-hématologique, les CCR sont rares en cytogénétique constitutionnelle : moins de 270 cas ont été rapportés. Dans leur grande majorité, ces réarrangements concernent des combinaisons de plusieurs translocations [
1]. |
Classification des remaniements chromosomiques complexes Élaborer une classification des CCR représente une vraie difficulté compte tenu de leur hétérogénéité. En 1982, Kleczkowska et al. [
2] en ont proposé une première classification basée sur leur mode de transmission, en distinguant les CCR hérités de ceux formés de novo. Cette première classification a révélé une nette prévalence de l’origine maternelle des CCR hérités. En 1987, Kousseff et al. [
3] proposèrent de classer les CCR en fonction du nombre de points de cassure. Cette classification met ainsi en évidence le fait que les anomalies transmises au travers des générations sont de plus faible complexité (avec moins de cinq points de cassure) que celles qui apparaissent de novo et qui font intervenir un nombre plus élevé de points de cassure. Mais, la classification la plus pertinente reste la classification structurale proposée par Kausch et al. [
4] en 1988. Elle distingue trois catégories de CCR (Figure 1) :
-
les remaniements appelés three-way, les plus fréquents, mais aussi les moins complexes : trois points de cassure sont identifiés, avec échange des segments chromosomiques distaux ;
-
les CCR exceptionnels, pour lesquels plus d’un point de cassure par chromosome est identifiable. Le degré de complexité de ce type de CCR n’a pas de limite. Ainsi, un caryotype à 15 points de cassure a été rapporté par Houge et al. [
5] ;
-
enfin, les remaniements doubles ou triples two-way, assez rares, qui correspondent à la coexistence chez un même individu de deux ou trois translocations indépendantes impliquant des chromosomes différents.
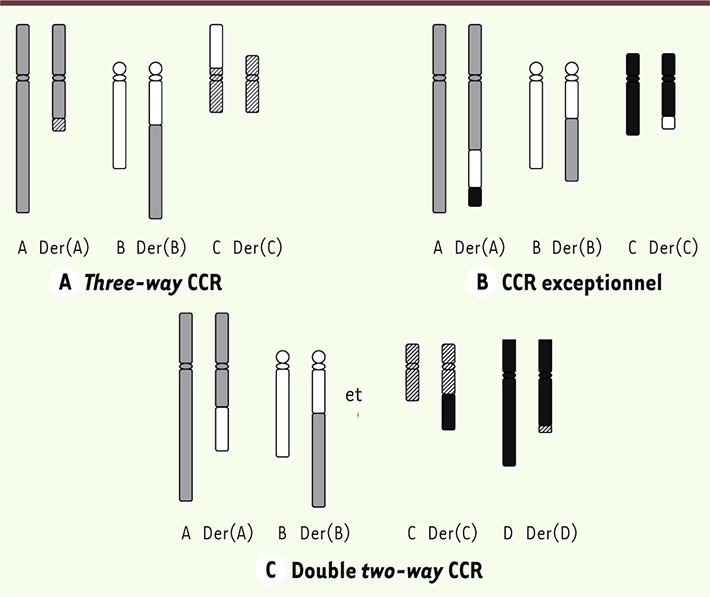
 | Figure 1.
Classification des CCR. A.
Three-way CCR : un point de cassure sur chacun des chromosomes A, B et C, donnant naissance à trois dérivés der(A), der(B) et der(C). B. CCR exceptionnel : plus de points de cassure que de chromosomes impliqués et aucune limite de complexité dans la formation des dérivés. C. Double two-way CCR : anomalies concomitantes mais indépendantes (d’après [ 1]). |
En 1997, Madan et al. [
6] ont étoffé cette classification structurale en y intégrant la présence additionnelle d’inversions, d’insertions ou de délétions. Cela a permis d’intégrer les translocations dites insertionnelles à la famille des CCR. En 2011, une analyse de 103 nouveaux cas de CCR équilibrés a conduit Madan et al. [
7] à proposer une ultime variante de cette classification structurale, distinguant quatre types de CCR en fonction du nombre de points de cassure et du mode de ségrégation. Cette difficulté à classifier les CCR traduit bien la complexité et la grande variabilité de ce type de remaniements. |
Aspects épidémiologiques et héritabilité des CCR La rareté de tels évènements chromosomiques rend difficile leur analyse épidémiologique. Concernant la présence ou non de phénotypes pathologiques chez les porteurs de CCR, les observations rapportées ont permis de constater que :
-
chez 70 % des sujets présentant un CCR, le phénotype est normal. L’examen chromosomique de ces sujets est généralement réalisé en raison d’antécédents de fausses-couches à répétition, d’une infertilité ou de l’enquête diagnostique déclenchée par l’observation d’un phénotype pathologique dans la descendance. Le caryotype de ces personnes est considéré comme équilibré, mais il faut garder à l’esprit que la présence de petits déséquilibres aux points de cassure (délétion/duplication/insertion) peut ne pas engendrer de phénotypes pathologiques ;
-
chez 20 à 25 % des patients porteurs de CCR, des atteintes congénitales sont présentes : syndrome polymalformatif et/ou retard mental. Leur génome est le plus souvent déséquilibré, même si le caryotype standard peut être apparemment équilibré. Quelques rares cas sont liés à des troubles psychiatriques ;
-
dans 5 à 10 % des cas, le diagnostic est porté en période anténatale.
Trente pour cent des cas de CCR sont familiaux et la transmission de l’anomalie est majoritairement d’origine maternelle. Une origine paternelle n’a été observée que dans quelques cas [
8]. Toutefois, la majorité des CCR, soit 70 %, surviennent de novo et l’origine de ces anomalies de novo serait préférentiellement paternelle. Leur découverte se fait généralement lors de l’exploration de troubles de la reproduction, bien que des cas exceptionnels de sujets fertiles porteurs de CCR aient été rapportés [
9]. Il est intéressant de souligner que les CCR survenant de novo se répartissent équitablement entre les sujets exprimant un phénotype pathologique (51 %) et les sujets non atteints (49 %). |
Évolution des techniques d’analyse des CCR Bien que l’analyse chromosomique classique, basée sur la réalisation du caryotype, ait permis d’identifier un grand nombre de CCR, cette approche reste limitée par un faible niveau de résolution (de 5 à 10 Mb). Fort heureusement, l’analyse chromosomique bénéficie aujourd’hui d’un large panel de techniques issues de la biologie moléculaire. En premier lieu, l’hybridation fluorescente in situ (FISH pour fluorescence in situ hybridization) a été déclinée par de multiples techniques - peinture chromosomique, FISH multicouleur, FISH télomérique ou locus spécifique, ou encore CGH (hybridation génomique comparative) -, et elle a contribué au diagnostic d’un nombre croissant de CCR [1]. Ces approches ont aussi permis aux cytogénéticiens de prendre la pleine mesure de la complexité de nombreux CCR, en révélant des microremaniements que le caryotype ne détectait pas. Un exemple flagrant concerne les translocations insertionnelles : leur incidence était estimée à 1/80 000 naissances en 2000, mais l’utilisation de la CGH a permis de la réévaluer à 1/500 en 2010, soit une proportion 160 fois plus élevée [
10]. Plus récemment, les techniques de puces à ADN (ACPA pour analyse chromosomique sur puces à ADN) et de séquençage à haut débit ont accru la résolution de l’analyse jusqu’au nucléotide, permettant ainsi de mieux décrypter les différents mécanismes de formation des CCR [
11]. Enfin, des stratégies originales ont été développées pour caractériser les points de cassure des CCR. Citons l’array painting basé sur la combinaison du tri chromosomique par cytométrie en flux et de l’ACPA des zones de cassures chromosomiques [
12]. Une conséquence de ce raffinement des techniques d’analyse est la découverte que 30 à 40 % des porteurs de CCR de novo apparemment équilibrés et présentant des anomalies phénotypiques, sont porteurs de microremaniements situés autour des points de cassure de leur CCR [
13]. Ainsi, de nombreux CCR se révèlent être bien plus complexes que ce que les analyses initiales permettaient d’envisager. Cette complexité accrue va de pair avec la découverte des multiples mécanismes de formation des CCR. |
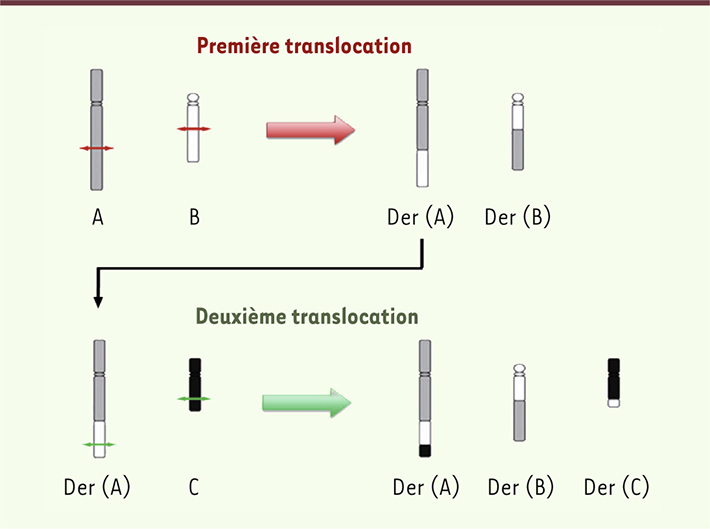
Plusieurs scénarios sont envisageables pour rendre compte de la formation d’un CCR. Le plus simple est de décomposer l’anomalie complexe en évènements chromosomiques simples. Ainsi, un CCR de type exceptionnel peut résulter d’un nombre restreint d’évènements chromosomiques indépendants, avec une première étape de translocation réciproque entre deux chromosomes A et B, donnant naissance à deux chromosomes remaniés appelés dérivés der(A) et der(B). Dans un second temps, une autre translocation réciproque se crée entre un troisième chromosome C et l’un des deux dérivés der(A) ou der(B), pour former les trois dérivés finalement observés dans le CCR : der(A), der(B) et der(C) (Figure 2). De cette manière, il est possible d’expliquer l’origine d’anomalies chromosomiques dont la complexité est a priori très élevée. Un autre scénario possible fait intervenir des translocations dites « sauteuses » dans lesquelles un segment chromosomique est transféré successivement sur plusieurs autres chromosomes hôtes. Mais il s’agit d’évènements rares se produisant au cours de plusieurs cycles cellulaires [
14].
 | Figure 2.
Décomposition théorique d’un CCR exceptionnel en deux étapes.
|
Parallèlement à ces modèles séquentiels, un mécanisme en une seule étape a été proposé : toutes les cassures chromosomiques se produiraient en même temps, soit au cours de la méiose chez l’un des parents, soit au cours des premières divisions cellulaires de l’embryon [
15]. Le fait que la majorité des CCR ait été diagnostiquée de novo, et donc qu’aucun des parents ne présente d’anomalies chromosomiques simples capables de se complexifier à la génération suivante, est en faveur de ce modèle. L’origine paternelle préférentielle des CCR de novo suggère que leur formation a lieu plutôt au cours de la spermatogenèse que de l’ovogenèse. Durant la spermatogenèse, le nombre très important de mitoses qui se succèdent (supérieur au nombre de mitoses survenant au cours de l’ovogenèse), ainsi que le manque d’efficacité des systèmes cellulaires de réparation de l’ADN, peuvent favoriser l’émergence de novo des CCR dans le sexe masculin. L’instabilité chromosomique au cours des premières divisions embryonnaires est un autre paramètre favorable à la formation des CCR [
16]. Là encore, la faiblesse des points de contrôle mitotiques a été mise en cause [
17]. Cette faiblesse pourrait s’expliquer par la rapidité des premières divisions des blastomères liée à la nécessité pour l’embryon de produire une masse cellulaire suffisante. |
Mécanismes moléculaires à l’origine des CCR La formation de réarrangements chromosomiques, qu’ils soient simples ou complexes, nécessite que se produise une cassure double-brin de l’ADN (DSB pour double strand break), suivie d’une réparation erronée des lésions de l’ADN. Les cassures double-brin de l’ADN sont des évènements nucléaires fréquents. Elles peuvent être induites par un certain nombre d’agents exogènes, dit clastogènes, tels que les radiations ionisantes, la chimiothérapie anticancéreuse, le stress oxydant, les infections virales, etc. Des mécanismes endogènes sont aussi à l’origine de DSB, comme l’action inappropriée d’enzymes nucléaires et le stress réplicatif qui se manifeste par la déstabilisation des fourches de réplication face à des obstacles rencontrés dans la structure de l’ADN. Les DSB interviennent aussi dans des mécanismes cellulaires physiologiques, tels que la recombinaison méiotique permettant le brassage de l’information génétique lors de la formation des gamètes [
41]. La réparation des cassures double-brin de l’ADN fait intervenir plusieurs enzymes et protéines complexes organisés en divers systèmes de réparation. La cellule utilisera celui qui est le mieux adapté à la situation. L’engagement enzymatique dans l’une de ces voies inhibe les autres systèmes possibles. Le choix d’un processus est guidé par l’espèce, le type cellulaire, la position chronologique de la cellule dans son cycle de vie et la présence éventuelle d’un fragment d’ADN homologue [
18]. Deux mécanismes principaux de réparation coexistent. Le système de réparation par jonction des extrémités non homologues (NHEJ pour non-homologous end joining) est un mécanisme dit biochimique, dans la mesure où il n’utilise pas et ne dépend pas de l’information génétique pour son action. Il consiste simplement à recoller les extrémités séparées par la cassure (Figure 3). Cette réparation de l’ADN est très rapide et peut survenir à n’importe quel stade du cycle cellulaire. Il s’agit du mode de réparation le plus fréquemment utilisé par la cellule, même si son niveau de fidélité est inférieur à celui des autres mécanismes, car il ne restitue pas intégralement la séquence originale de l’ADN. Les extrémités de la cassure double-brin sont souvent résorbées avant d’être recollées. Aussi, la séquence obtenue diffère souvent de la séquence d’origine. On parle de séquence cicatrice [
19].
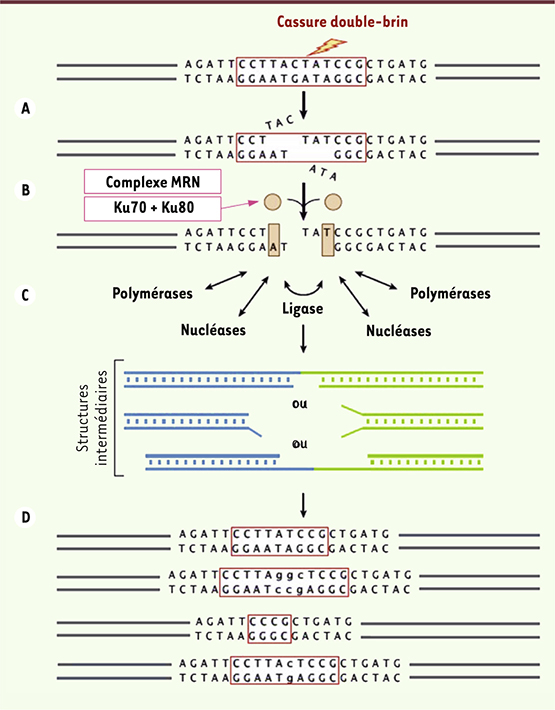
 | Figure 3.
Mécanisme de réparation de type NHJE des cassures double-brin. A. Événement déstabilisant et apparition d’une cassure double-brin. B. Le complexe MRN (Mre11, Rad50 and Nbs1) reconnaît la DSB. Les protéines Ku70 et Ku80 stabilisent la structure et favorisent l’action des enzymes de réparation. C. Intervention des différentes enzymes : nucléases (Artemis et DNA-PKc), polymérases (µ et λ) et ligases (XLF [XRCC4-like factor], XRCC4 [X-ray repair cross-complementing protein 4] et DNA ligase IV). D. Brins d’ADN réparés par NHEJ. Ce type de réparation présente un haut degré d’imprécision et est à l’origine de modifications du génome. |
Le deuxième mécanisme, appelé recombinaison homologue (HR pour homologous recombination) [
42], est un mécanisme dit génétique et conservateur dans la mesure où il restaure la séquence endommagée en utilisant une information similaire provenant généralement du chromosome homologue à celui qui est endommagé (Figure 4). Ce type de réparation ne peut donc être observé qu’après la réplication, au cours des phases S et G2 du cycle cellulaire. L’appareil enzymatique prend modèle sur la séquence indemne de la chromatide sœur pour réparer le brin cassé. Il s’opère une invasion du brin homologue par l’extrémité 3’ du brin à réparer, suivie de la formation de jonctions de Holliday réunissant les deux structures double-brin et permettant la réparation du brin d’ADN cassé. Une des propriétés de ce mode de réparation est sa grande fidélité, puisque l’ADN lésé est remplacé par une copie strictement identique [
20].
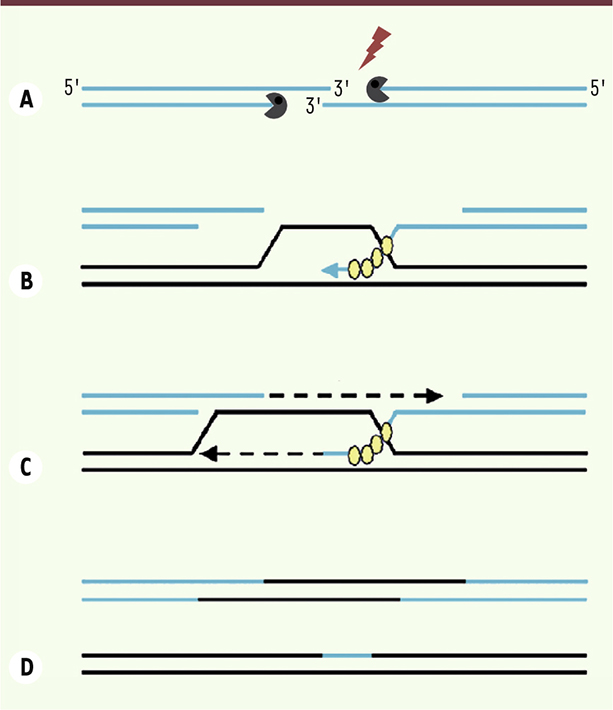
 | Figure 4.
Système de réparation des cassures double-brin par recombinaison homologue. A. Cassure double-brin et résection des extrémités 5’. B. Stabilisation des extrémités 5’ par les protéines RPA (replication protein A) et Rad51 et invasion d’une séquence homologue par le filament d’ADN simple-brin. C. Formation d’un hétéroduplex (appelé D-loop), réparation du brin d’ADN altéré et création d’une structure de Holliday après la ligation des extrémités. D. Démantèlement de la structure avec ou sans crossing-over. |
Un troisième mécanisme de réparation, mis en évidence récemment, se situe à mi-chemin entre les deux premiers types de réparation décrits. Appelé jonction des extrémités orientée par micro-homologie (MMEJ pour micro-homology mediated end joining), il permet de joindre les extrémités libres de l’ADN, à condition que les brins à apparier présentent une micro-homologie de séquence de 5 à 25 pb. Si cet appariement n’est pas suffisamment stable, il se produit une succession de désappariements et de réappariements de l’ADN, entraînant des variations de séquence dans la zone réparée. De ce fait, ce mécanisme peut conduire à une grande complexité des réarrangements [
21]. Les mécanismes de réparation des cassures double-brin de l’ADN ne sont pas infaillibles, et les erreurs commises par ces systèmes enzymatiques sont responsables de l’apparition des remaniements chromosomiques. Il est intéressant de classer les anomalies chromosomiques résultantes en fonction de leur récurrence. On peut alors définir trois classes d’anomalies.
-
Les réarrangements chromosomiques récurrents qui sont généralement dus à des erreurs de réparation de type recombinaison homologue. On parle alors de recombinaison homologue non allélique (NAHR pour non allelic homologous recombination). La séquence homologue prise comme matrice se trouve sur un locus chromosomique différent de celui à réparer. Ceci peut se produire lorsque la cassure double-brin survient au niveau de séquences répétées de l’ADN, appelées duplications segmentaires ou LCR (low copy repeat), ce qui permet d’expliquer la récurrence de certains réarrangements chromosomiques [
22]. Ce type d’erreur peut se produire lors de la mitose ou de la méiose. De nombreuses anomalies récurrentes sont la conséquence de ce type de recombinaison, comme le syndrome de Di George1 qui comporte une micro-délétion dans la région chromosomique 22q11.2.
-
Les réarrangements chromosomiques non récurrents, dont les points de cassure sont aléatoires. Ils sont dus à une erreur de réparation de l’ADN de type NHEJ à la suite de l’apparition d’au moins deux cassures double-brin [
23]. Il est possible de retrouver la signature de ces réarrangements grâce à la cicatrice nucléotidique qu’ils laissent au niveau des points de cassure.
-
Les réarrangements par stress réplicatif.
En raison de leur complexité, certaines aberrations ne peuvent pas être expliquées par les recombinaisons de type NAHR ou NHEJ. Aussi, plusieurs autres modèles de formation, basés sur les erreurs de réplication de l’ADN, ont été proposés.
-
Le processus FoSTeS (fork stalling and template switching) combine un arrêt de la fourche de réplication (le réplisome) avec un changement de brin modèle [
24]. On parle alors de stress réplicatif, que l’origine de ce stress soit exogène ou due à une conformation particulière de l’ADN forçant cet arrêt. Si cet arrêt est trop long, le réplisome finit par se démanteler. La partie 3’ du brin retardé peut alors envahir une autre fourche de réplication et la polymérase continue la synthèse du brin d’origine en prenant modèle sur le brin envahi. Des homologies de séquence de petite taille (4 à 15 pb) sont nécessaires pour que ce nouvel appariement ait lieu. Cet envahissement peut se faire entre deux fourches de réplication séparées par une grande distance génomique, et les réarrangements engendrés peuvent donc être de très grande taille. Ce processus de désengagement et d’envahissement peut se produire plusieurs fois à la suite, d’où son implication dans la genèse des CCR.
-
Le processus MMBIR (microhomology mediated break induced replication) [
25] nécessite aussi une mise en échec du réplisome aboutissant à une cassure double-brin. L’extrémité simple-brin en 3’ s’appariera à tout ADN mono-brin présentant une micro-homologie de séquence. Là encore, cette fourche secondaire peut se dissocier et la séquence libre peut chercher un autre brin présentant une micro-homologie. Au final, le brin réparé peut présenter des réarrangements très complexes de plus ou moins longue taille, en fonction de l’orientation et de la position des différents brins modèles rencontrés. Le même type de mécanisme, basé sur des homologies de grandes tailles, est appelé BIR (break induced replication). Ce mécanisme est plus particulièrement impliqué dans la maintenance des régions télomériques et la formation de remaniements dans ces régions [
26].
Les analyses de séquences réalisées au niveau des points de cassure de nombreux réarrangements ont montré la présence de séquences ou de motifs particuliers au voisinage de ces zones de cassure. Il peut s’agir d’éléments transposables ou de séquences répétées qui peuvent favoriser les phénomènes de recombinaison homologue non allélique (NAHR), ou simplement faciliter le contact et l’appariement des régions réarrangées. On note aussi la présence de séquences capables de créer des structures secondaires particulières, telles que les séquences palindromiques, voire des séquences pouvant induire des modifications de la conformation de la double hélice, comme les séquences polypurines ou polypyrimidines [
27]. Enfin, certaines séquences détectées correspondent à des sites de fixation de protéines particulières, comme les translines, retrouvées à proximité des points de cassure de nombreuses translocations, sans que leur rôle exact dans la cassure ou la réparation de la molécule d’ADN aient été élucidés [
28]. Beaucoup de ces éléments sont des facteurs d’instabilité génomique susceptibles de perturber la machinerie réplicative, d’exacerber la survenue de cassures double-brin ou de biaiser les mécanismes de réparation. La répartition de ces éléments sur le génome peut donner lieu à l’existence de zones de cassures privilégiées (hot spot), comme cela a été rapporté pour certains cancers. Cette notion conduit à prendre en compte le rôle de l’organisation nucléaire des chromosomes dans la formation des réarrangements complexes. La répartition des chromosomes en territoires est une notion admise et confirmée par de nombreuses analyses in situ [
29]. Plusieurs modèles d’organisation nucléaire ont été proposés en fonction de l’interpénétrabilité de ces territoires, mais tous admettent que leur répartition n’est pas due au hasard. Il s’agit de domaines très dynamiques dont les parties situées en surface peuvent interagir. Il est démontré que les gènes exprimés par une cellule se trouvent plus fréquemment situés à la surface de leurs territoires, dans une « bulle d’expression » où sont réunis les enzymes et les facteurs de transcription [
30]. Dans ces zones, la chromatine est particulièrement relâchée, donc plus sensible aux cassures double-brin. |
Comportement méiotique des CCR Comme toutes les anomalies chromosomiques de structure, les réarrangements complexes peuvent perturber le déroulement de la gamétogenèse et entraîner l’arrêt de la méiose ou la transmission d’une formule chromosomique déséquilibrée. Dans le cas des CCR, la combinaison de différents types de réarrangement et le nombre de chromosomes impliqués rendent délicates l’analyse et la prédiction du comportement méiotique de ces réarrangements. Les outils prédictifs, développés dans le but d’estimer le risque de déséquilibre à terme, ne sont hélas pas adaptés à l’analyse des réarrangements complexes [
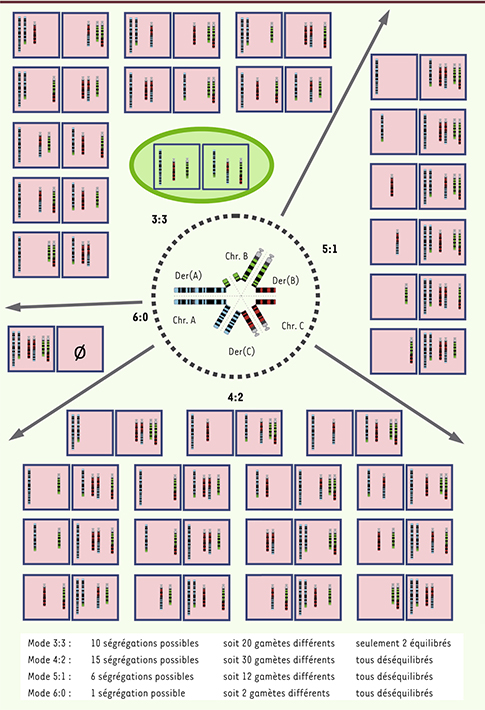
31]. L’exemple des translocations complexes de type three-way montre que le diagramme de pachytène formé pendant la méiose I est une structure hexavalente permettant l’appariement des chromosomes homologues. La ségrégation des chromosomes qui en découle peut théoriquement se faire selon les types 3:3, 4:2, 5:1 ou 6:0. Au total, 32 schémas de ségrégation méiotique peuvent se produire donnant 64 gamètes différents, dont seulement deux sont équilibrés (Figure 5). La survenue d’un crossing-over ne modifie en rien les produits de ségrégation méiotique. Dans le cas des enfants nés avec un caryotype déséquilibré, les modes de ségrégation méiotique de type 4:2 et 3:3 sont les plus fréquents, comme c’est le cas pour les translocations réciproques classiques [1]. Concernant les translocations complexes exceptionnelles, le nombre de ségrégations méiotiques possibles est encore plus élevé. Comme cette classe d’anomalies est hétérogène, le nombre de possibilités est variable et dépend du nombre de points de cassure et des partenaires chromosomiques impliqués. Kausch et al. [4] ont rapporté le cas d’un CCR dont la ségrégation méiotique pouvait donner plus de 70 types de gamètes déséquilibrés. Outre le mode de ségrégation, on peut observer l’apparition de nouveaux chromosomes recombinants à la suite d’un crossing-over, ce qui augmente encore les possibilités de déséquilibre. Paradoxalement, ces recombinaisons peuvent donner lieu à un caryotype moins complexe à la génération suivante [
32]. Enfin, pour les translocations double ou triple two-way, il est théoriquement plus simple de prédire le mode de ségrégation méiotique si on considère que les translocations en présence sont indépendantes. On peut alors calculer un risque de déséquilibre pour chaque anomalie, puis les combiner pour déterminer un risque global. Ces calculs supposent que les différentes anomalies n’interagissent pas entre elles. Il faut enfin noter l’intervention d’un autre phénomène capable d’augmenter encore le risque de déséquilibre méiotique : l’effet inter-chromosomique. Pendant la première division méiotique, la présence de la structure complexe en pachytène peut déstabiliser le plan équatorial du fuseau de division. Il peut en résulter la mauvaise ségrégation d’un chromosome non impliqué dans le CCR, entraînant ainsi une aneuploïdie supplémentaire [
33].
 | Figure 5.
Schéma de la ségrégation méiotique pour une translocation three-way. Les carrés bleus représentent les paires de gamètes formés après méiose. Les gamètes déséquilibrés sont dessinés sur fond rose et les gamètes équilibrés sur fond vert. |
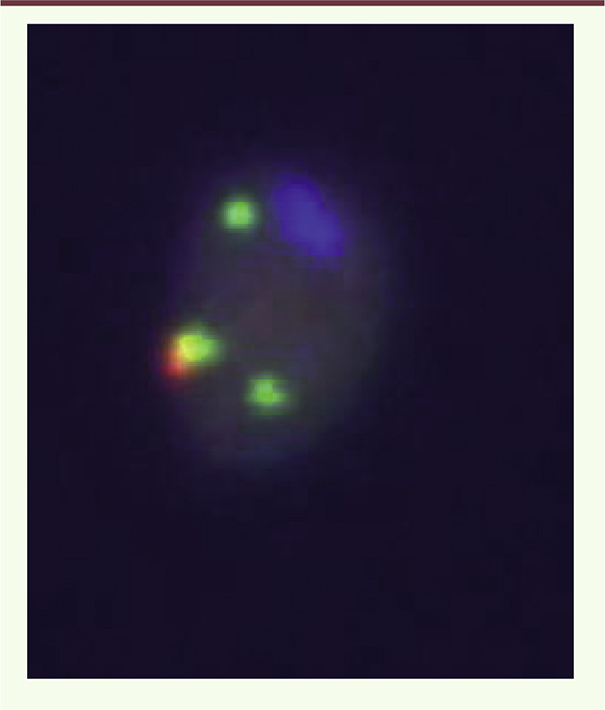
L’examen chromosomique direct des spermatozoïdes humains est particulièrement intéressant pour analyser la ségrégation méiotique des CCR. Cependant, très peu d’études de ce type ont été réalisées, du fait de l’hypofertilité de la plupart des sujets porteurs de CCR et des difficultés de mise en œuvre d’une telle technique sur les spermatozoïdes. Seules quatre études ont concerné des porteurs de CCR triple-way [1]. Dans tous les cas, l’analyse d’un nombre suffisant de spermatozoïdes (de 660 à 1900) par FISH a permis d’établir les profils de ségrégation des CCR (Figure 6). Des taux élevés de déséquilibres ont été observés (de 69 % à 85 %), ainsi qu’une part non négligeable de gamètes déséquilibrés secondairement à des recombinaisons multiples ou des erreurs de deuxième division méiotique (de 6 % à 9 %). Ces études ont confirmé la complexité de la ségrégation des CCR, mais elles ont aussi apporté la preuve que des patrimoines chromosomiques fortement déséquilibrés pouvaient progresser au travers de la gamétogenèse.
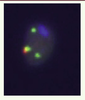 | Figure 6.
Exemple d’analyse chromosomique in situ par FISH sur spermatozoïdes de patients porteurs de CCR. Quatre sondes, marquées par quatre fluorochromes distincts, sont utilisées pour identifier les chromosomes impliqués dans le remaniement et déterminer la forme équilibrée ou déséquilibrée de l’anomalie, dans les noyaux de chaque spermatozoïde. Ainsi est établi le profil de ségrégation méiotique du remaniement. |
|
Conséquences sur la fertilité Si la découverte d’un réarrangement complexe de novo résulte le plus souvent d’une analyse chromosomique chez un homme, l’observation de cas familiaux de CCR montre que ceux-ci sont majoritairement transmis par la mère. On suppose donc que ces anomalies chromosomiques n’ont pas les mêmes conséquences sur la spermatogenèse et l’ovogenèse. Alors que les points de contrôle au cours de l’ovogenèse semblent être assez permissifs [
34], la spermatogenèse apparaît très sensible aux aberrations de structure. L’appariement des chromosomes homologues doit être parfait, sous peine de provoquer l’arrêt du processus méiotique. Échec de synapse2, hétérosynapse et association de segments autosomiques avec les gonosomes sont les principales causes d’interruption de la spermatogenèse. Plus le degré de complexité des anomalies chromosomiques est élevé, plus la proportion de segments asynaptiques augmente, entraînant une moindre qualité de l’appariement des chromosomes. De manière bien plus efficace que dans l’ovogenèse, ces anomalies de l’appariement du pachytène sont détectées dans le spermatocyte, pouvant alors conduire à l’arrêt de la méiose et à l’apoptose de la cellule. Les hommes porteurs de CCR présentent donc souvent des problèmes d’hypofertilité liés au nombre de gamètes (oligospermie ou azoospermie) [
43]. Si toutefois une grossesse survient, le risque d’anomalies de l’embryon est élevé. La très grande hétérogénéité des anomalies rencontrées fait qu’il est difficile d’évaluer quantitativement ce risque. On considère empiriquement que la probabilité de fausse-couche spontanée pour les couples porteurs de CCR se situe entre 50 % et 100 % des conceptions, et que le risque de donner naissance à un enfant avec un phénotype pathologique lié à un déséquilibre est de 20 % à 90 % [1, 7]. Des méthodes d’assistance médicale à la procréation (AMP) sont proposées aux couples concernés par des anomalies complexes du caryotype, notamment une fécondation in vitro (FIV) associée à une injection intracytoplasmique de spermatozoïde (ICSI) [
35]. Cependant, cette technique n’évalue pas la qualité chromosomique du spermatozoïde sélectionné, et le risque d’une conception déséquilibrée n’est donc pas maîtrisé. Le pourcentage des grossesses normales est faible et le recours à l’ISCI pose le problème de la propagation potentielle de l’anomalie complexe. Il est aussi possible de pratiquer un diagnostic pré-implantatoire (DPI). Une analyse chromosomique in situ par FISH de un ou deux blastomères prélevés permet de sélectionner un embryon ne présentant pas de déséquilibre. Là encore, le degré de complexité et le nombre de chromosomes impliqués conditionnent la faisabilité de la technique. Seuls six cas de DPI chez des couples porteurs de CCR ont été rapportés [1,
36] ; ils totalisent 129 embryons analysés par FISH pour seulement neuf embryons ayant une analyse chromosomique diagnostiquée comme normale ou équilibrée. À ce jour, seules deux grossesses ont été obtenues après transfert d’un embryon sélectionné par DPI. Récemment, Vanneste et al. [
37] ont réussi à mettre en œuvre une technique de puce à ADN appliquée au DPI pour les couples porteurs d’un CCR, afin de sélectionner les embryons selon leur caryotype moléculaire. |
Les CCR constituent une des manifestations les plus marquantes de l’instabilité génomique qui peut affecter le cycle de vie des cellules [
44], au gré de multiples facteurs de perturbation, exogènes ou endogènes. La diversité des mécanismes de réarrangement mis en œuvre et la détection de nombreux micro-remaniements supplémentaires témoignent de la complexité insoupçonnée des CCR. La découverte récente du phénomène de chromothripsis, qui correspond à la pulvérisation d’un ou de plusieurs segments chromosomiques et au réassemblage chaotique des fragments d’ADN au cours d’un seul évènement cellulaire, est venue apporter une nouvelle dimension à la complexité des mécanismes chromosomiques à l’origine des CCR [
38]. Initialement découvert dans les cancers, ce nouveau mode d’apparition de réarrangements chromosomiques complexes est considéré comme une voie d’aggravation du processus tumoral [
39]. Par ailleurs, ce phénomène de chromothripsis a aussi été observé en génétique constitutionnelle, chez des patients atteints de malformations congénitales [
40]. Face à cette complexité croissante, le développement de techniques d’analyse du génome de plus en plus fines permet aujourd’hui la caractérisation précise des CCR, l’étude de leur étiologie et de leur formation, ainsi que la prise en charge des patients porteurs de CCR. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Pellestor
F
,
Anahory
T
,
Lefort
G
, et al.
Complex chromosomal rearrangements: origin and meiotic behavior . Hum Reprod Update.
2011; ; 17 : :476.–494. 2.
Kleczkowska
A
,
Fryns
JP
,
Van den Berghe
H
. Complex chromosomal rearrangements (CCR) and their genetic consequences . J Genet Hum.
1982; ; 30 : :199.–214. 3.
Kousseff
BG
,
Nichols
P
,
Essig
YP
, et al.
Complex chromosome rearrangements and congenital anomalies . Am J Med Genet.
1987; ; 26 : :771.–782. 4.
Kausch
K
,
Haaf
T
,
Köhler
J
,
Schmid
M
. Complex chromosomal rearrangement in a woman with multiple miscarriages . Am J Med Genet.
1988; ; 31 : :415.–420. 5.
Houge
G
,
Liehr
T
,
Schoumans
J
, et al.
Ten years follow up of a boy with a complex chromosomal rearrangement: going from a > 5 to 15-breakpoint CCR . Am J Med Genet.
2003; ; 118 : :235.–240. 6.
Madan
K
,
Nieuwint
AWM
,
Van Bever
Y
. Recombination in a balanced complex translocation of a mother leading to a balanced reciprocal translocation in the child. Review of 60 cases of balanced complex translocations . Hum Genet.
1997; ; 99 : :806.–815. 7.
Madan
K
. Balanced complex chromosome rearrangements: reproductive aspects. A review . Am J Med Genet.
2012; ; 158 : :947.–963. 8.
Grasshoff
U
,
Singer
S
,
Liehr
T
, et al.
A complex chromosomal rearrangement with a translocation 4;10;14 in a fertile male carrier: ascertainment through an offspring with partial trisomy 14q24–1q22 and partial monosomy 4q27–q28 . Cytogenet Genome Res.
2003; ; 103 : :17.–23. 9.
Karadeniz
A
,
Mrasek
K
,
Weise
A
. Further delineation of complex chromosomal rearrangements in fertile male using multicolour banding . Mol Cytogenet.
2008; ; 1 : :17.. 10.
Kang
SH
,
Shaw
C
,
Ou
Z
, et al.
Insertional translocation detected using FISH confirmation or array-comparative genomic hybridization (aCGH) results . Am J Med Genet.
2010; ; 152 : :1111.–1126. 11.
Le Scouarnec
S
,
Gribble
SM
. Characterising chromosome rearrangements: recent technical advances in molecular cytogenetics . Heredity.
2012; ; 108 : :75.–85. 12.
Gribble
SM
,
Ng
BL
,
Prigmore
E
, et al.
Array painting: a protocol for the rapid analysis of aberrant chromosomes using DNA microarrays . Nat Protoc.
2009; ; 4 : :1722.–1736. 13.
Zhang
F
,
Dai
Y
,
Tu
Z
, et al.
Array-CGH detection of three cryptic submicroscopic imbalances in a complex chromosome rearrangement . J Genet.
2009; ; 88 : :369.–372. 14.
Reddy
KS
. The conundrum of a jumping translocation (JT) in CVS from twins and review of JTs . Am J Med Genet.
2010; ; 152 : :2924.–2936. 15.
Rosenberg
C
,
Knijnenburg
J
,
Chauffaille
M
, et al.
Array CGH detection of a cryptic deletion in a complex chromosome rearrangement . Hum Genet.
2005; ; 116 : :390.–394. 16.
Vanneste
E
,
Voet
T
,
Le Caignec
C
, et al.
Chromosome instability is common in human cleavage-stage embryos . Nat Med.
2009; ; 15 : :577.–583. 17.
Steuerwald
N
. Meiotic spindle checkpoints for assessment of aneuploidy oocytes . Cytogenet Genome Res.
2005; ; 111 : :256.–259. 18.
Tsai
AG
,
Lieber
MR.
, Mechanisms of chromosomal rearrangement in the human genome . BMC Genomics.
2010; ; 11 : :S1.. 19.
Lupski
JR
,
Stankiewicz
P.
, Genomic disorders: molecular mechanisms for rearrangements, conveyed phenotypes . PLoS Genet.
2005; ; 1 : :e49.. 20.
Hasting
PJ
,
Lupski
JR
,
Rosenberg
SM
,
Ira
G
. Mechanisms of change in gene copy number . Nat Rev Genet.
2009; ; 10 : :551.–564. 21.
McVey
M
,
Lee
SE
. MMEJ repair of double-strand breaks (director’s cut): deleted sequences and alternative ending . Trends Genet.
2008; ; 24 : :529.–538. 22.
George
CM
,
Alani
E
. Multiple cellular mechanisms prevent chromosomal rearrangements involving repetitive DNA . Critic Rev Biochem Mol Biol.
2012; ; 47 : :297.–313. 23.
Chen
JM
,
Cooper
DN
,
Férec
C
, et al.
Genomic rearrangements in inherited disease and cancer . Semin Cancer Biol.
2010; ; 20 : :222.–233. 24.
Lee
JA
,
Carvalho
CM
,
Lupski
JR
. A DNA replication mechanism for generating nonrecurrent rearrangements associated with genomic disorders . Cell.
2007; ; 131 : :1235.–1247. 25.
Hastings
PJ
,
Ira
G
,
Lupski
JR
. A microhomology-mediated break-induced replication model for the origin of human copy number variation . PLoS Genet.
2009; ; 5 : :e1000327.. 26.
Smith
CE
,
Llorente
B
,
Symington
LS
. Template switching during break-Induced replication . Nature.
2007; ; 447 : :102.–105. 27.
Kurahashi
H
,
Bolor
H
,
Kato
T
, et al.
Recent advance in our understanding of the molecular nature of chromosomal abnormalities . J Hum Genet.
2009; ; 54 : :253.–260. 28.
Jaendling
A
,
McFarlane
RJ
. Biological roles of translin and translin-associated factor-X: RNA metabolism comes to the fore . Biochem J.
2010; ; 429 : :225.–234. 29.
Cremer
T
,
Cremer
C
. Chromosome territories, nuclear architecture and gene regulation in mammalian cells . Nat Rev Genet.
2001; ; 2 : :292.–301. 30.
Osborne
CS
,
Chakalova
L
,
Brown
KE
, et al.
Active genes dynamically colocalize to shared sites of ongoing transcription . Nat Genet.
2004; ; 36 : :1065.–1071. 31.
Cohen
O
,
Mermet
MA
,
Demongeot
J
. HC Forum: a web site based on an international human cytogenetic database . Nucleic Acids Res.
2001; ; 29 : :305.–307. 32.
Tihy
F
,
Lemieux
N
,
Lemyre
E
. Complex chromosome rearrangement and recombinant balanced translocation in a mother and a daughter with the same phenotypic abnormalities . Am J Med Genet.
2005; ; 135 : :317.–319. 33.
Pellestor
F
,
Imbert
I
,
Andréo
B
, et al.
Study of the occurrence of interchromosomal effect in spermatozoa of chromosomal rearrangement carriers by fluorescence in situ hybridization and primed in situ labeling techniques . Hum Reprod.
2001; ; 16 : :1155.–1164. 34.
LeMaire-Adkins
R
,
Radke
K
,
Hunt
PA
. Lack of checkpoint control at the metaphase/anaphase transitiion: a mechanism of meiotic nondisjunction in mammalian females . J Cell Biol.
1997; ; 139 : :1611.–1619. 35.
Siffroi
JP
,
Benzacken
B
,
Straub
B
, et al.
Assisted reproductive technology and complex chromosomal rearrangements: the limites of ICSI . Mol Hum Reprod.
1997; ; 3 : :847.–851. 36.
Lim
CK
,
Cho
JW
,
Kim
JY
, et al.
A healthy live birth after successful preimplantation genetic diagnosis for carriers of complex chromosome rearrangements . Fertil Steril.
2008; ; 90 : :1680.–1684. 37.
Vanneste
E
,
Melotte
C
,
Voet
T
, et al.
PGD for a complex chromosomal rearrangement by array comparative genomic hybridization . Hum Reprod.
2011; ; 26 : :941.–949. 38.
Reynaud
C
,
Billaud
M
. La théorie de l’équilibre ponctué . Med Sci (Paris).
2011; ; 27 : :921.–923. 39.
Stephens
PJ
,
Greenman
CD
,
Fu
B
, et al.
Massive genomic rearrangement acquired in a single catastrophic event during cancer development . Cell.
2011; ; 144 : :27.–40. 40.
Klosterman
WP
,
Tavakoli-Yaraki
M
,
Van Roosmalen
M
, et al.
Constitutional chromothripsis rearrangements involve clustered double-stranded DNA breaks and nonhomologous repair mechnisms . Cell Rep.
2012; ; 1 : :648.–655. 41.
Baudat
F
,
de Massy
B
. SPO11 : une activité de coupure de l’ADN indispensable à la méiose . Med Sci (Paris).
2004; ; 20 : :213.–218. 42.
Buisson
R
,
Masson
JY
. Fonction des suppresseurs de tumeur PALB2 et BRCA2 dans la réparation des cassures double-brin de l’ADN . Med Sci (Paris).
2013; ; 29 : :301.–307. 43.
Coutton
C
,
Satre
V
,
Arnoult
C
,
Ray
P
. Génétique de l’infertilité masculine : les nouveaux acteurs . Med Sci (Paris).
2012; ; 28 : :497.–502. 44.
Le Borgne
M
,
Chartier
N
,
Billaud
M
. Instabilité chromosomique et cancer, enfin des CIN révélateurs . Med Sci (Paris).
2013; ; 29 : :807.–810. |