| |
| Med Sci (Paris). 2014 March; 30(3): 266–273. Published online 2014 March 31. doi: 10.1051/medsci/20143003014.Le chromothripsis
Une forme insoupçonnée de complexification extrême des remaniements chromosomiques Franck Pellestor,1* Vincent Gatinois,1 Jacques Puechberty,1 David Geneviève,1 and Geneviève Lefort1 1Laboratoire de génétique chromosomique, hôpital Arnaud de Villeneuve, CHRU de Montpellier, 371, avenue du doyen Gaston Giraud, 34295Montpellier, France |

Les remaniements chromosomiques complexes se définissent comme des anomalies de structure portant au minimum sur deux chromosomes et impliquant au moins trois points de cassure distincts [
1]. Lorsqu’ils sont constitutionnels, ces réarrangements complexes sont rares et associés à des manifestations pathologiques très variées, incluant des retards mentaux et des anomalies congénitales complexes [
2]. Les remaniements complexes acquis sont l’apanage de nombreux cancers et correspondent à des altérations de structure formées progressivement dans des cellules somatiques différenciées au cours de leur cycle de vie. Les réarrangements observés dans les cancers sont souvent bien plus complexes que les remaniements chromosomiques constitutionnels, dont le maintien doit faire face à une forte pression de sélection, de la conception à la naissance. Toutefois, les mécanismes moléculaires impliqués dans l’émergence de ces remaniements complexes à différentes périodes de la vie sont généralement considérés comme étant identiques. Les progrès des techniques d’investigation du génome humain ont largement contribué à améliorer notre connaissance de ces anomalies [
3]. Mais, alors que l’étiologie des remaniements complexes, leurs mécanismes de formation et leurs diagnostics semblaient être de mieux en mieux cernés, la découverte d’un nouveau type de réarrangement chromosomique complexe est venue bouleverser les schémas biologiques établis. |
La découverte du chromothripsis
La complexité des réarrangements chromosomiques atteint désormais son paroxysme avec une nouvelle variété de réarrangements complexes et massifs du génome, baptisée chromothripsis, dans laquelle de multiples réarrangements (jusqu’à plusieurs centaines) se produisent au cours d’un seul événement cellulaire, pour aboutir à la formation d’un ou de plusieurs chromosomes fortement remaniés, à l’image d’un patchwork (Figure 1) [
4].
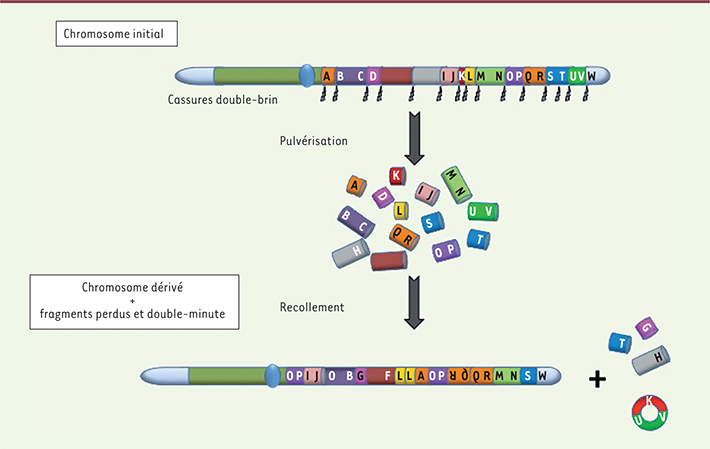
 | Figure 1.
Principe du chromothripsis. Au cours d’un seul événement catastrophique, de multiples cassures double-brin se produisent dans une région chromosomique, entraînant une pulvérisation en fragments chromosomiques. La plupart sont recollés de manière aléatoire afin de reformer une structure chromosomique chaotique (chromosome dérivé), alors que certains peuvent être perdus ou combinés sous forme de mini-chromosomes circulaires surnuméraires (double-minute), généralement porteurs d’oncogènes. |
Ce scénario catastrophe a été initialement proposé à la suite de la mise en évidence de 42 remaniements localisés sur le bras long d’un chromosome 4 chez un patient atteint de leucémie lymphoïde chronique [
5]. La découverte d’un si grand nombre de remaniements confinés à une seule région chromosomique a été rendue possible par l’utilisation combinée de l’analyse des polymorphismes par micro-array (single nucleotide polymorphism [SNP] array) et d’une nouvelle technique de séquençage massif à haut débit (paired-end sequencing) qui permet d’identifier tous les types de réarrangements génomiques avec une résolution de l’ordre de la paire de bases. L’analyse ultérieure de plus de 700 lignées cancéreuses et de 2 700 tumeurs a permis d’évaluer à 2-3 % la fréquence globale de ces réarrangements massifs dans les tumeurs, avec une incidence particulièrement élevée de 25 % dans les tumeurs osseuses [5]. Rapidement, ce phénomène a été identifié dans d’autres types de cancer, comme les cancers colorectaux [
6] et prostatiques [
7], les myélomes [
8], les neuroblastomes [
9] et les lymphomes de Hodgkin [
10]. Toutes ces observations ont remis en question les modèles classiques de cancérogenèse, basés sur l’acquisition progressive, ou par paliers, de multiples remaniements chromosomiques indépendants les uns des autres, et s’accumulant au fil du temps pour constituer une population de cellules malignes [
11]. Dans le même temps, le phénomène de chromothripsis a été découvert en cytogénétique constitutionnelle [
12]. On doit à Kloosterman et al. [
13] la description du premier cas de chromothripsis constitutionnel chez un enfant présentant un retard psychomoteur sévère et porteur d’une triple translocation t(1;10;4). L’analyse de séquences, menée conjointement chez l’enfant et ses parents, a révélé la présence de 12 réarrangements de novo inter- et intrachromosomiques à proximité des points de cassure sur les chromosomes 1, 4 et 10. Par la suite, une étude réalisée chez 17 sujets atteints d’un retard de développement associé à des anomalies cognitives diverses a identifié des profils complexes de remaniements, évocateurs de chromothripsis, et associant translocations, inversions, délétions et duplications. Ils sont là encore confinés à de petites régions d’un seul chromosome [
14]. Certains de ces remaniements étaient hérités de la mère alors que, chez la plupart des sujets, il s’agissait de remaniements de novo issus de la méiose paternelle. En 2012, deux nouvelles études ont confirmé l’existence de chromothripsis constitutionnels, d’une part chez des patients atteints de maladies congénitales et porteurs de réarrangements chromosomiques complexes [
15], mais aussi chez des sujets porteurs de translocations ou d’inversions simples [
16], avec, dans tous les cas, une nette prédominance de l’origine paternelle de ces remaniements. |
La signature moléculaire du chromothripsis
Toutes les études de chromothripsis rapportées font état, à la fois, de la multiplicité des remaniements et de l’unicité de l’événement de pulvérisation chromosomique aboutissant à de tels réarrangements massifs. Plusieurs caractéristiques communes confèrent une signature particulière au phénomène de chromothripsis et permettent de le distinguer des réarrangements chromosomiques complexes classiques [
17]. 1. Les réarrangements sont limités à une seule région d’un chromosome d’une paire d’homologues, ou à quelques chromosomes dont la structure est conjointement altérée. 2. Les points de cassure sont regroupés en clusters. On compte en moyenne de 5 à 10 cassures par segment de 50 kb. 3. Le nouvel agencement des fragments chromosomiques au sein du (ou des) chromosome(s) remanié(s) se fait de manière aléatoire et sans orientation précise de ces fragments. 4. Aucune mosaïque n’est observée dans la répartition cellulaire des réarrangements, ce qui serait logiquement attendu dans un modèle progressif du fait de l’accumulation de nouveaux remaniements au cours de la prolifération cellulaire. 5. Dans les zones remaniées, le nombre de copies des segments concernés oscille généralement entre un et deux, maintenant ainsi l’hétérozygotie allélique. Dans un modèle de formation progressive, toute délétion précoce d’un segment entraînerait une perte définitive d’hétérozygotie, et le nombre de copies d’autres segments devrait augmenter au fur et à mesure de l’accumulation des remaniements. Dans les tumeurs, les zones de chromothripsis se caractérisent par une juxtaposition de zones avec un faible nombre de copies et de segments où le nombre de copies est fortement augmenté. Mais, à l’issue de la pulvérisation chromosomique, des fragments peuvent aussi être perdus ou amplifiés pour donner naissance à des mini-chromosomes surnuméraires (chromosomes double-minutes) contenant des oncogènes [5]. Les chromothripsis constitutionnels, quant à eux, présentent un état relativement équilibré en dépit de la présence de nombreuses cassures et réarrangements pouvant être répartis sur plusieurs chromosomes. |
Facteurs causals du chromothripsis
L’existence d’un tel phénomène cataclysmique au sein d’une cellule suscite de nombreuses interrogations quant à sa formation et à son impact sur le fonctionnement cellulaire et la pathogenèse. La première question est de savoir quels facteurs peuvent induire la pulvérisation d’un ou de plusieurs chromosomes. Parmi les causes exogènes de cassures chromosomiques multiples, les drogues génotoxiques, les molécules réactives (ions à oxygène, radicaux libres) ou les radiations ionisantes sont classiquement évoquées en cancérologie [
18]. D’autres facteurs exogènes, comme certaines infections virales, peuvent aussi induire des cassures multiples de l’ADN [
19]. Au niveau cellulaire, divers processus endogènes peuvent être à l’origine de cassures chromosomiques multiples. Ainsi, des dysfonctionnements d’enzymes essentielles, comme les topo-isomérases ou les ADN polymérases, sont capables de provoquer de telles lésions. D’autres phénomènes intracellulaires, comme l’érosion des régions télomériques, ont aussi été mis en cause. Cet amenuisement progressif des télomères conduit à des processus cycliques de fusion télomérique/pont anaphasique/cassure qui aboutissent à de nombreuses fractures dans les régions télomériques et subtélomériques [
20]. Or, beaucoup d’exemples de chromothripsis concernent ces régions [5, 14]. Des similitudes ont aussi été notées entre le chromothripsis et le phénomène de condensation prématurée des chromosomes qui peut se produire lors de fusions cellulaires ou de fécondations asynchrones. En effet, lorsque les chromosomes d’une cellule en interphase (comme le spermatozoïde mature) subissent une décondensation sous l’effet de facteurs contenus dans une cellule au stade métaphasique (comme l’ovocyte I lors de la fécondation), ils peuvent subir une condensation prématurée de leur chromatine sans phase préliminaire de réplication, pouvant entraîner leur pulvérisation partielle [19]. Tubio et Estivill [
21] ont proposé que le chromothripsis puisse aussi être causé par un processus d’apoptose incomplète ou abortive dans certaines cellules. Ainsi, la fragmentation de la chromatine déclenchée par l’apoptose pourrait être interrompue, voire inversée, comme cela a été démontré récemment [
22], ce qui permet à la cellule de réparer tant bien que mal les cassures de son ADN et de survivre (Figure 2). Jones et Jallepalli [
23] ont aussi suggéré que l’apparition du chromothripsis puisse résulter de l’effet combiné des erreurs mitotiques de ségrégation, de l’instabilité chromosomique générée et du stress réplicatif qui en découle. Ce stress peut créer des points de cassure au niveau des origines de réplication [
24]. Enfin, une étude récente a établi une corrélation entre l’inactivation du gène TP53, codant pour la protéine p53, et le chromothripsis chez des patients atteints du syndrome de Li-Fraumeni. Chez ces sujets, la signature moléculaire du chromothripsis a été retrouvée dans une forte proportion (36 %) des tumeurs qui se développent [
25]. Or, la protéine p53 intervient dans de nombreux secteurs clés de la machinerie cellulaire, comme le contrôle de la transition G2/M, l’érosion des télomères et le contrôle de l’apoptose. Il a été démontré que le dysfonctionnement de ce gène pouvait favoriser, voire induire, divers phénomènes, comme les fusions télomériques, la décondensation prématurée des chromosomes, la duplication des centrosomes, l’inhibition de l’apoptose et la survie des cellules dont l’ADN est endommagé, qui sont autant de facteurs impliqués dans les scénarios d’apparition du chromothripsis. Un taux élevé de remaniements complexes a aussi été noté chez des patients présentant conjointement des leucémies myéloïdes et des mutations du gène TP53 [25]. La question se pose donc du lien possible entre le dysfonctionnement du gène TP53 et l’initiation du chromothripsis, en particulier dans les cancers.
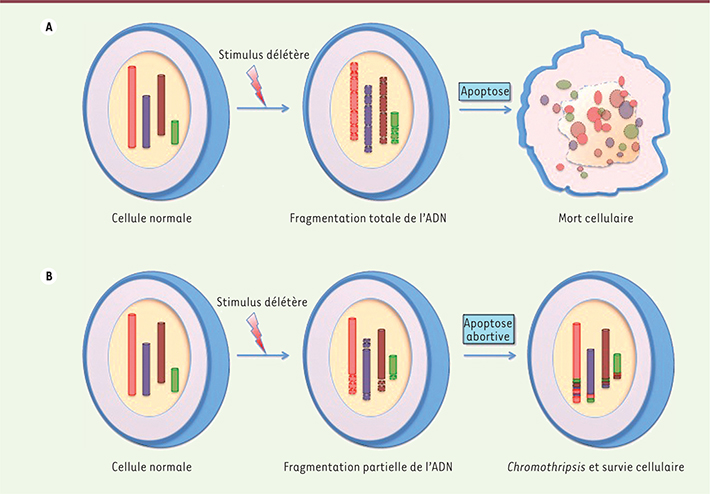
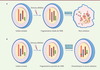 | Figure 2.
Hypothèse de l’apoptose abortive. A. Sous l’effet d’un stimulus délétère, la cellule entre en apoptose et subit une fragmentation progressive de son ADN aboutissant à sa mort. B. Si les dommages causés à l’ADN ne sont pas trop importants, le processus d’apoptose peut s’interrompre. La cellule peut réparer de manière chaotique la fragmentation de son ADN, et ainsi survivre. |
|
Scénario de réassemblage aberrant des fragments chromosomiques Si les causes de pulvérisation chromosomique sont multiples et encore largement débattues, les mécanismes de réparation impliqués dans la reformation des chromosomes fragmentés ont été en grande partie élucidés grâce à l’emploi des nouvelles méthodes de séquençage à haut débit. En effet, ces techniques permettent une analyse fine des zones de cassure des chromosomes recomposés. Dans beaucoup de cas de chromothripsis décrits en cancérologie, les points de cassure et de jonction présentent des zones de micro-homologie de quelques nucléotides, ou encore de petites insertions et délétions [5,
26]. Ces observations sont en faveur d’un réassemblage des fragments chromosomiques pulvérisés par le mécanisme de jonction des extrémités non homologues (NHEJ pour non homologous end-joining) ou par le mécanisme de jonction des extrémités orientées par micro-homologie (MHMEJ pour microhomology mediated end-joining). Il s’agit de processus simples et rapides de réparation des cassures double-brin qui peuvent être utilisés par la cellule à n’importe quel stade du cycle cellulaire, mais la fidélité de restitution des séquences réparées est assez faible. Ce type de réparation a été aussi identifié dans plusieurs exemples de chromothripsis constitutionnels [6, 15]. Mais de nombreux cas rapportés se caractérisent par la présence, au niveau des points de cassure, d’inversions, de duplications ou de triplications, ainsi que d’insertions de petites séquences qui sont homologues à d’autres segments du même ou d’un autre chromosome. Ces données suggèrent que les mécanismes de réparation intervenant lors du dysfonctionnement des fourches de réplication participent aussi au processus du chromothripsis [14, 16]. Les trois mécanismes identifiés, le FoSTes (fork stalling and template switching), le BIR (break induced replication) et le MMBIR (microhomology mediated break induced replication), combinent des arrêts des fourches de réplication avec des changements de brin servant de matrices pour les brins retardés. Ces processus de désengagement et d’envahissement, facilités ou non par de petites homologies de séquence entre les deux brins, peuvent se répéter plusieurs fois à la suite, d’où leur implication dans la genèse des remaniements chromosomiques complexes [
27]. Le regroupement des points de cassure sur un petit segment chromosomique peut être considéré comme un signe de l’intervention de ces mécanismes de réparation par stress réplicatif. Le déclenchement de ce type de réparation pourrait être lié à la stimulation inappropriée des origines de réplication dans la région remaniée [
28]. Toutefois, il faut se garder de privilégier cette voie de formation pour les chromothripsis constitutionnels, car la possibilité d’un biais d’analyse doit être prise en compte, dans la mesure où seules des formes viables de chromothripsis ont été identifiées et analysées jusqu’à présent [
29]. La majorité des chromothripsis constitutionnels se forment dans les cellules germinales ou au cours des premières divisions du zygote. L’origine paternelle préférentielle, qui est notée dans la plupart des cas rapportés, est en accord avec l’origine masculine attribuée à beaucoup d’anomalies de structure observées à terme [
30]. En fonction des modes de réparation impliqués, le phénomène de chromothripsis peut se dérouler au cours des divisions préméiotiques ou durant la première division méiotique, mais aussi lors de la phase de différenciation des spermatozoïdes (la spermiogenèse) qui comprend un ré-empaquetage protéique complet de l’ADN par des protamines. Or, cette opération engendre de nombreuses cassures de l’ADN dont la réparation ultérieure dans le zygote peut être incomplète [
31]. Cette fragmentation de l’ADN contenu dans les spermatozoïdes est un phénomène détectable in situ et analysé dans les bilans de stérilité, mais elle pourrait aussi être corrélée au chromothripsis. La survenue de chromothripsis au cours de l’ovogenèse doit aussi être prise en compte, du fait de la durée particulière de la méiose féminine et des risques d’exposition des chromosomes aux agents exogènes et endogènes à l’origine de cassures de l’ADN. Enfin, le chromothripsis peut avoir lieu au cours des premières divisions embryonnaires qui se caractérisent par une instabilité chromosomique accrue [
32], un contrôle du cycle cellulaire très permissif [
33], et de fréquentes anomalies de la ségrégation des chromosomes et du clivage des blastomères [
34]. L’apparition du chromothripsis au cours d’un seul événement cellulaire, les différents remaniements qui en résultent (translocations, inversions, duplications), ainsi que leur agencement aléatoire, ont conduit Kloosterman et Cuppen [29] à considérer que les chromothripsis constitutionnels relevaient du même mécanisme de formation que les translocations réciproques simples, basé sur le hasard de deux cassures double-brin et la proximité spatiale des deux chromosomes entre lesquels s’opère l’échange des deux fragments chromosomiques. Une extension connue de ce mécanisme est la formation des translocations réciproques à trois chromosomes et d’autres formes de remaniements chromosomiques complexes [1]. La cinétique de formation des chromothripsis constitutionnels apparaît similaire, avec un nombre bien plus grand de cassures double-brin entre des chromosomes physiquement proches au moment de la pulvérisation chromosomique initiale (Figure 3).
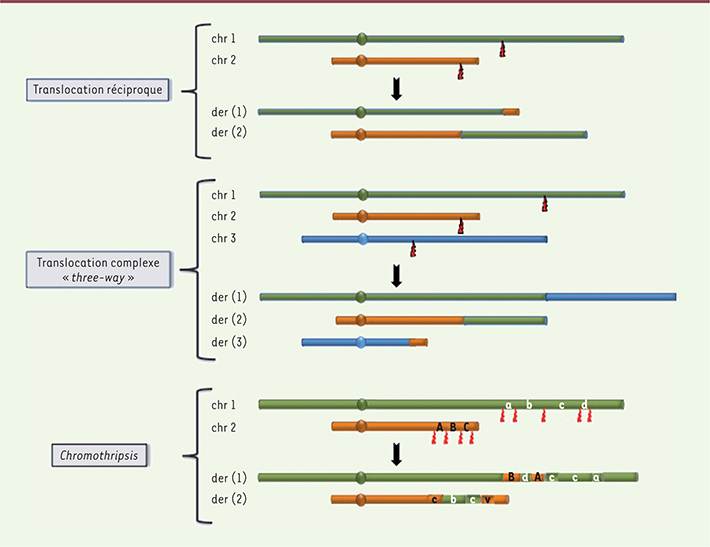
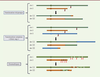 | Figure 3.
Similitudes des processus de formation des translocations réciproques, des remaniements complexes par translocation three-way et du chromothripsis, basées sur des cassures double-brin et des échanges de fragments chromosomiques entre plusieurs chromosomes.
|
Quelles sont les conditions pouvant inciter la cellule à utiliser ces deux voies de réparation malgré leur manque de fidélité ? Il est probable que le nombre de cassures double-brin et l’énergie nécessaire à la cellule pour mener à bien les réparations au cours d’un seul cycle soient des paramètres importants. Un excès de cassures double-brin (> 20) peut rapidement saturer les capacités de réparation de l’ADN par les voies classiques « error-free » [
35], ne laissant à la cellule que le recours à des modes de réparation « express », plus approximatifs. Toutefois, ces mécanismes (NHEJ ou stress réplicatif) ne sont nullement exclusifs, puisque l’utilisation des deux modes de réparation a été identifiée dans les chromothripsis tumoraux et constitutionnels. Dans les régions télomériques, les réarrangements peuvent résulter tout aussi bien de mécanismes de type NHEJ [
36] que de mécanismes de réparation réplicatifs [
37]. La localisation préférentielle du chromothripsis sur de courts segments chromosomiques laisse penser que le phénomène se produit lorsque les chromosomes sont les plus condensés, à savoir au cours de la mitose. Il est en effet difficile d’imaginer que tant de remaniements localisés sur de petits territoires chromosomiques puissent se produire en interphase, alors que les chromosomes sont amplement décondensés, à moins de considérer la répartition interphasique des chromosomes en territoires bien délimités. Comme pour les réarrangements chromosomiques complexes, divers éléments de la structure génomique pourraient être impliqués dans la survenue du chromothripsis. Ainsi, la présence de séquences répétées, comme les duplications segmentaires ou les séquences Alu, a été fréquemment observée au niveau des points de cassure des chromothripsis [6, 14]. La fréquence, la taille, l’agencement de ces séquences, mais aussi la distance les séparant, sont des paramètres intervenant dans la localisation des cassures double-brin [
38]. L’agencement des histones et leurs modifications peuvent aussi provoquer des cassures de l’ADN [
39], tout comme certaines configurations spatiales détectées dans l’environnement des points de cassure et qui pourraient favoriser les remaniements intrachromosomiques en créant des zones de fragilité et de susceptibilité aux cassures [
40]. Une explication intéressante au confinement du chromothripsis à un seul chromosome a été fournie par la découverte qu’un chromosome égaré au cours de l’anaphase et empaqueté dans un micronoyau pouvait y être pulvérisé et subir des réplications asynchrones [
41]. La formation d’un micronoyau peut résulter de divers mécanismes déjà évoqués, tels que la résolution des ponts chromosomiques formés à l’issue de fusion end-to-end des télomères, la décondensation prématurée de la chromatine, mais aussi la duplication des centrosomes et l’attachement mérotélique aux kinétochores [
42]. Les micronoyaux ainsi formés peuvent persister dans la cellule pendant plusieurs cycles cellulaires et, finalement, aboutir à la réintégration du chromosome remanié au sein du génome de la cellule [41]. Lorsque plusieurs chromosomes sont impliqués dans le chromothripsis, comme cela est le cas dans de nombreux cas de chromothripsis constitutionnels, l’organisation nucléaire des chromosomes en territoires doit être prise en compte. Là encore, la présence de configurations génomiques particulières ou de séquences répétées peut être importante pour la localisation du chromothripsis. Ces séquences peuvent servir de zones de micro-homologie et, ainsi, faciliter le changement de matrice dans la réparation réplicative. La juxtaposition des territoires chromosomiques au sein du noyau, le regroupement de différents locus ou de motifs particuliers dans ces territoires spécifiques [
43], ou encore leur proximité avec des zones de rétention des éléments de la machinerie de réparation ou de réplication (bulle de réplication) [
44], sont autant d’éléments qui peuvent constituer le fondement structural de la survenue d’un si grand nombre de remaniements dans des régions chromosomiques distinctes et du regroupement des points de cassure en cluster. |
Devant l’ampleur des remaniements observés, on peut penser que de tels événements catastrophiques ont des conséquences létales et entraînent inéluctablement la mort cellulaire. C’est vraisemblablement le cas pour la plupart des cellules subissant ce type de cataclysme. La localisation restreinte du chromothripsis à un seul chromosome, un segment chromosomique ou un petit territoire nucléaire pourrait constituer la limite d’acceptabilité du phénomène par la cellule. De nombreuses tumeurs ne possèdent pas de génomes stables et se caractérisent par de continuelles modifications de leur contenu chromosomique, commandées par l’instabilité génomique. Il est donc fort probable que des phénomènes de chromothripsis s’y produisent bien plus fréquemment que ce que laisse à penser leur incidence moyenne de 3 % [5]. Par ailleurs, les altérations génomiques associées au chromothripsis ne créent pas systématiquement les conditions d’un avantage sélectif pour la cellule. Mais lorsque ceci est le cas, divers mécanismes sont impliqués, comme la perte simultanée de gènes suppresseurs de tumeurs, l’amplification de gènes tumoraux, la formation d’oncogènes de fusion, ou la formation de mini-chromosomes surnuméraires porteurs d’oncogènes amplifiés [
45]. Cette forme particulière d’évolution rapide des cancers pourrait faire écho au modèle dit de « l’équilibre ponctué », caractérisé par l’accélération soudaine du développement tumoral sous l’impulsion d’un évènement cellulaire unique qui permet à la cellule de court-circuiter le processus graduel de transformation maligne [4,
46]. Pour les chromothripsis constitutionnels, la réorganisation complexe des chromosomes qui s’opère à la suite de leur fragmentation pourrait permettre à la cellule de conserver un statut équilibré à son génome. Le recours à des modes simples de réparation, comme le NHEJ ou le stress réplicatif, permet de résoudre rapidement la pulvérisation chromosomique au prix de la génération de remaniements ultra-complexes, qui peuvent parfois s’avérer stables et transmissibles. La découverte du phénomène de chromothripsis dans les remaniements hérités et de novo a mis en lumière la complexité insoupçonnée de nombreux réarrangements constitutionnels. Elle fournit de nouvelles pistes pour l’étude de pathologies congénitales complexes pour lesquelles l’étiologie restait encore inconnue et les mécanismes de formation mal compris. Compte tenu de ces hypothèses, Liu et al. [14] ont suggéré que l’appellation de chromoanasynthesis (chromo pour chromosome et anasynthesis pour re-synthèse) serait mieux adaptée pour exprimer la reconstruction chromosomique qui s’opère, alors que Holland et Cleveland [42] ont proposé le terme de chromoanagenesis pour évoquer la renaissance du chromosome ! Quoi qu’il en soit, les conséquences cliniques de telles catastrophes chromosomiques sont difficiles à appréhender, car elles sont sans doute très variées. En cancérologie, le phénomène a été associé à un mauvais pronostic et à une augmentation de l’agressivité des tumeurs [5, 8]. En cytogénétique constitutionnelle, les conséquences pathologiques du chromothripsis couvrent un large spectre de manifestations cliniques, compte tenu de la variété des altérations génomiques provoquées. Cependant, elles partagent une manifestation commune qui est le retard de développement neurologique, lié au déséquilibre génomique engendré par le phénomène. Il se peut que les similitudes observées dans les tableaux cliniques en rapport avec ces pathologies soient le reflet de la dimension multigénique des chromothripsis constitutionnels compatibles avec la vie. |
La découverte du phénomène de chromothripsis témoigne de la complexité insoupçonnée des phénomènes de restructuration chromosomique et démontre avec force que la mécanique chromosomique ne nous a pas encore livré tous ses secrets. Toutefois, il reste à apporter la preuve expérimentale de l’existence du chromothripsis, car comme le soulignent Liu et al. [
47], ainsi que Righolt et Mai [
48], d’autres mécanismes peuvent aussi conduire à une accumulation graduelle des cassures chromosomiques, par exemple la fragilité particulière d’une région chromosomique. Le chromothripsis pourrait ne pas être un événement unique et isolé, mais faire partie d’un mécanisme plus complexe et dynamique de gestion de l’instabilité génomique. Des expériences de simulation du chromothripsis dans des cellules en culture sont en cours [16], et des modèles in silico sont à l’étude pour mieux caractériser les chromothripsis et mieux comprendre les mécanismes qui pilotent leur apparition [
49]. D’un point de vue clinique, l’application des nouvelles techniques de séquençage à haut débit de l’ADN pour l’analyse des remaniements chromosomiques constitutionnels va sans doute révéler l’importance du chromothripsis dans l’émergence de nombreux désordres congénitaux, alors qu’en cancérologie, le lien découvert entre le chromothripsis et le dysfonctionnement du gène TP53 pourrait ouvrir de nouvelles voies thérapeutiques. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Pellestor
F
,
Anahory
T
,
Lefort
G
, et al.
Complex chromosomal rearrangements: origin and meiotic behavior . Hum Reprod Update.
2011; ; 17 : :476.–494. 2.
Madan
K
. What is a complex chromosome rearrangement ?
Am J Med Genet.
2013; ; 161 : :1181.–1184. 3.
Gatinois
V
,
Puechberty
J
,
Lefort
G
, et al.
Les remaniements chromosomiques complexes : un paradigme pour l’étude de l’instabilité chromosomique . Med Sci (Paris).
2013; ; 30 : :55.–63. 4.
Reynaud
C
,
Billaud
M
. La théorie de l’équilibre ponctué . Med Sci (Paris).
2011; ; 27 : :921.–944. 5.
Stephens
PJ
,
Greenman
CD
,
Fu
B
, et al.
Massive genomic rearrangement acquired in a single catastrophic event during cancer development . Cell.
2011; ; 144 : :27.–40. 6.
Kloosterman
WP
,
Hoogstraat
M
,
Paling
O
, et al.
Chromothripsis is a common mechanism driving genomic rearrangements in primary, metastatic colorectal cancer . Genome Biol.
2011; ; 12 : :R103.. 7.
Teles Alves
I
,
Hiltemann
S
,
Hartjes
T
, et al.
Gene fusions by chromothripsis of chromosome 5q in the VCaP prostate cancer cell line . Hum Genet.
2013; ; 132 : :709.–713. 8.
Magrangeas
F
,
Avet-Loiseau
H
,
Munshi
NC
,
Minvielle
S
. Chromothripsis identifies a rare and aggressive entity among newly diagnosed multiple myeloma patients . Blood.
2011; ; 118 : :675.–678. 9.
Molenaar
JJ
,
Koster
J
,
Zwijnenburg
DA
, et al.
Sequencing of neuroblastoma identifies chromothripsis and defects in neuritogenesis genes . Nature.
2012; ; 483 : :589.–593. 10.
Nagel
S
,
Meyer
C
,
Quentmeier
H
, et al.
Chromothripsis in Hodgkin lymphoma . Genes Chrom Cancer.
2013; ; 52 : :741.–747. 11.
Stratton
MR
,
Campbell
PJ
,
Futreal
PA
. The cancer genome . Nature.
2009; ; 458 : :719.–724. 12.
Reynaud
C
,
Billaud
M.
, Quand la chromothripsis devient germinale . Med Sci (Paris).
2011; ; 27 : :1083.. 13.
Kloosterman
WP
,
Guryev
V
,
van Roosmalen
M
, et al.
Chromothripsis as a mechanism driving complex de novo structural rearrangements in the germline . Hum Mol Genet.
2011; ; 20 : :1916.–1924. 14.
Liu
P
,
Erez
A
,
Sreenath Nagamani
SC
, et al.
Chromosome catastrophes involve replication mechanisms generating complex genomic rearrangements . Cell.
2011; ; 146 : :889.–903. 15.
Kloosterman
WP
,
Tavakoli-Yaraki
M
,
van Roosmalen
M
, et al.
Constitutional chromothripsis rearrangements involve clustered double-stranded DNA breaks and nonhomologous repair mechanisms . Cell Rep.
2012; ; 1 : :648.–655. 16.
Chiang
C
,
Jacobsen
JC
,
Ernst
C
, et al.
Complex reorganization and predominant non-homologous repair following chromosomal breakage in karyotypically balanced germline rearrangements and transgenic integration . Nat Genet.
2012; ; 44 : :390.–398. 17.
Korbel
JO
,
Campbell
PJ
. Criteria for inference of chromothripsis in cancer genomes . Cell.
2013; ; 152 : :1226.–1236. 18.
Okayasu
R
. Repair of DNA damage induced by accelerated heavy ions . Int J Cancer.
2012; ; 130 : :991.–1000. 19.
Stevens
JB
,
Abdallah
BY
,
Regan
SM
, et al.
Comparison of mitotic cell death by chromosome fragmentation to premature chromosome condensation . Mol Cytogenet.
2010; ; 3 : :20.–31. 20.
Luo
Y
,
Hermetz
KE
,
Jackson
JM
, et al.
Diverse mutational mechanisms cause pathogenic subtelomeric rearrangements . Hum Mol Genet.
2011; ; 20 : :3769.–3778. 21.
Tubio
JM
,
Estivill
X
. When catastrophe strikes a cell . Nature.
2011; ; 470 : :476.–477. 22.
Tang
HL
,
Tang
HM
,
Mak
KH
, et al.
Cell survival, DNA damage, and oncogenic transformation after a transient and reversible apoptotic response . Mol Biol Cell.
2012; ; 23 : :2240.–2252. 23.
Jones
MJK
,
Jallepalli
PV
. Chromothripsis: chromosomes in crisis . Dev Cell.
2012; ; 23 : :908.–917. 24.
Lukas
C
,
Savic
V
,
Bekker-Jensen
S
, et al.
53BP1 nuclear bodies form around DNA lesions generated by mitotic transmission of chromosomes under replication stress . Nat Cell Biol.
2011; ; 13 : :243.–253. 25.
Rausch
T
,
Jones
DTW
,
Zapatka
M
, et al.
Genome sequencing of pediatric medulloblastoma links catastrophic DNA rearrangements with TP53 mutations . Cell.
2012; ; 148 : :59.–71. 26.
Meyerson
M
,
Pellman
D
. Cancer genomes evolve by pulverizing single chromosomes . Cell.
2011; ; 144 : :9.–10. 27.
Lee
JA
,
Carvalho
CM
,
Lupski
JR
. A DNA replication mechanism for generating nonrecurrent rearrangements associated with genomic disorders . Cell.
2007; ; 131 : :1235.–1247. 28.
Green
BM
,
Finn
KJ
,
Li
JJ
. Loss of DNA replication control is a potent inducer of gene amplification . Science.
2010; ; 329 : :943.–946. 29.
Kloosterman
WP
,
Cuppen
E
. Chromothripsis in congenital disorders and cancer: similarities and differences . Curr Opin Cell Biol.
2013; ; 25 : :1.–8. 30.
Thomas
NS
,
Durkie
M
,
Van Zyl
B
, et al.
Parental and chromosomal origin of unbalanced de novo structural chromosome abnormalities in man . Hum Genet.
2006; ; 119 : :444.–450. 31.
Leduc
F
,
Nkoma
GB
,
Boissonneault
G
. Spermiogenesis and DNA repair; a possible etiology of human infertility and genetic disorders . Syst Biol Reprod Med.
2008; ; 54 : :3.–10. 32.
Vanneste
E
,
Voet
T
,
Le Caigec
C
, et al.
Chromosome instability is common in human cleavage-stage embryos . Nat Med.
2009; ; 15 : :577.–583. 33.
Kurahashi
H
,
Bolor
H
,
Kato
T
, et al.
Recent advance in our understanding of the molecular nature of chromosomal abnormalities . J Hum Genet.
2009; ; 54 : :253.–260. 34.
Mantzouratou
A
,
Delhanty
JD
. Aneuploidy in the human cleavage stage embryo . Cytogenet Genome Res.
2011; ; 133 : :141.–148. 35.
Gudjonsson
T
,
Altmeyer
M
,
Savic
V
, et al.
TRIP12 and UBR5 suppress spreading of chromatin ubiquitylation at damaged chromosomes . Cell.
2012; ; 150 : :697.–709. 36.
Palm
W
,
de Lange
T
. How shelterin protects mammalian telomeres . Annu Rev Genet.
2008; ; 42 : :301.–334. 37.
Yatsenko
SA
,
Hixson
P
,
Roney
EK
, et al.
Human subtelomeric copy number gains suggest a DNA replication mechanism for formation: beyond breakage-fusion-bridge for telomere stabilization . Hum Genet.
2012; ; 131 : :1895.–1910. 38.
Liu
P
,
Lacaria
M
,
Zhang
F
, et al.
Frequency of nonallelic homologous recombination is correlated with length of homology: evidence that ectopic synapsis precedes ectopic crossing-over . Am J Hum Genet.
2011; ; 89 : :580.–588. 39.
George
CM
,
Alani
E
. Multiple cellular mechanisms prevent chromosomal rearrangements involving repetitive DNA . Crit Rev Bioch Mol Biol.
2012; ; 47 : :297.–313. 40.
Halazonetis
TD
,
Gorgoulis
VG
,
Bartek
J
. An oncogene-induced DNA damage model for cancer development . Science.
2008; ; 319 : :1352.–1355. 41.
Crasta
K
,
Ganem
NJ
,
Dagher
R
, et al.
DNA break and chromosome pulverization from errors in mitosis . Nature.
2012; ; 482 : :53.–58. 42.
Holland
AJ
,
Cleveland
DW
. Chromoanagenesis and cancer: mechanisms and consequences of localized, complex chromosomal rearrangements . Nat Med.
2012; ; 18 : :1630.–1638. 43.
Zhang
Y
,
Patton McCord
R
,
Ho
YJ
, et al.
Spatial organization of the mouse genome and its role in recurrent chromosomal translocations . Cell.
2012; ; 148 : :908.–921. 44.
Howarth
KD
,
Pole
JC
,
Beavis
JC
, et al.
Large duplications at reciprocal translocation breakpoints that might be the counterpart of large deletions and could arise from stalled replication bubbles . Genome Res.
2011; ; 21 : :525.–534. 45.
Maher
CA
,
Wilson
RK
. Chromothripsis and human disease: piecing together the shattering process . Cell.
2012; ; 148 : :29.–32. 46.
Eldredge
N
,
Gould
SJ
. Punctuated equilibria: an alternative to phyletic gradualism . Models Paleobiol.
1972; ; 82 : :82.–115. 47.
Liu
P
,
Carvalho
CMB
,
Hastings
PJ
,
Lupski
JR
. Mechanisms for recurrent and complex human genomic rearrangements . Curr Opin Genet Dev.
2012; ; 22 : :211.–220. 48.
Righolt
C
,
Mai
S
. Shattered and stitched chromosomes-chromothripsis and chromoanasynthesis-manifestations of a new chromosome crisis ?
Genes Chrom Cancer.
2012; ; 51 : :975.–981. 49.
Greenman
CD
,
Pleasance
ED
,
Newman
S
, et al.
Estimation of rearrangement phylogeny for cancer genomes . Genome Res.
2012; ; 22 : :346.–361. |