| |
| Med Sci (Paris). 2014 August; 30(8-9): 790–796. Published online 2014 September 1. doi: 10.1051/medsci/20143008018.Rôle des longs ARN non codants dans le développement normal et pathologique Ève-Lyne Mathieu,1,2 Mohamed Belhocine,1,2 Lan T.M. Dao,1,2 Denis Puthier,1,2,3 and Salvatore Spicuglia1,2* 1Inserm UMR1090 TAGC (technological advances for genomics and clinics), 13288Marseille, France 2Université Aix-Marseille, UMR1090 TAGC, 13288Marseille, France 3Polytech Marseille, parc scientifique de Luminy, Marseille, France |

L’avènement des analyses génomiques à grande échelle a profondément modifié notre compréhension de l’organisation fonctionnelle du génome. En particulier, des études récentes ont mis en évidence qu’au moins 70 % du génome humain peut être transcrit [
1]. De ce fait, il est maintenant admis que la complexité du transcriptome s’étend bien au-delà des transcrits codant pour des protéines, puisqu’elle comprend aussi une myriade d’ARN non codants ayant des profils d’expression et de régulation complexes. Ces ARN non codants comprennent, d’une part, des types d’ARN régulateurs dont les fonctions et les mécanismes d’action impliqués sont relativement bien définis comme les miARN (micro-ARN) et les piARN (piwi-interacting RNA) et, d’autre part, des classes d’ARN moins bien caractérisées (comme les longs ARN non codants, lncRNA). Bien que la fonction de la grande majorité des lncRNA reste, à l’heure actuelle, largement inexplorée, il semble que ces transcrits jouent un rôle important dans plusieurs processus physiologiques, tels que la régulation épigénétique de l’expression génique ou la maturation des ARN messagers [
2–
6]. De plus, plusieurs travaux récents convergent pour indiquer que certains d’entre eux seraient impliqués dans le développement de pathologies humaines. Dans cette synthèse, nous nous proposons de passer en revue la littérature récente traitant des lncRNA et de leurs fonctions dans le développement normal et pathologique chez les mammifères. |
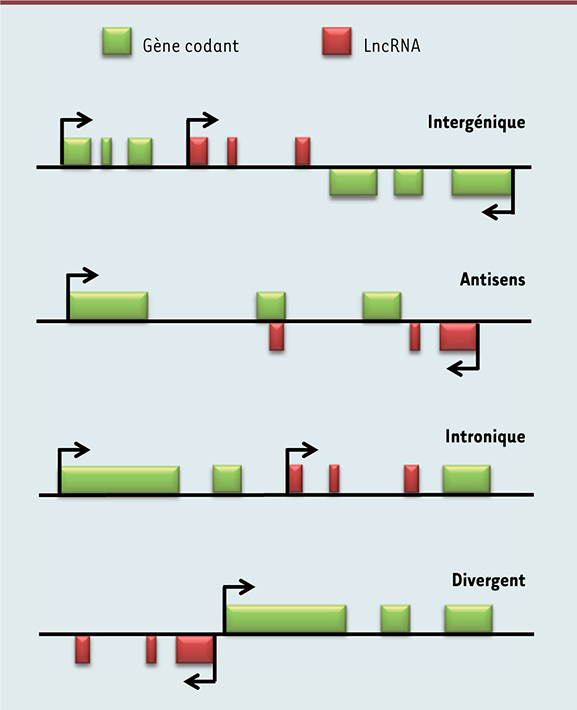
Une panoplie d’ARN longs non codants Les lncRNA sont des transcrits de plus de 200 nucléotides qui ne codent pas pour des protéines. Bien que cette définition soit en apparence arbitraire, elle permet de distinguer les lncRNA des petits ARN régulateurs, tels que les miARN, les piARN et les autres petits ARN nucléaires. Les lncRNA, transcrits généralement par l’ARN polymérase II, constituent un groupe de taille très hétérogène, certains pouvant s’étendre sur plusieurs dizaines de kilobases (kb). Les gènes de IncRNA partagent plusieurs caractéristiques avec les gènes codant pour des protéines, comme des profils épigénétiques similaires, la présence de signaux d’épissage et de polyadénylation, ainsi que la taille des exons et des introns [
3]. Toutefois, et comparativement aux ARNm, les lncRNA sont généralement plus enrichis dans le noyau et montrent une plus faible conservation de séquence, bien que certains d’entre eux soient fortement conservés [3]. De plus, les gènes de lncRNA s’expriment plus faiblement que les gènes codants et leur expression est remarquablement spécifique de certains tissus. En fonction de leur position par rapport aux gènes codants, les gènes de lncRNA peuvent être divisés en deux grandes catégories : les lncRNA intergéniques et les lncRNA intragéniques (Figure 1). Les lncRNA intergéniques, localisés par définition dans les régions non annotées du génome, sont généralement dénommés lincRNA. Ils représentent, à l’heure actuelle, la classe de lncRNA la mieux étudiée [3]. Les IncRNA intragéniques, quant à eux, peuvent être subdivisés en fonction de la façon dont ils chevauchent les gènes codants ou de leur orientation par rapport à ceux-ci (antisens, intronique, etc.). Il est à noter que de nombreux gènes de lncRNA intergéniques ont un site d’initiation de la transcription proche de celui d’un gène codant, la transcription se faisant sur le brin opposé (transcription divergente). Nous avons récemment démontré que les gènes associés à ces transcrits divergents codent fréquemment pour des régulateurs transcriptionnels impliqués dans le développement et la différenciation cellulaire [
7]. Enfin, certains lncRNA chevauchent de petits ARN, tels que des petits ARN nucléaires (snoRNA) [
46] ou des miARN, avec des liens fonctionnels potentiels, comme dans le cas de régions soumises à l’empreinte génomique [6].
 | Figure 1.
Représentation schématique des principales catégories de lncRNA. Les gènes de lncRNA sont définis par leur position relative aux gènes codants qui sont situés à leur proximité. Les gènes lncRNA intergéniques (lincRNA) sont des lncRNA localisés dans une région non annotée du génome. Les gènes lncRNA antisens sont des lncRNA transcrits dans la direction opposée d’un gène codant et dont la séquence chevauche en partie ou totalement le gène codant lui étant associé. Les lncRNA introniques sont des lncRNA contenus dans l’intron d’un gène codant. Les lncRNA divergents sont transcrits de façon divergente au promoteur d’un gène codant. Les exons de gènes sont représentés par des boîtes ; l’orientation et le site d’initiation de la transcription sont représentés par des flèches. Les gènes codants sont représentés en vert, les lncRNA sont en rouge. |
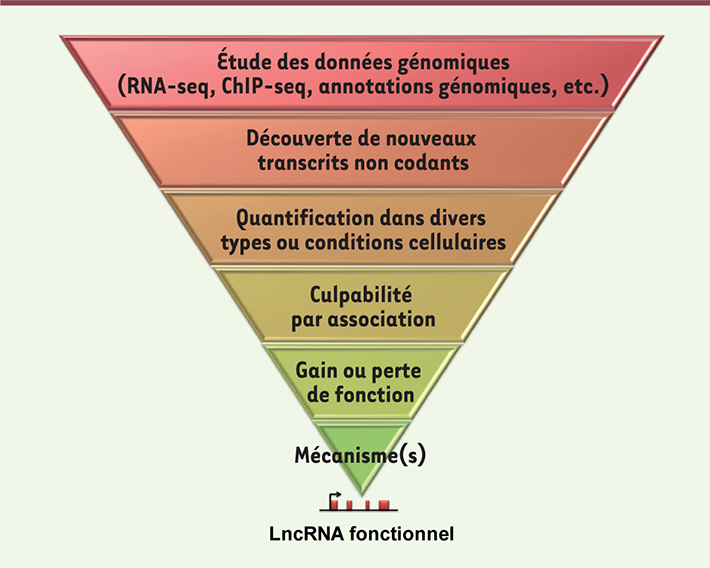
De multiples lncRNA contiennent des éléments répétés, comme des LINE (long interspersed nuclear elements) ou des SINE (short interspersed nuclear elements), avec des implications fonctionnelles potentielles [
8,
9]. Cependant, pris dans leur ensemble, les lncRNA n’ont pas de séquence ou de structure conservée qui pourrait être indicative d’une fonction particulière [3]. De ce fait, la plupart des études visant à identifier des lncRNA potentiellement pertinents dans un contexte physiologique ou pathologique donné sont basées sur des analyses de coexpression ou de corégulation. On transpose alors la fonction des gènes voisins ayant un profil similaire aux lncRNA dont la fonction est à définir [
10]. Ces approches sont aussi connues sous le terme « culpabilité par association » (Figure 2).
 | Figure 2.
Approches expérimentales menant à la caractérisation fonctionnelle des lncRNA. Les méthodes actuelles combinent des approches expérimentales et bio-informatiques afin de construire un pipeline robuste pour la découverte, l’annotation et la caractérisation fonctionnelle des lncRNA. RNA-seq : RNA-sequencing ; ChIP-seq : chromatin immunoprecipitation-sequencing. |
|
L’étude à grande échelle des lncRNA Au cours des deux dernières décennies, le séquençage systématique des banques d’ADNc a permis l’identification de divers transcrits, dont un certain nombre sont aujourd’hui considérés comme des lncRNA. De même, des études pionnières basées sur des puces à ADN ou sur l’analyse de profils épigénétiques ont révélé la présence de régions transcrites produisant un certain nombre de lncRNA intergéniques (lincRNA) [3]. En particulier, des analyses basées sur la combinaison de certaines marques de chromatine ont permis les premières caractérisations à haut débit des lincRNA (voir par exemple [
11]). Il a toutefois fallu attendre le développement généralisé du séquençage à haut débit de l’ARN (RNA-sequencing, RNA-seq), pour que des analyses systématiques du transcriptome révèlent l’existence de lncRNA de manière non ambiguë et permettent d’apprécier leur complexité chez les mammifères [
12,
13]. Cependant, étant donné que la plupart des lncRNA sont faiblement exprimés et/ou ont une structure exon/intron très complexe, il est parfois difficile d’identifier les différents transcrits produits à partir d’un gène lncRNA. Il est alors nécessaire de combiner une approche de RNA-seq classique avec d’autres techniques, telles que des analyses épigénétiques (certaines marques indiquent la présence de la région promotrice ou s’étendent sur l’ensemble du gène), du séquençage ciblé des transcrits d’intérêt après leur capture sur une puce d’ADN, et des analyses d’identification précise des extrémités 5’ ou 3’ des transcrits, de façon à pouvoir déterminer sans ambiguïté la structure des différents variants découlant de la transcription d’un gène lncRNA [12]. Ces diverses approches, couplées à des analyses bio-informatiques poussées, ont permis l’identification d’un très grand nombre de lncRNA exprimés dans une variété de tissus et de lignées cellulaires. Notamment, le projet ENCODE (encyclopedia of DNA elements), à travers la base de donnée GENCODE (http://www.gencodegenes.org), maintient à jour la liste la plus exhaustive de lncRNA exprimés chez l’homme [
14]. Actuellement, la version V19 de GENCODE compte 13 870 lncRNA détectés chez l’humain, dont 7 114 correspondent à des lincRNA. De plus, les lncRNA connus sont regroupés dans plusieurs catalogues publics, tels que « lncrnadb » (http://www.lncrnadb.org) et « noncode » (http://www.noncode.org), lesquels comportent une description détaillée de leurs structures génomiques. |
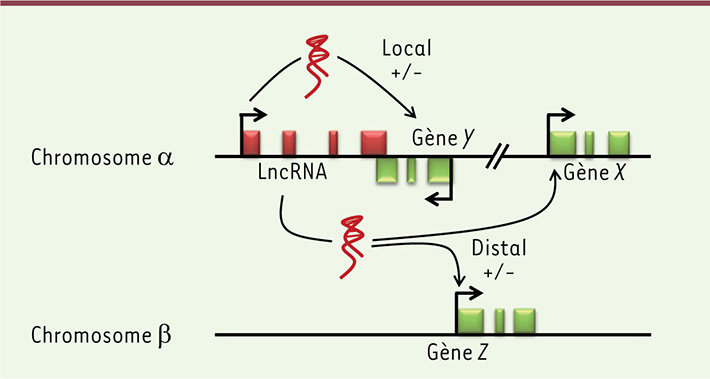
Fonctions des lncRNA dans les processus physiologiques Comme mentionné précédemment, contrairement aux protéines qui possèdent souvent des domaines fonctionnels bien définis, il est pour l’instant impossible de prédire la fonction des lncRNA à partir de leur séquence. Toutefois, il apparaît que les lncRNA agissent essentiellement en modulant l’expression des gènes [
15] (
Tableau I
). Cette fonction peut s’exercer localement, lorsque les lncRNA agissent en cis sur les gènes voisins, ou de façon distale, lorsque leurs fonctions se produisent indépendamment de la localisation des gènes cibles (Figure 3). En particulier, il existe une classe de lncRNA ayant une activité de type « enhancer » et qui peuvent activer de façon transcriptionnelle des gènes voisins [
16]. Plus généralement, des études de gain de fonction et de perte de fonction ont permis de mettre en évidence que les lncRNA sont potentiellement impliqués dans divers processus biologiques chez les mammifères [2, 15]. Ces processus incluent, par exemple, le maintien de la pluripotence des cellules souches embryonnaires, la différenciation cellulaire, la régulation du cycle cellulaire et la réponse immunitaire.
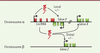 | Figure 3.
Régulation de l’expression génique par les lncRNA. Les lncRNA peuvent contrôler l’expression des gènes voisins (action locale) ou indépendamment de la localisation de leurs gènes cibles (action distale). Ce contrôle peut avoir un impact positif ou négatif (+/-) sur l’expression des gènes cibles. |
Tableau I.
| Noms |
Fonctions |
Mécanismes |
Références |
|
Linc-MD1
|
Contrôle temporel du programme de différenciation musculaire |
Séquestration des miR-133 et miR-135 |
[
39] |
|
HOTAIR
|
Répression transcriptionnelle des gènes HOXD
|
Recrutement des complexes répresseurs PRC2 et LSD1 |
[
18,
19] |
|
HOTTIP et Mistral
|
Activation transcriptionnelle de gènes HOXA
|
Recrutement du complexe activateur WDR5/MLL |
[
20,
21] |
|
UCHL1-as
|
Contrôle de la traduction de l’ARNm du gène UCHL1
|
Ciblage de l’ARNm vers le ribosome |
[
22] |
|
XIST
|
Répression mono-allélique du chromosome X chez les mammifères femelles |
En partie via le recrutement des complexes répresseurs |
[
17] |
Exemples de lncRNA impliqués dans des processus biologiques physiologiques.
Linc-MD1 : lincRNA muscle differentiation 1.
|
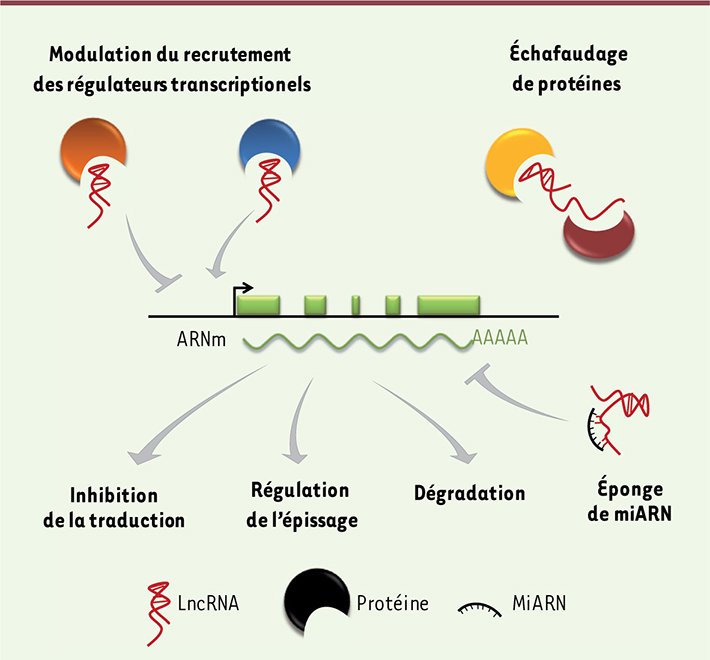
Les lncRNA peuvent réguler l’expression des gènes par divers mécanismes. Les aspects moléculaires de ces mécanismes ont été détaillés dans des revues récentes [2,
4, 15]. Les lncRNA peuvent potentiellement lier l’ADN, des protéines ou d’autres ARN, formant des plateformes d’échafaudage et permettant ainsi l’interaction entre diverses molécules fonctionnelles (
Figure 4
). Certains lncRNA peuvent modifier le contexte chromatinien près de leurs gènes cibles, en recrutant des facteurs de transcription ou des acteurs de la modification d’histones ou de la réorganisation de la chromatine, favorisant ou réprimant ainsi la transcription de gènes cibles, selon le contexte. Parmi les lncRNA qui ont été fonctionnellement caractérisés se trouve XIST (X-inactive specific transcript), un transcrit de 19-kb, dont le gène est localisé sur le chromosome X [17]. XIST est directement impliqué dans l’inactivation du chromosome X chez les mammifères femelles. Une fois transcrit, XIST est retenu dans le noyau et recouvre le chromosome X inactif. De plus, il interagirait avec le complexe répresseur Polycomb 2 (PRC2), permettant le recrutement ciblé de ce complexe et contribuant ainsi au maintien de l’inactivation du chromosome X [17]. De façon intéressante, XIST est à son tour régulé par d’autres lncRNA, tels que TSIX et XITE (X-inactivation intergenic transcription element) [17]. D’autres exemples de lncRNA localisés dans des régions génomiques soumises à l’empreinte parentale, comme AIRN (antisens of IGF2R RNA non-protein coding), H19 ou KCNQ1OT1, sont également impliqués dans l’inactivation de l’expression génique, via leurs associations avec des complexes répresseurs associés à la chromatine [6]. De même, le lncRNA HOTAIR (Hox antisense intergenic RNA), dont le gène est localisé dans le locus HOXC, servirait d’échafaudage pour les complexes PRC2 et LSD1 (lysine specific demethylase 1), deux complexes reliés à la répression transcriptionnelle [18, 19], et favoriserait leur recrutement au sein du locus HOXD. Au contraire, les lncRNA Mistral et HOTTIP (HOXA transcript at the distal Tip) faciliteraient l’expression des gènes HOXA en recrutant le complexe épigénétique activateur WDR5 (WD repeat-containing protein 5)/MLL (mixed lineage leukemia) [20, 21].  | Figure 4.
Exemples de mécanismes d’action des lncRNA. Les lncRNA peuvent favoriser l’interaction de protéines, guider des complexes protéiques vers leurs gènes cibles, séquestrer des protéines ou des miARN (éponge de miARN) et influencer les processus post-transcriptionnels liés aux ARNm, comme la traduction, l’épissage et la dégradation de l’ARNm. Un gène codant (en vert) est représenté à titre indicatif. Les mécanismes d’action favorisant positivement ou négativement l’expression des gènes sont représentés par des flèches grises. |
Les lncRNA sont aussi largement impliqués dans les processus post-transcriptionnels liés à la biogenèse des ARNm, tels que l’épissage, le transport, la traduction et la dégradation des ARNm (Figure 4). Par exemple, UCHL1-as (ubiquitin carboxyl-terminal hydrolase isozyme L1-antisens), un lncRNA antisens qui chevauche partiellement la partie 5’ du gène UCHL1, contribue positivement à la traduction de l’ARNm du gène UCHL1 [22]. Par ailleurs, des lncRNA peuvent agir comme des « éponges » pour prévenir la liaison de miARN à leurs ARNm cibles. Un cas extrême est représenté par CDR1-as/ciRS-7 (circular RNA sponge for miR-7), un lncRNA circulaire exprimé chez l’humain, qui possède 70 sites de liaison pour miR-7 [
23]. Bien que le mécanisme d’action de nombreux lncRNA soit maintenant établi, il reste néanmoins possible que l’expression de certains lncRNA soit plutôt le reflet d’une structure particulière de la chromatine ou d’un sous-produit d’un mécanisme impliquant le processus transcriptionnel. Cela est le cas de l’expression des transcrits germinaux produits au cours du processus de réarrangement somatique des gènes codant les récepteurs pour l’antigène [
24]. De plus, certains ARN non codants, appelés enhancer ARN (eARN), sont produits à partir d’éléments cis-régulateurs distaux [16]. À l’heure actuelle, le rôle de ces eARN sur l’activité transcriptionnelle du gène cible n’est pas encore clairement définie, puisque ceux-ci pourraient également être tout simplement des sous-produits d’éléments régulateurs actifs. En ce sens, nous avons démontré récemment que les couples lncRNA/ARNm transcrits de façon divergente sont le reflet d’un mécanisme spécialisé de la régulation transcriptionnelle impliquant des promoteurs bidirectionnels [7]. |
Le rôle des lncRNA dans le développement des maladies et des cancers Étant donné leurs contributions aux processus physiologiques, une altération du niveau d’expression des lncRNA pourrait mener au développement de pathologies, comme le cancer. En effet, de nombreuses études démontrant une dérégulation des lncRNA dans divers types de cellules cancéreuses suggèrent que les lncRNA peuvent agir comme des suppresseurs de tumeurs ou des oncogènes potentiels [4] (Tableau II). Parmi les exemples de lncRNA associés au cancer, le rôle de HOTAIR a été le plus étudié. Plusieurs publications ont rapporté une surexpression de HOTAIR dans différents cancers, où il participerait parfois à la formation de métastases [
25,
26]. MALAT1 (metastasis associated lung adenocarcinoma transcript 1) est un autre lncRNA très étudié ; sa surexpression est associée à l’état métastatique de tumeurs. Son utilisation comme marqueur pronostique dans le cancer du poumon a d’ailleurs été suggérée [4]. De même, des fonctions oncogéniques ont été suggérées pour plusieurs autres lncRNA dans, entre autres, le cancer du foie (HULC, highly up-regulated in liver cancer) [
27], de la prostate (PCA3, prostate cancer antigen 3) [
28,
29] et du rein (MVIH, microvascular invasion in HCC) [
30]. Finalement, certains lncRNA, tels que LincRNA-p21 et Meg3 (maternally expressed 3), ont été impliqués dans la modulation de la réponse p53 [
31,
32].
Tableau II.
| Noms |
Pathologies |
Réf. |
|
CDKN2B-as1
|
Artériosclérose, risques cardiovasculaires, divers cancers |
[
33–
35] |
|
BACE1-as
|
Alzheimer |
[
40] |
|
DBE-T
|
Dystrophie facio-scapulo-humérale |
[
41] |
|
MVIH
|
Carcinome hépatique |
[30] |
|
HOTAIR
|
Cancer du sein, cancer colorectal |
[25, 26] |
|
HULC
|
Cancer du foie |
[27] |
|
lncRNA HYMAI
|
Diabète néonatal transitoire |
[
42] |
|
PCA3
|
Cancer de la prostate |
[28] |
|
LincRNA-p21
|
Sarcome, lymphome, cancer du poumon |
[31] |
|
MALAT1
|
Cancer du poumon, de l’utérus, du sein, du pancréas, du rein, du côlon et de la prostate |
[
43-
45] |
Exemples de lncRNA associés à des pathologies.
BACE1-as : β-amyloid converting enzyme 1-antisense ; DBE-T : D4Z4 binding element transcript ; CDKN2B-as1 : (ANRIL), un lncRNA impliqué dans divers cancers ; MVIH : microvascular invasion ; HOTAIR : Hox antisense intergenic RNA ; HULC : highly up-regulated in liver cancer ; PCA3 : prostate cancer antigen 3 ; MALAT1 : metastasis associated lung adenocarcinoma transcript 1. |
La participation des lncRNA au développement de pathologies ne se limite cependant pas aux cancers. Le nombre d’études suggérant l’implication de lncRNA dans le développement de diverses maladies ne cesse d’augmenter [4]. Ainsi, CDKN2B-as1 (ANRIL), un lncRNA impliqué dans plusieurs cancers [33], est aussi associé à l’artériosclérose [
34] et à des risques cardiovasculaires accrus [35]. De la même manière, le développement de diverses pathologies, comme la maladie d’Alzheimer, le diabète néonatal transitoire et la dystrophie facio-scapulo-humérale, pourrait découler de la dérégulation de l’expression d’un lncRNA (Tableau II). Enfin, outre la dérégulation de l’expression des lncRNA, plusieurs études génétiques ont révélé la présence de mutations dans leurs séquences primaires [4]. Étant donné que la plupart des variants génétiques identifiés par des études d’association pangénomiques (GWAS) sont localisés en dehors des gènes codants, on peut s’attendre à ce qu’un grand nombre de ces mutations affectent des lncRNA [1], bien que les conséquences de ces mutations sur les fonctions des lncRNA doivent être démontrées. |
Les lncRNA comme agents thérapeutiques et biomarqueurs Étant donné leur rôle clé dans la régulation de l’expression génique, les lncRNA représentent des cibles thérapeutiques potentielles. Bien qu’ils soient encore à leur début, des essais cliniques utilisant des agents thérapeutiques à base d’ARN sont déjà à l’étude [15]. La plupart d’entre eux implique de petits ARN interférents (siRNA) ou des oligonucléotides antisens (ASO) qui mènent à la dégradation de leurs ARN cibles. Par exemple, l’utilisation in vivo chez la souris d’un ASO dirigé contre le lncRNA Bdnf-as (brain-derived neutrophic factor) lève la répression de Bdnf et permet la prolifération neuronale [
36]. Alternativement, les ASO pourraient agir comme agents bloquants en prévenant la liaison d’un lncRNA avec une protéine ou une séquence d’ADN ou d’ARN. Cependant, il est à noter que la distribution et la délivrance des ASO dans les organismes multicellulaires restent, jusqu’à ce jour, un obstacle majeur au développement de ces agents thérapeutiques [15]. Puisque plusieurs de ces lncRNA ont été associés au développement de métastases ou à la progression de tumeurs, les lncRNA peuvent servir de biomarqueurs potentiels pour le dépistage et le pronostic de cancers. Quelques-uns, comme PCA3 et HULC, sont déjà utilisés pour le diagnostic du cancer de la prostate et du carcinome hépatocellulaire [27, 28]. De façon intéressante, des acides nucléiques (ARN et ADN) très stables peuvent être détectés dans les fluides corporels, comme le sang, le plasma et l’urine [
37]. Il semblerait qu’il y ait une bonne corrélation entre les niveaux d’acides nucléiques circulants et les modifications génomiques, épigénétiques ou transcriptionelles associées aux tumeurs. Ces acides nucléiques seraient sécrétés ou relâchés dans le sang par des cellules cancéreuses en apoptose ou en nécrose. Notamment, des lncRNA, de par leur structure secondaire ou leur présence dans des microparticules, ont été détectés dans les fluides corporels [27, 28]. |
Les lncRNA apparaissent comme des régulateurs clés de l’expression génique. Les quelques exemples qui ont été étudiés jusqu’à présent démontrent clairement leurs rôles dans plusieurs processus physiologiques fondamentaux. De même, la mise en évidence de leur implication dans de nombreuses pathologies, dont notamment le cancer, ne cesse d’augmenter. Leurs utilisations potentielles comme biomarqueurs et cibles thérapeutiques sont, par ailleurs, prometteuses. Le défi majeur à l’heure actuelle reste la caractérisation fonctionnelle de la grande majorité des lncRNA. Toutefois, l’identification et la cartographie de la totalité des lncRNA présents dans le génome humain devraient accélérer l’atteinte de cet objectif. Cela implique des efforts conjoints entre la génomique fonctionnelle, l’épigénomique et la bio-informatique. La mise en place de consortiums internationaux, tels que le projet ENCODE et le projet européen « A BLUEPRINT of haematopoietic epigenome » [
38], partenaire de l’IHEC (international human epigenome consortium), joue un rôle déterminant dans ce contexte. On peut déjà être certain qu’avec tous les nouveaux questionnements découlant des fonctions associées aux lncRNA, l’intérêt des chercheurs à l’égard des lncRNA n’est pas près de s’estomper ! |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Nous remercions le Dr Jean Imbert pour la lecture critique de cette synthèse. Le travail de recherche dans le laboratoire de S. Spicuglia est financé par l’Union européenne (FP7 programme n° 282510-BLUEPRINT), l’ARC (projet n° SFI20111203756) et A*MIDEX (projet n° ANR-11-IDEX-0001-02). L.T.M. Dao est financée par le ministère de l’Éducation et de la formation du Vietnam. Nous nous excusons auprès de ceux dont les travaux, pour des raisons de place, n’apparaissent pas dans cette synthèse.
|
1.
Derrien
T
,
Guigo
R
,
Johnson
R.
. The long non-coding RNAs: A new (p)layer in the dark matter . Front Genet.
2011; ; 2 : :107.. 2.
Hu
W
,
Alvarez-Dominguez
JR
,
Lodish
HF
. Regulation of mammalian cell differentiation by long non-coding RNAs . EMBO Rep.
2012; ; 13 : :971.–983. 3.
Ulitsky
I
,
Bartel
DP
. lincRNAs: genomics, evolution, and mechanisms . Cell.
2013; ; 154 : :26.–46. 4.
Shi
X
,
Sun
M
,
Liu
H
, et al.
Long non-coding RNAs: a new frontier in the study of human diseases . Cancer Lett.
2013; ; 339 : :159.–166. 5.
Esteller
M
. Non-coding RNAs in human disease . Nat Rev Genet.
2011; ; 12 : :861.–874. 6.
Koerner
MV
,
Pauler
FM
,
Huang
R
,
Barlow
DP
. The function of non-coding RNAs in genomic imprinting . Development.
2009; ; 136 : :1771.–1783. 7.
Lepoivre
C
,
Belhocine
M
,
Bergon
A
, et al.
Divergent transcription is associated with promoters of transcriptional regulators . BMC Genomics.
2013; ; 14 : :914.. 8.
Kelley
DR
,
Transposable
Rinn JL.
. elements reveal a stem cell specific class of long noncoding RNAs . Genome Biol.
2012; ; 13 : :R107.. 9.
Kapusta
A
,
Kronenberg
Z
,
Lynch
VJ
, et al.
Transposable elements are major contributors to the origin, diversification, regulation of vertebrate long noncoding RNAs . PLoS Genet.
2013; ; 9 : :e1003470.. 10.
Guttman
M
,
Rinn
JL
. Modular regulatory principles of large non-coding RNAs . Nature.
2012; ; 482 : :339.–346. 11.
Guttman
M
,
Amit
I
,
Garber
M
, et al.
Chromatin signature reveals over a thousand highly conserved large non-coding RNAs in mammals . Nature.
2009; ; 458 : :223.–227. 12.
Spicuglia
S
,
Maqbool
MA
,
Puthier
D
,
Andrau
JC
. An update on recent methods applied for deciphering the diversity of the noncoding RNA genome structure and function . Methods.
2013; ; 63 : :3.–17. 13.
Derrien
T
,
Guigo
R
. De longs ARN non codants activateurs de la transcription des gènes . Med Sci (Paris).
2011; ; 27 : :359.–361. 14.
Derrien
T
,
Johnson
R
,
Bussotti
G
, et al.
The GENCODE v7 catalog of human long noncoding RNAs: analysis of their gene structure, evolution, and expression . Genome Res.
2012; ; 22 : :1775.–1789. 15.
Yang
L
,
Froberg
JE
,
Lee
JT
. Long noncoding RNAs: fresh perspectives into the RNA world . Trends Biochem Sci.
2014; ; 39 : :35.–43. 16.
Orom
UA
,
Shiekhattar
R
. Long noncoding RNAs usher in a new era in the biology of enhancers . Cell.
2013; ; 154 : :1190.–1193. 17.
Lee
JT
. Gracefully ageing at 50, X-chromosome inactivation becomes a paradigm for RNA and chromatin control . Nat Rev Mol Cell Biol.
2011; ; 12 : :815.–826. 18.
Rinn
JL
,
Kertesz
M
,
Wang
JK
, et al.
Functional demarcation of active and silent chromatin domains in human HOX loci by noncoding RNAs . Cell.
2007; ; 129 : :1311.–1323. 19.
Tsai
MC
,
Manor
O
,
Wan
Y
, et al.
Long noncoding RNA as modular scaffold of histone modification complexes . Science.
2010; ; 329 : :689.–693. 20.
Bertani
S
,
Sauer
S
,
Bolotin
E
,
Sauer
F
. The noncoding RNA Mistral activates Hoxa6 and Hoxa7 expression and stem cell differentiation by recruiting MLL1 to chromatin . Mol Cell.
2011; ; 43 : :1040.–1046. 21.
Wang
KC
,
Yang
YW
,
Liu
B
, et al.
A long noncoding RNA maintains active chromatin to coordinate homeotic gene expression . Nature.
2011; ; 472 : :120.–124. 22.
Carrieri
C
,
Cimatti
L
,
Biagioli
M
, et al.
Long non-coding antisense RNA controls Uchl1 translation through an embedded SINEB2 repeat . Nature.
2012; ; 491 : :454.–457. 23.
Memczak
S
,
Jens
M
,
Elefsinioti
A
, et al.
Circular RNAs are a large class of animal RNAs with regulatory potency . Nature.
2013; ; 495 : :333.–338. 24.
Spicuglia
S
,
Zacarias-Cabeza
J
,
Pekowska
P
,
Ferrier
P.
. Epigenetic regulation of antigen receptor gene rearrangement . F1000 Biol Rep.
2010; ; 2 : :23.. 25.
Kogo
R
,
Shimamura
T
,
Mimori
K
, et al.
Long noncoding RNA HOTAIR regulates polycomb-dependent chromatin modification and is associated with poor prognosis in colorectal cancers . Cancer Res.
2011; ; 71 : :6320.–6326. 26.
Gupta
RA
,
Shah
N
,
Wang
KC
, et al.
Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis . Nature.
2010; ; 464 : :1071.–1076. 27.
Panzitt
K
,
Tschernatsch
MM
,
Guelly
C
, et al.
Characterization of HULC, a novel gene with striking up-regulation in hepatocellular carcinoma, as noncoding RNA . Gastroenterology.
2007; ; 132 : :330.–342. 28.
Fradet
Y
,
Saad
F
,
Aprikian
A
, et al.
uPM3, a new molecular urine test for the detection of prostate cancer . Urology.
2004; ; 64 : :311.–316. 29.
Ferreira
LB
,
Palumbo
A
,
de Mello
KD
, et al.
PCA3 noncoding RNA is involved in the control of prostate-cancer cell survival, modulates androgen receptor signaling . BMC Cancer.
2012; ; 12 : :507.. 30.
Yuan
SX
,
Yang
F
,
Yang
Y
, et al.
Long noncoding RNA associated with microvascular invasion in hepatocellular carcinoma promotes angiogenesis and serves as a predictor for hepatocellular carcinoma patients’ poor recurrence-free survival after hepatectomy . Hepatology.
2012; ; 56 : :2231.–241. 31.
Huarte
M
,
Guttman
M
,
Feldser
D
, et al.
A large intergenic noncoding RNA induced by p53 mediates global gene repression in the p53 response . Cell.
2010; ; 142 : :409.–419. 32.
Zhou
Y
,
Zhong
Y
,
Wang
Y
, et al.
Activation of p53 by MEG3 non-coding RNA . J Biol Chem.
2007; ; 282 : :24731.–24742. 33.
Pasmant
E
,
Laurendeau
I
,
Sabbagh
A
, et al.
ANRIL ou l’étrange histoire d’un grand ARN non codant . Med Sci (Paris).
2010; ; 26 : :564.–566. 34.
Holdt
LM
,
Beutner
F
,
Scholz
M
, et al.
ANRIL expression is associated with atherosclerosis risk at chromosome 9p21 . Arterioscler Thromb Vasc Biol.
2010; ; 30 : :620.–627. 35.
Liu
Y
,
Sanoff
HK
,
Cho
H
, et al.
INK4/ARF transcript expression is associated with chromosome 9p21 variants linked to atherosclerosis . PLoS One.
2009; ; 4 : :e5027.. 36.
Modarresi
F
,
Faghihi
MA
,
Lopez-Toledano
MA
, et al.
Inhibition of natural antisense transcripts in vivo results in gene-specific transcriptional upregulation . Nat Biotechnol.
2012; ; 30 : :453.–459. 37.
Tong
YK
,
Lo
YM
. Diagnostic developments involving cell-free (circulating) nucleic acids . Clin Chim Acta.
2006; ; 363 : :187.–196. 38.
Adams
D
,
Altucci
L
,
Antonarakis
SE
, et al.
Blueprint to decode the epigenetic signature written in blood . Nat Biotechnol.
2012; ; 30 : :224.–226. 39.
Cesana
M
,
Cacchiarelli
D
,
Legnini
I
, et al.
A long noncoding RNA controls muscle differentiation by functioning as a competing endogenous RNA . Cell.
2011; ; 147 : :358.–369. 40.
Faghihi
MA
,
Modarresi
F
,
Khalil
AM
, et al.
Expression of a noncoding RNA is elevated in Alzheimer’s disease and drives rapid feed-forward regulation of beta-secretase . Nat Med.
2008; ; 14 : :723.–730. 41.
Cabianca
DS
,
Casa
V
,
Bodega
B
, et al.
A long ncRNA links copy number variation to a polycomb/trithorax epigenetic switch in FSHD muscular dystrophy . Cell.
2012; ; 149 : :819.–831. 42.
Temple
IK
,
Shield
JP
. Transient neonatal diabetes, a disorder of imprinting . J Med Genet.
2002; ; 39 : :872.–875. 43.
Ji
P
,
Diederichs
S
,
Wang
W
, et al.
Malat-1, a novel noncoding RNA, and thymosin beta4 predict metastasis and survival in early-stage non-small cell lung cancer . Oncogene.
2003; ; 22 : :8031.–8041. 44.
Yamada
K
,
Kano
J
,
Tsunoda
H
, et al.
Phenotypic characterization of endometrial stromal sarcoma of the uterus . Cancer Sci.
2006; ; 97 : :106.–112. 45.
Lin
R
,
Maeda
S
,
Liu
C
, et al.
A large noncoding RNA is a marker for murine hepatocellular carcinomas and a spectrum of human carcinomas . Oncogene.
2007; ; 26 : :851.–858. 46.
Abel
Y
,
Clerget
G
,
Bourguignon-Igel
V
, et al.
Les petits ARN nucléolaires nous surprennent encore !
Med Sci (Paris).
2014; ; 30 : :297.–302. |