| |
| Med Sci (Paris). 2014 December; 30(12): 1081–1084. Published online 2014 December 24. doi: 10.1051/medsci/20143012009.L’architecture moléculaire complète de l’ADN gyrase révélée par cryo-microscopie électronique Nouvelles informations sur le mécanisme de surenroulement négatif de l’ADN gyrase Julie Papillon,1 Jean-François Ménétret,1 Claire Batisse,1 Reynald Hélye,2 Patrick Schultz,1 Noëlle Potier,2 and Valérie Lamour1,3* 1IGBMC, départment de biologie structurale intégrative, CNRS UMR7104, Inserm U964, université de Strasbourg, 1, rue Laurent Fries, 67400Illkirch, France 2Institut de chimie de Strasbourg, université de Strasbourg, CNRS UMR7177, 67000Strasbourg, France 3hôpitaux universitaires de Strasbourg, fédération de médecine translationnelle de Strasbourg, 67000Strasbourg, France MeSH keywords: Antigènes néoplasiques, Bactéries, enzymologie, Sites de fixation, DNA gyrase, composition chimique, métabolisme, ultrastructure, ADN topoisomérases de type II, ADN superhélicoïdal, Protéines de liaison à l'ADN, Microscopie électronique, Structure moléculaire |
Les ADN topoisomérases de type 2 (Top2) sont des protéines nucléaires conservées de la bactérie à l’homme, essentielles à la survie cellulaire. Elles interviennent lors des processus de réplication, de transcription et de recombinaison en modulant la topologie de l’ADN. Ces enzymes sont capables d’introduire et d’éliminer des tours dans la double hélice d’ADN en catalysant le passage d’un double brin d’ADN à travers la coupure transitoire d’une autre molécule d’ADN [
1]. Les Top2 procaryotes et eucaryotes font l’objet d’une recherche intensive comme cibles pharmacologiques respectivement d’antibiotiques et d’agents anticancéreux, dans un contexte où le développement de résistances aux traitements est parfois corrélé à l’apparition de mutations dans ces enzymes [
2]. Notre travail a porté sur la Top2 procaryote, l’ADN gyrase, la seule Top2 capable de générer un ADN surenroulé négativement, une activité essentielle pour le maintien de la compaction du génome bactérien [
3]. L’ADN gyrase est composée de 2 polypeptides, gyrase A et gyrase B, organisés en un hétérotétramère A2B2
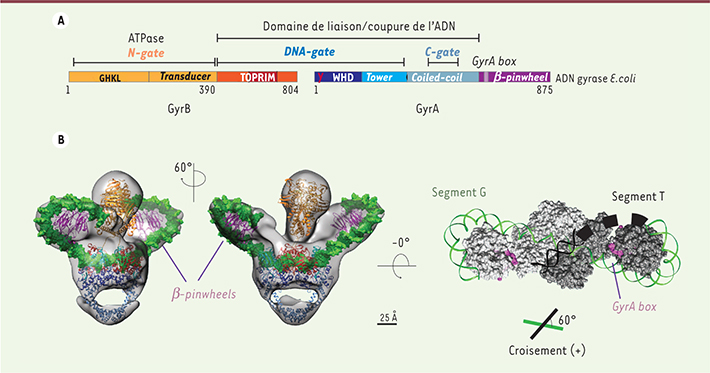
(Figure 1A). La partie centrale comprend trois interfaces protéiques majeures ou « portes », constituées d’un domaine de fixation de l’ATP et d’un domaine de liaison/coupure de l’ADN, à travers lesquelles l’ADN est transporté. Cette interface dimérique est flanquée de deux domaines carboxy-terminaux adoptant un repliement tridimensionnel en forme de disque (β-pinwheel), indispensables à l’introduction de courbures dans l’ADN [
4]. L’accumulation des données biochimiques et structurales sur plus de quatre décennies a permis de mettre en évidence l’implication des différents domaines dans le mécanisme de surenroulement de l’ADN. Dès qu’une première portion d’ADN (segment G) est fixée à l’interface dimérique, la séquence contiguë (segment T) s’enroule autour d’un domaine β-pinwheel et forme un croisement au dessus du segment G. L’enzyme fixe alors deux molécules d’ATP et ferme l’interface dimérique au-dessus du croisement d’ADN. Ceci permet successivement la coupure du segment G, le passage du segment T à travers la coupe double brin transitoire, puis l’expulsion du segment T, générant ainsi un tour de surenroulement [
5].
 | Figure 1.
Organisation des domaines catalytiques de l’ADN gyrase (numérotation selon la référence de l’ADN gyrase de E. coli). A. Dans la sous-unité B, le domaine ATPase est formé des deux régions GHKL (site de liaison de l’ATP commun aux protéines gyrase, Hsp90, histidine kinase, MutL) et transducer formant la porte N (ou N-gate). Le domaine de liaison/clivage de l’ADN (ou DNA-gate) est composé du domaine TOPRIM de la sous-unité B, et des domaines WHD (winged helix domain portant la tyrosine conservée catalytique) et Tower de la sous-unité A. La troisième porte ou « C-gate » est constituée par deux domaines coiled-coil. Les domaines carboxy-terminaux CTD possèdent un repliement b-pinweel avec le motif conservé GyrA box. B. Reconstruction tridimensionnelle en cryo-microscopie électronique du complexe de l’ADN gyrase avec un ADN de 155 pb en complexe avec l’ADPNP et la ciprofloxacine. Deux vues de la densité électronique (surface grise) sont représentées avec les modèles moléculaires des domaines placés en densité. Les couleurs pour chaque domaine correspondent à celles du schéma ci-dessus, l’ADN est représenté en surface verte. Le schéma de droite représente une vue de dessus de la surface du complexe sans le domaine ATPase et avec une extrapolation de la trajectoire de l’ADN à l’interface de l’enzyme (segment T en noir). La GyrA box (violet) fait face au croisement positif d’ADN formé par les segments T et G. Le sens de ce croisement est inversé en surenroulement négatif après transport du segment T à travers la coupure du segment G. |
La caractérisation structurale de ces complexes nucléoprotéiques représente un vrai défi scientifique et technique, de par la taille (~320 kDa) et la flexibilité de cet édifice moléculaire. Les études structurales, jusqu’à présent cantonnées aux domaines catalytiques isolés des Top2 en présence d’oligonucléotides et d’inhibiteurs [
6–
8], fournissent des informations très partielles sur les conformations adoptées par ces protéines modulaires au cours du cycle catalytique, et ne permettent pas d’étudier le positionnement des longues séquences d’ADN sur l’enzyme. Nous avons entrepris l’analyse structurale par cryo-microscopie électronique d’une ADN gyrase en complexe avec un ADN long de 155pb et un inhibiteur poison de la famille des quinolones, la ciprofloxacine (CFX) [
9]. |
étude de l’architecture moléculaire des complexes nucléoprotéiques de l’ADN gyrase par microscopie électronique Nous avons construit une protéine chimérique en fusionnant les deux sous-unités B/A afin de limiter la dissociation du complexe A2B2. Cette chimère mime l’organisation structurale homodimérique des Top2 eucaryotes, mais conserve ses propriétés catalytiques procaryotes, notamment de surenroulement de l’ADN [
10]. Nous avons confirmé l’état oligomérique de ces complexes et suivi par spectrométrie de masse supramoléculaire la formation du complexe avec un ADN de 155pb, la CFX et 2 molécules d’ADPNP, un analogue non hydrolysable de l’ATP. Le complexe en solution a été vitrifié dans l’éthane liquide puis des images de microscopie électronique ont été enregistrées grâce à un microscope à transmission (Polara FEG 300keV, FEI). La reconstruction tridimensionnelle du complexe met en évidence pour la première fois l’organisation structurale de l’ADN gyrase entière en présence d’un ADN s’enroulant autour de l’enzyme (Figure 1B). Le modèle moléculaire basé sur la carte de microscopie électronique montre une organisation orthogonale des domaines de liaison à l’ATP et à l’ADN conduisant à un entrelacement des deux sous-unités en présence d’ADPNP. Cet arrangement tridimensionnel a été également récemment décrit pour une Top2 de levure et semble donc conservé à travers l’évolution [
11]. Dans la conformation que nous observons, les domaines carboxy-terminaux β-pinwheel sont positionnés de façon asymétrique de part et d’autre du domaine ATPase. Nous avons pu placer plus de 130 pb d’ADN centré à l’interface entre le domaine ATPase et le domaine de liaison/coupure de l’ADN et s’enroulant autour des β-pinwheels. Ces domaines participent activement à la liaison et à la torsion de l’ADN lors du surenroulement, avec un angle de torsion total de plus de 240°. L’extrapolation du trajet de l’ADN suggère que le domaine β-pinwheel positionne le segment T à l’interface entre les domaines ATPase et de liaison à l’ADN en formant un croisement d’ADN de 60° sous les hélices carboxy-terminales du domaine ATPase (Figure 1B). Le croisement d’ADN se trouverait alors positionné en face d’un motif propre aux β-pinwheels de l’ADN gyrase, la « GyrA-box ». Ce motif conservé, initialement identifié comme impliqué dans la stabilisation de la structure β-pinwheel, semble donc également un élément important de la stabilisation du croisement d’ADN dans le domaine de liaison/coupure de l’ADN et renforcerait l’hypothèse d’une étude récente selon laquelle la « GyrA-box » serait un élément de communication inter-domaine [
12]. |
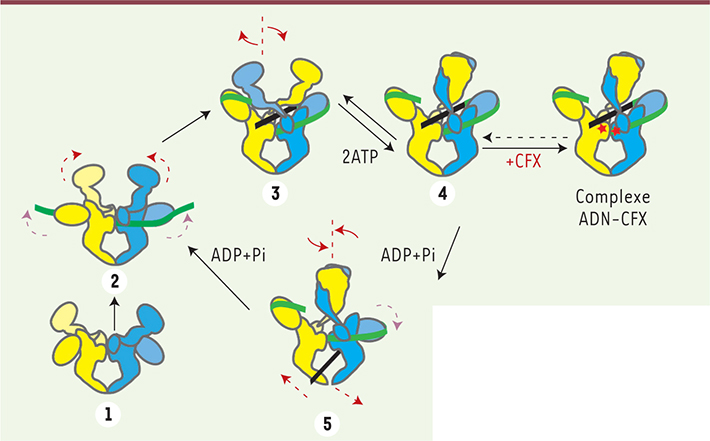
Coordination des domaines lors du cycle catalytique La disposition relative des différents domaines dans la conformation que nous observons en présence de l’ADN suggère que les domaines β-pinwheel et ATPase agissent de concert pour capturer le segment T et maintenir un croisement d’ADN à l’interface dimérique. Pour vérifier cette hypothèse, nous avons effectué des expériences de surenroulement de l’ADN avec des enzymes tronquées de leur domaine ATPase, ou bien en absence d’ATP empêchant le domaine ATPase de se refermer. Ces expériences montrent que l’ADN gyrase est toujours capable de courber l’ADN grâce à son domaine β-pinwheel, mais qu’elle perd la capacité de produire des croisements correctement orientés et compatibles avec le passage d’un ADN à travers l’autre pour produire un surenroulement négatif. Nos résultats suggèrent donc que le domaine ATPase, en plus de la fonction d’hydrolyse de l’ATP catalysant le transport de l’ADN, remplit un rôle structural dans les premières étapes du surenroulement négatif en permettant la capture et l’orientation du segment T conjointement avec un des domaines β-pinwheel. L’entrelacement des sous-unités induit alors le croisement des sous-unités du domaine ATPase qui se referme comme un piège sur le croisement d’ADN dans le domaine de liaison/coupure de l’ADN. La capture et le transport de l’ADN s’effectuent séquentiellement et s’accompagnent de nombreux réarrangements structuraux de l’enzyme. L’observation de l’organisation quaternaire des domaines dans nos reconstructions tridimensionnelles de l’ADN gyrase nous permet de proposer un modèle de coordination des domaines lors du cycle catalytique menant au surenroulement négatif de l’ADN (Figure 2).
 | Figure 2.
Mécanisme de surenroulement négatif par l’ADN gyrase révisé avec les nouvelles informations structurales. Les monomères de l’ADN gyrase sont colorés en jaune et bleu. (1-2) un premier segment T d’ADN se fixe à l’interface dimérique de l’ADN gyrase s’enroulant autour des domaines C-terminaux, faisant remonter les b-pinweel et l’ADN à proximité d’un des monomères du domaine ATPase. (3) Le segment T contigu au segment G est capturé à l’interface de l’enzyme et forme un croisement d’ADN stabilisé par l’entrelacement des monomères du domaine ATPase. (4) La fixation de deux molécules d’ATP entraîne la dimérisation du domaine ATPase et la fermeture du « piège à ADN » au dessus du croisement positif des segments T et G. (4-5) L’hydrolyse successive des molécules d’ATP entraîne des réarrangements structuraux qui accompagnent le transport du segment T à travers la coupure double brin du segment G. Après éjection du segment T, l’enzyme est alors prête pour un nouveau cycle catalytique. Les mouvements effectués par l’ADN gyrase peuvent être assimilés à ceux d’un nageur de crawl. L’antibiotique ciprofloxacine (CFX, étoiles rouges) s’intercale dans les coupures double brin du segment G, empêchant le passage du segment T et bloquant l’enzyme au début du cycle catalytique dans une conformation de « pré-transport » du segment T (DNA-cipro bound complex). |
Nos modèles montrent que le domaine ATPase une fois refermé peut adopter plusieurs orientations par rapport au domaine de liaison à l’ADN. Il est possible d’imaginer que lors de l’hydrolyse des ATP, ces rotations pourraient ainsi accompagner l’ouverture et la fermeture des interfaces protéiques. D’autre part, le positionnement asymétrique des β-pinwheels en présence d’ADN et la flexibilité reportée de ces domaines suggèrent qu’ils peuvent adopter différentes positions le long de l’enzyme, accompagnant le transport de l’ADN à travers l’interface dimérique. Enfin, l’élargissement de la densité électronique que nous observons au niveau de la dernière « porte » protéique en présence d’ADN pourrait refléter un mélange d’intermédiaires conformationnels ouverts en présence d’ADN. Ainsi, l’ADN gyrase pourrait se comparer à un nageur de « crawl » où le domaine ATPase joue le rôle de la tête, les β-pinwheels les bras et la dernière interface protéique les jambes du nageur, permettant une synchronisation optimale des différents domaines afin de générer un surenroulement négatif efficace. |
Conformations de l’enzyme bloquée par l’antibiotique La conformation que nous observons en présence d’ADN est stabilisée par l’antibiotique Ciprofloxacine que nous avons ajouté pour bloquer l’enzyme (
Figure 2
, complexe ADN-CFX). Cet antibiotique « poison » inhibe l’étape de religation de l’ADN lors du cycle catalytique. La conformation de l’ADN gyrase observée dans cette étude correspond à l’étape initiale de capture de l’ADN et de formation du croisement. Nous pouvons déduire de cette analyse que la ciprofloxacine bloque le passage du segment T à travers la coupure du segment G en piégeant une conformation de l’ADN gyrase au début du cycle catalytique, juste avant le transport du segment T. |
La reconstruction tridimensionnelle par microscopie électronique de l’ADN gyrase entière a révélé pour la première fois l’architecture entrelacée de ses complexes nucléoprotéiques. Cette étude permet de définir l’implication et la coordination des différents domaines de l’enzyme dans les premières étapes du mécanisme de surenroulement négatif de l’ADN. Ces travaux ouvrent de nombreuses perspectives pour l’étude de ces complexes modulaires et flexibles et apportent un éclairage inédit sur les états conformationnels de l’ADN gyrase visés par les antibiotiques lors du cycle catalytique. ‡ |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Wang
JC
. Cellular roles of DNA topoisomerases: a molecular perspective . Nat Rev Mol Cell Biol.
2002; ; 3 : :430.–440. 2.
Pommier
Y
,
Leo
E
,
Zhang
H
, et al.
DNA topoisomerases and their poisoning by anticancer and antibacterial drugs . Chem Biol.
2010; ; 17 : :421.–433. 3.
Schoeffler
AJ
,
Berger
JM
. DNA topoisomerases: harnessing and constraining energy to govern chromosome topology . Q Rev Biophys.
2008; ; 41 : :41.–101. 4.
Kramlinger
VM
,
Hiasa
H
. The ‘‘GyrA-box’’ is required for the ability of DNA gyrase to wrap DNA and catalyze the supercoiling reaction . J Biol Chem.
2006; ; 281 : :3738.–3742. 5.
Roca
J
,
Berger
JM
,
Harrison
SC
, et al.
DNA transport by a type II topoisomerase: direct evidence for a two-gate mechanism . Proc Natl Acad Sci USA.
1996; ; 93 : :4057.–4062. 6.
Dong
KC
,
Berger
JM
. Structural basis for gate-DNA recognition and bending by type IIA topoisomerases . Nature.
2007; ; 450 : :1201.–1205. 7.
Laponogov
I
,
Sohi
MK
,
Veselkov
DA
, et al.
Structural insight into the quinolone-DNA cleavage complex of type IIA topoisomerases . Nat Struct Mol Biol.
2009; ; 16 : :667.–669. 8.
Lamour
V
,
Hoermann
L
,
Jeltsch
JM
, et al.
An open conformation of the Thermus thermophilus gyrase B ATP-binding domain . J Biol Chem.
2002; ; 277 : :18947.–18953. 9.
Papillon
J
,
Ménétret
JF
,
Batisse
C
, et al.
Structural insight into negative DNA supercoiling by DNA gyrase, a bacterial type 2A DNA topoisomerase . Nucleic Acids Res.
2013; ; 41 : :7815.–7827. 10.
Trigueros
S
,
Roca
J
. A GyrB-GyrA fusion protein expressed in yeast cells is able to remove DNA supercoils but cannot substitute eukaryotic topoisomerase II . Genes Cells.
2002; ; 7 : :249.–257. 11.
Schmidt
BH
,
Osheroff
N
,
Berger
JM
. Structure of a topoisomerase II-DNA-nucleotide complex reveals a new control mechanism for ATPase activity . Nat Struct Mol Biol.
2012; ; 19 : :1147.–1154. 12.
Lanz
MA
,
Klostermeier
D
. The GyrA-box determines the geometry of DNA bound to gyrase and couples DNA binding to the nucleotide cycle . Nucleic Acids Res.
2012; ; 21: :10896.–10903. |