| |
| Med Sci (Paris). 2015 January; 31(1): 17–19. Published online 2015 February 6. doi: 10.1051/medsci/20153101005.L’investissement parental des espèces animales prédit leur diversité génétique Nicolas Galtier1,2* and Jonathan Romiguier1,2 1Institut des sciences de l’évolution, CNRS UMR5554, université Montpellier2, place E. Bataillon CC64, 34095Montpellier, France 2département d’écologie et d’évolution, université de Lausanne, Suisse MeSH keywords: Animaux, Comportement animal, Drosophila, Fécondité, Dérive génétique, Variation génétique, Génétique des populations, Comportement maternel, Comportement de nidification, Comportement paternel, physiologie |
Si l’on compare la séquence d’ADN d’un de vos chromosomes à celle d’un des chromosomes de votre voisin, on détectera une différence toutes les 5 000 paires de bases environ. Si l’on applique la même comparaison à deux individus de l’espèce Drosophila simulans, on enregistrera une différence toutes les 300 paires de bases. La mouche du vinaigre présente une diversité génétique beaucoup plus élevée que celle de notre espèce. Pour quelles raisons ? Il est remarquable que cette question historique [1] soit restée essentiellement sans réponse jusqu’à très récemment, et cela malgré le rôle central joué par la diversité génétique en biologie des populations et dans les politiques de préservation des espèces [2]. |
Taille de population et dérive génétique La diversité génétique attendue dans une population panmictique – c’est-à-dire sans isolement géographique marqué – est proportionnelle au produit de deux paramètres : le taux de mutation et la taille efficace des populations. Le premier de ces deux facteurs se comprend aisément : si un plus grand nombre de mutations apparaissent à chaque génération, la variation génétique augmente. L’autre effecteur majeur, la taille des populations, est sans doute moins intuitif. Il agit non pas sur le taux d’apparition des mutations, mais sur leur durée de vie à l’état polymorphe, plus courte dans les populations à faible effectif. Pour bien le comprendre, il faut réaliser que le devenir d’une mutation neutre (qui ne procure ni avantage ni désavantage à ses porteurs) est incertain, et lié au hasard de la reproduction. Si, par chance, les porteurs d’un allèle mutant venaient à avoir un grand nombre de descendants, la prévalence de cet allèle augmenterait mécaniquement à la prochaine génération – et inversement en cas de faible nombre de descendants. Cet effet aléatoire, appelé dérive génétique, est plus marqué dans les petites que dans les grandes populations : un variant malchanceux peut disparaître en quelques générations d’une population de très petite taille. En raccourcissant la durée de vie des mutations polymorphes, la dérive tend donc à diminuer la diversité génétique dans les petites populations [3]. Or, il est communément admis que la taille des populations naturelles est variable dans le temps. La démographie des espèces est affectée par des processus climatiques, écologiques et anthropiques qui font fluctuer leurs abondances. Ainsi, la paléoécologie nous a appris qu’un grand nombre d’espèces européennes étaient confinées il y a 100 000 ans, lors du dernier maximum glaciaire, à quelques « zones refuges » (péninsules ibérique, italienne et balkanique) [4]. Ces phases de forte réduction d’effectif, appelées « goulots d’étranglement », ont un impact durable sur la diversité génétique : en intensifiant soudainement la dérive, un goulot fait disparaître de nombreux variants, qui ne se régénéreront que lentement par mutation une fois l’épisode passé. Ces fluctuations temporelles d’effectif sont typiquement invoquées pour expliquer le caractère erratique de la diversité génétique : si elle reflète avant tout le passé des espèces, comment la prédire sur la base des données actuelles ? Une synthèse récente n’a pas permis de corréler de manière convaincante la diversité génétique de 167 espèces distinctes à quelque variable que ce soit, écologique, taxonomique ou biologique [5]. |
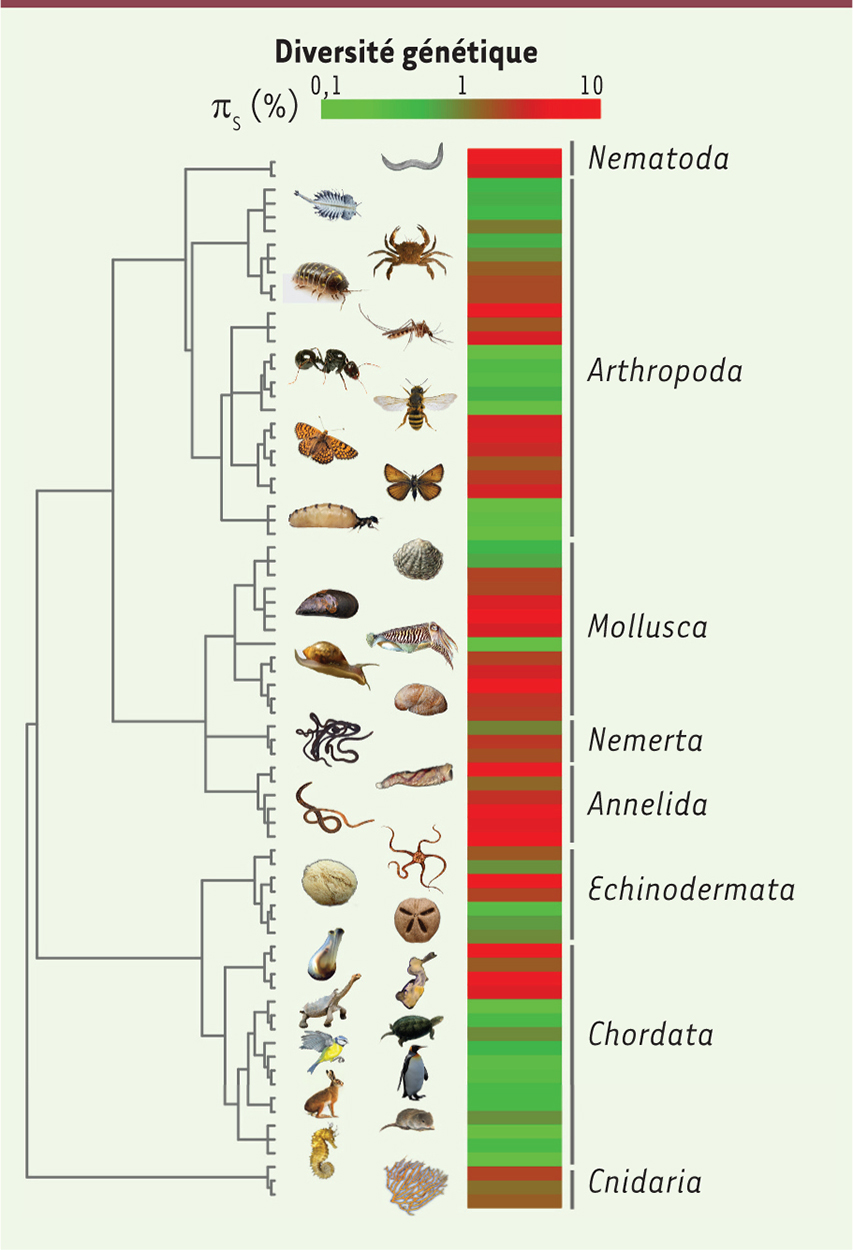
Le rôle clé de l’investissement parental Il faut toutefois noter que les données compilées à ce jour sont typiquement basées sur une couverture génomique très faible, de l’ordre de quelques gènes (à l’exception de quelques organismes modèles), et sur un échantillonnage taxonomique biaisé, avec une nette surreprésentation des mammifères [6]. Nous avons donc décidé de reconsidérer la question en explorant la majorité des grands groupes d’animaux (mammifères, mais aussi oiseaux, reptiles, poissons, urochordés, échinodermes, insectes, crustacés, nématodes, mollusques, annélides, némertes, cnidaires), et en mettant en œuvre des technologies de séquençage à haut débit pour accéder à l’échelle génomique. Ces techniques de lecture massive de fragments courts offrent la possibilité de séquencer simultanément de très nombreux gènes (ici, des ARN messagers), au prix de défis méthodologiques nouveaux pour l’identification des variants [7]. Notre jeu de données final contient 76 espèces, pour lesquelles 800 à 20 000 séquences géniques ont été séquencées chez 2 à 10 individus. Ces données ont fourni deux résultats inattendus [8]. Nous montrons tout d’abord que la diversité génétique d’une espèce est raisonnablement bien prédite par la taxonomie. Une fourmi, un oiseau, une tortue ou un mollusque céphalopode seront très probablement des espèces peu polymorphes. Un moustique, une ascidie, un ver de terre ou un mollusque bivalve seront très probablement des espèces fortement polymorphes. Ceci est illustré par la
figure 1
, qui montre que les espèces d’une même famille, regroupées en blocs, ont tendance à montrer des niveaux de diversité similaires. Cette découverte suffit à elle seule à mettre en doute l’idée que la fluctuation erratique des tailles de population au cours du temps effacerait tout signal. En effet, les espèces d’une même famille ne partagent clairement pas la même histoire : notre échantillon contient des bivalves atlantiques et d’autres pacifiques, des oiseaux européens et d’autres antarctiques, des moustiques à répartition mondiale et d’autres localisés, etc. Aucune variable potentiellement associée à l’histoire des populations, telle que la latitude ou l’étendue de l’aire de répartition, n’est d’ailleurs significativement corrélée à la diversité génétique dans notre jeu de données. Il apparaît donc que la biologie même d’une espèce suffit à prédire en bonne partie son niveau de diversité génétique, et ce malgré les aléas de son histoire récente.
 | Figure 1.
Distribution de la diversité génétique des espèces animales. La variable mesurée, πS (code couleur), s’interprète comme la proportion attendue de différences entre deux chromosomes échantillonnés aléatoirement dans la population, pour les sites neutres seulement. |
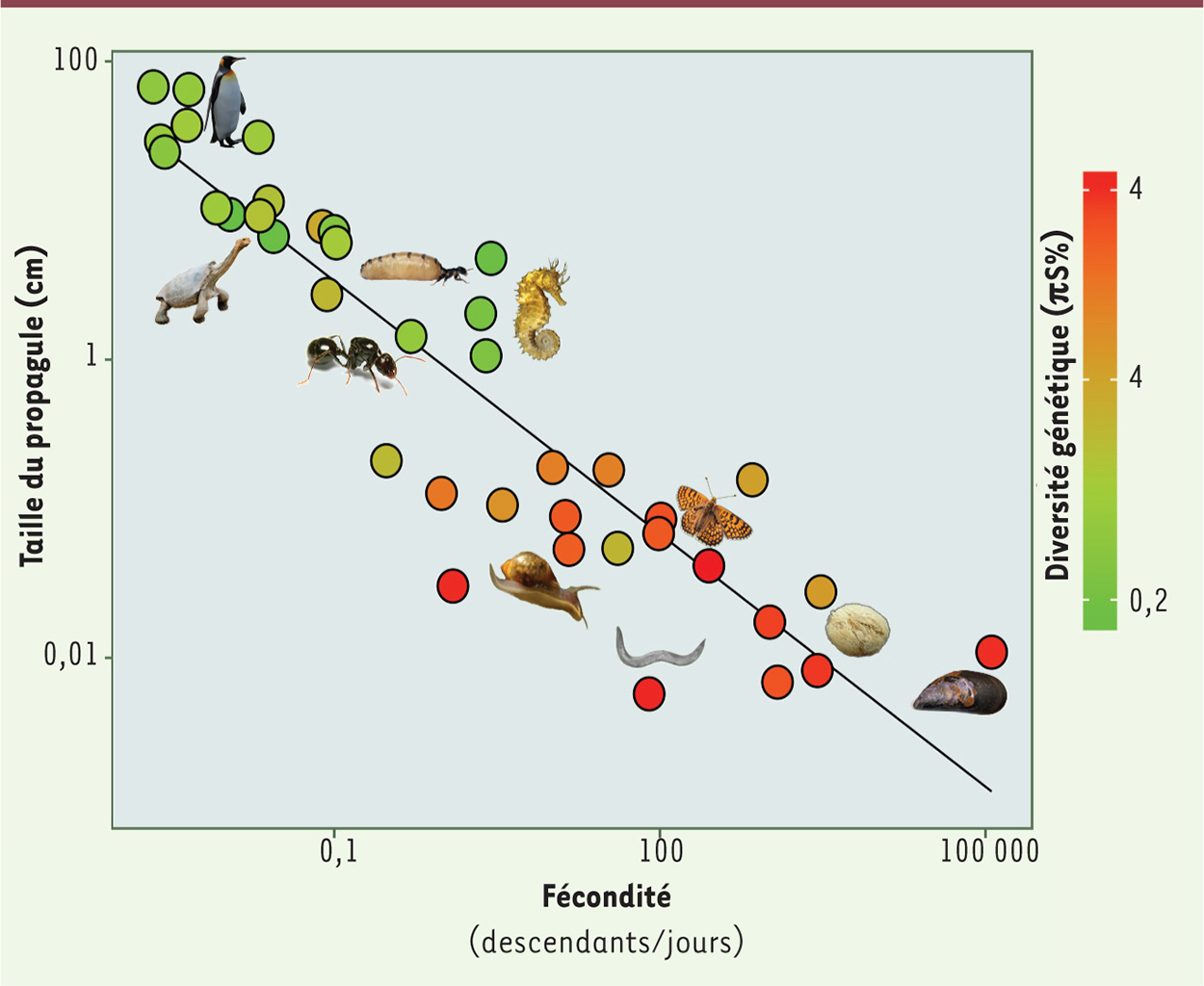
Pour avancer dans cette exploration, nous avons essayé d’identifier les caractéristiques biologiques ou écologiques susceptibles d’expliquer la distribution de la diversité génétique entre espèces – c’est là la deuxième surprise. La variable prédisant le mieux, et de loin, les variations de diversité entre espèces animales, s’avère être la taille du stade qui quitte la mère et se disperse, le « propagule » – œuf, larve ou juvénile selon les cas. La
figure 2
illustre un compromis bien connu entre quantité (axe horizontal) et taille (axe vertical) des descendants. Pour se reproduire, certaines espèces, dites à « stratégie r », misent sur le nombre ; c’est le cas des moules et des oursins, qui libèrent dans la colonne d’eau des dizaines de milliers de gamètes microscopiques. D’autres, au contraire, parient sur la qualité. Les parents investissent plus dans chacun de leurs descendants, qui sont donc moins nombreux mais ont chacun une probabilité plus élevée de survie jusqu’à l’état adulte – c’est la « stratégie K », employée notamment par le manchot, le termite et l’hippocampe. Elle est fréquemment associée à une longévité élevée et un temps de génération plus long. La
figure 2
montre clairement que ces espèces à fort investissement parental sont peu polymorphes comparées aux espèces « r », possiblement en raison de leur robustesse plus élevée aux changements environnementaux, qui leur permettrait de tolérer de très forts goulots d’étranglement sans pour autant s’éteindre.
 | Figure 2.
Investissement parental et diversité génétique. Les espèces à stratégie r (en bas à droite), qui produisent beaucoup de petits descendants, sont plus polymorphes que les espèces à stratégie K (en haut à gauche), qui produisent peu de gros descendants. |
|
Notre échantillonnage à la Prévert et notre couverture génomique étendue ont donc permis de mettre en évidence un lien jusqu’alors inconnu entre stratégie écologique et diversité génétique. Ces résultats soulèvent de nouvelles questions et ont une portée de long terme, fondamentale et appliquée. Ils suggèrent que les espèces r et K répondent différemment aux perturbations environnementales, et démontrent que de nombreux invertébrés présentent une diversité génétique aussi faible que celles des vertébrés, qui sont pourtant à ce jour les bénéficiaires quasi exclusifs de la biologie de la conservation. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Lewontin R. The genetic basis of evolutionary change . New York: : Columbia University Press; , 1974. 2.
Nair P. Conservation genomics . Proc Natl Acad Sci USA. 2014; ; 111 : :569.. 3.
Raymond M, Thomas F, Lefevre T. Biologie évolutive . Bruxelles: : De Boeck; , 2010. 4.
Hewitt G. The genetic legacy of the quaternary ice ages . Nature. 2000; ; 405 : :907.–913. 5.
Leffler EM, Bullaughey K, Matute DR, et al. Revisiting an old riddle: what determines genetic diversity levels within species? PLoS Biol. 2012; ; 10 : :e1001388.. 6.
Perry G, Melsted P, Marioni JC, et al. Comparative RNA sequencing reveals substantial genetic variation in endangered primates . Genome Res. 2012; ; 22 : :602.–610. 7.
Gayral P, Melo-Ferreira J, Glémin S et al. Reference-free population genomics from next-generation transcriptome data and the vertebrate-invertebrate gap . PLoS Genet. 2013; ; 9 : :e10003457.. 8.
Romiguier J, Gayral P, Ballenghien M et al. Comparative population genomics in animals uncovers the determinants of genetic diversity . Nature. 2014; ; 515 : :261.–263. |