| |
| Med Sci (Paris). 2015 February; 31(2): 159–167. Published online 2015 March 4. doi: 10.1051/medsci/20153102012.Approches de gene silencing pour le traitement de la maladie de Huntington Nicolas Merienne1* and Nicole Déglon1** 1Lausanne university hospital (CHUV), department of clinical neurosciences (DNC), laboratory of cellular and molecular neurotherapies (LCMN), avenue de Beaumont, Pavillon 3, 1011Lausanne, Suisse ; Lausanne university hospital (CHUV), neuroscience research center (CRN), LCMN, Lausanne, Suisse |
La maladie de Huntington (MH) est une maladie neurodégénérative de transmission autosomique dominante affectant 5-7/100 000 personnes dans le monde [1]. Il y a 20 ans, les progrès en biologie moléculaire, et plus particulièrement les prémisses du séquençage d’ADN, ont permis d’identifier l’anomalie génétique à l’origine de cette maladie [2]. Celle-ci, localisée sur le premier exon du gène codant pour la huntingtine (HTT), est caractérisée par une expansion pathologique de triplets CAG codant pour la glutamine [39]. Cette répétition CAG est présente chez tous les humains, mais sa taille diffère entre les sujets sains et les patients atteints de la maladie. Si le nombre des répétitions est supérieur à 39, la protéine adopte une conformation anormale qui entraîne l’apparition de la maladie. Malgré l’expression de la HTT mutée (mHTT) dès les premiers stades développementaux, les premiers symptômes de la maladie de Huntington ne se déclarent qu’à l’âge adulte. La symptomatologie est assez complexe, mais les principaux déficits observés sont moteurs, principalement caractérisés par des mouvements choréiques [3]. Les symptômes s’aggravent avec l’avancée de la pathologie pour atteindre ensuite les sphères psychiatrique et cognitive. La mHTT confère une vulnérabilité sélective aux neurones épineux de taille moyenne (MSN, voir Glossaire) du striatum, une région cérébrale interne des ganglions de la base [4]. Ces neurones sont les premiers à dégénérer dans la maladie, mais l’effet de la mHTT s’étend ensuite à d’autres régions du cerveau. Les mécanismes précis causant la dégénérescence sélective des neurones épineux de taille moyenne du striatum n’ont pas été totalement élucidés. La HTT saine est impliquée dans la régulation de nombreuses fonctions cellulaires, allant du métabolisme énergétique à la régulation de la transcription du génome. L’expansion de polyglutamine modifie la structure tridimensionnelle de la HTT, lui conférant de nouvelles propriétés toxiques (gain de fonctions pathologiques), et altérant partiellement certaines de ses fonctions normales (perte partielle de fonctions). Une des conséquences est l’apparition d’inclusions nucléaires ou neuritiques, marqueur typique de la pathologie, bien que la contribution de ces agrégats de HTT à la maladie reste controversée. Dans les stades précoces, la mHTT induit principalement des dysfonctionnements cellulaires majeurs, mais réversibles. Un traitement réduisant l’impact de la mHTT dans les premiers stades de la maladie permettrait donc de stopper la progression de cette dernière, mais aussi d’induire une récupération fonctionnelle. Les médicaments actuellement disponibles pour traiter la maladie de Huntington agissent sur les symptômes. Ces molécules vont des psychotropes généraux à des traitements plus ciblés. La tétrabénazine est, par exemple, utilisée pour son efficacité sur les symptômes moteurs [5]. Cependant, cette maladie reste incurable et la mort survient environ 15 à 20 ans après l’apparition des premiers signes. Le développement et la validation de stratégies thérapeutiques permettant de ralentir l’évolution de la pathologie restent un défi. Des approches neuroprotectrices basées sur la modulation de certaines voies intracellulaires par des molécules chimiques, par l’expression de facteurs trophiques ou par la transplantation de cellules embryonnaires [40], ont été développées ou sont actuellement en essais cliniques chez les patients [6–8]. Une stratégie particulièrement attractive pour le traitement de cette maladie héréditaire est basée sur le blocage de l’expression de la mHTT qui permettrait, en principe, de stopper la progression de la pathologie. |
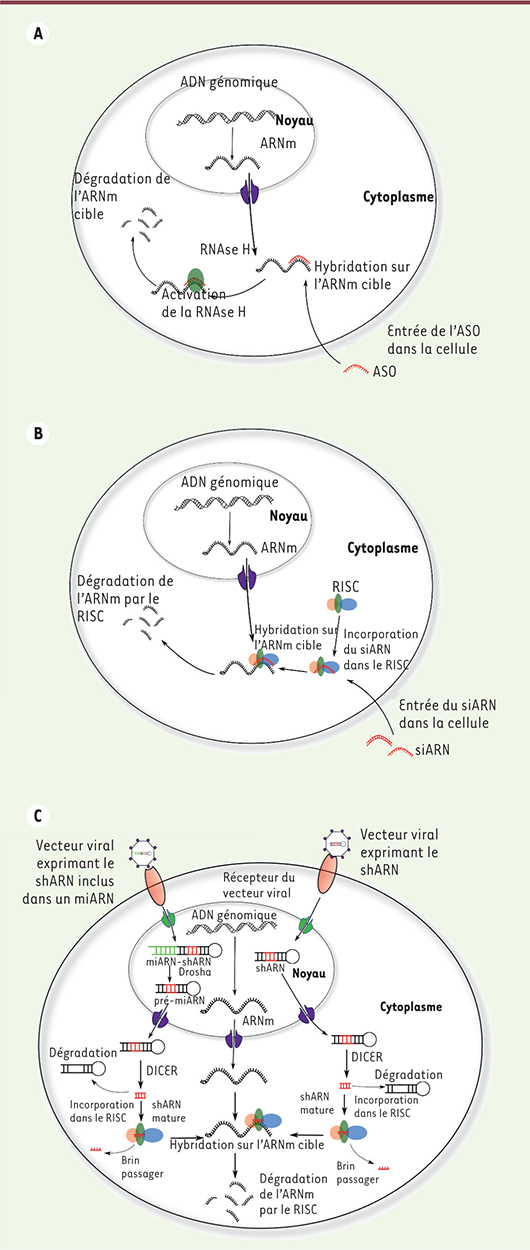
Acides nucléiques non codants pour le gene silencingL’approche thérapeutique de gene silencing est basée sur le transfert, dans la cellule, d’une petite séquence d’acides nucléiques synthétique (12-25 nucléotides d’ADN ou d’ARN simple- ou double-brins) capable de reconnaître un ARNm cible pour induire sa dégradation et, ainsi, bloquer la synthèse de la protéine correspondante. Deux approches de gene silencing ont été développées : l’une est basée sur la dégradation de l’ARNm par une RNAse endogène (antisense oligonucleotide [ASO, allele-specific oligonucleotide]), et l’autre sur l’ARN interférence (small-interfering RNA [siARN] et small-hairpin RNA [shARN]). Régulation de l’expression d’un gène par des acides nucléiques non codants exogènes
-
Les ASO sont des petites séquences synthétiques d’ADN simple brin de 12 à 25 nucléotides reconnaissant et liant un ARNm afin de moduler sa fonction (Figure 1A) [9]. Après hybridation de l’ASO, une enzyme endogène, la RNAse H, induit le clivage du complexe ADN-ARN, et entraîne une dégradation de l’ensemble de l’ARNm. Des modifications chimiques ont été apportées aux ASO afin d’augmenter leur demi-vie, et donc leur efficacité [10]. Des modifications de la structure du squelette ou des sucres présents dans les nucléotides ont été testées. En particulier, les modifications de squelette permettent d’augmenter la résistance des ASO aux nucléases cellulaires, alors que les modifications au niveau des sucres améliorent l’affinité du brin d’ADN pour l’ARNm cible. Cependant, ces changements peuvent induire une résistance de l’ASO à la RNAse H et, donc, modifier l’efficacité d’inactivation.
 | Figure 1.
Mécanismes d’action des ASO, des siARN et des shARN. A. Les ASO pénètrent dans la cellule grâce à la semi-perméabilité de la membrane plasmique. Une fois dans le cytoplasme, ils s’hybrident sur l’ARNm cible, ce qui active la RNAse H endogène et permet la dégradation de l’ARNm. B. L’entrée des siARN simple- ou double-brins dans la cellule se fait de la même façon que celle des ASO. Dans le cytoplasme, ils sont incorporés dans le complexe RISC et peuvent alors reconnaître l’ARNm cible. La dégradation de l’ARNm est alors produite grâce au RISC. C. L’entrée des shARN seuls ou inclus dans un miARN (miARN-shARN) se fait par l’interaction du vecteur viral avec son récepteur à la surface de la membrane plasmique. Le matériel génétique est alors transféré dans le noyau où il est transcrit. Dans le cadre des shARN, la tige-boucle transcrite est exportée vers le cytoplasme, où elle est clivée par l’enzyme Dicer. Une séquence double-brin contenant les 20 nucléotides complémentaires de l’ARNm cible est libérée et incorporée dans le complexe RISC, qui libère alors le brin passager pour ne conserver que la séquence complémentaire de l’ARNm cible. L’ARNm est dégradé par RISC après l’hybridation du shARN. La maturation cytoplasmique des miARN-shARN reste la même ; cependant, une étape supplémentaire a lieu dans le noyau. Après la transcription, le miARN-shARN est clivé par l’enzyme Drosha pour libérer un pré-miARN, qui est exporté vers le cytoplasme. |
-
Par opposition aux ASO, l’ARN interférence (ARNi) utilise d’autres mécanismes pour induire le silencing d’un gène. L’ARNi a été initialement découverte chez le pétunia en 1986. Ce n’est qu’au début des années 2000 que Craig Melo et Andrew Fire ont démontré l’existence d’ARNi chez les mammifères ; ils ont reçu le prix Nobel de physiologie et de médecine en 2006 pour cette découverte [41]. L’intérêt thérapeutique des petits ARN interférents a été rapidement reconnu et démontré avec les siARN et les shARN.
Les siARN sont des petites séquences d’ARN simple ou double brin d’environ 19-21 nucléotides ayant une action proche de celle des microARN (Figure 1B) [11]. Après leur introduction dans le cytoplasme de la cellule, les siARN sont reconnus par le complexe RISC (RNA-induced silencing complex) de la machinerie des microARN, s’hybrident sur l’ARNm mature du gène cible et induisent son clivage [42]. Tout comme les ASO, des modifications chimiques des séquences de nucléotides ont été testées afin d’augmenter la stabilité des molécules sans modifier leur mécanisme d’action. Cependant, l’administration des ASO/siARN dans le système nerveux central représente un défi majeur de par la présence de la barrière hémato-encéphalique (BHE) [38] et de la membrane plasmique des cellules. De plus, pour maintenir le bénéfice thérapeutique dans une pathologie progressive comme la maladie de Huntington, des injections répétées seraient nécessaires. Afin d’obtenir une expression continue, des systèmes basés sur des shARN artificiels ont été développés. Dans ce cas, la cellule cible produit elle-même le shARN thérapeutique. Ce dernier est synthétisé sous la forme d’une « tige-boucle » constitutivement exprimée par la cellule (Figure 1C) [12]. Pour cela, le shARN est transféré dans la cellule cible avec toutes les séquences nécessaires et, en particulier, un promoteur eucaryote permettant une expression stable et continue de la molécule. Le shARN est ensuite reconnu par la machinerie cellulaire (endoribonucléase Dicer) et le complexe RISC qui incorpore la séquence de 20 nucléotides complémentaire de celle de l’ARNm cible. Récemment, des shARN ont été incorporés dans le « squelette » d’un miARN afin de tirer parti de cette voie de synthèse et, ainsi, de permettre une maturation plus physiologique du shARN [13] Méthode de transfert de gènes dans le système nerveux central (SNC) Comme mentionné précédemment, l’administration d’ASO ou de si/shARN dans le cerveau représente un obstacle important au développement d’un traitement pour la maladie de Huntington. L’injection de matériel génétique en périphérie, par voie intraveineuse par exemple, nécessite l’utilisation de très hautes doses de molécules. De plus, ce procédé ne permet pas de sélectionner la zone cérébrale à traiter. Afin de contourner la barrière hémato-encéphalique [ 38] et d’améliorer la diffusion dans le système nerveux central, le matériel génétique peut être injecté directement dans la structure cérébrale à traiter (voie intraparenchymateuse) ou dans les ventricules cérébraux (voie intracérébroventriculaire, ICV). Dans le cas des ASO et des siARN, qui ne nécessitent pas de vecteurs d’expression, la solution la plus simple consiste à injecter la séquence génétique directement dans le cerveau. Grâce à la semi-perméabilité de la membrane plasmique, une quantité significative de matériel génétique est transférée dans la cellule cible. Afin d’augmenter la quantité d’ADN/ARN incorporée dans la cellule, les acides nucléiques peuvent être couplés à des composés chimiques pénétrant naturellement dans le cytoplasme, comme des liposomes ou le cholestérol, ce qui augmente la quantité de matériel transféré dans la cellule [ 14, 15]. Dans le cas des shARN, la séquence génétique doit être intégrée dans un vecteur d’expression permettant la transcription du shARN. Ce vecteur doit être transféré dans le noyau cellulaire, siège de l’expression des gènes. Des approches dites « virales » sont privilégiées pour le transfert des shARN dans le cerveau. Les virus ont la capacité naturelle de transférer du matériel génétique dans un vaste éventail de types cellulaires, indépendamment de l’espèce, de l’âge et du stade de développement [16] (Encadré 1). Parmi ceux-ci, les vecteurs dérivés des virus adéno-associés (AAV), des adénovirus (Ad) ou des lentivirus (LV) présentent un fort taux de transduction dans le cerveau et sont utilisés depuis plusieurs années pour le transfert de gènes [17]. |
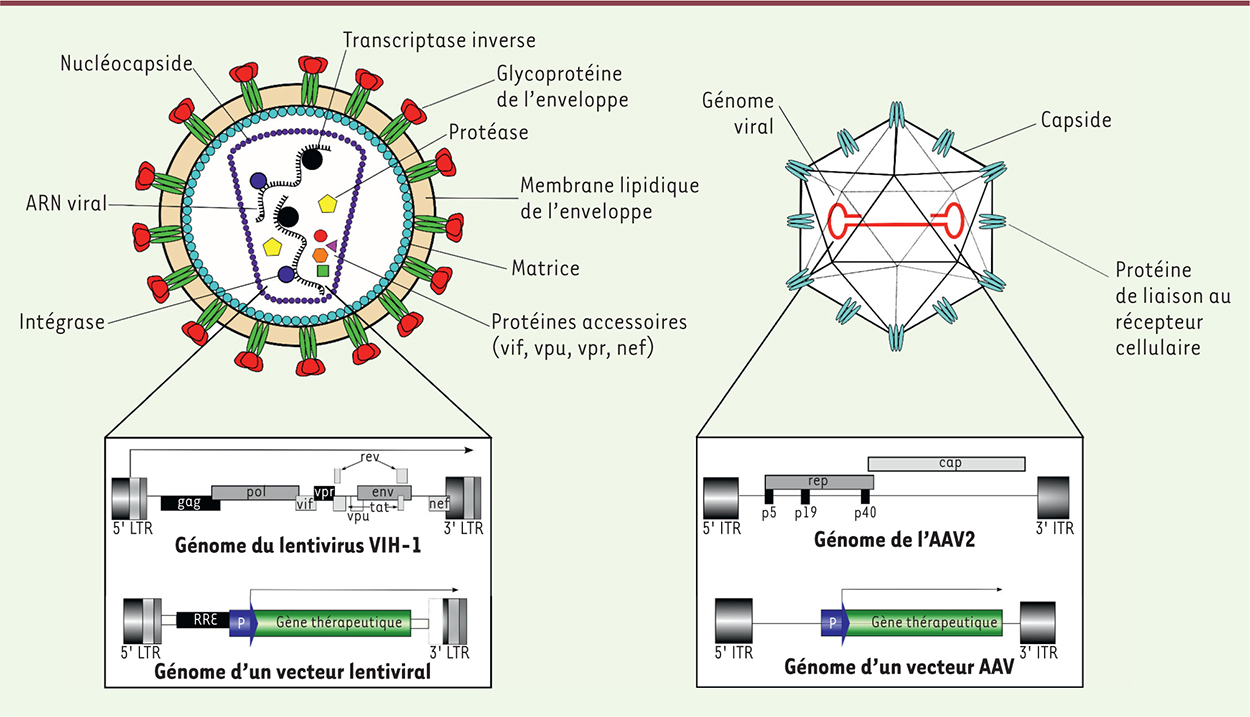
1. Les vecteurs viraux Les vecteurs viraux sont des virus génétiquement modifiés utilisés pour transporter une séquence nucléique dans une cellule. Un virus est une entité biologique composée d’une capside protéique et d’un génome viral contenu dans la capside (Figure 4). Parfois, la capside est elle-même encerclée par une enveloppe de glycoprotéines (virus enveloppés). Les protéines de la capside ou de l’enveloppe interagissent spécifiquement avec des protéines membranaires de certains types cellulaires et déterminent donc le tropisme du virus. Lors de l’infection, le virus transfère naturellement son génome viral dans la cellule hôte, qui peut alors exprimer les gènes codant pour les protéines structurales du virus (capside et/ou enveloppe) et les protéines essentielles à la multiplication virale. Les vecteurs viraux utilisent cette capacité des virus à transférer du matériel génétique dans un type cellulaire défini. Cependant, pour des raisons de biosécurité, l’intégralité des gènes viraux ont été éliminés et remplacés par le gène thérapeutique, tout en préservant les extrémités non codantes du génome (Figure 4) [16]. Ceci permet d’obtenir des vecteurs incapables de se répliquer par eux-mêmes. Dans la grande majorité des cas, la production des vecteurs viraux se fait in vitro en transférant dans les cellules une grande quantité d’ADN contenant le gène thérapeutique, ainsi que des séquences codant pour les protéines indispensables à la formation du virus (transfection transitoire). Ceci permet aux particules virales de s’assembler dans les cellules de production. Cependant, seuls les génomes contenant le transgène peuvent être encapsidés, assurant ainsi que les vecteurs produits ne contiennent que le gène thérapeutique. Les principaux vecteurs viraux pour le système nerveux central sont des vecteurs lentiviraux (LV) dérivés du virus de l’immunodéficience humaine, ou des vecteurs adéno-associés (AAV). Bien que présentant de nombreuses différences (virus enveloppé à ARN simple-brin de 10 kilobases (kb) pour le LV, virus non enveloppé à ADN simple-brin de 5-kb pour les AAV), AAV et LV présentent un fort taux de transduction dans le cerveau de multiples espèces y compris l’humain. L’application de stratégies basées sur les modifications d’enveloppes/sérotypes, sur la sélection de promoteurs spécifiques de certains types cellulaires et sur l’ajout d’éléments de régulation post-transcriptionnelle de l’expression du transgène, a permis de restreindre le tropisme de ces vecteurs pour un type cellulaire particulier, et permet donc de choisir le type cellulaire à traiter.
|
|
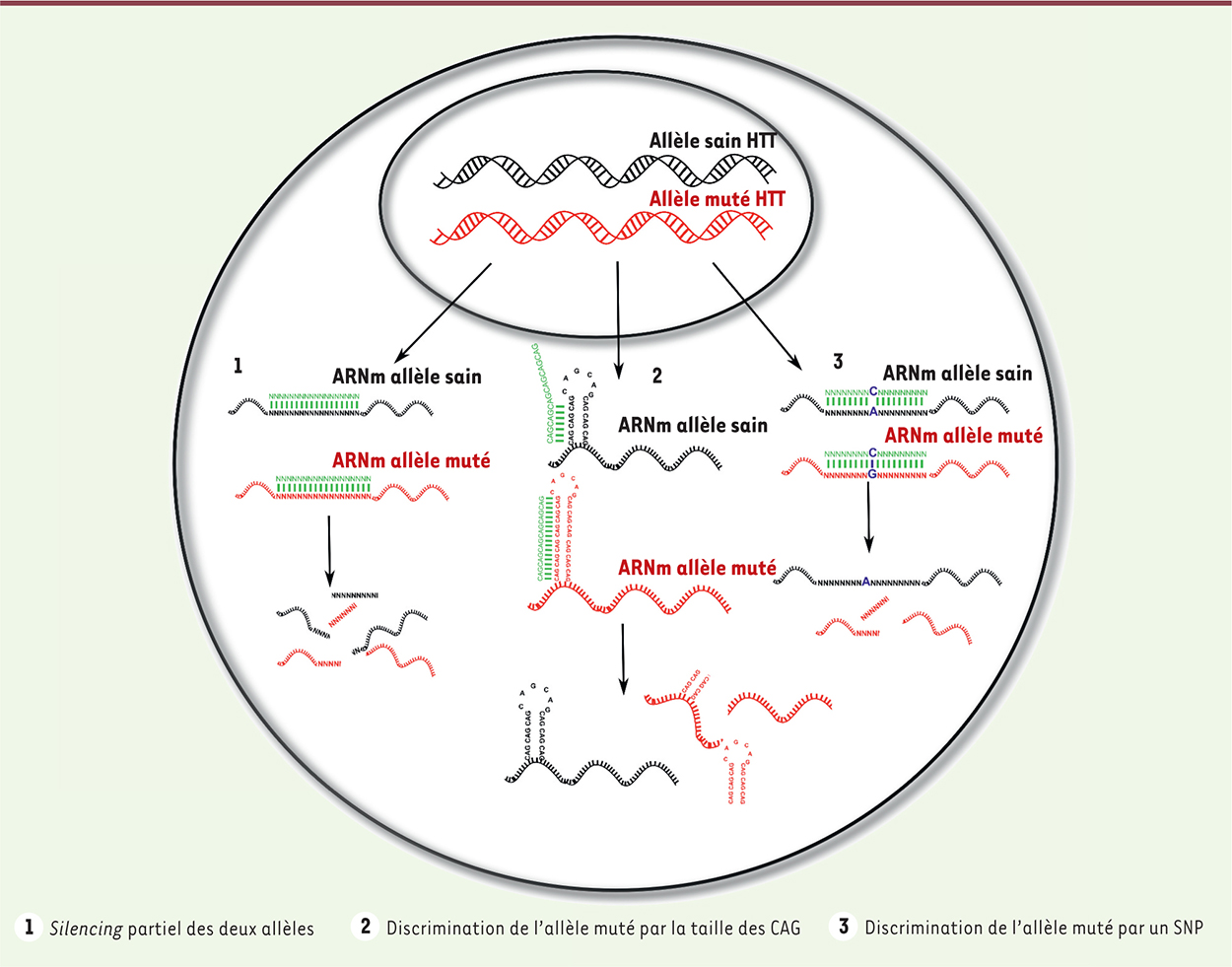
Vers un blocage de l’expression de la mHTT La plupart des patients atteints de maladie de Huntington sont hétérozygotes pour la mutation, ce qui signifie qu’un seul des deux allèles codant pour la HTT est muté. Les deux allèles du gène ne différent que par la longueur de l’expansion CAG et par la présence de variations ponctuelles d’un nucléotide entre les deux séquences (single nucleotide polymorphisms, SNP). La plupart des approches à ce jour ne discriminent pas les deux transcrits de l’HTT et induisent une baisse partielle de l’expression des deux allèles (Figure 2). Récemment, des stratégies permettant la reconnaissance exclusive de la mHTT grâce à la longueur de l’expansion CAG ou à certains SNP ont conduit à un silencing allélique de la huntingtine mutée [18].
 | Figure 2.
Stratégies de silencing de la mHTT. Trois stratégies de silencing de la mHTT ont été développées : les approches de silencing partiel des deux allèles (1), les approches discriminant l’allèle muté par la taille des CAG (2) et les approches discriminant l’allèle muté par un SNP (3). Dans la première approche, la séquence nucléique reconnaît et permet la dégradation aussi bien de l’allèle sain que de l’allèle muté. Les approches discriminant les deux allèles par la taille des CAG se basent sur le fait que l’hybridation de la séquence nucléique sera incomplète sur l’allèle sain du fait du faible nombre de CAG. Cela a pour conséquence d’introduire des non-homologies de séquences avec l’ARNm de l’HTT saine et d’inhiber la dégradation de l’ARNm. Enfin, la troisième approche permet un silencing sélectif des allèles de la mHTT en se servant de SNP différenciant les deux allèles. Le nucléotide du SNP étant différent entre l’allèle sain et muté, une non-homologie de séquence est introduite avec l’allèle sain, ce qui le préserve de la dégradation. |
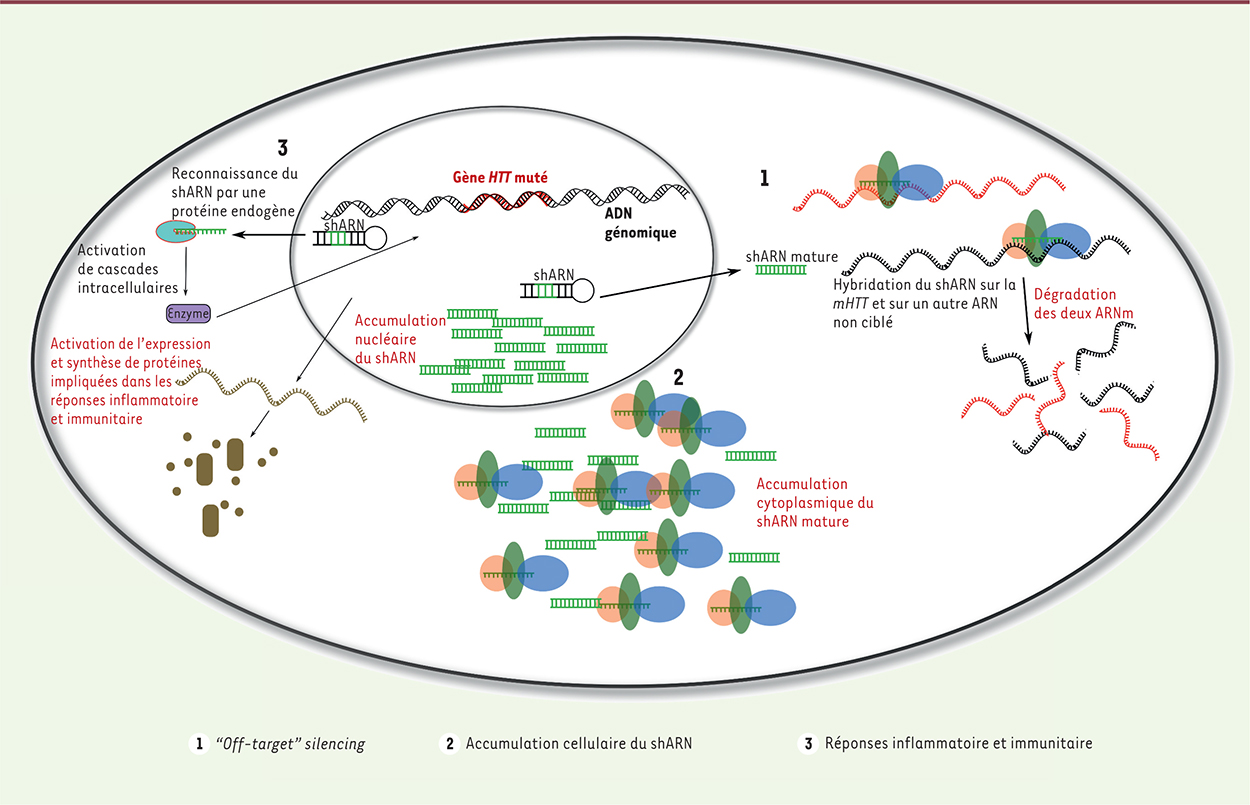
Approches recherchant une baisse partielle de la HTT Une des premières utilisations du gene silencing pour la maladie de Huntington a été décrite en 2005 (Figure 2) [ 14]. Un siARN ciblant l’ARNm de la mHTT humaine a été injecté par voie intracérébroventriculaire dans des souris transgéniques modèles de la maladie peu de temps après leur naissance. Une baisse de l’expression de la mHTT a été observée jusqu’à huit semaines, ainsi qu’une diminution du nombre d’agrégats de mHTT, une amélioration motrice et une extension de la durée de vie des souris [ 19]. Cette première preuve de principe a montré l’intérêt de l’approche, même si le traitement était transitoire. D’autres travaux, basés sur l’injection d’ASO ou de siARN visant la mHTT chez la souris adulte, ont confirmé l’intérêt du silencing dans cette indication [ 9, 15]. Une seule administration d’ASO en intracérébroventriculaire assure une large distribution dans le cerveau, et un silencing qui atteint plus de 60 % du taux de mHTT dans certaines régions. L’expression de la HTT retrouve son niveau basal 16 semaines après l’administration, mais les effets thérapeutiques persistent durant cinq mois. Ceci révèle qu’une baisse transitoire de l’expression de la mHTT induit des effets bénéfiques à moyen terme. Cependant, il a été observé que les molécules diffusent moins bien dans les couches profondes du cerveau, dont le striatum, après une injection par voie intracérébroventriculaire [ 20, 21] Des résultats similaires ont été obtenus avec des vecteurs AAV exprimant un shARN ou un miARN-shARN. Une réduction de l’expression de la mHTT dès les premières semaines post-infection a été observée dans le striatum de différents modèles animaux de la maladie [15, 22–27]. Cependant, contrairement aux approches basées sur les siARN et les ASO, l’intégration des shARN dans des vecteurs permet d’obtenir une expression constitutive et stable dans le temps, avec un blocage de l’expression de la mHTT et des effets thérapeutiques persistant jusqu’à neuf mois post-infection [27]. De plus, ces études ont montré qu’une baisse partielle de l’expression de la HTT saine ne semble pas induire d’effets délétères, et qu’un silencing partiel de la mHTT est suffisant pour procurer des effets thérapeutiques. La biosécurité et la tolérance d’un silencing partiel de l’HTT restent à démontrer, mais les premiers résultats chez le primate non humain montrent que cette approche est envisageable chez l’homme [28]. Silencing sélectif de l’allèle muté La grande similitude de séquence entre l’allèle sain et muté de la HTT limite considérablement les options permettant de discriminer les deux allèles. Cependant, des siARN et des ASO reconnaissant sélectivement les longues répétitions CAG ont été synthétisés (Figure 2) [ 11, 18]. Certaines de ces molécules permettent de discriminer les deux allèles de la HTT tout en conservant une bonne efficacité thérapeutique. Il a été observé que la sélectivité pour l’allèle muté diminue pour les formes les plus communément observées (40-45 CAG) du fait du nombre moins important de triplets [ 9]. De plus, ces molécules sont potentiellement capables de reconnaître les ARNm d’autres gènes contenant des expansions CAG (comme les transcrits de l’ataxine 1 ou de l’atrophine 1), ce qui pourrait induire des effets secondaires. Une autre stratégie pour obtenir un silencing sélectif de la mHTT est basée sur la présence de SNP dans le gène HTT (Figure 2). Ils sont au nombre d’environ 2 000 pour ce gène, dont certains ont une haute fréquence d’hétérozygotie et permettraient de traiter 75-88 % des patients si 3-5 SNP étaient ciblés [ 29– 31]. Dès lors, ces SNP peuvent être utilisés pour introduire des non-homologies de séquences ( mismatches) entre l’ASO/siARN/shARN et l’allèle HTT sain, ce qui prévient la dégradation de l’ARNm de l’allèle sain. Des ASO et des siARN chimiquement modifiés visant ces SNP permettent de baisser de 39 à 68 % l’expression de la mHTT en ayant un impact minimal sur l’allèle sain. Ces ASO et siARN spécifiques des allèles sont actuellement testés dans des études précliniques [ 9, 11, 32]. L’application de cette même stratégie avec un lentivirus exprimant un shARN ciblant sélectivement la mHTT humaine permet de réduire l’expression de la protéine mutée in vivo de près de 40-70 % dans un modèle animal de la maladie [ 33]. Une diminution des marqueurs de la pathologie a été observée consécutivement au traitement, et des améliorations fonctionnelles, dont une normalisation du trafic vésiculaire, ont été démontrées à la fois dans des modèles animaux et dans des cellules de patients. Ces premières données mettent en évidence la faisabilité, l’efficacité et la sélectivité d’un silencing de la mHTT. Cependant, ces approches nécessitent de génotyper les patients afin d’identifier les SNP qui leur sont spécifiques et de développer les produits couvrant l’ensemble de ces SNP. Effets secondaires du silencing de la HTT La principale difficulté d’une stratégie thérapeutique basée sur le gene silencing reste la spécificité du traitement. Trois types d’effets indésirables peuvent survenir : la présence d’effets hors cibles ( off-targets effects), la saturation de la machinerie cellulaire et la mise en place de réponses immunitaires/inflammatoires contre la séquence nucléique (Figure 3).
 | Figure 3.
Effets secondaires potentiels du gene silencing par un shARN. Il existe trois effets secondaires possibles du silencing de la mHTT : les effets hors cibles (off-targets effects) (1), la saturation de la machinerie cellulaire (2) et les réponses inflammatoires/immunitaires (3). Les effets hors cibles surviennent si une homologie partielle ou complète est observée entre la séquence nucléique (ici shARN) et un ARNm non ciblé. Cela entraîne une dégradation à la fois de l’ARNm cible et non ciblé. La saturation de la machinerie cellulaire survient quand une trop forte quantité de shARN est exprimée. La cellule ne peut alors plus effectuer ses propres régulations physiologiques. Enfin, une réponse immunologique de l’organisme pourrait survenir lors de l’expression d’une séquence nucléique. Le shARN est reconnu par une protéine cellulaire, ce qui conduit à l’activation de cascades intracellulaires favorisant la mise en place de mécanismes inflammatoires et de défense immunitaire. |
 | Figure 4.
Représentation d’un vecteur lentiviral dérivé du VIH-1 (virus de l’immunodéficience humaine 1) et d’un vecteur adéno-associé. A. Les vecteurs lentiviraux sont des virus enveloppés contenant au sein de leur capside deux molécules d’ARN simple brin composant le génome viral. En complément du matériel génétique, la capside contient les protéines impliquées dans la synthèse d’ADN complémentaire (ADNc) à partir de l’ARN viral (transcriptase inverse et protéase) et dans l’intégration de l’ADNc dans le génome de l’hôte (intégrase). L’ensemble des protéines accessoires sont également présentes. Le génome du VIH-1 contient des gènes codant pour les protéines impliquées dans le cycle viral (pol, tat, rev), les protéines structurales de la capside (gag), les protéines de l’enveloppe (env) et les protéines accessoires. Aux extrémités du génome, deux régions répétées (5’ LTR et 3 ’LTR) contiennent le promoteur viral (flèche) et le signal de fin de transcription. Pour construire un vecteur lentiviral, l’ensemble des gènes viraux sont supprimés et remplacés par le gène thérapeutique contrôlé par un promoteur eucaryote (P). Seuls les LTR et le site RRE (impliqué dans l’épissage alternatif du génome du vecteur lentiviral lors de la production) sont préservés. Cependant, pour assurer que la transcription ne s’initie qu’à partir du promoteur P, une partie du 3’ LTR contenant le promoteur interne du VIH-1 a été supprimée (lignes pointillées), ce qui permet d’augmenter la biosécurité de ces vecteurs viraux. B. Les vecteurs AAV sont issus de virus non enveloppés contenant uniquement le génome viral à l’intérieur de la capside. Ce génome est composé de deux répétitions terminales inversées (5’ ITR et 3’ ITR) et de deux cassettes d’expression (rep et cap) contrôlées par trois promoteurs (p5, p19 et p40) codant pour l’ensemble des gènes indispensables à la multiplication du virus. L’ensemble de ces éléments génétiques sont supprimés dans les vecteurs AAV et remplacé par le gène thérapeutique sous le contrôle d’un promoteur hétérologue. Cette configuration assure une grande biosécurité de ces vecteurs qui ne peuvent pas se multiplier par eux-mêmes. |
-
Il est important de contrôler la présence d’effets hors cibles, c’est-à-dire le silencing d’ARNm présentant une homologie partielle avec le shARN-HTT, ce qui induirait une hybridation partielle ou complète de la molécule thérapeutique avec ces ARNm. Des modèles mathématiques permettent d’estimer la probabilité de cibles indésirables d’une séquence nucléique [34]. Cependant, une potentielle homologie de séquence mise en évidence par ces algorithmes ne se traduit pas forcément par le silencing du gène dans une cellule. De plus, ces effets hors cibles sont dépendants de la séquence et de l’espèce ; des études précliniques dans des modèles proches de l’humain sont nécessaires pour réduire leur probabilité d’apparition.
-
Une trop forte dose d’un ASO/siARN/shARN peut également conduire à la saturation de la machinerie cellulaire, induisant une forte toxicité [35].
-
Enfin, la présence dans la cellule de matériel génétique étranger peut induire une réponse inflammatoire ou immunitaire. Cela a déjà été observé dans les organes périphériques, mais reste peu présent dans le système nerveux central du fait de la protection du cerveau par la barrière hémato-encéphalique [36]. Cependant, une quantité anormale de matériel génétique dans le cerveau peut conduire à l’expression par les neurones de protéines reconnues par le système immunitaire, et donc à une destruction des cellules traitées par l’organisme [37]. Il est donc primordial de bien contrôler ces paramètres, in vitro dans des cellules humaines, et in vivo dans des modèles primates, plus proches de l’homme, pour ces aspects de spécificité de séquence et de réponse de l’organisme au produit thérapeutique.
|
Le développement de petits ARN non codants a permis de mettre en place des stratégies de silencing efficaces pour la maladie de Huntington Les études précliniques menées à ce jour montrent qu’un blocage partiel de l’expression de l’HTT mutée permettrait de réduire significativement la progression de marqueurs pathologiques et d’améliorer la survie des neurones du striatum. À ce jour, l’efficacité du silencing reste équivalente que les approches soient spécifiques ou non spécifiques des allèles, suggérant que l’une ou l’autre de ces stratégies pourrait être appliquée en clinique. Bien que la HTT soit indispensable durant le développement embryonnaire, le silencing partiel des allèles sain et muté ne semble pas conduire à des déficits particuliers chez l’adulte. Une approche de silencing non spécifique des allèles semble donc envisageable, et des études sont en cours pour vérifier la biosécurité de l’approche. Un traitement spécifique d’allèle basé sur les 3-5 SNP permettrait de traiter 80-90 % des patients, mais nécessiterait de génotyper chaque sujet et de développer un traitement pour chacun des SNP. Une telle approche thérapeutique impliquerait l’évaluation de 6 à 10 traitements indépendamment. Nonobstant les contraintes pour le développement de ces traitements, le choix de cette stratégie permettrait de passer d’une médecine globale, dont l’approche thérapeutique cible tous les patients, à une médecine spécialisée, personnelle, basée sur l’élaboration d’une molécule spécifique à un patient et permettant de minimiser l’impact sur l’expression de l’HTT saine. Considérant l’absence d’effets secondaires suggérés par le silencing partiel de l’HTT saine chez le primate, une approche non spécifique des allèles pourrait donc être envisagée en complément pour les 10-20 % des patients ne pouvant bénéficier du traitement spécifique des allèles. À ce jour, aucune différence d’efficacité n’a été observée entre les ASO, siARN et les shARN pour le traitement de la maladie de Huntington. Le recul obtenu dans les différents essais cliniques réalisés avec des ASO/siARN pour d’autres maladies [43] conforte ces approches et suggère qu’un silencing global de l’HTT avec un ASO/siARN pourrait être évalué chez l’humain d’ici quelques années. Cependant, les shARN délivrés par un vecteur viral ont la capacité de s’exprimer à long terme, et donc d’induire un effet thérapeutique sans nécessiter de traitements répétés. Cet avantage, combiné à la tolérance du cerveau humain aux injections de vecteurs viraux, suggère que cette stratégie permettrait d’induire des effets thérapeutiques sur plusieurs années chez le patient et, peut-être, d’altérer significativement la progression de la maladie de Huntington. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Glossaire
|
ARN interférence (ARNi) :
|
principe par lequel des petits ARN non codants bloquent l’expression d’un gène en dégradant son ARN messager en utilisant la machinerie cellulaire des microARN. |
|
Barrière hémato-encéphalique (BHE) :
|
barrière physiologique présente chez tous les vertébrés au niveau des vaisseaux sanguins permettant une séparation physique entre le système nerveux central et la circulation sanguine. La BHE permet de filtrer les molécules et cellules entrant dans le cerveau afin de protéger les neurones de la périphérie. |
|
Complexe RISC (RNA-induced silencing complex) :
|
complexe de protéines impliquées dans la machinerie des miARN. RISC reconnaît et incorpore les miARN, ce qui permet l’hybridation entre le miARN et l’ARNm cible. Suite à cette liaison, RISC induit la dégradation de l’ARNm ou bloque sa traduction. Le complexe RISC est également impliqué dans le fonctionnement des siARN et des shARN. |
|
MicroARN (miARN) :
|
petits ARN non codants endogènes de 20 nucléotides impliqués dans la régulation fine de l’expression des gènes. Les miARN sont d’abord reconnus et chargés dans un complexe protéique, le RNA-induced silencing complex (RISC), pour ensuite s’hybrider dans la région 3’ non traduite des gènes cibles, afin d’induire leur dégradation ou le blocage de la traduction. |
|
Neurone épineux de taille moyenne (MSN, medium-spiny neurons) :
|
principal type de neurones (90-95 %) présent dans le striatum des mammifères. Les MSN présentent un soma de 20-25 µm avec des dendrites épineuses de 200 à 400 µm recevant des afférences principalement corticales et nigrales, et un axone de 200 à 400 µm projetant vers le globus pallidus et la substance noire réticulée chez les rongeurs. Les MSN sont impliqués dans la régulation fine de la coordination motrice. |
|
Off-target silencing :
|
effet secondaire du silencing dû à la reconnaissance et la dégradation par la séquence nucléique d’un ARNm non ciblé. |
|
Single nucleotide polymorphism (SNP) :
|
variations d’un nucléotide entre deux allèles d’un même gène permettant une discrimination allélique. |
|
1.
Walker FO. Huntington’s disease . Lancet. 2007; ; 369 : :218.–228. 2.
The Huntington’s disease collaborative research group . A novel gene containing a trinucleotide repeat that is expanded and unstable on Huntington’s disease chromosomes . Cell. 1993; ; 72 : :971.–983. 3.
Youssov K, Dolbeau G, Maison P, et al. Unified Huntington’s disease rating scale for advanced patients: validation and follow-up study . Mov Disord. 2013; ; 28 : :1717.–1723. 4.
Vonsattel JP, Myers RH, Stevens TJ, et al. Neuropathological classification of Huntington’s disease . J Neuropathol Exp Neurol. 1985; ; 44 : :559.–577. 5.
Ross CA, Tabrizi SJ. Huntington’s disease: from molecular pathogenesis to clinical treatment . Lancet Neurol. 2011; ; 10 : :83.–98. 6.
Saudou F, Humbert S. La cystéamine rétablit les dynamiques intracellulaires et la sécrétion du BDNF dans la maladie de Huntington . Med Sci (Paris). 2006; ; 22 : :906.–908. 7.
Bloch J, Bachoud-Levi AC, Deglon N, et al. Neuroprotective gene therapy for Huntington’s disease, using polymer-encapsulated cells engineered to secrete human ciliary neurotrophic factor: results of a phase I study . Hum Gene Ther. 2004; ; 15 : :968.–975. 8.
Bachoud-Levi AC, Gaura V, Brugieres P, et al. Effect of fetal neural transplants in patients with Huntington’s disease 6 years after surgery: a long-term follow-up study . Lancet Neurol. 2006; ; 5 : :303.–309. 9.
Southwell AL, Skotte NH, Bennett CF, Hayden MR. Antisense oligonucleotide therapeutics for inherited neurodegenerative diseases . Trends Mol Med. 2012; ; 18 : :634.–643. 10.
Deleavey GF, Damha MJ. Designing chemically modified oligonucleotides for targeted gene silencing . Chem Biol. 2012; ; 19 : :937.–954. 11.
Yu D, Pendergraff H, Liu J, et al. Single-stranded RNAs use RNAi to potently and allele-selectively inhibit mutant huntingtin expression . Cell. 2012; ; 150 : :895.–908. 12.
Boudreau RL, Davidson BL. RNAi therapeutics for CNS disorders . Brain Res. 2010; ; 1338 : :112.–121. 13.
Shin KJ, Wall EA, Zavzavadjian JR, et al. A single lentiviral vector platform for microRNA-based conditional RNA interference and coordinated transgene expression . Proc Natl Acad Sci USA. 2006; ; 103 : :13759.–13764. 14.
Wang YL, Liu W, Wada E, et al. Clinico-pathological rescue of a model mouse of Huntington’s disease by siRNA . Neurosci Res. 2005; ; 53 : :241.–249. 15.
DiFiglia M, Sena-Esteves M, Chase K, et al. Therapeutic silencing of mutant huntingtin with siRNA attenuates striatal and cortical neuropathology and behavioral deficits . Proc Natl Acad Sci USA. 2007; ; 104 : :17204.–17209. 16.
Kay MA, Glorioso JC, Naldini L. Viral vectors for gene therapy: the art of turning infectious agents into vehicles of therapeutics . Nat Med. 2001; ; 7 : :33.–40. 17.
Lentz TB, Gray SJ, Samulski RJ. Viral vectors for gene delivery to the central nervous system . Neurobiol Dis. 2012; ; 48 : :179.–188. 18.
Zhang Y, Friedlander RM. Using non-coding small RNAs to develop therapies for Huntington’s disease . Gene Ther. 2011; ; 18 : :1139.–1149. 19.
Crook ZR, Housman D. Huntington’s disease: can mice lead the way to treatment? Neuron. 2011; ; 69 : :423.–435. 20.
Kordasiewicz HB, Stanek LM, Wancewicz EV, et al. Sustained therapeutic reversal of Huntington’s disease by transient repression of huntingtin synthesis . Neuron. 2012; ; 74 : :1031.–1044. 21.
Davidson BL. Taking a break from huntingtin . Mol Ther Nucleic Acids. 2012;; 1; doi: 10.1038/mtna. 22.
Harper SQ, Staber PD, He X, et al. RNA interference improves motor and neuropathological abnormalities in a Huntington’s disease mouse model . Proc Natl Acad Sci USA. 2005; ; 102 : :5820.–5825. 23.
Rodriguez-Lebron E, Denovan-Wright EM, Nash K, et al. Intrastriatal rAAV-mediated delivery of anti-huntingtin shRNAs induces partial reversal of disease progression in R6/1 Huntington’s disease transgenic mice . Mol Ther. 2005; ; 12 : :618.–633. 24.
Boudreau RL, McBride JL, Martins I, et al. Nonallele-specific silencing of mutant and wild-type huntingtin demonstrates therapeutic efficacy in Huntington’s disease mice . Mol Ther. 2009; ; 17 : :1053.–1063. 25.
Stanek LM, Sardi SP, Mastis B, et al. Silencing mutant huntingtin by adeno-associated virus-mediated RNA interference ameliorates disease manifestations in the YAC128 mouse model of Huntington’s disease . Hum Gene Ther. 2014; ; 25 : :461.–474. 26.
Franich NR, Fitzsimons HL, Fong DM, et al. AAV vector-mediated RNAi of mutant huntingtin expression is neuroprotective in a novel genetic rat model of Huntington’s disease . Mol Ther. 2008; ; 16 : :947.–956. 27.
Drouet V, Perrin V, Hassig R, et al. Sustained effects of nonallele-specific Huntingtin silencing . Ann Neurol. 2009; ; 65 : :276.–285. 28.
McBride JL, Pitzer MR, Boudreau RL, et al. Preclinical safety of RNAi-mediated HTT suppression in the rhesus macaque as a potential therapy for Huntington’s disease . Mol Ther. 2011; ; 19 : :2152.–2162. 29.
Pfister EL, Kennington L, Straubhaar J, et al. Five siRNAs targeting three SNPs may provide therapy for three-quarters of Huntington’s disease patients . Curr Biol. 2009; ; 19 : :774.–778. 30.
Lombardi MS, Jaspers L, Spronkmans C, et al. A majority of Huntington’s disease patients may be treatable by individualized allele-specific RNA interference . Exp Neurol. 2009; ; 217 : :312.–319. 31.
Sah DW, Aronin N. Oligonucleotide therapeutic approaches for Huntington disease . J Clin Invest. 2011; ; 121 : :500.–507. 32.
Ostergaard ME, Southwell AL, Kordasiewicz H, et al. Rational design of antisense oligonucleotides targeting single nucleotide polymorphisms for potent and allele selective suppression of mutant Huntingtin in the CNS . Nucleic Acids Res. 2013; ; 41 : :9634.–9650. 33.
Drouet V, Ruiz M, Zala D, et al. Allele-specific silencing of mutant huntingtin in rodent brain and human stem cells . PloS One. 2014; ; 9 : :e99341.. 34.
Naito Y, Yoshimura J, Morishita S, Ui-Tei K. siDirect 2.0: updated software for designing functional siRNA with reduced seed-dependent off-target effect . BMC Bioinformatics. 2009;; 10 : :392.. 35.
Grimm D, Streetz KL, Jopling CL, et al. Fatality in mice due to oversaturation of cellular microRNA/short hairpin RNA pathways . Nature. 2006; ; 441 : :537.–541. 36.
Judge AD, Sood V, Shaw JR, et al. Sequence-dependent stimulation of the mammalian innate immune response by synthetic siRNA . Nat Biotechnol. 2005; ; 23 : :457.–462. 37.
Davidson BL, Boudreau RL. RNA interference: a tool for querying nervous system function and an emerging therapy . Neuron. 2007; ; 53 : :781.–788. 38.
Gosselet F, Candela P, Cecchelli R, et al. La barrière hémato-encéphalique. Med Sci (Paris). 2011; ; 27 : :987.–992. 39.
Lebre AS, Brice A. Maladies par expansion de polyglutamine : données moléculaires et physiopathologiques . Med Sci (Paris). 2001; ; 17 : :1149.–1157. 40.
Aubry L, Peschanski M, Perrier A. Des cellules souches embryonnaires humaines pour la thérapie cellulaire de la maladie de Huntington . Med Sci (Paris). 2009; ; 25 : :333.–335. 41.
Harel-Bellan A.. Prix Nobel de médecine 2006: Andrew Z. Fire et Craig C. Mello. Silence, on désactive les gènes . Med Sci (Paris). 2006; ; 22 : :993.–994. 42.
Dunoyer P. La bataille du silence. Med Sci (Paris). 2009; ; 25 : :505.–512. 43.
Adams D, Lozeron P, Algalarrondo V. Utilisation de l’ARN interférence dans le traitement de l’amylose héréditaire due à une mutation de la transthyrétine . Med Sci (Paris). 2014; ; 30 : :345.–347. |