| |
| Med Sci (Paris). 2015 March; 31(3): 236–238. Published online 2015 April 8. doi: 10.1051/medsci/20153103003.Projet européen Blueprint
Vers une caractérisation épigénomique du système immunitaire Lydie C. Pradel,1,2 Laurent Vanhille,1,2 and Salvatore Spicuglia1,2* 1Inserm, UMR1090 TAGC, Techniques avancées pour la génomique et la clinique, 163, avenue de Luminy, Marseille, F-13288, France 2Aix-Marseille Université, UMR1090 TAGC, Marseille, F-13288, France MeSH keywords: Reprogrammation cellulaire, Cartographie chromosomique, Épigénomique, Europe, Glucose, Humains, Système immunitaire, Tolérance immunitaire, Immunité innée, Coopération internationale, Voies et réseaux métaboliques, génétique, méthodes, organisation et administration, métabolisme |
Définition des cartes épigénomiques : le consortium Blueprint
Malgré la diversité des types cellulaires dans le corps, chaque cellule a la même information génétique. En effet, le décodage de l’information génétique varie selon le type cellulaire, les signaux extérieurs perçus et le développement d’éventuelles pathologies. Cette différence est en partie déterminée par la façon dont l’ADN est organisé dans les chromosomes. Ainsi, l’épigénétique s’intéresse aux changements d’état de la transcription des gènes ; ces changements sont héritables au cours des divisions cellulaires, mais n’impliquent aucune modification de la séquence d’ADN génomique. Au niveau moléculaire, la régulation épigénétique est reflétée par des modifications constitutives au niveau de la chromatine, structure constituée de l’ADN enroulé autour des protéines histones. D’une part, la méthylation de l’ADN est généralement associée à un état réprimé. D’autre part, l’analyse des combinaisons de modifications post-traductionnelles au niveau des histones permet d’identifier très précisément la nature des séquences (éléments régulateurs, gènes, etc.) mais aussi leur état (activé, réprimé). Grâce à l’essor du séquençage à haut débit, il est maintenant possible de caractériser l’ensemble de ces modifications à l’échelle du génome, et ceci pour chaque type cellulaire d’intérêt ; on parle alors de cartographie épigénomique. En Europe, le consortium Blueprint (www.blueprint-epigenome.eu) vise à définir, chez l’homme, les cartes épigénomiques et les profils de transcription d’une grande variété de cellules primaires saines et néoplasiques du sang [1, 2]. Il s’agit d’un projet d’envergure financé par la communauté européenne et regroupant 41 instituts et laboratoires de recherche qui collaborent avec des partenaires industriels. Ce projet, coordonné par le Pr. Hendrik Stunnenberg à l’université de Radboud (Nijmegen, Pays-Bas), s’est donné comme objectif principal de déchiffrer l’épigénome de plus de 100 types cellulaires hématopoïétiques différents. Dans l’édition du journal Science du 26 septembre 2014, trois publications du consortium Blueprint illustrent l’utilité de ce type d’approche et les implications possibles en termes de santé publique [3–6]. En particulier, deux de ces études révèlent l’existence de programmes épigénétiques distincts selon la nature des stimulus induisant la différenciation des monocytes en macrophages, acteurs essentiels de l’immunité innée [4, 5]. |
Reprogrammation épigénétique et mémoire immunitaire innée Les monocytes et les macrophages sont des phagocytes mononucléés du système immunitaire inné. Ils reconnaissent des molécules simples à la surface des pathogènes et agissent rapidement pour éliminer ces derniers. Les monocytes circulants ont un rôle clé au cours d’une infection. Ils peuvent infiltrer le tissu infecté et se différencier en macrophages : ceux-ci élimineront l’infection en phagocytant les pathogènes et en recrutant les cellules du système immunitaire adaptatif, notamment en induisant une inflammation locale. Les macrophages résidents des tissus ont, quant à eux, un rôle important au cours du développement et du contrôle de l’homéostasie, ainsi que dans la résolution de l’inflammation. Traditionnellement, l’action de ces cellules était considérée comme aspécifique et n’impliquant pas de mémoire immunologique, contrairement à celle que développent les cellules de l’immunité adaptative. Toutefois, il a été remarqué que des vaccins ciblant des pathogènes spécifiques protégeaient les souris d’autres infections microbiennes, suggérant l’intervention d’une forme de mémoire immune innée [7]. En effet, durant certaines infections (comme avec Candida albicans), ou en réponse à une exposition courte à certains stimulus, le système immunitaire inné peut répondre plus fortement à un second stimulus. La réponse au premier stimulus décline rapidement une fois l’infection résolue. Cependant, au décours de l’infection, la réponse immune innée ne revient pas à son état basal initial, et les cellules demeurent activées pendant une période plus ou moins longue, et ce grâce à une « reprogrammation » fonctionnelle épigénomique. Cette reprogrammation a notamment été identifiée dans les macrophages. Une étude récente a ainsi identifié des éléments cis-régulateurs cryptiques que seul un stimulus donné rendait accessibles, mais qui se maintenaient dans une conformation ouverte même après extinction du stimulus [8]. Cela représente donc une mémoire de facto de l’immunité innée, d’où l’apparition récente du terme « immunité innée éduquée ». Au contraire, au cours de certaines infections et du sepsis sévère, après une forte réponse à un premier stimulus, le système immun inné non seulement ne retrouve pas son activité basale, mais celle-ci est négativement modulée. Cette période d’activité réduite est dite « tolérante ». Les cellules réagissent alors beaucoup moins efficacement à une seconde invasion de pathogènes et l’hôte est plus vulnérable ; on parle alors d’immunoparalysie. |
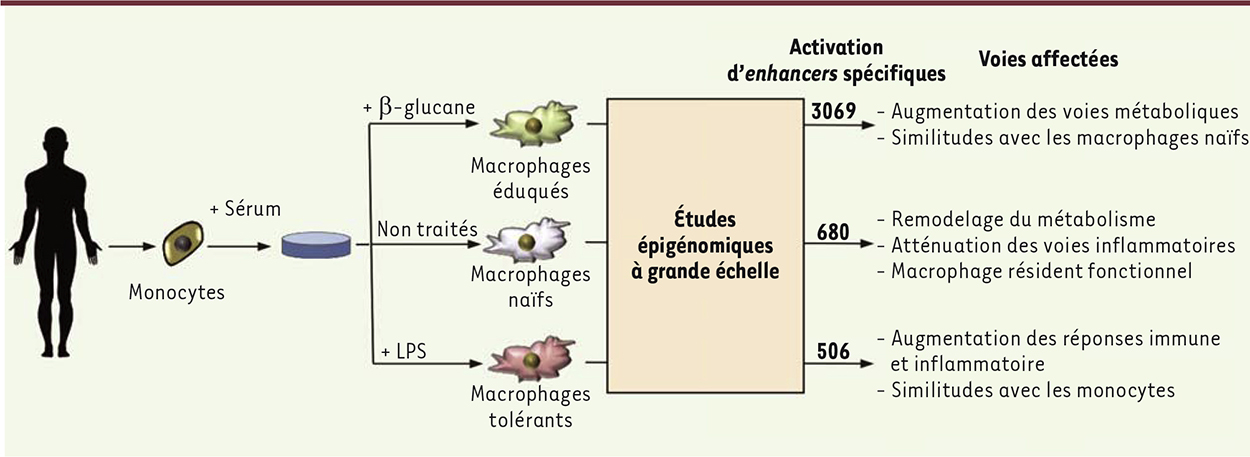
Tolérance immunitaire et immunité éduquée impliquent des programmes épigénétiques spécifiques L’une des études du projet Blueprint démontre qu’il existe des programmes épigénétiques distincts permettant d’instaurer un état soit de tolérance immunitaire, soit d’immunité innée éduquée [4]. Cette étude, dirigée de façon conjointe par Colin Logie, Mihai Netea et Hendrick Stunnenberg, décrit les voies spécifiques qui induisent ces processus. Les auteurs ont pour cela étudié les profils épigénétiques de monocytes humains circulants, ou différenciés in vitro en macrophages. Les monocytes circulants ont été différenciés soit en macrophages naïfs - en les cultivant dans un milieu supplémenté avec du sérum humain -, soit en macrophages tolérants - en les exposant à des lipopolysaccharides (LPS) bactériens, ou enfin en macrophages éduqués via leur exposition au β-glucane (composant de la levure Candida albicans) (Figure 1). Les auteurs ont ensuite établi une cartographie très complète de l’état initial de l’épigénome (monocytes circulants) et des trois types de macrophages dont la différenciation était induite dans les différentes conditions. Pour cela, ils ont réalisé des expériences d’immuno-précipitation de chromatine couplée au séquençage à haut débit (ChIP-seq) ciblant plusieurs modifications d’histones, des expériences d’hypersensibilité à la DNase I (DNase-seq), ainsi qu’une analyse complète du transcriptome par la technique de RNA-seq. Ces approches ont permis d’identifier de façon extrêmement précise les régions cis-régulatrices proximales (promoteurs) et distales (appelées aussi enhancers) se trouvant activées ou réprimées selon les différentes conditions de stimulation, et de les corréler aux changements d’expression des gènes qui leur sont associés (Figure 1). Les auteurs ont pu ainsi identifier les signatures épigénétiques des macrophages naïfs, tolérants ou éduqués. Il est frappant de voir que la plupart des changements spécifiques à chaque condition sont localisés au niveau de séquences cis-régulatrices distales. L’analyse de l’expression des gènes associés a révélé que les macrophages naïfs possèdent une activité métabolique différente de celle des monocytes circulants, et que l’expression des voies inflammatoires, certainement nécessaires à leur établissement en tant que macrophages résidents, y est atténuée. Les macrophages stimulés par le β-glucane ressemblent aux macrophages naïfs, mais surexpriment certains modules de gènes, notamment ceux qui codent pour des enzymes métaboliques. Ceci suggère l’expression dans les macrophages éduqués, à l’instar des macrophages naïfs, d’une signature épigénétique spécifique. Par ailleurs, la stimulation avec du LPS, mimant l’état d’immunoparalysie observé dans le sepsis, induit plutôt des gènes impliqués dans les réponses immune et inflammatoire. Cette plasticité épigénétique est soulignée par une autre étude chez la souris, montrant que les profils épigénétiques des macrophages résidents divergent selon leur provenance tissulaire, suggérant donc un rôle important de l’environnement dans la mise en place d’un programme épigénétique spécifique [9].
 | Figure 1.
Reprogrammation épigénétique lors des différents états de différenciation des macrophages. L’épigénome, le transcriptome et les régions accessibles à la DNase I ont été caractérisés dans les monocytes circulants et dans des macrophages naïfs, tolérants ou éduqués, différenciés in vitro [ 4]. Cette étude a permis d’identifier les enhancers spécifiquement activés dans les différents états de différenciation des macrophages. La réponse épigénétique la plus massive est induite par le traitement au β-glucane, qui permet d’activer spécifiquement 3069 enhancers et qui permet la différenciation des macrophages éduqués. Bien que de façon plus modérée, la différenciation des monocytes en macrophages naïfs et tolérants induit, elle aussi, l’activation d’un répertoire spécifique d’ enhancers (680 et 506, respectivement). L’identification de ces signatures épigénétiques, associée à l’analyse du transcriptome, a révélé des voies métaboliques associées aux différents états de différenciation des macrophages. Ainsi, les macrophages éduqués, bien que présentant certaines similitudes avec les macrophages naïfs, se caractérisent par un renforcement significatif des voies métaboliques. Les macrophages tolérants, plus proches des monocytes, se distinguent quant à eux par une élévation des voies liées à la réponse immune et à l’inflammation. |
|
Reprogrammation du métabolisme du glucose et immunité innée éduquée Une deuxième étude du consortium Blueprint a comparé de façon similaire l’immunité innée éduquée et tolérante [5]. Ces travaux démontrent un lien direct entre la différenciation des macrophages et le métabolisme du glucose. En effet, les chercheurs ont découvert qu’au cours de la différenciation, plusieurs locus de gènes impliqués dans la glycolyse subissent des changements épigénétiques conduisant à leur expression. Ainsi, une nouvelle dimension - métabolique - de l’établissement de cette mémoire de l’immunité innée a été identifiée. Cette modification du métabolisme interne dans les monocytes différenciés en macrophages éduqués se traduit par une forte consommation de glucose, une production abondante de lactate, et par une valeur élevée du rapport nicotinamide adénine dinucléotide (NAD+) sur sa forme réduite (NADH), témoin d’un changement métabolique avec une augmentation de la glycolyse dépendante de la voie mTOR (mammalian target of rapamycin) [5]. L’immunité innée éduquée pourrait donc être induite par la mise en place d’une glycolyse aérobie via la voie Akt-mTOR-HIF-1a (hypoxia inducible factor-1a). En effet, des expériences chez la souris montrent que l’inhibition de l’un des composants de cette voie abroge complètement la mise en place de l’immunité innée éduquée. Ainsi, le changement du métabolisme du glucose - de la phosphorylation oxydative à la glycolyse - permet une augmentation rapide de la production d’énergie pour la cellule. Ce changement métabolique permettrait aux macrophages éduqués de produire l’énergie nécessaire à une activité accrue lors de la lutte contre les pathogènes. |
L’importance de l’environnement dans la réponse immunitaire innée Dans l’ensemble, ces deux études montrent que la réponse immunitaire innée est beaucoup plus fortement régulée par des facteurs environnementaux qu’envisagé précédemment. Ceci conduit à des différences très marquées du point de vue épigénétique et ouvre la voie à des études visant à manipuler ou moduler la réponse immunitaire. Au-delà de leur seule description, ces nouvelles données représentent l’un des premiers résultats significatifs obtenu en exploitant les données épigénétiques produites grâce au consortium Blueprint. Librement accessibles, ces données seront d’une grande valeur pour les chercheurs souhaitant mieux comprendre les mécanismes de différenciation des acteurs de l’immunité innée, dans des contexte normaux et pathologiques, et, au-delà, ceux qui contrôlent d’autres processus, comme l’hématopoïèse, une autre cible d’étude privilégiée du consortium Blueprint [1, 3]. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Martens JH, Stunnenberg HG. BLUEPRINT: mapping human blood cell epigenomes . Haematologica. 2013; ; 98 : :1487.–1489. 2.
Adams D, Altucci L, Antonarakis SE et al. BLUEPRINT to decode the epigenetic signature written in blood . Nat Biotechnol. 2012; ; 30 : :224.–226. 3.
Chen L, Kostadima M, Martens JH et al. Transcriptional diversity during lineage commitment of human blood progenitors . Science. 2014; ; 345 : :1251033.. 4.
Saeed S, Quintin J, Kerstens HH et al. Epigenetic programming of monocyte-to-macrophage differentiation and trained innate immunity . Science. 2014; ; 345 : :1251086.. 5.
Cheng SC, Quintin J, Cramer RA et al. mTOR- and HIF-1alpha-mediated aerobic glycolysis as metabolic basis for trained immunity . Science. 2014; ; 345 : :1250684.. 6.
Pennisi E. Immunology Metabolic shift may train immune cells . Science. 2014; ; 345 : :1550.–1551. 7.
Netea MG. Training innate immunity: the changing concept of immunological memory in innate host defence . Europ J Clin Invest. 2013; ; 43 : :881.–884. 8.
Ostuni R, Piccolo V, Barozzi I et al. Latent enhancers activated by stimulation in differentiated cells . Cell. 2013; ; 152 : :157.–171. 9.
Lavin Y, Winter D, Blecher-Gonen R et al. Tissue-resident macrophage enhancer landscapes are shaped by the local microenvironment . Cell. 2014; ; 159 : :1312.–1326. |