| |
| Med Sci (Paris). 2015 March; 31(3): 247–249. Published online 2015 April 8. doi: 10.1051/medsci/20153103007.Criblages à haut débit d’ARN interférents Quand les petits ARN interférents se prennent pour des micro-ARN Roger Meier,1a Magalie Mazelier,2b and Pierre-Yves Lozach2c 1ScopeM, ETH Zürich, Suisse 2CellNetworks, cluster of excellence and department of infectious diseases, virology, university hospital Heidelberg, Im Neuenheimer Feld 324, 69120Heidelberg, Allemagne MeSH keywords: Faux positifs, Séquençage nucléotidique à haut débit, Humains, microARN, Interférence par ARN, Petit ARN interférent, physiologie |
L’interférence par ARN a été découverte dans les années 1990, valant à Fire et Mello le prix Nobel de physiologie et de médecine en 2006. Le terme d’interférence par ARN désigne la capacité de petits ARN simple ou double brins d’induire la diminution de l’expression d’une protéine en liant spécifiquement l’ARN messager (ARNm) correspondant [1]. Deux types de petites molécules d’ARN, dont la taille est comprise entre 21 et 23 nucléotides (nt), sont capables de provoquer un tel phénomène : les microARN (miARN) et les petits ARN interférents (ARNi) [2, 3]. Leur liaison à l’ARNm entraîne la dégradation de ce dernier par la ribonucléase DICER, et, par conséquent, conduit à une réduction de la traduction associée. Très largement répandue chez les eucaryotes, l’interférence par ARN a très probablement été retenue lors de l’évolution comme mécanisme de régulation de l’expression génique, ou encore, comme défense contre l’introduction de génomes étrangers, notamment viraux [1]. |
Criblages à haut débit de petits ARNi Les progrès de l’informatique et de la robotique ont permis des avancées majeures dans le séquençage de génomes de grande taille, et ont abouti, au milieu des années 2000, à la possibilité de cribler des librairies d’ARNi contre des génomes entiers, dont celui de l’homme. Ces progrès ouvrirent de nouvelles perspectives, non seulement pour l’étude de la génomique fonctionnelle, mais également pour la compréhension des interactions moléculaires entre cellules hôtes et pathogènes. Ainsi, en éteignant sélectivement chaque gène du génome humain, plusieurs groupes ont révélé l’implication de centaines de protéines cellulaires dans l’infection par des virus, comme ceux de l’immunodéficience humaine et de la grippe. De façon assez similaire à ce qui s’était passé lors de l’avènement des puces à ADN lors de la décennie précédente, beaucoup pensèrent que les criblages à haut débit d’ARNi allaient répondre à toutes les questions que posent la microbiologie et la virologie. Malheureusement, le chevauchement modeste entre les candidats cellulaires identifiés dans des criblages indépendants réalisés avec un même micro-organisme, combiné à la complexité de l’analyse de la masse de données générées par ces approches, ont douché l’enthousiasme général. Pourtant, si on s’attarde aux fonctions cellulaires identifiées plutôt qu’aux protéines elles-mêmes, les similitudes sont bien plus grandes. La désillusion est aujourd’hui excessive, de même que les attentes suscitées par les criblages d’ARNi étaient trop grandes à leurs débuts. Nous avons récemment criblé deux librairies indépendantes d’ARNi dirigés contre l’ensemble du génome humain dans des cellules infectées par divers micro-organismes, dont le virus Uukuniemi (VUUK) et les bactéries Brucella abortus et Salmonella thyphimurium [4, 5]. Au-delà des centaines de facteurs cellulaires révélés pour leur implication potentielle dans ces infections, ces travaux apportent avant tout des réponses sur les origines possibles des divergences observées entre criblages d’ARNi pour un même pathogène, ainsi que des solutions pour limiter ce phénomène. |
Mais d’où proviennent les faux positifs ? Pour répondre à cette question, prenons l’exemple de nos deux criblages d’ARNi avec le VUUK, un virus transmis par les tiques et utilisé comme modèle pour les bunyavirus depuis plusieurs décennies [5, 6]. Des analyses initiales avaient identifié environ 800 candidats cellulaires ayant un rôle dans l’infection. Après avoir déterminé par puces à ADN l’identité de tous les ARNm présents dans notre modèle cellulaire (une lignée HeLa), il est apparu qu’un peu plus de 200 de nos candidats ARNi ciblaient en fait des gènes non exprimés dans ces cellules. Cette démonstration, que certains ARNi agissent de façon non spécifique, renforce le sentiment général d’un niveau élevé de faux positifs dans les listes de candidats obtenus par criblage d’ARNi. Après avoir retiré ces faux positifs de notre analyse, la fraction des candidats commune aux deux criblages n’excédait pas 10 à 15 %. Contrairement aux criblages publiés antérieurement pour d’autres virus, nous avions pourtant utilisé la même lignée cellulaire, la même souche virale, et des procédures expérimentales identiques. Seuls les fournisseurs de librairies d’ARNi différaient. Mais alors pourquoi une si faible concordance ? Dans nos criblages, nous avons employé une librairie avec quatre ARNi indépendants par gène (Qiagen), et une autre constituée d’un mélange de quatre ARNi contre chaque gène (Dharmacon). Il est cependant peu probable que la conception même des ARNi ainsi que le type de librairies choisi et développé par les différents fournisseurs, soient les causes principales de cette faible concordance. En analysant les résultats obtenus avec la librairie de Qiagen, nous avons pu démontrer que certains ARNi inhibaient le cycle viral du VUUK par des mécanismes semblables à ceux des miARN. Autrement dit, seulement 6-7 nt de la séquence externe (position 2-3 à 8) de l’ARNi servent à cibler l’ARNm, au lieu de la totalité des 21-23 nt habituels (Figure 1A–B). Avec une région d’appariement aussi courte, il est plus facile de comprendre que la spécificité des ARNi s’en trouve diminuée. Le nombre d’ARNm indésirables reconnus par de tels ARNi est probablement comparable à celui que les miARN équivalents reconnaissent eux-mêmes, typiquement plusieurs centaines.
 | Figure 1.
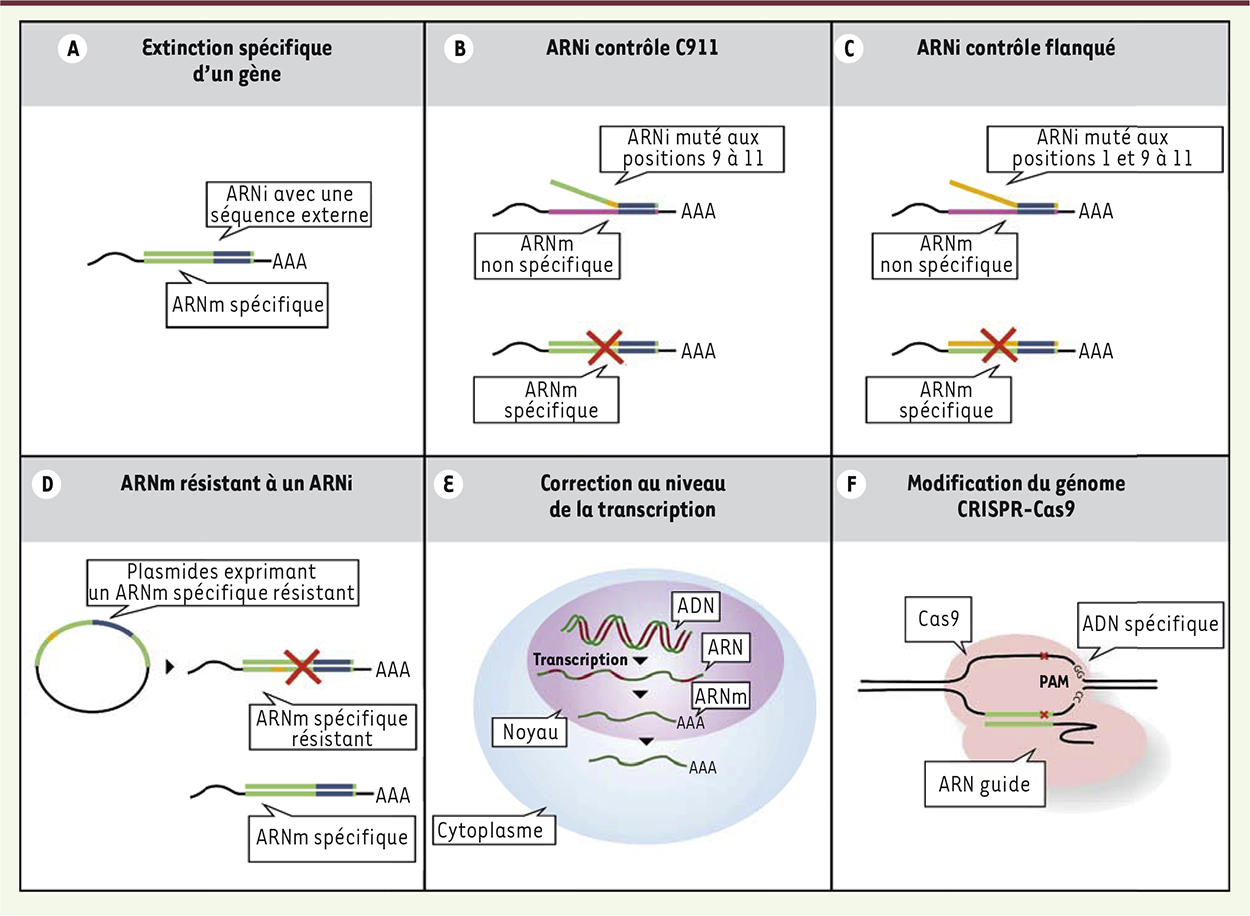
Stratégies de validation des candidats identifiés dans les criblages d’ARNi. A. Extinction spécifique d’un gène par un ARNi. L’expression du gène ciblé est éteinte suite à une complémentarité parfaite et totale entre l’ARNm et l’ARNi. B. Méthode C911 - Un contrôle ARNi C911 consiste au remplacement des bases 9 à 11 sur le brin anti-sens de l’ARNi par des bases complémentaires alors que la séquence externe de 6-7 nt en position 2-3 à 8 est conservée. À la différence de l’ARNi original, le contrôle ARNi C911 ne peut donc pas s’apparier de façon optimale sur l’ARNm cible et provoquer l’extinction du gène correspondant. En revanche, un tel contrôle permet de mettre en évidence de potentiels effets non spécifiques dus à la séquence externe de l’ARNi. C. Méthode de l’ARNi flanqué. Un contrôle ARNi flanqué consiste à remplacer de manière aléatoire les bases de l’ARNi en positions 1-2 et 9 à 21 afin de conserver la séquence externe de 6-7nt en position 2-3 à 8. Ce contrôle est donc incapable de s’apparier à l’ARNm cible. En revanche, il est possible de comparer l’effet de l’ARNi original et celui dû spécifiquement à sa séquence externe. Si l’extinction est spécifique, le phénotype observé pour un ARNi ne devrait plus être visible avec le contrôle ARNi flanqué. D. ARNm rendu résistant à des ARNi. En mutant directement l’ARNm cible dans les cellules, l’ARNi correspondant ne peut plus s’apparier. En cas de ciblage spécifique, le phénotype observé pour un ARNi ne devrait pas être observé dans ces cellules. E. Correction basée sur l’expression des ARNm dans la cellule. Le profil d’expression des ARNm est déterminé dans le modèle cellulaire pour le criblage d’ARNi, par exemple, par puce à ADN. Seuls les gènes transcrits sont pris en considération dans l’analyse des résultats. F. Méthode CRISPR-Cas9. La modification du génome par la technologie CRISPR-Cas9 rend possible la génération de lignées cellulaires dépourvues d’un gène candidat, ou d’en réprimer l’expression. Un tel modèle cellulaire permet de valider un phénotype observé avec l’ARNi correspondant au gène candidat dans une lignée standard. |
Notre démonstration s’appuie essentiellement sur l’emploi de miARN synthétiques dont les séquences externes de 6-7 nt correspondent à celles que nous avons prédites pour inhiber l’infection dans nos criblages d’ARNi. Le facteur de corrélation entre nos prédictions et l’effet des miARN synthétiques mesuré sur l’infection est supérieur à 0,8. Pour résumer simplement, parmi les 72 773 ARNi de la librairie de Qiagen, au moins 5 257 inhibent l’infection par le VUUK en se liant à des ARNm autres que ceux souhaités. Après l’élimination de tous les ARNi présentant un risque de fonctionner comme des miARN, la fraction des candidats commune aux deux librairies d’ARNi atteignait 25 %. Les criblages que nous avons réalisés avec des bactéries ont conduit à des résultats et interprétations similaires [4]. |
Conclusions et perspectives Le nombre important de candidats identifiés dans les criblages d’ARNi rend quasiment impossible une validation systématique par des approches conventionnelles. Des choix sont en général faits, et le risque de travailler sur un faux positif en bout de ligne est important. Nos études ont permis de comprendre l’origine de certaines des causes des faux positifs et offrent des moyens d’optimiser les futurs criblages, mais aussi de corriger ceux déjà existants. Nos résultats devraient également aider au développement de nouvelles librairies d’ARNi, notamment avec des séquences externes plus neutres. Il est évident qu’il y a d’autres raisons à la présence de faux positifs, et en limiter l’importance dans les résultats de criblages d’ARNi demeure un enjeu majeur [7–9]. Cela passe inéluctablement par le développement de nouvelles méthodes qui permettent la validation rapide d’un nombre important de candidats, si ce n’est l’intégralité des candidats révélés. Certaines existent déjà (C911, CRISPR, etc.) ou sont en cours d’adaptation aux contraintes du haut débit [1, 10]. La liste n’est bien entendu pas exhaustive, et quelques-unes de ces solutions sont brièvement présentées dans la
Figure 1
. Le dévelopement des criblages d’ARNi à haut débit étant très récent - moins de dix ans -, ces technologies ont une marge réelle d’amélioration et de progression. Ces criblages sont un outil scientifique parmi d’autres, et ne peuvent répondre, seuls, à toutes les questions. Ils doivent impérativement être complétés par une série d’approches complémentaires. Bien contrôlés, ils ont déjà démontré leur intérêt en biologie cellulaire et microbiologie et peuvent encore nous en apprendre beaucoup sur la cellule et ses parasites. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Mohr SE, Smith JA, Shamu CE, et al. RNAi screening comes of age: improved techniques and complementary approaches . Nat Rev Mol Cell Biol. 2014; ; 15 : :591.–600. 2.
Zamore PD, Tuschl T, Sharp PA, Bartel DP. RNAi: double-stranded RNA directs the ATP-dependent cleavage of mRNA at 21 to 23 nucleotide intervals . Cell. 2000; ; 101 : :25.–33. 3.
Carthew RW, Sontheimer EJ. Origins and mechanisms of miRNAs and siRNAs . Cell. 2009; ; 136 : :642.–655. 4.
Franceschini A, Meier R, Casanova A, et al. Specific inhibition of diverse pathogens in human cells by synthetic microRNA-like oligonucleotides inferred from RNAi screens . Proc Natl Acad Sci USA. 2014; ; 111 : :4548.–4553. 5.
Meier R, Franceschini A, Horvath P, et al. Genome-wide small interfering RNA screens reveal VAMP3 as a novel host factor required for Uukuniemi virus late penetration . J Virol. 2014; ; 88 : :8565.–8578. 6.
Meier R, Helenius A, Lozach PY. DC-SIGN, un récepteur des phlébovirus : dynamique des interactions virus-récepteur . Med Sci (Paris). 2012; ; 28 : :16.–18. 7.
Birmingham A, Anderson EM, Reynolds A, et al. 3’ UTR seed matches, but not overall identity, are associated with RNAi off-targets . Nat Methods. 2006; ; 3 : :199.–204. 8.
Echeverri CJ, Beachy PA, Baum B, et al. Minimizing the risk of reporting false positives in large-scale RNAi screens . Nat Methods. 2006; ; 3 : :777.–779. 9.
Jackson AL, Bartz SR, Schelter J, et al. Expression profiling reveals off-target gene regulation by RNAi . Nat Biotechnol. 2003; ; 21 : :635.–637. 10.
Buehler E, Chen YC, Martin S. C911: a bench-level control for sequence specific siRNA off-target effects . PloS One. 2012; ; 7 : :e51942.. |