| |
| Med Sci (Paris). 2015 June; 31(6-7): 667–673. Published online 2015 July 7. doi: 10.1051/medsci/20153106021.Modulation pharmacologique de la réponse au stress du réticulum endoplasmique
Potentiel thérapeutique en cancérologie Saïd Taouji1,2,3* and Éric Chevet1,2 1Inserm U1053, Université de Bordeaux Segalen, 146, rue Léo Saignat, 33076Bordeaux Cedex, France 2BMYscreen, Bordeaux, France 3Adresse actuelle : Inserm U916, CRLCC Institut Bergonié, 229, cours de l’Argonne, 33076Bordeaux Cedex, France |

Le réticulum endoplasmique (RE) est le principal compartiment subcellulaire impliqué dans le repliement et la maturation des protéines membranaires ou sécrétées. Environ un tiers du protéome total y transite. De nombreux stress cellulaires peuvent altérer la fonction de cet organite, ce qui conduit à l’accumulation de protéines ayant un défaut de repliement. La surcharge du RE en protéines mal conformées représente un risque protéotoxique pour la cellule, et active une série de mécanismes d’adaptation qui sont connus sous le nom de réponse aux protéines mal conformées ou unfolded protein response – UPR [1]. L’UPR, qui est une réponse adaptative, a pour but de restaurer l’homéostasie du RE via (1) l’arrêt de la synthèse protéique globale, (2) la synthèse accrue de chaperons et de foldases impliqués dans le repliement des protéines, et (3) l’augmentation de la clairance des protéines accumulées, soit par export dans la voie de sécrétion, soit par dégradation (ER-associated degradation, ERAD). De plus, si le stress ne peut être résorbé, ni par conséquent l’homéostasie du RE restaurée, la signalisation de l’UPR conduit à la mort cellulaire [2]. L’UPR est généralement associée au maintien de l’homéostasie de la voie de sécrétion dans des cellules sécrétrices, telles que les plasmocytes, les glandes salivaires ou les cellules bêta pancréatiques. En outre, des modèles de souris knock-out pour les gènes codant les composants essentiels de l’UPR ont révélé que ces composés exercent certaines fonctions dans de nombreux processus physiologiques qui ne sont pas directement liés au repliement des protéines. Il s’agit notamment du métabolisme des lipides et du cholestérol, de l’équilibre énergétique, de l’inflammation et de la différenciation cellulaire [3]. Par ces nouvelles fonctions, l’UPR intervient dans le développement de pathologies telles que le cancer, le diabète ou les maladies neurodégénératives. Il est donc logique de développer des stratégies thérapeutiques ciblant l’UPR, même si cette stratégie n’en est encore qu’à un stade embryonnaire. Dans les faits, plusieurs sociétés pharmaceutiques, ainsi que des laboratoires académiques, élaborent et développent de nouvelles molécules capables de moduler sélectivement chaque branche de signalisation de l’UPR. Les premières communications scientifiques disponibles décrivent des effets thérapeutiques manifestes, démontrant qu’une « manipulation » de la réponse au stress du RE via un ciblage de l’UPR pourrait s’avérer pertinente dans certaines pathologies. Dans cet article, nous présenterons les dernières avancées obtenues dans l’identification et la mise en place de stratégies intégrant les approches de chimie-biologie pour le ciblage de l’UPR et de l’homéostasie du RE. La chimie-biologie met en œuvre l’usage de petites molécules pour déchiffrer le vivant et développer des outils pharmacologiques et potentiellement des candidats médicaments. Nous avons précédemment décrit en détails les étapes de cette voie UPR, et nous renvoyons le lecteur à plusieurs revues parues dans médecine/sciences [4, 5] et à la Figure 1 qui en résume les principaux aspects.
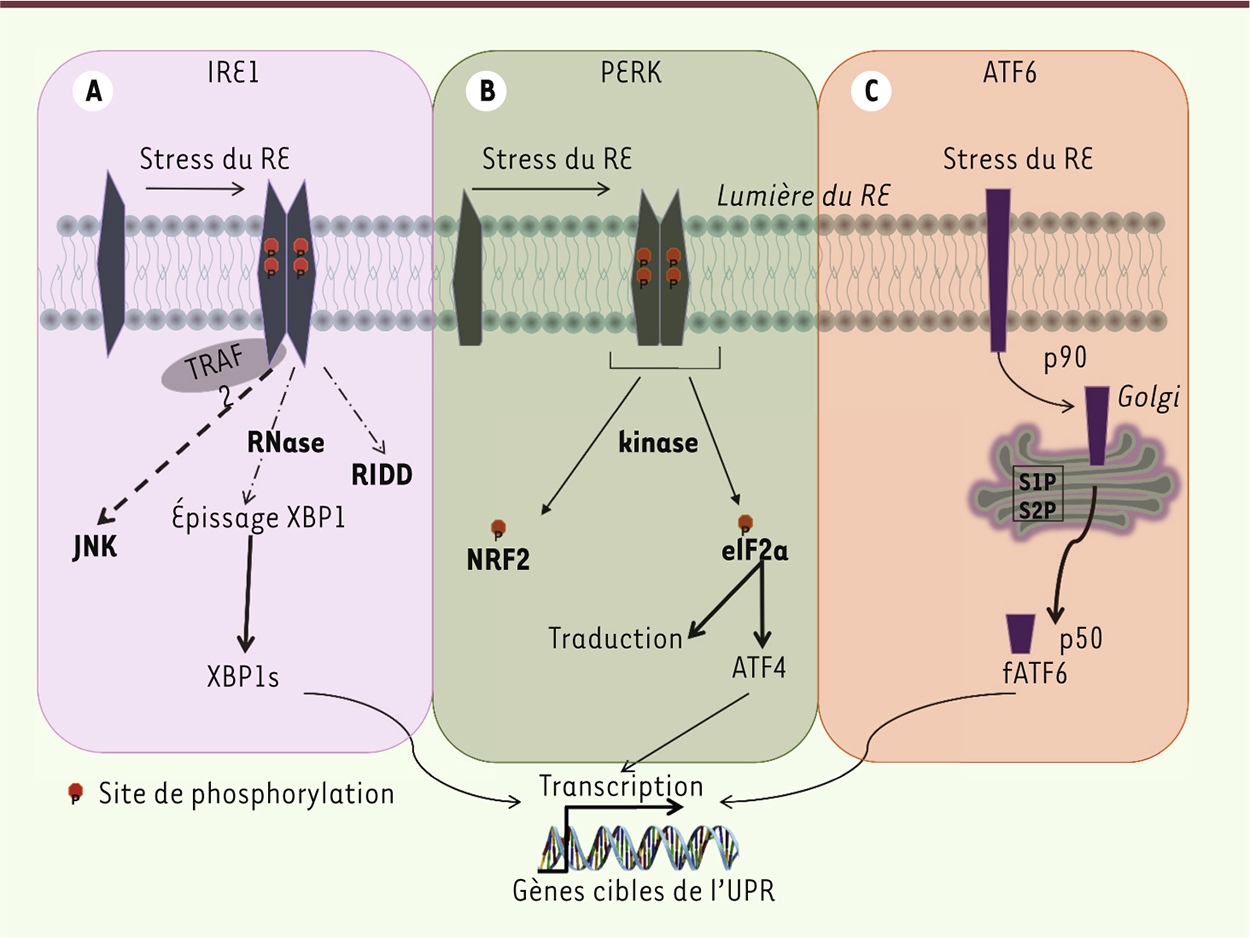
 | Figure 1.
La réponse au stress lié à l’accumulation de protéines mal repliées dans le réticulum endoplasmique (RE). Le stress du RE induit une réponse adaptative connue sous le nom de l’UPR. Trois grands senseurs du stress incluant IRE1α, PERK et ATF6, contrôlent les réponses dépendantes de l’UPR. Ces protéines transmembranaires du RE sont directement impliquées dans la transduction de signaux vers le cytosol et le noyau pour rétablir la capacité de repliement des protéines. A. L’activité RNAse d’IRE1α clive spécifiquement l’ARNm codant pour XBP1 pour générer un facteur de transcription qui active un sous-ensemble de gènes cibles de l’UPR liés au repliement, l’ERAD, le contrôle qualité des protéines, et la biogenèse des organites. IRE1α dégrade également certains ARNm et active les voies JNK et NF-kB via des interactions avec des protéines adaptatrices. B. L’activation de PERK atténue la synthèse des protéines par phosphorylation générale du facteur d’initiation de la traduction eIF2α. La phosphorylation de eIF2α permet la traduction sélective de l’ARNm d’ATF4, qui code pour un facteur de transcription induisant l’expression de gènes impliqués dans les réponses anti-oxydatives, du métabolisme des acides aminés et de l’apoptose. C
. Dans des conditions basales, ATF6 est localisée dans le RE et contient dans son domaine cytosolique un facteur de transcription de type bZIP. Dans les cellules soumises à un stress du RE, ATF6 migre vers l’appareil de Golgi pour y être activée par deux protéases (S1P et S2P), libérant son domaine cytosolique (ATF6f). ATF6f active certains gènes cibles de l’UPR notamment ceux codant pour des chaperons. |
L’activation de la signalisation de l’UPR engage à la fois des composants pro-survie ou pro-apoptotique. Cibler ces protéines pourrait donc, soit réduire ou atténuer les défauts de repliement des protéines induits par des états pathologiques, soit, dans le cas d’un stress aigu, entraîner la cellule vers une des voies antiproliférative et pro-apoptotique. Nous définissons deux grandes classes de composés, ceux qui favorisent l’adaptation au stress, et ceux qui affaiblissent cette voie adaptative. La plupart des molécules pharmacologiques disponibles ciblant l’UPR ont été testées dans des modèles de cancer, raison pour laquelle notre discussion portera surtout sur l’implication de la voie du stress du RE dans les différents aspects de la biologie du cancer. |
Les modulateurs de la signalisation de l’UPR Outre son rôle dans la différenciation, la migration des cellules et l’angiogenèse, l’activation de l’UPR a été proposée comme un facteur de survie majeur facilitant la croissance tumorale. La plupart des approches cherchant à inhiber l’activité de cette voie dans un contexte tumoral se sont focalisées sur des composés qui bloquent l’activité d’IRE1α (inositol–requiring enzyme–1a) ou de PERK (PRKR-like endoplasmic reticulum kinase) (Figure 2). En outre, dans cette même logique, des inhibiteurs de chaperons, de foldases du RE, ou des mécanismes de contrôle de qualité et de l’ERAD ont été testés pour leur capacité à réguler le stress du RE de cellules cancéreuses. Nous discutons ci-dessous des composés disponibles les plus prometteurs qui bloquent les effets prosurvie et d’adaptation de la signalisation de l’UPR.
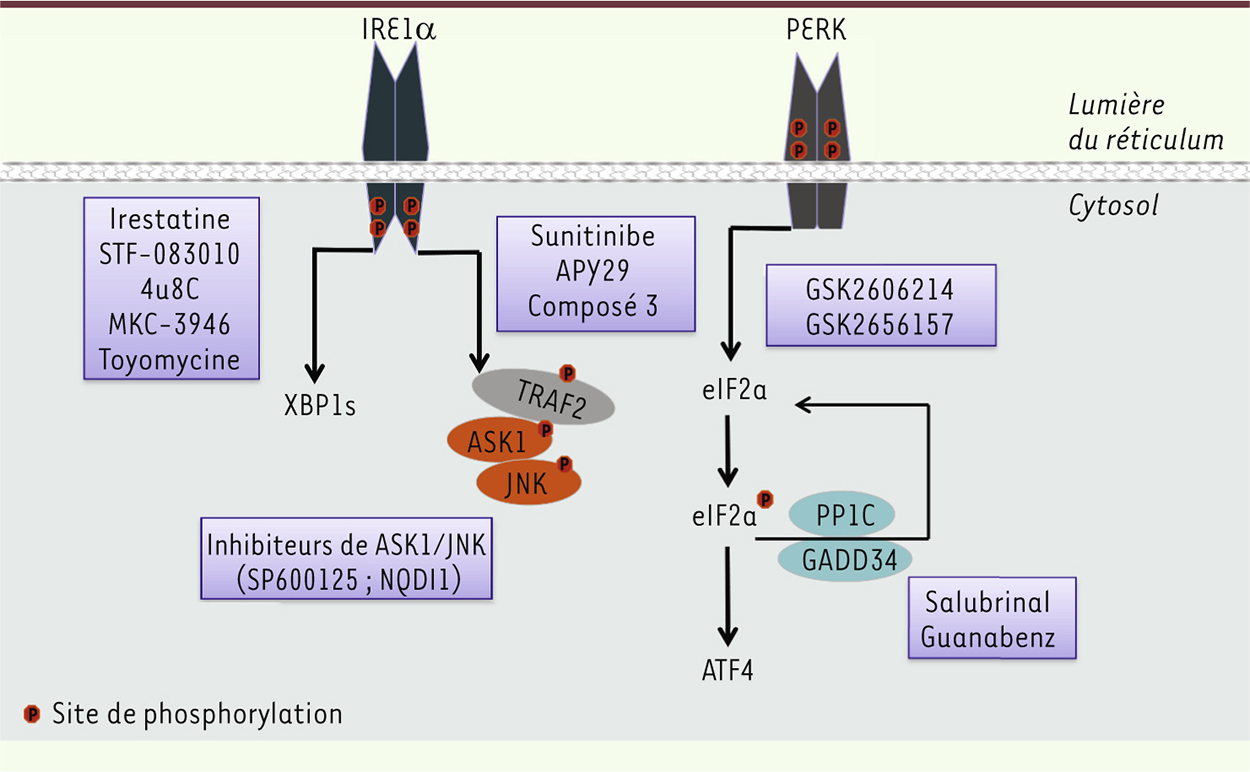
 | Figure 2.
Les modulateurs de la signalisation de l’UPR.
|
Les sites catalytiques d’IRE1, cibles doublement pertinentes Les composés actuels ciblent deux sites actifs d’IRE1 : la poche de liaison de l’ATP du domaine kinase, ou le noyau catalytique du domaine RNase (voir Figure 2). Les inhibiteurs de l’activité RNase Ce groupe d’inhibiteurs identifiés par criblage à haut débit comprend plusieurs espèces chimiques dont le 4μ8C (IC 50 = 76 nM), les salicylates (en particulier le 3-méthoxy-6-bromo-salicylate), le MKC-3946, et le STF-083010. Ces molécules ciblent l’activité RNase sans interférer avec l’activité kinase [ 6]. La molécule 4μ8C se fixe à la lysine-907, formant une imine stable dans le site catalytique du domaine RNase, et bloque l’épissage de XBP-1 (X-box binding protein 1) et la dégradation des messagers par la voie RIDD (regulated Ire1-dependent decay). D’autres inhibiteurs de ce groupe peuvent avoir un mode d’action similaire en raison de leur structure ou de leur capacité à bloquer par compétition la liaison 4μ8C à la lysine 907 de IRE1. Certains inhibiteurs ont été testés in vivo. Par exemple, le 3-méthoxy-6-bromo-salicylate se fixe de façon réversible à IRE1 et bloque l’épissage de l’ARNm codant pour XBP-1 dans différents organes (rein, foie, rate) d’animaux auxquels de la tunicamycine (inducteur de stress du RE) a été administrée. Le composé STF-083010 a été évalué dans un modèle de xénogreffe de myélome multiple humain [7]. Le myélome multiple est caractérisé au niveau moléculaire par une suractivation de l’activité RNase de IRE1 α conduisant à une forte expression de XBP1, un facteur de transcription central impliqué dans la différenciation plasmocytaire. Le STF-083010 induit une cytotoxicité sélective pour les plasmocytes malins du myélome multiple, mais pas pour les cellules issues d’un donneur sain. Ce composé est en phase préclinique dans des modèles de myélome multiple et de cancer du sein. De même, le MKC-3946 induit la mort des cellules du myélome multiple en combinaison avec le Bortezomib1, (Velcade). Le MKC-3946 seul a une faible toxicité sur les cellules du myélome multiple, mais, en inhibant l’épissage de XBP1, il renforce l’action du Bortezomib qui induit un stress du RE. Le MKC-3946 est actif in vivo dans un modèle de xénogreffe de myélome multiple, et son action est renforcée par le Bortezomib [6]. MKC204 et MKC-3946 sont en phase préclinique dans cette hémopathie. Le 4μ8C n’a pas été testé in vivo probablement en raison d’une mauvaise pharmacocinétique. Fait remarquable observé en culture cellulaire, même si le 4μ8C bloque l’épissage de XBP1 et l’activité RIDD, il n’a pas d’impact sur la survie des cellules lors d’un stress aigu du RE. Un autre criblage de petites molécules a identifié - dans un test utilisant le gène rapporteur luciférase-XBP1 – la toyocamycine comme répresseur de l’activité RNase d’IRE1α. Ce composé, produit par une souche d’Actinomycète, n’affecte pas in vitro la phosphorylation d’IRE1α, mais bloque spécifiquement l’activité RNase. L’effet de la toyocamycine est sélectif car ce composé n’a pas d’incidence sur ATF6α ni sur la signalisation de PERK. En synergie avec le Bortezomib, la toyocamycine induit, à des concentrations nanomolaires, l’apoptose de cellules de myélome multiple et retarde la croissance de xénogreffes chez la souris. D’autres composés ont été identifiés dans le même criblage, tels que la trierixine et la quinotrierixine (isolés à partir de souches de Streptomyces), agissant comme des répresseurs possibles de l’épissage de l’ARNm XBP1 sans que leur mécanisme d’action n’ait encore été exploré [8]. Enfin, le groupe de Laurie Glimcher (Weill Corneil medical college, États-Unis) a réussi à réduire la formation tumorale de xénogreffes de cancers du sein triple négatifs en inhibant l’expression de XBP1, en association avec un anti-mitotique (doxorubicine) [9]. Les inhibiteurs de l’activité kinase La seconde classe de modulateurs de l’IRE1 (sunitinib et APY29) se compose d’inhibiteurs compétitifs de kinases de type I. Ils stabilisent la forme active du domaine kinase en se fixant à la région charnière du site de fixation de l’ATP. Cette conformation stabilisée entraîne de façon allostérique une activation du domaine RNase d’IRE1. Toutefois, des résultats d’inhibition, comme de stimulation, de l’épissage de l’ARNm de XBP-1 par le sunitinib ont été publiés (Figure 2). La troisième classe de modulateurs est représentée par « le composé 3 », un inhibiteur de kinase de type II qui stabilise la conformation inactive et inhibe l’activité kinase, l’oligomérisation et l’activité RNase comme le fait le STF-083010 [10] (Figure 2). Le composé 3 dérive de l’inhibiteur APY29, dans lequel le noyau pyrazolopyrimidine a été remplacé par l’imidazopyrazine substitué à la position C-3 par un radical naphthylamine. L’effet de ces composés sur la signalisation de la voie ASK1 (apoptosis signal-regulating kinase 1)/JNK dépendante d’IRE1 est inconnu à ce jour. Néanmoins, un tel effet est déjà décrit pour certains inhibiteurs compétitifs de l’ATP disponibles, tels que le NQDI-1 identifié par criblage virtuel, ainsi que le composé MSC2032964A qui montre une bonne sélectivité. La molécule SD-0006 est un inhibiteur compétitif de l’ATP récemment identifié qui cible l’activité de la kinase p38 en aval de ASK1. Un certain nombre d’inhibiteurs compétitifs de l’ATP de JNK1, possédant à la fois des propriétés activatrices ou inhibitrices de la branche IRE1, ont été décrits dans des revues récentes [11]. D’autres modulateurs de la kinase ASK1 ont été identifiés par criblage et s’avèrent des outils pertinents pour moduler l’apoptose en cas de stress chronique du RE. Les inhibiteurs de PERK Récemment, un inhibiteur du domaine kinase de PERK a été identifié, doué de propriétés antitumorales et antiangiogéniques [ 12]. Un criblage de petites molécules a permis d’identifier plusieurs autres composés ciblant la phosphorylation de PERK. GSK2606414 a été découvert et optimisé grâce à la chimie médicinale, qui a permis de générer le composé GSK265157 [ 13]. Le GSK265157 est un inhibiteur compétitif de PERK puissant et très sélectif. Ce composé a été identifié par criblage et modifié par chimie médicinale assistée par des études de co-cristallisation du complexe inhibiteur-PERK. Le GSK2606414 est actif par voie orale et diminue la croissance tumorale dans un modèle de xénogreffe du cancer du pancréas. Des résultats similaires ont été obtenus pour le GSK2656157. De façon remarquable, le test in vivo a montré que GSK2656157 affecte l’autophosphorylation de PERK dans le pancréas. Le GSK2656157 inhibe également la croissance tumorale dans plusieurs modèles de xénogreffes de souris. Dans cette étude [ 13], plusieurs paramètres ont été corrélés avec l’activité anticancéreuse du GSK2656157, dont un métabolisme altéré des acides aminés, une diminution de la densité des vaisseaux sanguins et de la perfusion vasculaire. Les agonistes et antagonistes de la clairance des protéines du RE Une inhibition des voies de clairance des protéines du RE impliquées dans la dégradation des protéines mal conformées déclenche l’activation de l’UPR et diminue la survie des tumeurs dépendantes de la voie de sécrétion (revue [ 14]). Une inhibition des voies de l’autophagie et de l’ERAD ( endoplasmic reticulum-associated protein degradation) par des petites molécules peut être obtenue, soit en ciblant directement le protéasome (ce que fait par exemple le Bortezomib ou le MG132), soit en bloquant la dislocation des protéines « clientes » du RE par des inhibiteurs de l’ATPase p97/VCP tels que l’Eeyarestatine, les alkylsulfanyle -1,2,4- triazolés et le DBEQ. Le Bortezomib et le MG132 sont des peptides pseudomimétiques respectivement protégés sur la partie amino-terminale par l’acide boronique et par un aldéhyde, qui se lient à la sous-unité β1 du protéasome 26S inhibant ainsi l’activité catalytique. De multiples inhibiteurs de deuxième génération ont été développés à la suite du succès du Bortezomib dans le traitement du myélome multiple [ 15, 16]. Les causes de la mort cellulaire induite par le Bortezomib sont encore incomplètement comprises, mais pourraient impliquer plusieurs voies, dont celle du stress du RE [ 17]. L’ATPase AAA+ p97/VCP, qui est nécessaire à la rétro-translocation des substrats de l’ERAD, peut être inhibée par l’Eeyarestatine, un inhibiteur de première génération, ou des inhibiteurs réversibles récemment identifiés tels que les quinazolines comme le DBEQ, ML240, Ml241, dont l’efficacité et la spécificité d’action sur p97/VCP sont plus importantes que sur d’autres ATPases [ 18, 19]. Le DBeQ bloque également l’autophagie, expliquant probablement la puissance accrue de ces composés comme agents antitumoraux par rapport aux inhibiteurs du protéasome. Les modulateurs de l’activité des chaperons et du contrôle qualité des protéines du RE La dépendance vis-à-vis des chaperons moléculaires est une caractéristique commune des cellules transformées, et une diminution de l’activité des chaperons moléculaires a été envisagée dans le traitement des cancers. BiP est un chaperon essentiel du RE, qui exerce des rôles clés dans la survie des cellules. L’expression de BiP favoriserait la progression tumorale, la survie cellulaire, le développement de métastases et de résistances à la chimiothérapie. BiP a même été proposé comme biomarqueur de la progression tumorale. Ainsi, les foldases du RE constituent de nouvelles cibles émergentes et pertinentes pour la découverte de médicaments contre le cancer. Les stratégies visant à diminuer l’expression de BiP dans des modèles de culture de cellules tumorales, ou l’utilisation d’inhibiteurs de la liaison de BiP à l’ATP ont un fort potentiel cytotoxique [20]. L’inhibition transcriptionnelle ou post-traductionnelle de BiP pourrait également avoir des effets anticancéreux potentiels [21]. Parmi les inhibiteurs de chaperons, citons les inhibiteurs de HSP90 (heat shock protein) tels que le 17-AAG, dérivé moins toxique de l’antibiotique geldanamycine, l’inhibiteur canonique de HSP90, le radicicol, et des inhibiteurs de HSP70. Chacun de ces inhibiteurs se lie au domaine amino-terminal de fixation de l’ATP sur sa cible, inhibant ainsi la fonction du chaperon, et provoquant la mort cellulaire [22, 23]. La protéine GRP94, résidente du RE et homologue de HSP90, a également été ciblée par la geldanamycine. Cette molécule inhibe fortement la croissance des cellules tumorales dans la leucémie lymphoïde B chronique [24]. Des approches ont aussi été développées pour modifier, grâce à des outils pharmacologiques, la capacité de repliement des protéines du RE. Les PDI sont des cibles de plus en plus intéressantes en raison de leur rôle émergent dans plusieurs pathologies humaines. Les inhibiteurs de PDIA1 (protein disulfide isomerase family A, member 1) ont une grande efficacité antitumorale dans des modèles de mélanome et de gliome [25, 26], probablement due à l’inhibition des effets prosurvie de l’UPR dans le cancer. De façon inattendue, un criblage cherchant à identifier des composés inhibant la toxicité de la forme mutante de la huntingtine a sélectionné les inhibiteurs de PDIA1 en raison de leurs propriétés neuroprotectrices puissantes [27]. Bien que les chaperons du RE apparaissent comme des cibles intéressantes pour modifier l’homéostasie protéique du RE, il existe toujours un besoin accru pour le développement de composés plus spécifiques et mieux caractérisées in vivo. Les atténuateurs de la réponse au stress du RE Plusieurs stratégies ont cherché à améliorer la capacité d’adaptation des cellules ayant un stress du RE, ou réduire le défaut de repliement des protéines dans le RE. Dans cette section, nous discutons des approches les plus pertinentes pour réduire les effets délétères du stress chronique du RE relevés dans plusieurs pathologies humaines.
Les amplificateurs de la phosphorylation d’eIF2a Les composés ciblant les phosphatases d’eIF2α ont été identifiés à partir de tests cellulaires détectant ceux qui favorisent la survie en situation de stress. Le salubrinal inactive indirectement à la fois les complexes eIF2α-PP1-GADD34 ( growth arrest and DNA damage-inducible protein) et PP1-CReP (CreP est un régulateur constitutif de la phosphorylation d’eIF2α) probablement par désassemblage du complexe. Cet effet augmente le taux de phosphorylation d’eIF2α, réduit le taux de traduction et active en aval la signalisation d’ATF4 [ 28]. La molécule guanabenz se lie directement à GADD34 mais pas à CreP, empêchant l’assemblage du complexe GADD34-PP1 ; elle augmente la phosphorylation d’eIF2α et la signalisation en aval uniquement en cas de stress ( Figure 2). Ce composé est également un agoniste du récepteur α2-adrénergique, et il est approuvé cliniquement dans le traitement de l’hypertension artérielle ; les effets secondaires doivent donc être interprétés avec soin. La sélectivité du guanabenz, qui n’agit sur la phosphorylation d’eIF2α qu’en présence d’un stress du RE, peut être un avantage et faire préférer ce composé au salubrinal, qui, par son inhibition traductionnelle incontrôlée, pourrait induire des effets indésirables à long terme. En effet, si les souris déficientes pour GADD34 se développent normalement, les animaux dépourvus à la fois de CReP et GADD34 meurent au cours du développement embryonnaire [ 29], ce qui suggère qu’une inhibition constitutive des deux phosphatases d’eIF2α peut être délétère pour l’ensemble de l’organisme. Les chaperons chimiques Les chaperons chimiques représentent un groupe de composés de faible poids moléculaire qui stabilisent le repliement des protéines et tamponnent (dans le sens chimique du terme) les agrégations anormales des protéines. Les chaperons chimiques améliorent la fonction du RE probablement via l’atténuation du défaut de repliement des protéines, ce qui réduit le stress du RE [ 30]. Les chaperons chimiques les plus étudiés in vivo dans un contexte de pathologie sont le 4-phénylbutyrate (4-PBA), l’acide tauro-urso-désoxycholique (TUDCA), et le tréhalose ; tous ont tous été approuvés aux États-Unis par la Food and Drug Administration (FDA) pour leur utilisation chez des patients. Les chaperons chimiques réduisent également le stress du RE dans le foie de modèles murins d’obésité, améliorent la sensibilité à l’insuline et l’homéostasie du glucose, et peuvent restaurer la sensibilité à la leptine [ 31]. Le 4-PBA permet également d’améliorer la tolérance au glucose chez des patients insulino-résistants, et le TUDCA restaure partiellement la sensibilité à l’insuline dans le foie et les muscles, mais pas dans le tissu adipeux de patients obèses. L’administration de chaperons chimiques dans un modèle expérimental d’ischémie-reperfusion cérébrale atténue le stress du RE, en corrélation avec les effets neuroprotecteurs. Des résultats similaires ont également été publiés dans des modèles expérimentaux de lésions de la moelle épinière et d’altérations des photorécepteurs [ 32]. Les chaperons chimiques protègent également le foie de la stéatose et de l’ischémie associée au stress du RE [ 33]. Ainsi, les chaperons chimiques offrent une possibilité thérapeutique intéressante pour réduire le stress du RE dans un large éventail de maladies ; cependant, leur mode d’action demeure peu clair. |
Perspectives : valider la « druggabilité » des cibles Le déséquilibre de l’homéostasie du RE est actuellement reconnu comme un facteur important conduisant à diverses pathologies humaines, et, par conséquent, il représente une niche pertinente pour la découverte de nouveaux médicaments. La voie adaptative du RE offre des cibles intéressantes pour moduler de nombreux aspects de la physiologie cellulaire, tels que les mécanismes de survie et de mort. Selon le contexte pathologique, la stratégie thérapeutique peut avoir plusieurs objectifs : (1) l’atténuation de la réponse au stress du RE, (2) l’inactivation des composants pro-apoptotiques de l’UPR, ou (3) l’amélioration de la réponse UPR pour une meilleure adaptation au stress. Dans le cancer, les sondes chimiques devraient permettre une activation ou une amélioration sélectives de certaines voies, telles que celles de IRE1α/XBP1 ou d’ATF6α. Les inhibiteurs des domaines RNase ou kinase d’IRE1α ont montré une efficacité notable dans le cancer. À l’instar des success stories que représentent les découvertes de nouvelles drogues ciblant les domaines kinases, les activités kinase IREα et PERK constituent des cibles thérapeutiques privilégiées par de nombreux laboratoires. De même, puisque l’activité RIDD devient un effecteur important de l’UPR, avec des implications dans le diabète, la synthèse des lipides, la migration cellulaire, de nouveaux efforts de criblage de molécules seront nécessaires pour identifier des composés qui ciblent sélectivement le RIDD ou l’épissage de XBP1. Mais il est encore nécessaire de caractériser plus en détail la sélectivité des inhibiteurs d’IRE1α et PERK. Les effets secondaires que pourrait induire une administration chronique de ces composés n’ont pas encore été établis. Les données expérimentales disponibles indiquent que l’inhibition durable d’IRE1α déclenche des effets secondaires considérables sur la fonction du pancréas, le système immunitaire et le foie. La même conclusion peut être anticipée lors de l’utilisation d’inhibiteurs de PERK. L’hépatotoxicité doit être explorée avec soin, car IRE1α et XBP1 ont des rôles importants dans plusieurs fonctions clés du foie, y compris le métabolisme du cholestérol et des lipides et des mécanismes de détoxification [34]. Au-delà du rôle établi de l’UPR dans la réponse à l’accumulation de protéines mal conformées, cette voie est aussi impliquée dans des fonctions physiologiques importantes et dans divers organes. Les effets secondaires de molécules chimiques ciblant l’UPR seront probablement très complexes à prédire. Dans le cadre du traitement d’un cancer, de faibles doses d’agents anticancéreux pourraient, si elles sont associées à une inhibition temporaire de l’UPR, réduire ainsi le risque de toxicité. Dans d’autres contextes, les stratégies thérapeutiques peuvent nécessiter une modulation partielle de l’activité de l’UPR. Les transducteurs de signaux de l’UPR sont modulés par des régulateurs positifs et négatifs via de nombreuses interactions physiques, et le ciblage de certains composants de cet « interactome » des voies de l’UPR pourrait permettre d’ajuster finement l’homéostasie protéique dans le RE, évitant ainsi les effets délétères d’une inhibition totale de cette voie. D’autres études seront nécessaires pour identifier les sites allostériques des composants de l’UPR ainsi que les mécanismes de la régulation fine de l’UPR. Prévoir et définir les effets secondaires possibles (off targets) de la modulation de la réponse l’UPR demeure un objectif important pour valider l’activité thérapeutiques des cibles. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Walter P, Ron D. The unfolded protein response: from stress pathway to homeostatic regulation . Science. 2011; ; 334 : :1081.–1086. 2.
Tabas I, Ron D. Integrating the mechanisms of apoptosis induced by endoplasmic reticulum stress . Nat Cell Biol. 2011; ; 13 : :184.–190. 3.
Rutkowski DT, Hegde RS. Regulation of basal cellular physiology by the homeostatic unfolded protein response . J Cell Biol. 2010; ; 189 : :783.–794. 4.
Bouchecareilh M, Chevet E. Stress du réticulum endoplasmique : une réponse pour éviter le pIRE . Med Sci (Paris). 2009; ; 25 : :281.–287. 5.
Foufelle F, Ferré P. La réponse UPR : son rôle physiologique et physiopathologique . Med Sci (Paris). 2007; ; 23 : :291.–296. 6.
Mimura N, Fulciniti M, Gorgun G, et al. Blockade of XBP1 splicing by inhibition of IRE1alpha is a promising therapeutic option in multiple myeloma . Blood. 2012; ; 119 : :5772.–5781. 7.
Papandreou I, Denko NC, Olson M, et al. Identification of an Ire1alpha endonuclease specific inhibitor with cytotoxic activity against human multiple myeloma . Blood. 2011; ; 117 : :1311.–1314. 8.
Sugiura K, Muro Y, Futamura K, et al. The unfolded protein response is activated in differentiating epidermal keratinocytes . J Invest Dermatol. 2009; ; 129 : :2126.–2135. 9.
Chen X, Iliopoulos D, Zhang Q, et al. XBP1 promotes triple-negative breast cancer by controlling the HIF1alpha pathway . Nature. 2014; ; 508 : :103.–107. 10.
Wang L, Perera BG, Hari SB, et al. Divergent allosteric control of the IRE1alpha endoribonuclease using kinase inhibitors . Nat Chem Biol. 2012; ; 8 : :982.–989. 11.
Siddiqui MA, Reddy PA. Small molecule JNK (c-Jun N-terminal kinase) inhibitors . J Med Chem. 2010; ; 53 : :3005.–3012. 12.
Atkins C, Liu Q, Minthorn E, et al. Characterization of a Novel PERK kinase inhibitor with antitumor and anti-angiogenic activity . Cancer Res. 2013; ; 73 : :1993.–2002. 13.
Axten JM, Medina JR, Feng Y, et al. Discovery of 7-methyl-5-(1-{[3-(trifluoromethyl)phenyl]acetyl}-2,3-dihydro-1H-indol-5-yl)-7H-p yrrolo[2,3-d]pyrimidin-4-amine (GSK2606414), a potent and selective first-in-class inhibitor of protein kinase R (PKR)-like endoplasmic reticulum kinase (PERK) . J Med Chem. 2012; ; 55 : :7193.–7207. 14.
Dejeans N, Pluquet O, Lhomond S, et al. Autocrine control of glioma cells adhesion and migration through IRE1alpha-mediated cleavage of SPARC mRNA . J Cell Sci. 2012; ; 125 : :4278.–4287. 15.
Kardosh A, Golden EB, Pyrko P, et al. Aggravated endoplasmic reticulum stress as a basis for enhanced glioblastoma cell killing by bortezomib in combination with celecoxib or its non-coxib analogue, 2, 5-dimethyl-celecoxib . Cancer Res. 2008; ; 68 : :843.–851. 16.
D’Arcy P, Brnjic S, Olofsson MH, et al. Inhibition of proteasome deubiquitinating activity as a new cancer therapy . Nat Med. 2011; ; 17 : :1636.–1640. 17.
Suraweera A, Munch C, Hanssum A, Bertolotti A. Failure of amino acid homeostasis causes cell death following proteasome inhibition . Mol Cell. 2012; ; 48 : :242.–253. 18.
Chou TF, Li K, Frankowski KJ, et al. Structure-activity relationship study reveals ML240 and ML241 as potent and selective inhibitors of p97 ATPase . ChemMedChem. 2013; ; 8 : :297.–312. 19.
Polucci P, Magnaghi P, Angiolini M, et al. Alkylsulfanyl-1, 2, 4-triazoles, a new class of allosteric valosine containing protein inhibitors. Synthesis and structure-activity relationships . J Med Chem. 2013; ; 56 : :437.–450. 20.
Pyrko P, Schonthal AH, Hofman FM, et al. The unfolded protein response regulator GRP78/BiP as a novel target for increasing chemosensitivity in malignant gliomas . Cancer Res. 2007; ; 67 : :9809.–9816. 21.
Li J, Ni M, Lee B, et al. The unfolded protein response regulator GRP78/BiP is required for endoplasmic reticulum integrity and stress-induced autophagy in mammalian cells . Cell Death Differ. 2008; ; 15 : :1460.–1471. 22.
Goloudina AR, Demidov ON, Garrido C. Inhibition of HSP70: a challenging anti-cancer strategy . Cancer Lett. 2012; ; 325 : :117.–124. 23.
Neckers L, Workman P. Hsp90 molecular chaperone inhibitors: are we there yet? Clin Cancer Res. 2012; ; 18 : :64.–76. 24.
Jones DT, Addison E, North JM, et al. Geldanamycin and herbimycin A induce apoptotic killing of B chronic lymphocytic leukemia cells and augment the cells’ sensitivity to cytotoxic drugs . Blood. 2004; ; 103 : :1855.–1861. 25.
Goplen D, Wang J, Enger PO, et al. Protein disulfide isomerase expression is related to the invasive properties of malignant glioma . Cancer Res. 2006; ; 66 : :9895.–9902. 26.
Lovat PE, Corazzari M, Armstrong JL, et al. Increasing melanoma cell death using inhibitors of protein disulfide isomerases to abrogate survival responses to endoplasmic reticulum stress . Cancer Res. 2008; ; 68 : :5363.–5369. 27.
Hoffstrom BG, Kaplan A, Letso R, et al. Inhibitors of protein disulfide isomerase suppress apoptosis induced by misfolded proteins . Nat Chem Biol. 2010; ; 6 : :900.–906. 28.
Boyce M, Bryant KF, Jousse C, et al. A selective inhibitor of eIF2alpha dephosphorylation protects cells from ER stress . Science. 2005; ; 307 : :935.–939. 29.
Harding HP, Zhang Y, Scheuner D, et al. Ppp1r15 gene knockout reveals an essential role for translation initiation factor 2 alpha (eIF2alpha) dephosphorylation in mammalian development . Proc Natl Acad Sci USA. 2009; ; 106 : :1832.–1837. 30.
Lindquist SL, Kelly JW. Chemical and biological approaches for adapting proteostasis to ameliorate protein misfolding and aggregation diseases: progress and prognosis . Cold Spring Harbor Perspect Biol. 2011; ; 3 : :pii a004507.. 31.
Ozcan L, Ergin AS, Lu A, et al. Endoplasmic reticulum stress plays a central role in development of leptin resistance . Cell Metab. 2009; ; 9 : :35.–51. 32.
Mizukami T, Orihashi K, Herlambang B, et al. Sodium 4-phenylbutyrate protects against spinal cord ischemia by inhibition of endoplasmic reticulum stress . J Vasc Surg. 2010; ; 52 : :1580.–1586. 33.
Ben Mosbah I, Alfany-Fernandez I, Martel C, et al. Endoplasmic reticulum stress inhibition protects steatotic and non-steatotic livers in partial hepatectomy under ischemia-reperfusion . Cell Death Dis. 2010; ; 1 : :e52.. 34.
Lee AH, Scapa E, Cohen D, Glimcher L. Regulation of hepatic lipogenesis by the transcription factor XBP1 . Science. 2008; ; 320 : :1492.. |