| |
| Med Sci (Paris). 2015 August; 31(8-9): 709–712. Published online 2015 September 4. doi: 10.1051/medsci/20153108002.Valeur prédictive des génotypes et phénotypes bactériens fécaux dans la détection précoce des cancers colorectaux Charlotte Gagnière1* 1Service de gastroentérologie, Université Paris Est Créteil-Val de Marne, équipe universitaire EC2M3, hôpital Henri Mondor-APHP, 51, avenue du maréchal de Lattre de Tassigny, 94010Créteil, France MeSH keywords: Bactéries, Tumeurs colorectales, Détection précoce de cancer, Fèces, Microbiome gastro-intestinal, Génotype, Humains, Phénotype, Valeur prédictive des tests, génétique, diagnostic, microbiologie |
Le cancer colorectal (CCR) est le troisième cancer le plus fréquent chez l’homme et le second chez la femme avec près de 1,4 millions de nouveaux cas diagnostiqués en 2012 dans le monde. Bien que le taux de mortalité soit en baisse dans de nombreux pays, ce cancer reste responsable de près de 700 000 décès pour cette même année [1]. Chaque année en France, 40 000 nouveaux cas sont diagnostiqués et environ 17 000 décès sont directement imputables à l’évolution du cancer colorectal. Tous sexes confondus, il s’agit du 3e cancer le plus fréquent, après ceux de la prostate et du sein. Sa fréquence élevée, le pronostic encore réservé et le coût important pour la société, ont justifié la mise en place d’une politique de dépistage au sein de la population. |
Cancer colorectal, enjeu de santé publique La majorité (plus de 90 %) des cancers colorectaux sont sporadiques, favorisés par notre mode de vie occidental, alors que, dans près de 5 % des cas, une mutation génique constitutionnelle est responsable de la survenue de ce cancer. Ces formes héréditaires imposent chez le porteur de la mutation une surveillance régulière et précoce du côlon ou de plusieurs organes à la recherche de l’émergence d’une tumeur. À défaut d’une colectomie préventive avant l’apparition du cancer, le traitement précoce des tumeurs améliore la survie. Dans la forme sporadique, le dispositif de dépistage par la pratique d’un test biologique biennal est moins astreignant qu’une coloscopie, cette dernière restant le gold standard pour le dépistage. Néanmoins, la survenue de cancers dits « d’intervalle », entre deux tests de dépistage ou deux coloscopies consécutives, et le nombre très important de coloscopies successives normales nous obligent à mieux comprendre la genèse du cancer colorectal et à en faire une détection précoce avec un coût acceptable pour la société. C’est pourquoi, il semble pertinent d’améliorer l’estimation du risque de survenue de cancer afin de mieux programmer les coloscopies et les études de prévention chez les sujets les plus exposés, et expliquer pourquoi, même en cas de mutation constitutionnelle, pour une mutation donnée, le cancer peut survenir à des âges différents d’une famille à une autre, d’un individu à un autre. Les formes héréditaires ne seraient qu’une caricature d’un phénomène plus général où l’environnement joue le rôle déterminant [2]. L’analyse exhaustive de tous les facteurs de l’environnement n’est guère possible. L’étude du microbiote pourrait permettre d’étudier un de ces éléments environnementaux. |
Le microbiote, l’outil d’analyse de l’impact de l’environnement Le microbiote intestinal est une population complexe faite de plus de 1 000 espèces bactériennes différentes dont l’équilibre peut être considéré comme unique ; 80 % de ces espèces ne sont pas cultivables. Cela rend nécessaire l’analyse des séquences nucléotidiques d’ADN pour leur identification. L’identité génique elle-même permet l’estimation des fonctions dominantes. L’approche par analyse phénotypique du microbiote intestinal permet de rendre compte de l’ensemble des facteurs de l’hôte et de l’environnement impliqués dans la genèse des maladies [3]. Dans le domaine du cancer, une étude française préliminaire a pu rapporter pour la première fois que le profil phylogénique des bactéries fécales des sujets présentant un cancer colorectal était très différent de celui des témoins [4]. Cette donnée nous a rassurés sur le fait qu’il existait une dysbiose fécale dans le cancer colorectal, et que l’installation de ce processus était sans doute étalé dans le temps [5]. Ainsi, au-delà d’une recherche d’intérêt cognitif, nous pouvions suspecter que ce champ d’investigation aboutirait à l’émergence de nouveaux marqueurs de dépistage du cancer colorectal. |
Dépistage du cancer colorectal, un objectif de santé publique La réduction de la mortalité spécifique due au cancer colorectal passe avant tout par le dépistage des lésions précancéreuses et des formes précoces, dont l’élimination par des procédés endoscopiques et/ou chirurgicaux amène la guérison. Le dépistage organisé des cancers colorectaux entraîne une réduction de la mortalité de 25 à 30 % en France ou aux États-Unis, si au moins 50 % de la population cible y adhère [6]. La survie globale sera ainsi meilleure et le coût économique moindre pour la société. Malheureusement, encore plus d’un tiers des cancers colorectaux sont diagnostiqués tardivement, parfois au stade métastatique. Si le taux de survie à 5 ans dans les formes localisées est estimé à plus de 80 %, celui des formes diagnostiquées au stade métastatique atteint à peine 10 %. Dans la plupart des pays développés, des programmes de dépistage en population générale ont été mis en place. En France, le test de dépistage recommandé reposait - de 2009 à 2014 - sur la détection de sang occulte dans les selles par des tests Hemoccult. Depuis janvier 2015, le test d’Hemoccult est remplacé par une quantification d’hémoglobine appelée « test immunologique » [7]. Les tests sont proposés à des individus asymptomatiques (soit 80 % de la population), sans antécédents de tumeurs familiales ni de maladie inflammatoire chronique intestinale personnelle, âgés de 50 à 74 ans. Cependant, bien qu’il s’agisse d’un test non invasif et peu coûteux, il reste peu spécifique et surtout peu sensible, ne permettant de détecter que 50 % des cancers et 20 à 30 % des adénomes à haut risque. Il est donc, à l’heure actuelle, légitime de rechercher d’autres méthodes de dépistage. |
Un lien entre microbiote et cancer colorectal Une des premières études à montrer un lien entre le cancer colorectal et le microbiote est celle de Moore et al., qui, en utilisant des méthodes de culture, a observé que l’augmentation de Bacteroides et Bifidobacteria majorait le risque de polypes [8]. Plus récemment, les nouvelles méthodes de pyroséquençage et PCR ont permis de grandes avancées dans l’étude du microbiote. Ainsi, Marchesi et al. ont pu comparer, chez le même patient, des prélèvements réalisés sur les pièces opératoires au sein du tissu tumoral ou du tissu sain [9]. Ils ont observé que certaines espèces étaient surreprésentées, telles que Coriobacteridae, Roseburia, Fusobacterium et Faecalibacterium, considérées comme commensales de l’intestin. Cette surreprésentation en Fusobacterium a été confirmée par plusieurs autres études [10]. Nous avons également montré cette modification du microbiote (augmentation du groupe Bacteroides/Prevotella) chez les patients atteints de cancer colorectal. Cette dysbiose semblait en rapport avec un recrutement plus important des cellules immunes productrices d’interleukine (IL)-17 dans la muqueuse normale des patients atteints d’un cancer colorectal [4]. Pour déterminer le caractère causal de ces anomalies, nous avons procédé à un transfert de flore chez des souris axéniques. Les souris colonisées par le microbiote de patients atteints de cancer colorectal ont développé des lésions précancéreuses (indice de prolifération intestinale ; cryptes aberrantes coliques) et une activation du système de transduction du signal dans la voie NOTCH [11]. Le rôle fondamental du microbiote dans la survenue et le développement des cancers colorectaux sporadiques, via l’implication de certaines sous-classes bactériennes commensales et des mécanismes relatifs à la dysrégulation des systèmes métabolique et immunitaire, a été analysé dans une revue récente [12]. |
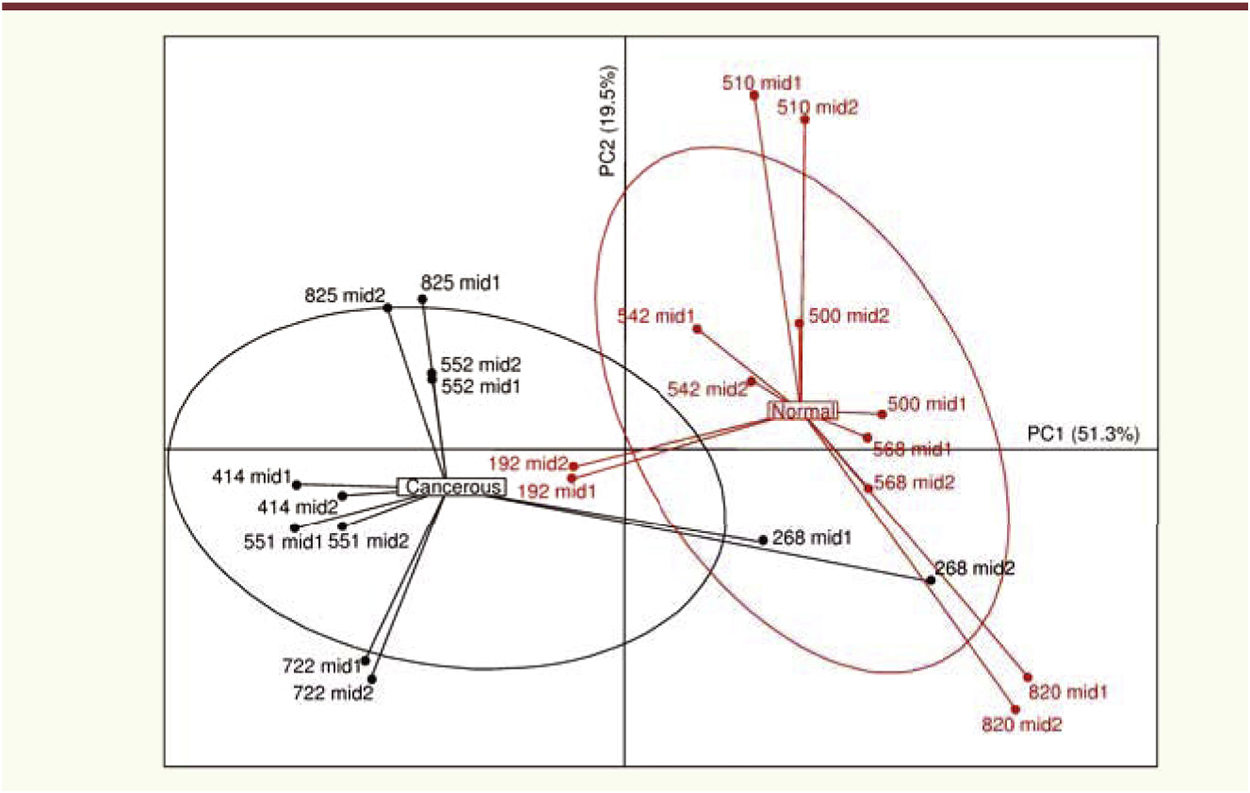
Contribution du profil de microbiote au dépistage Des modifications du microbiote sont ainsi observées dès les premières étapes de la carcinogenèse, et pourraient être détectées précocement si des marqueurs microbiologiques existaient. Notre étude s’est donc intéressée à la faisabilité d’un test de dépistage basé sur l’analyse du microbiote [13]. Notre équipe a entrepris l’analyse de l’ensemble des bactéries retrouvées dans les selles de patients atteints d’un cancer colique ou rectal, et comparé les résultats à celle réalisée chez des individus dont la coloscopie était normale et chez des patients porteurs de lésions intermédiaires, sous forme de polypes adénomateux. Notre équipe a été la première à rapporter en 2011 des différences majeures entre les profils du noyau phylogénique du microbiote du cancer colorectal par rapport à celui de sujets sains (Figure 1). Trois populations ont été définies pour la présente étude :
 | Figure 1.
Le microbiote colique des sujets porteurs d’un cancer colorectal et des sujets normaux (N). L’étude est faite sur la base du séquençage du gène de l’ARN 16S des bactéries fécales et de la comparaison phylogénique de composantes communes (figure reprise de [ 4], © PLoS One). |
1. la population française F, issue d’une cohorte de sujets explorés au CHU Henri Mondor-APHP (Créteil, France) pour une coloscopie de dépistage, recrutés entre 2004 et 2006. Ces individus devaient réaliser un test Hemoccult de dépistage avant la coloscopie, ainsi qu’un recueil de selles pour une étude métagénomique du microbiote. Au total, 156 échantillons de selles (53 patients ayant un cancer colorectal, 42 ayant un adénome et 61 contrôles ayant une coloscopie normale) ont été sélectionnés, l’ADN fécal extrait, et un séquençage métagénomique réalisé. De plus, chez tous ces individus, une quantification de la méthylation du promoteur du gène WIF1 (régulant la principale voie de carcinogenèse colique) a été effectuée sur un échantillon sanguin [14, 15]. Pour 129 de ces patients, une amplification d’ADNr 16S a pu être réalisée en vue d’un pyroséquençage de la région 16s. 2. La population G, issue de l’étude internationale (Allemagne et États-Unis) Colocare, qui incluait 38 patients avec un diagnostic récent de cancer colorectal porté au vu des résultats de l’examen coloscopique et de l’analyse histologique, a été utilisée pour valider les résultats obtenus à partir de la population F. Le recueil des selles était effectué avant la chirurgie. 3. Enfin, la population H concernait 297 individus (Allemands, Danois et Espagnols) témoins, indemnes de toute pathologie tumorale (N = 282 normaux et 25 patients atteints d’une maladie chronique inflammatoire intestinale) chez lesquels une étude métagénomique complète colique a été réalisée. L’analyse métagénomique de la population F a montré une modification de l’abondance des différents taxons chez les patients atteints de cancer. Ainsi, il existait une augmentation des phylums des Fusobacteria, Proteobacteria, et Bacteroidetes, tandis que les populations d’Actinobacteria et de Firmicutes étaient diminuées. Une évaluation du lien prédictif entre la composition en taxons et le cancer colorectal a ensuite été réalisée. Plus de la moitié des différences étaient liées à quatre bactéries dont deux du groupe des Fusobacteria. Les données récentes de la littérature suggérant que les fusobactéries sont associées à ce cancer, nous avons retenu, par le test métagénomique, les 2 sous-espèces F. nucleatum vincentii et F. nucleatum animalis comme les plus pertinents. Lorsqu’on compare les performances intrinsèques du score bactérien dans la population F, ses valeurs de sensibilité et de spécificité paraissent meilleures que celles obtenues avec l’Hemoccult ou le test sanguin de détection basé sur la méthylation Wif-1, comme en atteste l’aire sous la courbe (AUC : area under the curve) qui illustre généralement la variation simultanée de sensibilité et de spécificité (ici à 0,84 dépassant de manière significative celle générée par Hemoccult ou par le Wif-1). La combinaison du test Hemoccult et du test métagénomique permettait d’améliorer le test de dépistage en augmentant l’AUC à 0,87. De plus, la sensibilité du test était identique dans les cancers localisés et les cancers avancés ou métastatiques. Ces résultats étaient confirmés après validation croisée avec les populations G et H (AUC = 0,85), témoignant qu’un test de détection métagénomique fiable et reproductible est possible. Afin de nous assurer de l’impact relatif de ces bactéries, la composition du microbiote fécal ainsi que celle des bactéries adhérentes (s’attachant à la muqueuse par opposition à celles qui sont libres dans la lumière) ont été déterminées par une méthode de qPCR, ciblant les gènes codant pour les ARN ribosomaux 16S bactériens, notamment la région V4 du gène. Ce séquençage a été réalisé à partir d’échantillons de tissus (tumoral et sain) des patients opérés d’un cancer colorectal (population G), et à partir d’échantillons de selles (populations F et H). L’implication d’une dizaine de bactéries dont la surexpression pourrait être considérée comme un facteur favorisant le processus de carcinogenèse colique a ainsi été identifiée et, en premier lieu, la surreprésentation de deux espèces de fusobactéries. Une prédominance du microbiote oral (ensemble des bactéries présentes dans la bouche) a été notée dans les selles des patients atteints de cancer colorectal comme de maladies inflammatoires, ce qui a déjà été rapporté dans d’autres maladies digestives. De façon fort intéressante, il existe un chevauchement entre la signature bactérienne de cancer colorectal et celle, plus spécifique, des maladies inflammatoires chroniques de l’intestin. |
Profil fonctionnel bactérien dans le cancer colorectal Une autre approche intéressante était la recherche des modifications des fonctions du microbiote, par quantification des modules procaryotiques à partir des banques internationales de données géniques (KEGG-Kyoto Encyclopedia of genes and genomes), et enzymatiques (CAZy-Carbohydrate active enzymes) dans le métagénome. L’analyse de ces fonctions a montré une tendance à l’utilisation des ressources énergétiques des cellules de l’hôte et des protéines animales dans le cas du microbiote associé au cancer colorectal, avec une augmentation de la capacité à métaboliser certains acides aminés via la voie de la putréfaction. Une majoration du métabolisme des lipopolysaccharides a également été observée. Un test de détection du cancer colorectal, basé sur ces modifications, a également été réalisé ; l’AUC mesurée était de 0,77, plus faible que celle obtenue avec le test génomique. L’utilisation combinée des tests génomique et fonctionnel donne une AUC à 0,87. Le test combiné permettrait probablement d’améliorer la détection précoce des tumeurs. En conclusion, les données récentes de la littérature montrent l’association étroite qui existe entre les modifications quantitatives et fonctionnelles du microbiote et le développement d’un cancer colorectal. Les tests de dépistage actuels pourraient être améliorés si on y intégrait les données de ce nouveau champ d’investigation. En outre, les modifications fonctionnelles relatives aux bactéries vont ouvrir de nouvelles pistes d’étude de la carcinogenèse colique. |
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Torre LA, Bray F, Siegel RF, et al. Global cancer statistics, 2012 . CA Cancer J Clin. 2015; ; 65 : :87.–108. 2.
Samadder NJ, Curtin K, Tuohy TM, et al. Increased risk of colorectal neoplasia among family members of patients with colorectal cancer: a population-based study in Utah . Gastroenterology. 2014; ; 147 : :814.–821. 3.
McLean MH, Dieguez D Jr, Miller LM, et al. Does the microbiota play a role in the pathogenesis of autoimmune diseases? Gut. 2015; ; 64 : :332.–341. 4.
Sobhani I, Tap J, Roudot-Thoraval F, et al. Microbial dysbiosis in colorectal cancer (CRC) patients . PLoS One. 2011; ; 6 : :e16393.. 5.
Jobin C. Microbiome Un nouveau facteur de risque de cancer colorectal ? Med Sci (Paris). 2013; ; 29 : :582.–585. 6.
Faivre J, Dancourt V, Lejeune C, et al. Reduction in cancer colorectal mortality by fecal occult blood screening in a French controlled study . Gastroenterology. 2004; ; 126 : :1674.–1680. 8.
Moore WE, Moore LH. Intestinal floras of populations that have a high risk of colon cancer . Appl Environ Microbiol. 1995; ; 61 : :3202.–3207. 9.
Marchesi JR, Dutilh BE, Hall N, et al. Towards the 661 human colorectal cancer microbiome . PLoS One. 2011; ; 6 : :e20447.. 10.
Warren RL, Freeman DJ, Pleasance S, et al. Co-occurrence of anaerobic bacteria in colorectal carcinomas . Microbiome. 2013; ; 1 : :16.. 11.
Sobhani I, Amiot A, Le Baleur Y, et al. Microbial dysbiosis and colon carcinogenesis: could colon cancer be considered a bacteria-related disease? Therap Adv Gastroenterol. 2013; ; 6 : :215.–229. 12.
Keku TO, Dulal S, Deveaux A, et al. The gastrointestinal microbiota and colorectal cancer . Am J Physiol Gastrointest Liver Physiol. 2015; ; 308 : :G351.–G363. 13.
Zeller G, Tap J, Voigt AY, et al. Potential of fecal microbiota for early-stage detection of colorectal cancer . Mol Syst Biol. 2014; ; 10 : :766.. 14.
Amiot A, Mansour H, Baumgartner I, et al. The detection of methylated Wif-1 gene is more accurate for identifying advanced adenomas and colorectal cancer than faecal occult blood test . PLoS One. 2014; ; 9 : :e99233.. 15.
Roperch JP, Incitti R, Forbin S, et al. Aberrant methylation of NPY, PENK, and WIF1 as a promising marker for blood-based diagnosis of colorectal cancer . BMC Cancer. 2013; ; 13 : :566.. |