| |
| Med Sci (Paris). 2015 August; 31(8-9): 770–776. Published online 2015 September 4. doi: 10.1051/medsci/20153108016.Potentiel des ARN non codants comme biomarqueurs dans l’insuffisance cardiaque
Florence Pinet1* and Christophe Bauters1 1Inserm U1167, Institut Pasteur de Lille, université de Lille, CHRU de Lille, faculté de médecine, 1, rue du Professeur Calmette, 59019Lille, France |
Vignette (Photo © Inserm-Daniel Caro). Les maladies cardiovasculaires sont la principale cause de mortalité dans les pays industrialisés avec 1 900 000 morts dans l’Union européenne entraînant un coût estimé à 196 milliards d’euros [1]. En ce qui concerne l’insuffisance cardiaque (IC), sa prévalence a été estimée à 1-2 % de la population adulte des pays industrialisés et augmente avec l’âge puisqu’elle dépasse 10 % chez les individus âgés de plus de 70 ans [2]. En dépit d’importants progrès thérapeutiques, la mortalité des patients insuffisants cardiaques demeure élevée [3], en particulier pour les formes les plus sévères de la maladie. Il existe deux formes d’insuffisance cardiaque : l’insuffisance cardiaque systolique, qui est caractérisée par un dysfonctionnement systolique du ventricule gauche (VG) accompagné d’une réduction de la fraction d’éjection (FE, pourcentage du sang contenu dans la cavité qui est éjecté au cours d’un battement cardiaque), et l’insuffisance cardiaque avec fraction d’éjection préservée, qui se développe classiquement chez les patients âgés, hypertendus, et/ou diabétiques. Après un diagnostic d’insuffisance cardiaque, la survie à 5 ans est estimée à 50 % [4]. L’évaluation pronostique fait donc partie intégrante de la prise en charge du patient insuffisant cardiaque. En effet, les patients identifiés à « haut risque » sont éligibles pour les thérapeutiques de recours que sont l’assistance et/ou la transplantation cardiaque. L’évaluation pronostique du patient insuffisant cardiaque fait classiquement appel à plusieurs paramètres parmi lesquels le stade de la maladie selon la classification NYHA (New York Heart Association), la fonction ventriculaire gauche, la fonction ventriculaire droite, le taux de peptide natriurétique de type B (BNP pour brain natriuretic peptide, responsable de l’excrétion de sodium par les reins) et les paramètres issus de l’épreuve d’effort métabolique (pic de VO2) [5, 6]. Il persiste toutefois une importante variabilité dans le pronostic de l’insuffisance cardiaque, des patients considérés à faible risque présentant une évolution défavorable, et des patients évalués à haut risque ayant, de manière surprenante, une survie très prolongée. De plus, certains paramètres pronostiques puissants, comme ceux de l’épreuve d’effort métabolique par exemple, ne sont pas toujours disponibles en pratique clinique. Il existe donc un besoin important de nouveaux indicateurs pronostiques dans l’insuffisance cardiaque. Idéalement, il devrait s’agir de marqueurs non invasifs, facilement disponibles, ne dépendant pas de l’opérateur, et relativement peu coûteux. Les biomarqueurs circulants correspondent à ce « cahier des charges ». À l’heure actuelle, le BNP est le seul biomarqueur circulant qui est utilisé en routine dans cette indication [7]. En effet, chez les patients ayant une insuffisance cardiaque systolique stable, la détermination du BNP permet une meilleure information pronostique, indépendante de la fraction d’éjection ou du pic de consommation d’oxygène [8], et elle est utile pour la stratification du risque. En revanche, il n’existe pas à l’heure actuelle de biomarqueurs permettant de prédire le pronostic des patients insuffisants cardiaques dont la fraction d’éjection est préservée. L’enjeu est alors d’anticiper l’évolution vers le stade terminal de la maladie afin d’adapter les thérapeutiques, et notamment d’envisager l’éventualité d’une assistance ventriculaire ou d’une transplantation cardiaque. Le manque de greffons, les délais d’attente et les complications de la greffe justifient néanmoins de sélectionner au mieux les patients qui bénéficieront de cette thérapeutique. Pour cela, le suivi d’un patient insuffisant cardiaque sévère nécessite l’évaluation régulière d’un certain nombre de marqueurs pronostiques. Cependant, à l’heure actuelle, ces marqueurs ne permettent pas d’apprécier le risque évolutif de ces patients, soulignant le réel besoin de nouveaux marqueurs pronostiques indicateurs de mortalité précoce. |
Découverte des ARN non codants comme biomarqueurs de l’insuffisance cardiaque Durant les dernières années, plusieurs candidats biomarqueurs ont été étudiés dans le champ de l’insuffisance cardiaque. Une revue de E. Braunwald montre que les biomarqueurs de l’insuffisance cardiaque identifiés se divisent en 7 catégories de protéines/peptides : des marqueurs de l’inflammation, du stress oxydant, du remodelage de la matrice extracellulaire, de lésion cardiaque, de stress du myocyte, des neurohormones et des marqueurs de dysfonction rénale [9]. Plus récemment, de nombreuses études dans le domaine de la recherche sur les ARN ont mis en évidence l’importance des ARN non codants (ncARN) dans la régulation de l’expression génique [35](→). Ces ncARN sont d’importants régulateurs cellulaires qui se divisent en plusieurs catégories, telles que les micro-ARN (miARN), de taille d’environ 22 nucléotides, et les ARN longs non codants (lncARN) (de taille supérieure à 200 nucléotides). Les miARN et lncARN sont d’importants régulateurs intracellulaires de l’expression génique, mais ils peuvent aussi réguler de manière directe ou indirecte les protéines.
(→) Voir la Synthèse de T. Pedrazzini, m/s n°3, mars 2015, page 261
Plus de 2 000 miARN ont été isolés et, récemment, plus de 40 miARN ont été identifiés comme étant impliqués dans les pathologies cardiovasculaires [10, 11]. En effet, les miARN sont très stables et donc détectables dans le sang et d’autres fluides biologiques tels que l’urine [12]. Ils peuvent donc être utilisés pour le dépistage, le pronostic et l’amélioration du diagnostic des maladies cardiovasculaires telles que le remodelage du ventricule gauche post-infarctus (Figure 1A) et l’insuffisance cardiaque. Malgré leur potentiel, les miARN ne s’avèrent pas supérieurs aux marqueurs classiques utilisés du fait de plusieurs limitations, notamment leur faible concentration dans le plasma ou le sérum [10]. Jusqu’à présent, 23 études ont testé l’intérêt des miARN comme biomarqueurs, mais, dans l’insuffisance cardiaque, un grand nombre de ces études ont été effectuées sur de faibles effectifs de patients (< 50) [10]. Or, il est très important de tester un grand nombre de patients pour les études de biomarqueurs. Ainsi, récemment, les taux circulants de deux miARN décrits comme biomarqueurs potentiels de l’insuffisance cardiaque, les miR-133a et miR-423-5p, ont été quantifiés chez 246 patients inclus dans le cadre de l’étude REVE2. Les patients étaient suivis pendant un an par échographie après un infarctus du myocarde inaugural (i.e. cet infarctus n’était précédé d’aucune douleur prémonitoire) et des prélèvements sanguins ont été réalisés pendant l’hospitalisation, puis à 1 mois, 3 mois et 12 mois après l’infarctus [13]. Bien que des variations des taux des miR-133a et miR-423-5p aient été observées en fonction du temps, aucune association avec les taux circulants de BNP, ni avec le remodelage ventriculaire gauche quantifié par échographie n’était observée [14]. La disponibilité d’une cohorte européenne HOMAGE, regroupant plus de 40 000 sujets atteints d’insuffisance cardiaque provenant de 8 pays européens, devrait permettre d’identifier des miARN biomarqueurs de cette pathologie [15]. La caractérisation des miARN est donc en plein essor et leur potentiel de biomarqueur, mis en évidence dans le champ de l’oncologie [36, 37] (→), devrait pouvoir s’appliquer à un très large spectre de maladies [16].
(→) Voir la Synthèse de Y. Ladeiro et J. Zucman-Rossi, m/s n° 5, mai 2009, page 467 et celle de A. Gougelet et S. Colnot, m/s n° 10, octobre 2013, page 861
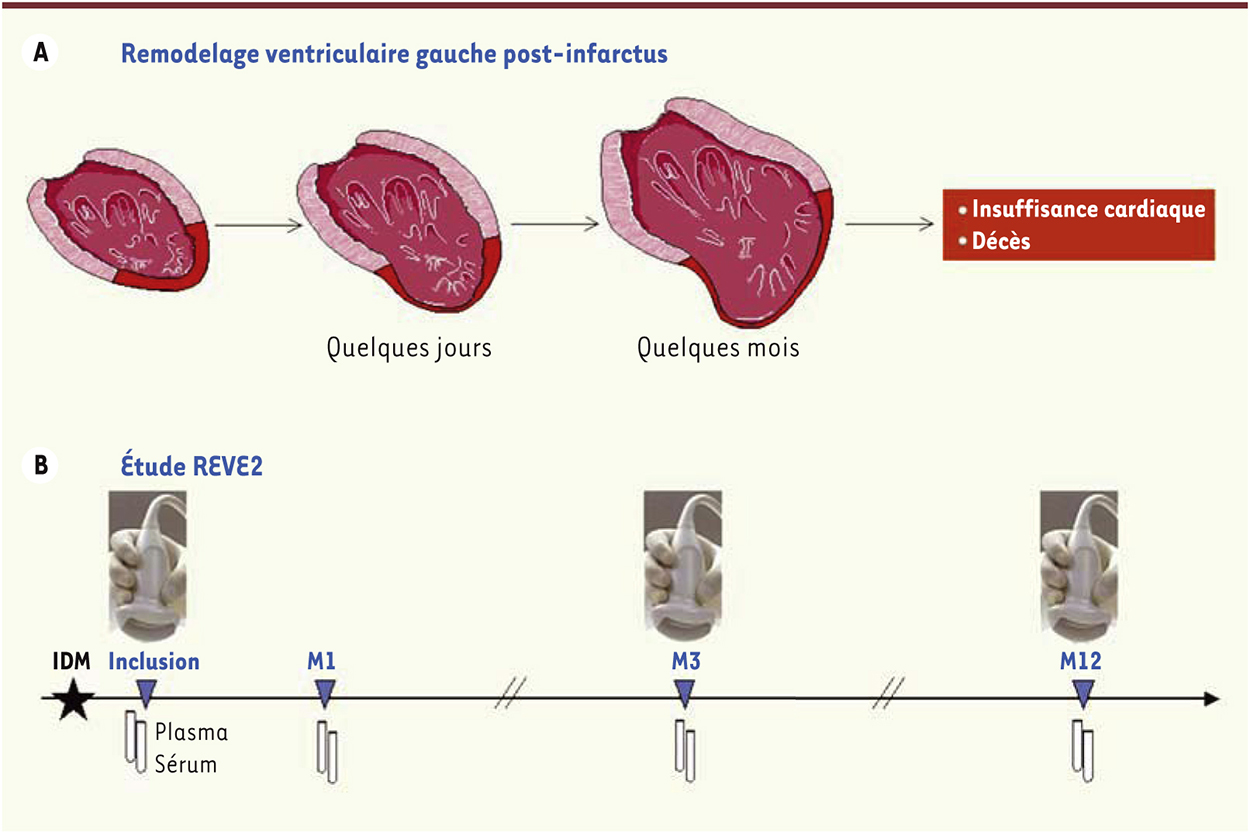
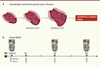 | Figure 1.
Représentation schématique du remodelage ventriculaire gauche après un infarctus du myocarde. A. Le remodelage ventriculaire gauche apparaît en quelques jours après un infarctus et peut se développer en quelques mois pour devenir délétère et conduire à l’insuffisance cardiaque et au décès des patients. Il peut être quantifié par imagerie en calculant le delta entre la mesure effectuée en post-infarctus et en basal au moment de l’hospitalisation. B. Plan de l’étude REVE 2 conçue pour la recherche de biomarqueurs du remodelage ventriculaire gauche par suivi échocardiographique et prélèvements sériés de plasma et de sérum des patients recrutés après un premier infarctus et suivis pendant un an [ 13]. IDM : infarctus du myocarde ; M : mois. |
L’autre catégorie de IncARN qui a émergé récemment est celle des lncARN, qui interviennent dans la régulation du génome aussi bien au niveau transcriptionnel que post-transcriptionnel [17], et qui ont été impliqués dans diverses pathologies [18]. Les lncARN sont des transcrits de plus de 200 nucléotides, qui ne sont pas traduits en protéines, dont la spécificité d’espèce est très importante, et qui sont donc beaucoup moins conservés que les gènes codant pour des protéines. Le projet ENCODE (encyclopedia of DNA elements), à travers la base de données GENCODE (http://www.gencodegenes.org), maintient à jour la liste de lncARN exprimés chez l’homme (15 877 lncARN détectés dans la version V21) [19]. Entre 500 et 700 de ces lncARN ont été identifiés comme régulés dans le tissu ventriculaire de patients insuffisants cardiaques [20]. Ces données récentes suggèrent l’utilité des lncARN circulants comme nouveaux biomarqueurs des pathologies cardiovasculaires. La question qui se pose est la suivante : les taux plasmatiques des lncARN pourraient-ils prédire la mortalité précoce chez des patients insuffisants cardiaques et permettre d’envisager une prise en charge appropriée et bénéfique pour ces patients. |
Stratégie utilisée pour découvrir et sélectionner des lncARN comme biomarqueurs circulants dans l’insuffisance cardiaque La stratégie la plus fréquemment utilisée pour découvrir des biomarqueurs est d’effectuer une étude cas/contrôle et ensuite de valider les biomarqueurs identifiés dans une autre cohorte de patients. Dans le domaine de l’insuffisance cardiaque, plusieurs approches peuvent être utilisées comme nous l’avons précédemment publié pour la recherche de biomarqueurs de l’insuffisance cardiaque par analyse protéomique [21]. Nous avons choisi de travailler avec des patients chez lesquels existe un remodelage ventriculaire gauche post-infarctus, le remodelage étant fréquemment un stade de transition évoluant vers une insuffisance cardiaque. Deux études, REVE [22] et REVE2 [13], ont montré que ce remodelage, témoin d’une dilatation progressive du ventricule gauche, est observé après un infarctus du myocarde chez 30 % des patients, et ce en dépit des meilleurs traitements connus actuellement. Ces patients sont à haut risque de développer une insuffisance cardiaque (Figure 1A). Le remodelage ventriculaire gauche1, peut être aisément quantifié par les techniques d’imagerie (échographie ou IRM cardiaques) et des prélèvements plasmatiques sériés, obtenus pendant le suivi de l’infarctus (Figure 1B), permettent d’analyser la cinétique des taux de biomarqueurs, comme nous avons pu le mettre en évidence pour les marqueurs conventionnels cardiaques [13]. L’autre intérêt de faire ces études pendant la phase de remodelage ventriculaire gauche est que les biomarqueurs identifiés comme associés à un remodelage VG élevé sont aussi retrouvés dans l’IC chronique [23]. L’approche utilisée a été un criblage par puces des lncARN présents dans le plasma. Les résultats ont été comparés chez deux groupes de patients (n = 15 dans chaque groupe) appartenant à la cohorte REVE2, présentant un fort ou un faible remodelage ventriculaire gauche (Figure 2A) [13]. Pour chaque groupe, les échantillons plasmatiques de 5 patients, prélevés 5 jours après l’infarctus, ont été groupés avant l’extraction ARN. Au total, 33 045 lncARN ont été criblés sur 3 échantillons de 5 patients présentant un fort remodelage ventriculaire gauche et 3 échantillons de 5 patients présentant un faible remodelage ventriculaire gauche (Figure 2B). Ce criblage a permis de détecter 768 lncARN dont 550 étaient surexprimés et 218 sous-exprimés chez les patients présentant un fort remodelage ventriculaire gauche. Près de 80 % de ces lncARN étaient d’origine génomique mitochondriale. Le taux de 15 de ces 768 lncARN était modulé d’un facteur trois entre les 2 groupes de patients, et suffisant pour pouvoir être détecté de manière fiable. Seuls 7 de ces 15 lncARN, tous d’origine mitochondriale, ont pu être amplifiés individuellement chez chacun des patients testés dans ces 2 groupes et étaient significativement sous-exprimés chez les patients présentant un fort remodelage ventriculaire gauche [24].
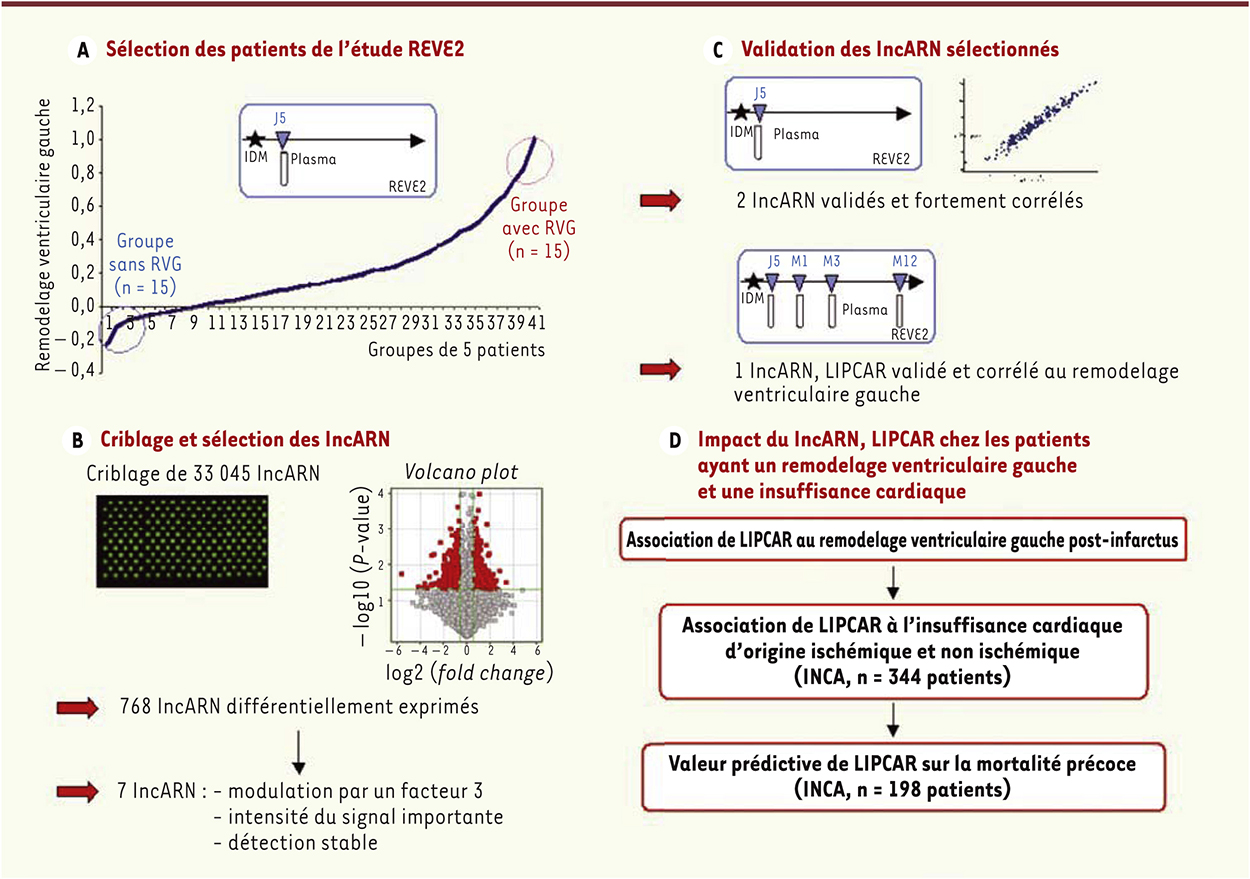
 | Figure 2.
Schéma récapitulatif de l’approche utilisée pour découvrir un lncARN, biomarqueur circulant de l’insuffisance cardiaque. A. Les patients de l’étude REVE2 ont été regroupés par 5 en fonction du remodelage ventriculaire gauche (RVG) déterminé avec l’échographie effectuée 1 an après l’infarctus du myocarde (IDM). Le prélèvement plasmatique obtenu à 5 jours post-infarctus dans les 3 groupes de patients sans RVG et avec RVG extrême a été utilisé pour le criblage des lncARN. B. À partir de 33 045 lncARN criblés, 768 lncARN ont été détectés comme différentiellement exprimés. Les sept lncARN dont le taux était modulé au moins par un facteur 3, une intensité de signal importante et une stabilité de détection ont été sélectionnés pour validation. C. Deux des sept lncARN quantifiés dans le prélèvement à J5 chez les 246 patients de l’étude REVE 2 ont été sélectionnés pour validation. Le lncARN présentant la plus forte modulation, nommé LIPCAR, a été quantifié dans les 4 prélèvements des 226 patients de l’étude REVE2 et son taux était corrélé au remodelage ventriculaire gauche. D. L’impact de LIPCAR a été recherché chez 344 patients présentant une insuffisance cardiaque d’origine ischémique ou non ischémique, ainsi que sa valeur prédictive sur la mortalité précoce des patients insuffisants cardiaques (99 décédés à 3 ans, 99 survivants) après ajustement sur les marqueurs classiques tels le stade NYHA, la fraction d’éjection du ventricule gauche, les taux de BNP et le pic de VO2. |
Les 7 lncARN ont ensuite été quantifiés dans le prélèvement initial effectué chez les 246 patients de l’étude REVE2 (Figure 2C). Bien que les taux plasmatiques de ces 7 lncARN soient corrélés positivement entre eux, seuls deux d’entre eux, dont les taux étaient significativement sous-exprimés dans le prélèvement initial, se sont donc avérés prédictifs du remodelage ventriculaire gauche quantifié 1 an après l’infarctus du myocarde. Nous avons sélectionné le lncARN uc022bqs.1 qui a été nommé LIPCAR (pour long intergenic non coding RNA predicting cardiac remodeling). Les taux plasmatiques de LIPCAR 5 jours après un infarctus sont significativement inférieurs chez les patients qui développeront un fort remodelage ventriculaire gauche (> 20%) (voir note 1). Des prélèvements sanguins sériés durant cette période ont permis de mettre en évidence le fait que, alors que les taux plasmatiques de LIPCAR étaient sous-exprimés juste après l’infarctus, ils évoluaient et devenaient significativement plus élevés à 1 mois, 3 mois et un an post-infarctus chez les patients qui présentaient un fort remodelage ventriculaire gauche (Figure 2D). Le suivi échocardiographique effectué parallèlement a permis d’évaluer le remodelage. Du fait de ces taux plus élevés un an après l’infarctus, nous avons émis l’hypothèse selon laquelle les taux du lncARN LIPCAR seraient encore plus élevés chez les patients présentant une insuffisance cardiaque chronique [24]. Nous avons testé cette hypothèse chez les patients recrutés dans l’étude INCA, coordonnée par le Dr Pascal de Groote, qui sont adressés dans le service pour une évaluation pronostique d’un dysfonctionnement systolique du ventricule gauche. Ce bilan incluait une évaluation de leur classe NYHA, une échocardiographie pour évaluer la fraction d’éjection du VG, une épreuve d’effort métabolique, et un dosage du BNP. Pour être inclus, les patients devaient être stables et avoir une fraction d’éjection du VG inférieure à 45 %. Au moment de l’entrée dans l’étude, un prélèvement sanguin était réalisé. Entre janvier 2006 et mai 2010, nous avons mesuré les taux plasmatiques de LIPCAR chez 344 patients consécutifs, d’âge similaire à celui des patients de l’étude REVE2, inclus dans la cohorte INCA et montré qu’ils étaient significativement plus élevés (p < 0,0001) que les taux mesurés à 1 an post-infarctus chez les patients présentant un fort remodelage ventriculaire gauche (Figure 3A). Ces taux plus élevés de LIPCAR ont été retrouvés aussi bien chez les patients ayant une insuffisance cardiaque d’origine ischémique que non ischémique (Figure 3A). Parmi ces 344 patients, 43 sont décédés de cause cardiovasculaire au cours des 3 ans de suivi, et nous avons trouvé une association statistiquement significative entre les taux de LIPCAR à l’inclusion du patient dans la cohorte et la mortalité cardiovasculaire (odds ratio de 1,42 [1,02-2,01], p = 0,04) (Figure 2D). Nous avons ensuite testé la valeur pronostique de LIPCAR dans l’insuffisance cardiaque sur une autre série de patients de la cohorte INCA recrutés entre novembre 1998 et décembre 2005. Le groupe cas était constitué de 99 patients qui sont décédés de cause cardiaque dans les 3 ans qui suivirent leur entrée dans l’étude, et le groupe témoin était constitué de 99 patients appariés individuellement pour l’âge, le sexe, et l’étiologie de l’insuffisance cardiaque et survivants après 3 ans. Nous avons observé que les taux de LIPCAR étaient significativement plus élevés chez les patients décédés pendant les 3 ans de suivi que chez les patients non décédés (Figure 3B). Nous avons montré, après ajustement de l’âge, du sexe, de l’étiologie de l’insuffisance cardiaque, du diabète, du stade NYHA, de la fraction d’éjection du ventricule gauche, du taux de BNP et du pic de VO2, que le taux de LIPCAR était un facteur indépendant de prédiction de la mortalité cardiovasculaire à 3 ans (odds ratio de 4,16 [2,67-6,90], p < 0,0001) (Figure 2D).
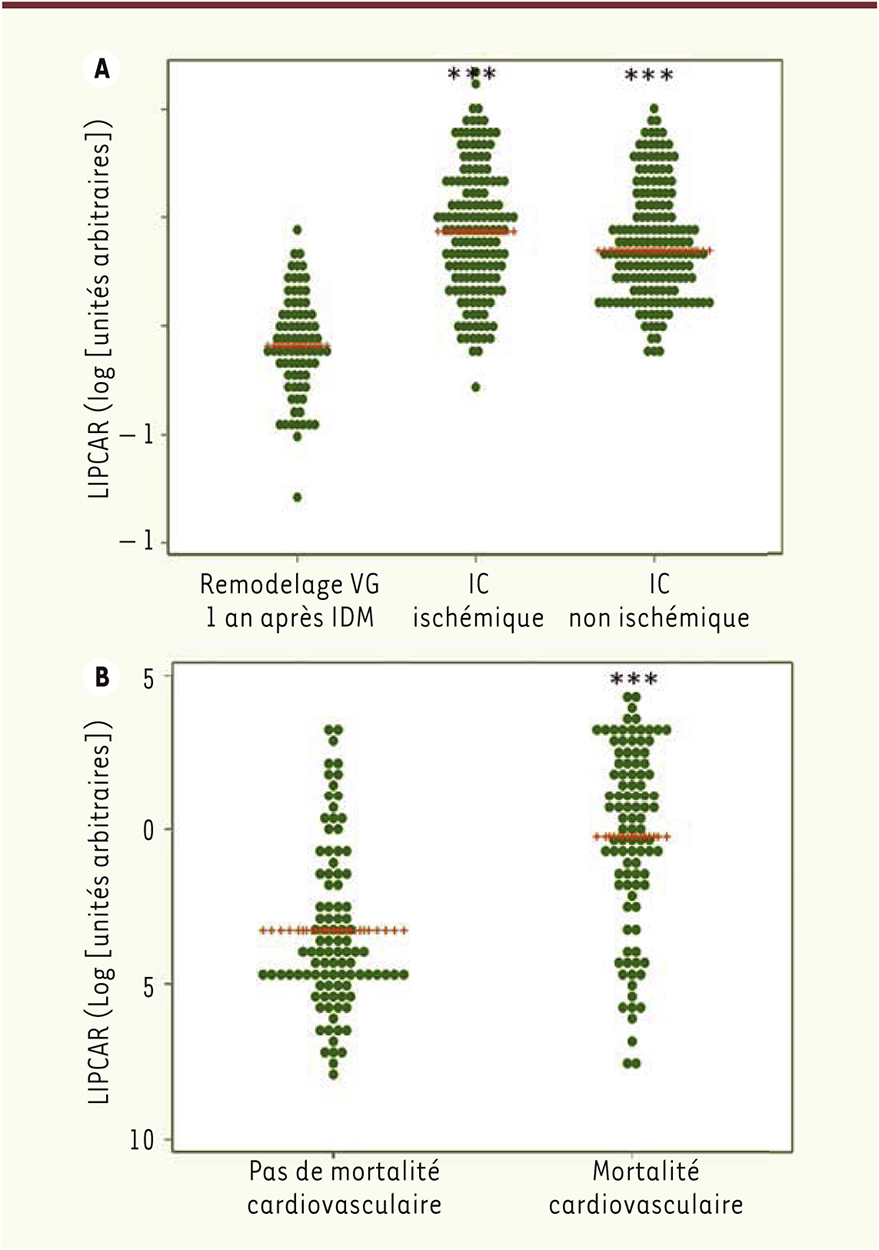
 | Figure 3.
Taux plasmatiques de l’ARN long non codant (lncARN) LIPCAR (long intergenic non-coding RNA predicting cardiac remodeling) chez les patients insuffisants cardiaques. A. Valeurs individuelles des taux plasmatiques de LIPCAR mesurés par qRT-PCR un an post-infarctus chez les 87 patients de l’étude REVE2 présentant un remodelage ventriculaire gauche élevé (VG) et chez les patients ayant une insuffisance cardiaque (IC) de l’étude INCA d’origine ischémique (n = 164) et non ischémique (n = 180). ***p < 0,0001 versus remodelage VG. B. Valeurs individuelles des taux plasmatiques de LIPCAR mesurés lors de l’évaluation de l’IC chez les patients de l’étude cas-contrôle INCA, présentant (n = 99) ou non (n = 99) une mortalité cardiovasculaire pendant le suivi à 3 ans. ***p < 0,0001 versus pas de mortalité cardiovasculaire. IDM : infarctus du myocarde (figure adaptée de la figure 2 de [ 24]). |
Cette étude a permis d’identifier le lncARN LIPCAR comme un nouveau biomarqueur du remodelage cardiaque et de prédiction d’une mortalité précoce chez les patients IC [24]. |
Autres approches de criblage de lncARN dans l’insuffisance cardiaque Une autre approche de criblage est d’utiliser le séquençage profond d’ARN (ARN Seq) [35] à partir de tissus cardiaques provenant de patients insuffisants cardiaques [25]. Il a été montré que le taux d’expression de 10 % de ces transcrits se normalise chez les patients ayant un dispositif d’assistance ventriculaire. Comme ceux du plasma, la majorité des lncARN détectables dans le tissu cardiaque étaient d’origine mitochondriale. De manière intéressante, le profil d’expression de ces lncARN permet de distinguer les cardiomyopathies d’origine ischémique ou non, suggérant que ces lncARN possèdent des fonctions distinctes selon le mécanisme en cause dans l’insuffisance cardiaque [25]. Dans une autre étude, faite dans un modèle murin expérimental d’infarctus du myocarde, un criblage des IncARN par la même technique sur des échantillons de cœur, a mis en évidence des groupes de lncARN associés à un remodelage pathologique également identifiés chez l’homme [26]. À partir d’une analyse transcriptomique sur puce, les auteurs ont mis en évidence que le lncARN AK048451, qu’ils ont nommé CHRF (cardiac hypertrophy related factor), peut se lier au miARN miR-489 et, ainsi, réguler son niveau d’expression. Or miR-489 contrôle lui-même l’expression de Myd88 (myeloid differentiation primary response gene 88) dans les cardiomyocytes hypertrophiques, faisant du trio lncARN AK048451 – miR-489 – Myd88 une voie de régulation de l’hypertrophie cardiaque [27]. L’autre démarche pour déterminer si les IncARN peuvent représenter de bons biomarqueurs est d’utiliser les données de la littérature sur les lncARN identifiés comme étant associés aux pathologies cardiaques [35]. Pour cela, 5 lncARN (aHIF, hypoxia inducible factor 1A antisense RNA 2 ; ANRIL, cyclin-dependent kinase inhibitor 2B antisense RNA 1 ; KCNQ10T1, potassium voltage-gated channel, KQT-like subfamily, member 1 opposite strand/antisense transcript 1 ; MIAT, myocardial infarction-associated transcript ; et MALAT1, metastasis-associated lung adenocarcinoma transcript 1) ont été quantifiés par PCR quantitative dans les cellules sanguines isolées chez des patients ayant eu un infarctus, puis suivis par échographie 4 mois après pour déterminer le dysfonctionnement ventriculaire gauche, et chez des volontaire sains. Les taux de aHIF, KCNQ10T1 et MALAT1 étaient plus élevés, et ceux d’ANRIL moins élevés chez les patients ayant eu un infarctus que chez les volontaires sains. Les taux de MIAT n’étaient pas différents entre les deux groupes. En revanche, aucun de ces 5 lncARN ne prédisait une dysfonction ventriculaire gauche [28]. Toutefois, certains des lncARN testés ont été impliqués dans d’autres pathologies, telles que le cancer, et ne sont donc pas spécifiques des pathologies cardiaques. Pour cibler spécifiquement les lncARN impliqués dans le développement cardiaque et les pathologies associées, une étude récente a combiné les données de séquençage profond d’ARN obtenues à partir de cœurs de souris et les données disponibles dans les bases de données de séquençage. Cette étude a permis de définir, en utilisant la bioinformatique, un sous-groupe de 117 lncARN spécifiquement exprimés dans le cœur comparé au cœur embryonnaire. Une analyse à différents stades de développement (fœtal versus adulte) et de pathologie (sain versus hypertrophié) a mis en évidence 157 lncARN différentiellement exprimés dans le cœur embryonnaire comparé au cœur adulte dont seulement 17 étaient présents dans le cœur adulte hypertrophié. Cette approche a permis de mettre en évidence le rôle des lncARN cardiaques comme modulateurs de différentes voies transcriptionnelles [29]. |
Nouveaux rôles des lncARN dans les pathologies cardiovasculaires Très récemment, il a été montré chez la souris que le lncARN Myheart (Mhrt) était exprimé de manière spécifique et abondante au niveau du cœur adulte et qu’il protégeait de l’hypertrophie cardiaque en interagissant avec des facteurs de remodelage chromatinien. De manière intéressante, les auteurs ont montré dans un modèle murin d’IC, que le lncARN Mhrt était réprimé par le complexe chromatinien Brg1-Hdac-Parp, conduisant à une répression de la chaîne lourde de la myosine 6 (Myh6) et une expression de Myh7, isoforme moléculaire caractéristique d’une myopathie. Inversement, Mhrt peut empêcher ce dernier de reconnaître ses cibles chromatiniennes en se liant à son domaine hélicase. Ceci témoigne de la subtilité de la régulation en fonction du stress cardiaque [30]. C’est la première observation du contrôle de la structure chromatinienne par lncARN. De manière intéressante, ce mécanisme pourrait être conservé chez l’homme, car les structures primaire et secondaire du lncARN Mhrt humain sont similaires à celles du lncARN murin. La surexpression de Mhrt, induisant une action cardioprotectrice, ouvre des perspectives thérapeutiques utilisant une modification chimique de l’ARN puisqu’une molécule d’ARN peut être modifiée chimiquement pour pouvoir être administrée comme un médicament. En revanche, nous n’avons pas encore d’information sur la présence de ce lncARN dans le plasma et de son potentiel de biomarqueur. |
Afin de mieux identifier le rôle du lncARN LIPCAR comme biomarqueur de l’insuffisance cardiaque, de nombreuses études seront encore nécessaires. Il sera important, d’une part, d’identifier les cibles (protéines, ncARN) régulées par LIPCAR, et, d’autre part, de déterminer son origine subcellulaire, noyau ou mitochondrie, ainsi que les mécanismes permettant à ce lncARN d’affecter le métabolisme et de répondre à un stress myocardique [31]. D’autres études prospectives devront également évaluer si les taux plasmatiques de LIPCAR peuvent prédire spécifiquement le remodelage cardiaque et la mortalité précoce des patients présentant une insuffisance cardiaque. La fiabilité de ces taux circulants pour identifier les patients à haut risque d’événements délétères dans l’insuffisance cardiaque, un critère important pour considérer LIPCAR comme un biomarqueur utile, devra être établie. Le besoin clinique d’avoir de nouveaux marqueurs pronostiques pour apprécier le risque évolutif et la mortalité précoce des patients insuffisants cardiaques est réel. L’avantage d’utiliser LIPCAR comme biomarqueur est que sa détection utilise une technique très simple, la RT-PCR quantitative, qui peut être adaptée à une utilisation clinique. Il ne faut cependant pas oublier que pour déterminer un profil à haut risque de mortalité précoce chez les patients insuffisants cardiaques, plusieurs cibles génomiques, protéomiques, métabolomiques et environnementales seront sûrement nécessaires. Très récemment, en utilisant une approche protéomique sans a priori sur les groupes cas-témoin de la cohorte INCA ainsi que différentes méthodes de régression statistique, nous avons établi un score de risque qui prédit la mortalité précoce chez les patients insuffisants cardiaques [32]. Ces résultats, et d’autres à venir, suggèrent qu’il sera important de déterminer une signature moléculaire pour chaque patient, combinant plusieurs biomarqueurs et nécessitant des modélisations mathématiques basées sur un modèle de réseau dynamique pour chaque pathologie [33]. Ces approches devraient permettre de développer une « médecine personnalisée » qui serait prédictive et aussi préventive, bien que ce concept soit encore flou [34, 38] (→).
(→) Voir le Forum de M. Billaud et X. Guchet, page 797 de ce numéro
|
Les auteurs sont co-inventeurs d’un brevet : Kumarswamy K, Thum T, Pinet F, de Groote P, Bauters C. Mitochondrial non-coding RNAs for predicting disease progression in heart failure and myocardial infarction (EP14160577, le 19 mars 2014).
|
Le travail de recherche dans le laboratoire est financé par l’Union européenne (E.U. FP7 HOMAGE (305507). Nous nous excusons auprès de ceux dont les travaux, pour des raisons de place, n’apparaissent pas dans cette synthèse.
|
Footnotes |
1.
Nichols M, Townsend N, Luengo-Fernandez R, et al. European cardiovascular disease statistics 2012 . Brussels: : European Heart Network; . Sophia Antipolis : European Society of Cardiology., 2012. 2.
Mosterd A, Hoes AW. Clinical epidemiology of heart failure . Heart. 2007; ; 93 : :1137.–1146. 3.
Jhund PS, Macintyre K, Simpson CR, et al. Long-term trends in first hospitalization for heart failure and subsequent survival between 1986 and 2003: a population study of 5.1 million people . Circulation. 2009; ; 119 : :515.–523. 4.
Roger VL. Epidemiology of heart failure . Circ Res. 2013; ; 113 : :646.–659. 5.
De Groote P, Millaire A, Foucher-Hossein C, et al. Right ventricular ejection fraction is an independent predictor of survival in patients with moderate heart failure . J Am Coll Cardiol. 1998; ; 32 : :948.–954. 6.
De Groote P, Fertin M, Goéminne C, et al. Right ventricular systolic function for risk stratification in patients with stable left ventricular systolic dysfunction: comparison of radionuclide angiography to echoDoppler parameters . Eur Heart J. 2012; ; 33 : :2672.–2679. 7.
Lee DS, Vasan RS. Novel markers for heart failure diagnosis and prognosis . Curr Opin Cardiol. 2005; ; 20 : :201.–210. 8.
De Groote P, Dagorn J, Soudan B, et al. B-type natriuretic peptide and peak exercise oxygen consumption provide independent information for risk stratification in patients with stable congestive heart failure . J Am Coll Cardiol. 2004; ; 43 : :1584.–1589. 9.
Braunwald E. Biomarkers in heart failure . N Engl J Med. 2008; ; 358 : :2148.–2250. 10.
Kumarswamy R, Thum T. Non-coding RNAs in cardiac remodeling and heart failure . Circ Res. 2013; ; 113 : :676.–689. 11.
Braunwald E. Research advances in heart failure . Circ Res. 2013; ; 113 : :633.–645. 12.
Tong YK, Lo YM. Diagnostic developments involving cell-free (circulating) nucleic acids . Clin Chem Acta. 2006; ; 363 : :187.–196. 13.
Fertin M, Hennache B, Hamon M, et al. Usefulness of serial assessment of B-type natriuretic peptide, troponin I, and C-reactive protein to predict left ventricular remodeling after acute myocardial infarction (from the REVE-2 study) . Am J Cardiol. 2010; ; 106 : :1410.–1416. 14.
Bauters C, Kumarswamy R, Holzmann A, et al. Circulating miR-133a and miR-423-5p fail as biomarkers for left ventricular remodeling after myocardial infarction . Int J Cardiol. 2013; ; 168 : :1837.–1840. 15.
Jacobs L, Thijs L, Jin Y, et al. Heart omics in AGEing (HOMAGE): design, research objectives and characteristics of the common database . J Biomed Res. 2014; ; 28 : :349.–359. 16.
Baulande S, Criqui A. Duthieuw. Les microARN circulants, une nouvelle classe de biomarqueurs pour la médecine . Med Sci (Paris). 2014; ; 30 : :289.–296. 17.
Mathieu EL, Belhocine M, Dao LTM, et al. Rôle des longs ARN non codants dans le développement normal et pathologique . Med Sci (Paris). 2014; ; 30 : :790.–796. 18.
Kung JTY, Colognori D, Lee JT. Long non coding RNAs: past, present, and future . Genetics. 2013; ; 193 : :651.–669. 19.
Derrien T, Johnson R, Bussotti G, et al. The Gencode v7 catalog of human long non coding RNAs: analysis of their gene structure, evolution, and expression . Genome Res. 2012; ; 22 : :1775.–1789. 20.
Thum T. Noncoding RNAs and myocardial fibrosis . Nat Rev Cardiol. 2014; ; 11 : :655.–663. 21.
Dubois E, Fertin M, Burdese J, et al. Cardiovascular proteomics: translational study to develop novel biomarkers in heart failure and left ventricular remodeling . Proteomics Clin Appl. 2011; ; 5 : :57.–66. 22.
Savoye C, Equine O, Tricot O, et al. Left ventricular remodeling after anterior wall acute myocardial infarction in modern clinical practice (from the REmodelage VEntriculaire [REVE] study group) . Am J Cardiol. 2006; ; 98 : :1144.–1149. 23.
Fertin M, Dubois E, Belliard A, et al. Usefulness of circulating biomarkers for the prediction of left ventricular remodeling after myocardial infarction . Am J Cardiol. 2012; ; 110 : :277.–283. 24.
Kumarswamy R, Bauters C, Volkmann I, et al. The circulating long non-coding LIPCAR predicts survival in heart failure patients . Circ Res. 2014; ; 114 : :1569.–1575. 25.
Yang KC, Yamada KA, Patel AY, et al. Deep RNA sequencing reveals dynamic regulation of myocardial noncoding RNAs in failing human heart and remodeling with mechanical circulatory support . Circulation. 2014; ; 129 : :1009.–1021. 26.
Ounzain S, Micheletti R, Beckmann T, et al. Genome-wide profiling of the cardiac transcriptome after myocardial infarction identifies novel heart-specific long non-coding RNAs . Eur Heart J. 2015; ; 36 : :353.–368. 27.
Wang K, Liu F, Zhou LY, et al. The long noncoding RNA CHRF regulates cardiac hypertrophy by targeting miR-489 . Circ Res. 2014; ; 114 : :1377.–1388. 28.
Vausort M, Wagner DR, Devaux Y. Long non-coding RNAs in patients with acute myocardial infarction . Circ Res. 2014; ; 115 : :668.–677. 29.
Matkovich SJ, Edwards JR, Grossenheider TC, et al. Epigenetic coordination of embryonic heart transcription by dynamically regulated long noncoding RNAs . Proc Natl Acad Sci USA. 2014; ; 111 : :12264.–12269. 30.
Han P, Li W, Lin CH, et al. A long noncoding RNA protects the heart from pathological hypertrophy . Nature. 2014; ; 514 : :102.–106. 31.
Dorn GW. LIPCAR: a mitochondrial lnc in the noncoding RNA chain? Circ Res. 2014; ; 114 : :1548.–1550. 32.
Lemesle G, Maury F, Beseme O, et al. Multi-marker proteomic profiling for the prediction of cardiovascular mortality in patients with chronic heart failure . PLoS One. 2015; ; 10 : :e0119265.. 33.
Gustafsson M, Nestor CE, Zhang H, et al. Modules, networks and systems medicine for understanding disease and aiding diagnosis . Genome Med. 2014; ; 6 : :82.. 34.
Bateman S. Médecine personnalisée : un concept flou, des pratiques diversifiées . Med Sci (Paris). 2014; ; 30 : :8.–13. 35.
Le Pedrazzini T. cœur des ARN non codants : un long chemin à découvrir . Med Sci (Paris). 2015; ; 31 : :261.–267. 36.
Ladeiro Y, Zucman-Rossi J. Micro-ARN (miARN) et cancer : le cas des tumeurs hépatocellulaires . Med Sci (Paris). 2009; ; 25 : :467.–472. 37.
Gougelet A, Colnot S. Les microARN dans le cancer du foie à l’orée de nouvelles thérapies ciblées ? Med Sci (Paris). 2013; ; 29 : :861.–867. 38.
Billaud M, Guchet X. L’invention de la médecine personnalisée : entre mutations technologiques et utopie . Med Sci (Paris). 2015; ; 31 : :797.–803. |