| |
| Med Sci (Paris). 2015 November; 31(11): 947–950. Published online 2015 November 17. doi: 10.1051/medsci/20153111003.Le résistome pulmonaire Fabienne Brégeon1,2* and Jean-Marc Rolain1,3 1Unité de recherche sur les maladies infectieuses et tropicales émergentes, IHU méditerranée infection, UMR CNRS-IRD 6236, IFR48, Aix-Marseille Université, Marseille, France 2Service d’explorations fonctionnelles respiratoires, Pôle CVT, CHU Nord, APHM, Marseille, France 3Fédération de microbiologie clinique, hôpital de La Timone, Marseille, France MeSH keywords: Résistance bactérienne aux médicaments, Génome bactérien, Humains, Poumon, Microbiote, Maladies de l'appareil respiratoire, génétique, métabolisme, microbiologie, traitement médicamenteux |
Les malades atteints de maladies respiratoires chroniques associées à une colonisation bronchique, tels que les asthmatiques, les patients porteurs de bronchopneumopathies chroniques obstructives et ceux atteints de mucoviscidose, portent une flore pulmonaire qui est non seulement polymocrobienne, mais aussi variable en quantité et en qualité au cours du temps. Il est alors particulièrement difficile de connaître le ou les seuls agents responsables des épisodes d’aggravation infectieuse. Trop large, un traitement antibiotique risque d’étendre la pression de sélection à un grand nombre d’espèces, trop étroit il risque de manquer le microbe responsable de l’aggravation. Le suivi du résistome chez un même patient pourrait permettre un ajustement thérapeutique individualisé, sur la base d’un risque identifié d’émergence des résistances antibiotiques de l’ensemble de son microbiome. Ceci permettrait de proposer des thérapies individuelles ciblées, comme on le voit aujourd’hui dans certaines stratégies de lutte contre le cancer. |
La lutte pharmacologique contre les maladies infectieuses implique non seulement le développement de nouvelles molécules, mais également de connaître les moyens de défense que déploient les bactéries contre ces molécules. Une telle approche est permise, dans l’idéal, par une étude exhaustive, pour l’ensemble des microorganismes d’un écosystème, des mécanismes de résistance aux antibiotiques et des systèmes de transmission des gènes responsables. Le « résistome » est un terme qui désigne l’ensemble des gènes de résistance portés par l’ensemble des bactéries pathogènes, ou commensales, d’un milieu (microbiome) ou d’un système (organisme entier animal ou végétal, partie d’un organisme tel que le microbiote intestinal, milieu environnemental, etc.). Ce terme a été proposé la première fois par Gerard D. Wright [1, 2] et ne fait, depuis, l’objet que d’environ 150 publications indexées dans PubMed. On l’emploie aussi bien pour décrire l’ensemble des gènes de résistance dans un milieu, tous germes confondus, que ceux d’une espèce bactérienne dans un contexte donné, ou encore pour les gènes supportant les mécanismes de résistance vis-à-vis d’une molécule antibiotique donnée. La description du résistome fait appel à des données de biologie moléculaire et de séquençage, plus ou moins associées à des tests de sensibilité in vitro, son interprétation en reste cependant malaisée compte tenu de la multitude d’informations à traiter et du rôle encore mal connu de nombreux gènes. |
Le résistome dans les microbiomes humains Contrairement au milieu environnemental, la description du résistome chez l’homme n’a pour l’instant été que peu publiée. Elle porte à ce jour essentiellement sur le milieu digestif, aboutissant aux conclusions que l’exposition massive à des antibiotiques, que ce soit via leur utilisation dans l’industrie agro-alimentaire [3] ou pour la décontamination digestive sélective des patients de réanimation [4], transforme la flore digestive en un véritable réservoir de gènes de résistance aux antibiotiques. Seules deux revues de la littérature abordent ce sujet dans le poumon, dont une publiée dans The Lancet en août 2014 [5]. Compte tenu de l’utilisation répétée d’antibiotiques chez les patients porteurs chroniques de bactéries pathogènes, il apparaît plausible que le poumon soit un réservoir de gènes de résistance transmissibles. |
Le microbiome pulmonaire et la résistance aux antibiotiques Par l’inhalation de l’air inspiré et des aérosols qu’il contient, l’appareil broncho-pulmonaire est en contact permanent avec l’environnement. Il se trouve par ailleurs en contigüité anatomique avec les structures oro-digestives. La flore microbienne extérieure peut donc, logiquement, atteindre plus au moins profondément les voies aériennes inférieures. Pourtant, c’est seulement récemment que le milieu pulmonaire, jusqu’alors dit normalement stérile, attire l’attention pour sa flore microbienne commensale [20, 21] (→). On y trouve, par exemple, des espèces communes avec la cavité buccale, mais aussi des espèces non communes comme les Enterobacteriaceae, Haemophilus, Methylobacterium et l’espèce Ralstonia [6]. L’idée qu’il existe des interactions entre les microbiomes digestif et pulmonaire fait de plus en plus de chemin, de même que l’hypothèse de leur rôle conjoint dans l’inflammation et l’immunité locale [7]. (→) Voir la Nouvelle de L. Nguyen et L. Delhaes, et la Synthèse de C. Andréjak et L. Delhaes, pages 945 et 971 de ce numéro
Avec l’avènement d’outils technologiques innovants de microbiologie à haut-débit et de séquençage, il a été possible de révéler la présence de germes chez le sujet sain, dont l’identification était mise en défaut par les techniques de culture conventionnelles. Ces outils ont prouvé la présence d’une flore microbienne pulmonaire, dont l’origine apparaît mixte, environnementale et digestive, et dont la variété s’étend au monde non bactérien, viral (virome) et fongique (mycobiome). Les travaux récents convergent vers l’hypothèse que le microbiome pulmonaire, et en particulier le mycobiome, joue un rôle dans la physiopathologie des maladies respiratoires chroniques telles que l’asthme, la bronchite chronique obstructive, la mucoviscidose et autres maladies chroniques des bronches [7, 8]. Les patients chez qui la clairance des voies aériennes est altérée, sont exposés à des colonisations bronchiques chroniques. L’équilibre de l’écosystème microbien local et les défenses immunitaires se trouvent menacés, favorisant les attaques par des bactéries, qui évoluent vers des phénotypes de résistances aux antibiotiques au fur et à mesure des traitements. |
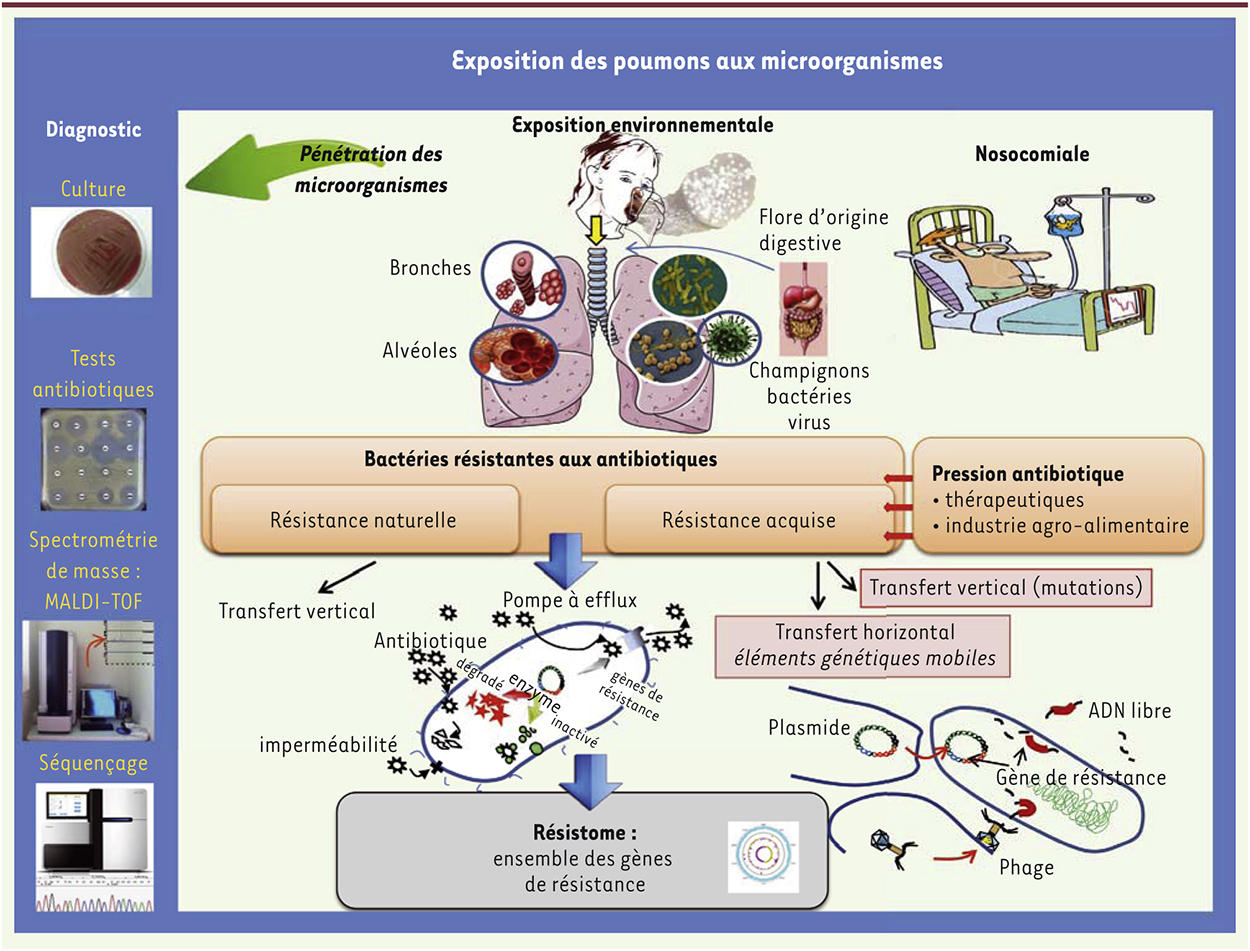
La résistance des bactéries aux antibiotiques peut résulter de différents mécanismes qui n’épargnent pas le microbiome pulmonaire. Certains sont naturels et dépendent des caractéristiques structurelles et fonctionnelles de l’agent infectieux, ainsi que du mécanisme d’action de l’antibiotique. D’autres, apparaissant au cours du développement de souches bactériennes qui modifient leur structure au point ciblé, se mettent à produire une enzyme modifiant ou détruisant l’antibiotique, ou encore, se rendent imperméables à la pénétration de la molécule. Ces résistances acquises résultent d’une information portée au sein du génome de la bactérie, découlant d’une mutation génétique au niveau de son chromosome ou de l’acquisition d’éléments génétiques mobiles (transposons, cassettes de gènes, plasmides, etc.). Ces éléments génétiques, communément regroupés sous le terme « mobilome », sont porteurs de gènes de résistance provenant d’une autre bactérie. Ainsi, la transmission de gènes de résistance peut se faire de façon soit horizontale, par un élément génétique allant d’une bactérie à l’autre, soit verticale, transmis d’une bactérie à sa descendance lors de sa multiplication clonale. La résistance acquise pose des problèmes majeurs de santé publique. Elle apparaît de façon inopinée et met médecins et patients devant des impasses thérapeutiques. Des souches multi-résistantes, voire pan-résistantes1, émergent régulièrement alors qu’aucune nouvelle famille d’antibiotiques n’a été découverte depuis les années 1970. Les maladies chroniques des bronches sont particulièrement concernées [9]. Le rôle de l’appareil respiratoire dans l’évolution du résistome antibiotique mérite donc d’être pris en compte (Figure 1). Dans ce cadre, le poumon du patient atteint de mucoviscidose apparaît particulièrement pertinent eu égard au nombre et à la variété des agents infectieux isolés. Ce modèle est également pertinent en raison du fait que ces patients reçoivent, depuis leur plus jeune âge, des associations complexes d’agents antimicrobiens, tant à visée préventive que curative, aussi bien sous formes de cures au cours des exacerbations, que de traitements au long cours, et incluant parfois des administrations locales par voie aérosolisée telle que préconisée par les recommandations d’experts [10].
 | Figure 1.
Hypothèses actuelles de l’existence et du développement du résistome pulmonaire.
|
Avec l’augmentation de la survie de ces patients, un profil cinétique commun d’évolution de la flore microbienne pulmonaire dans la mucoviscidose a pu être établi. Les bactéries fréquemment retrouvées sont Pseudomonas aeruginosa, Staphylococcus aureus, le complexe Burkholderia cepacia, Haemophilus influenzae et des mycobactéries atypiques. De plus, grâce aux méthodes de biologie moléculaire à haut débit, on sait qu’une flore anaérobie y occupe également une part non négligeable [11]. Avec l’âge, la diversité microbienne diminue et l’installation de P. aeruginosa apparaît corrélée à la dégradation fonctionnelle respiratoire, ainsi qu’à la prépondérance de bactéries multi- voire pan-résistantes. La voie aérosolisée délivrant de la tobramycine, de la colistine ou de l’aztreonam, des antibiotiques qui ciblent particulièrement P. aeruginosa, permet d’augmenter la concentration locale d’antibiotiques au risque d’augmenter la pression de sélection d’isolats résistants. La sévérité de l’atteinte pulmonaire est plus marquée chez les sujets porteurs de germes multirésistants que chez les patients porteurs de souches sensibles [12]. L’évolution du microbiome respiratoire au cours des traitements antibiotiques du patient atteint de mucoviscidose commence à être documentée [13-15]. L’étude du résistome serait un complément utile à ce type d’approche microbiologique. Elle permettrait en effet de suivre l’évolution des gènes de résistance chez un même patient et ainsi de dépister le tournant évolutif de la flore, au moment de son évolution génique vers un profil à risque de résistance avec, en perspective, la possibilité d’un diagnostic et d’un traitement personnalisé, adapté au cours du temps. Dans une étude pilote très récente conduite auprès de patients porteurs de mucoviscidose, Lim et al. ont séquencé le métagénome des expectorations et rapportent l’évolution du résistome chez 3 d’entre eux [14]. Le premier prélèvement correspondait à un épisode d’exacerbation. Des antibiotiques étaient ensuite administrés par voie intraveineuse en complément des traitements de fond, selon les standards de prise en charge thérapeutique, et associés à des procédés de drainage bronchique physiques et pharmacologiques. Par la suite, jusqu’à quatre autres échantillons d’expectorations étaient prélevés pour des analyses métagénomiques à des périodes englobant différentes phases du régime antibiotique et de l’évolution clinique. Dans ce travail, 50 % des gènes de résistance aux antibiotiques identifiés étaient candidats pour un mécanisme de résistance portant sur les pompes à efflux. Les gènes codant pour des β-lactamases étaient cependant les plus nombreux. De façon intéressante, il a été observé que le nombre et les cibles des gènes de résistance identifiés variaient rapidement dans le temps et étaient influencés par le régime antibiotique. De plus, les auteurs retrouvent une grande quantité de gènes codant pour les β-lactamases au sein du virome, laissant supposer une responsabilité des bactériophages hébergés par le microbiome dans la dissémination des gènes de résistance. Le rôle des bactériophages associés au microbiome pulmonaire des patients atteints de mucoviscidose, et leur possible responsabilité dans l’évolution du résistome pulmonaire, est discuté dans une revue générale [15]. Le poumon de ces patients semble être un milieu complexe et adaptatif au sein duquel virus, bactéries et champignons évoluent en réseaux interconnectés, favorisant les recombinaisons génomiques. La promiscuité et la variété des microorganismes semblent propices aux transferts latéraux de gènes, comme cela est décrit pour le microbiome intestinal [16] ou environnemental marin [17]. Depuis que l’intégralité du génome de nombreuses bactéries a pu être séquencée, la fréquence à laquelle les bactéries hébergent un ou plusieurs prophages (c’est-à-dire des bactériophages dans leur forme tempérée ou lysogénique)2 a pu être évaluée [18]. Étant donné l’apport de matériel génétique qu’ils représentent, ces prophages seraient des acteurs importants de modifications génotypique et phénotypique des bactéries et pourraient favoriser l’apparition de phénotypes de résistance aux antibiotiques [19]. Des travaux restent cependant à conduire pour préciser l’effet des antibiotiques sur l’induction des prophages du microbiome pulmonaire et leur contribution à la modification du résistome au cours des épisodes d’exacerbation et des traitements antibiotiques. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
D’Costa VM, McGrann KM, Hughes DW, Wright GD. Sampling the antibiotic resistome . Science. 2006; ; 311 : :374.–377. 2.
Wright GD. The antibiotic resistome: the nexus of chemical and genetic diversity . Nat Rev Microbiol. 2007; ; 5 : :175.–186. 3.
Hu Y, Yang X, Lu N, et al. The abundance of antibiotic resistance genes in human guts has correlation to the consumption of antibiotics in animal . Gut Microbes. 2014; ; 5 : :245.–249. 4.
Buelow EJ, Gonzalez TB, Versluis D, et al. Effects of selective digestive decontamination (SDD) on the gut resistome . Antimicrob Chemother. 2014; ; 69 : :2215.–2223. 5.
Sherrard LJ, Tunney MM, Elborn JS. Antimicrobial resistance in the respiratory microbiota of people with cystic fibrosis . Lancet. 2014; ; 384 : :703.–713. 6.
Morris A, Beck JM, Schloss PD, et al. Comparison of the respiratory microbiome in healthy nonsmokers and smokers . Am J Respir Crit Care Med. 2013; ; 187 : :1067.–1075. 7.
Nguyen LD, Viscogliosi E, Delhaes L. The lung mycobiome: an emerging field of the human respiratory microbiome . Front Microbiol. 2015; ; 6 : :89.. 8.
Goleva E, Jackson LP, Harris JK, et al. The effects of airway microbiome on corticosteroid responsiveness in asthma . Am J Respir Crit Care Med. 2013; ; 188 : :1193.–1201. 9.
Bassetti M, Ginocchio F, Mikulska M. New treatment options against gram-negative organisms . Crit Care. 2011; ; 15 : :215.. 10.
Flume PA, O’Sullivan BP, Robinson KA, et al. Cystic fibrosis pulmonary guidelines: chronic medications for maintenance of lung health . Am J Respir Crit Care Med. 2007; ; 176 : :957.–969. 11.
Bittar F, Richet H, Dubus JC, et al. Molecular detection of multiple emerging pathogens in sputa from cystic fibrosis patients . PLoS One. 2008; ; 3 : :e2908.. 12.
Lechtzin N, John M, Irizarry R, et al. Outcomes of adults with cystic fibrosis infected with antibiotic resistant Pseudomonas aeruginosa . Respiration. 2006; ; 73 : :27.–33. 13.
Zhao J, Schloss PD, Kalikin LM, et al. Decade-long bacterial community dynamics in cystic fibrosis airways . Proc Natl Acad Sci USA. 2012; ; 109 : :5809.–5814. 14.
Lim YW, Evangelista JS3rd, Schmieder R, et al. Clinical insights from metagenomic analysis of sputum samples from patients with cystic fibrosis . J Clin Microbiol. 2014; ; 52 : :425.–437. 15.
Rolain JM, Fancello L, Desnues C, et al. Bacteriophages as vehicles of the resistome in cystic fibrosis . Antimicrob Chemother. 2011; ; 66 : :2444.–2447. 16.
Kurokawa K, Itoh T, Kuwahara T, et al. Comparative metagenomics revealed commonly enriched gene sets in human gut microbiomes . DNA Res. 2007; ; 14 : :169.–181. 17.
Sobecky PA, Hazen TH. Horizontal gene transfer and mobile genetic elements in marine systems . Methods Mol Biol. 2009; ; 532 : :435.–453. 18.
Willner D, Furlan M, Haynes M, et al. Metagenomic analysis of respiratory tract DNA viral communities in cystic fibrosis and non-cystic fibrosis individuals . PLoS One. 2009; ; 4 : :e7370.. 19.
Rolain JM, Francois P, Hernandez D, et al. Genomic analysis of an emerging multiresistant Staphylococcus aureus strain rapidly spreading in cystic fibrosis patients revealed the presence of an antibiotic inducible bacteriophage . Biol Direct. 2009; ; 4 : :1.. 20.
Nguyen L, Delhaes L. Un nouveau concept : le mycobiome pulmonaire . Med Sci (Paris). 2015; ; 31 : :945.–947. 21.
Andréjak C, Delhaes L. Le microbiome pulmonaire en 2015 . Med Sci (Paris). 2015; ; 31 : :971.–978. |