| |
| Med Sci (Paris). 2015 November; 31(11): 971–978. Published online 2015 November 17. doi: 10.1051/medsci/20153111011.Le microbiome pulmonaire en 2015 Une fenêtre ouverte sur les pathologies pulmonaires chroniques Claire Andréjak1* and Laurence Delhaes2,3,4** 1Service de pneumologie et réanimation respiratoire, CHU d’Amiens, EA 4294, université de Picardie Jules Verne, 80000Amiens, France 2Centre d’immunologie et d’infectiologie de Lille (BDEEP), Inserm U1019, CNRS UMR 8204, Institut Pasteur de Lille, université de Lille Nord de France, 1, rue du Professeur Calmette, 59019Lille Cedex, France 3Laboratoire de parasitologie-mycologie, CHRU de Lille, faculté de médecine, université Lille2, Lille, France 4Adresse actuelle : Service de parasitologie- Mycologie, CHU de Bordeaux, université de Bordeaux, France |

La littérature sur le microbiome humain (Figure 1) est en constante augmentation grâce notamment au développement récent des techniques moléculaires de séquençage à haut-débit. Jusqu’à récemment, les données publiées concernaient essentiellement les microbiomes digestif, cutané, buccal ou génital et leurs liens avec différentes pathologies comme par exemple le diabète. Mais, très récemment, de nombreux microorganismes (bactéries, virus, champignons) ont également été mis en évidence dans les voies aériennes inférieures des sujets en bonne santé, rendant caduque le concept classique de stérilité des voies aériennes normales [1–5]. Plusieurs questions se posent aujourd’hui : ces germes sont-ils des « touristes » ou des résidents permanents du tractus respiratoire ? Quel est leur rôle chez le sujet en bonne santé ? Comment interfèrent-ils avec les pathologies pulmonaires et leur prise en charge thérapeutique ? Cela pose la question éternelle « colonisation versus infection ». In fine, ces nouvelles techniques moléculaires de séquençage à haut débit pourraient expliquer des situations respiratoires aujourd’hui sans réponse et ouvrent donc de nouvelles perspectives pour aborder différemment les phénomènes physiopathologiques impliqués dans les pathologies respiratoires chroniques (et donc participer à leur compréhension).
 | Figure 1.
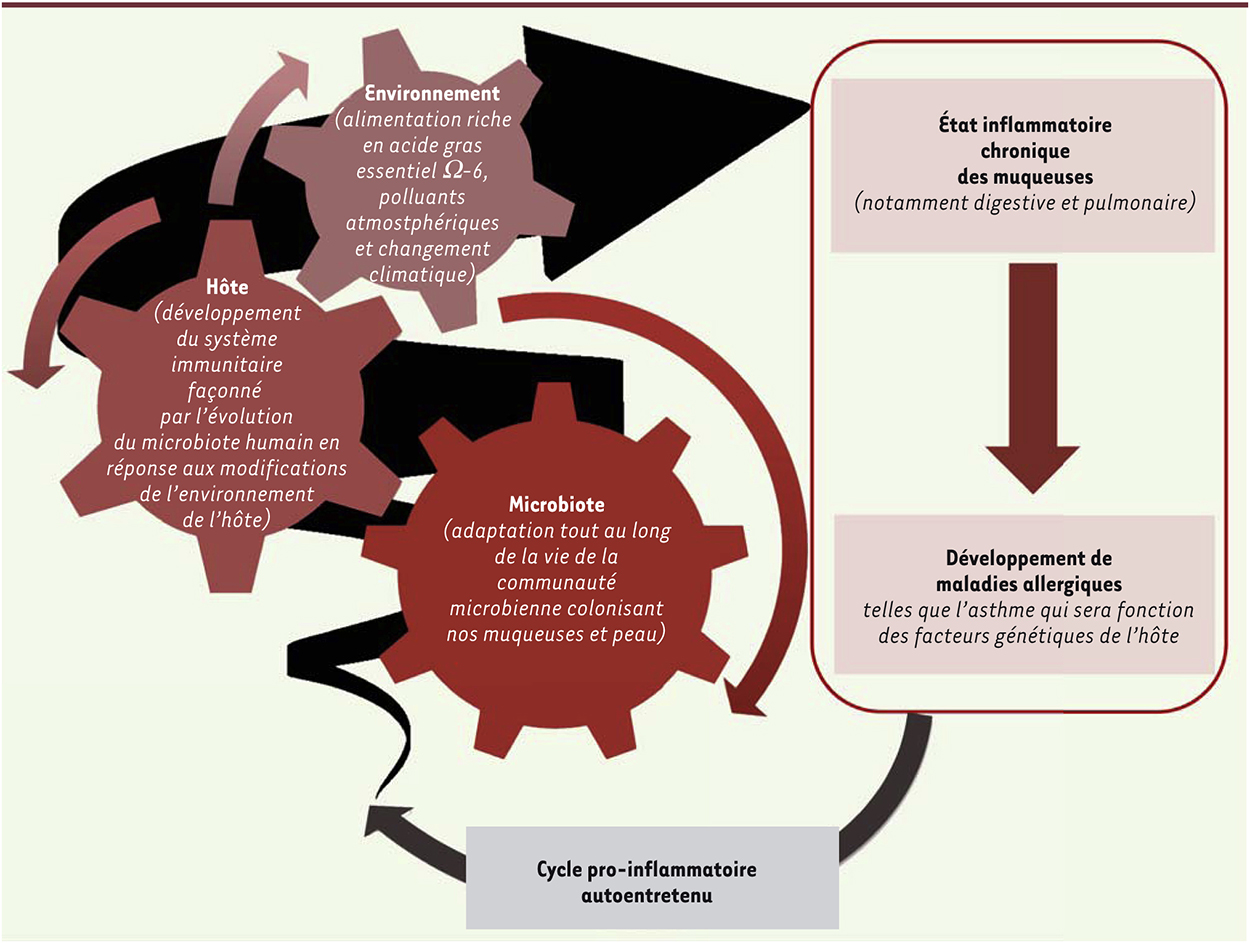
Hypothèses actuelles de l’implication du microbiote humain dans les maladies respiratoires allergiques.
|
Si le microbiome semble être dépendant de l’environnement de l’hôte, de son système immunitaire, de son génotype, de son style de vie et de la présence transitoire de certains germes [6], une meilleure connaissance du microbiome pulmonaire du sujet sain et du sujet ayant une pathologie respiratoire chronique permettrait de mettre en évidence une (des) différence(s) ou dysbiose(s) et ainsi d’élaborer de nouvelles hypothèses thérapeutiques [2, 3]. Cependant, en 2015, les connaissances sur le microbiome pulmonaire du sujet sain ou en présence de pathologie respiratoire restent très parcellaires et aucune conclusion pratique n’est actuellement possible. Les données actuelles tendent à montrer un lien entre microbiome pulmonaire et statut clinique du patient, évolution de la pathologie respiratoire ou encore impact des thérapeutiques [2, 3, 7]. Ainsi, nos connaissances dans ce nouveau domaine de recherche ont permis d’établir l’existence d’un microbiome pulmonaire essentiellement composé de bactéries (ou bactériome), mais complété d’une communauté fongique (ou mycobiome) et de virus et de phages (virome), pour lesquels les connaissances sont très limitées (voir Glossaire) [2, 3, 7]. Même si les microbiotes et microbiomes pulmonaires doivent être plus largement étudiés et mieux définis de façon à comprendre leurs rôles éventuels dans des pathologies et leurs exacerbations, notamment les différentes pathologies respiratoires chroniques telles que la mucoviscidose, la broncho-pneumopathie chronique obstructive (BPCO) ou l’asthme, nous présentons ici une synthèse des données bibliographiques concernant le microbiote/microbiome pulmonaire et ses perspectives en matière de santé humaine. En tenant compte des nouveaux concepts de mycobiome/mycobiote et de résistome [47, 48] (→), nous proposons d’aborder la flore polymicrobienne pulmonaire en tant qu’entité unique dans laquelle les interactions entre microorganismes (comme les phénomènes de transferts horizontaux de gène de résistance) et le rôle des micromycètes sont discutés. (→) Voir les Nouvelles de L. Nguyen et L. Delhaes, et de F. Brégeon et J.M. Rolain, pages 945 et 947 de ce numéro
|
Le microbiome pulmonaire humain : de l’abandon d’un dogme vers la naissance d’un nouveau concept Historiquement, les poumons humains étaient considérés comme un organe stérile et aseptique, selon des études essentiellement fondées sur les méthodes microbiologiques conventionnelles (examen direct et cultures microbiologiques de prélèvements respiratoires). Ce n’est que récemment, grâce au développement des techniques moléculaires de séquençage à haut-débit, qu’il a été démontré la présence d’une flore polymicrobienne appelée microbiote, probablement transitoire dans l’appareil respiratoire de l’homme. Dans une étude publiée en 2010 utilisant le séquençage à haut débit de l’ADN ribosomique 16S, Erb-Downward et ses collègues ont comparé les échantillons provenant de différentes régions de poumons de sujets sains non-fumeurs, de fumeurs sains (sans signes cliniques) et de fumeurs atteints de broncho-pneumopathie chronique obstructive (BPCO) [1]. Les auteurs ont montré que le microbiome pulmonaire était constitué d’un grand nombre de bactéries et que la diversité de la communauté bactérienne diminuait chez les patients atteints de BPCO par rapport aux sujets sains. Cette étude et d’autres ont conduit à proposer une nouvelle conception de notre appareil respiratoire, colonisé par des microorganismes, qui possèderait donc un microbiome au même titre que les microbiomes humains intestinal, cutané ou vaginal. Bien que le nombre de microorganismes retrouvés dans le poumon ne soit pas aussi élevé que pour le microbiome intestinal ou buccal, le microbiome pulmonaire est maintenant considéré comme un écosystème spécifique où l’abondance et la grande diversité des différents microorganismes le composant est unique pour chaque individu [2, 3]. Le microbiome pulmonaire est actuellement un domaine de recherche en pleine émergence avec de nombreuses études centrées sur la comparaison entre les communautés bactériennes pulmonaires de sujets sains et celles de patients atteints de maladies pulmonaires chroniques comme l’asthme, la BPCO, la mucoviscidose, la fibrose pulmonaire idiopathique, le cancer pulmonaire, ou ayant subi une greffe de poumon [1, 4–27]. En dépit de résultats variables selon les publications, Proteobacteria, Firmicutes et Bacteroidetes ont été identifiés comme les phylums les plus fréquents. Au niveau du genre bactérien, Pseudomonas, Streptococcus, Prevotella, Fusobacteria, Porphyromonas et Veillonella sont prédominants ; d’autres pathogènes potentiels tels que Haemophilus et Neisseria y sont moins représentés [1, 4–9, 22, 23, 25–27]. Même si le nombre des articles dans la littérature étudiant la composition et la structure du microbiote pulmonaire dans des contextes différents de maladies respiratoires est croissant, les connaissances actuelles, en particulier sur sa composition fongique et virale, et sur sa fonction, restent encore très limitées. Selon les études publiées (Tableau I), les modifications du microbiote pulmonaire seraient corrélées à l’évolution clinique des patients (par exemple : dans la mucoviscidose, une perte de diversité bactérienne et fongique peut être associée à une diminution de la fonction respiratoire) [7, 9–11, 14]. De plus, comme le microbiome intestinal, le microbiome pulmonaire jouerait un rôle dans la régulation de la réponse immunitaire [3]. En effet, une perte de la diversité du microbiome pulmonaire provoquerait un déséquilibre de l’homéostasie immunologique qui pourrait participer à la genèse des maladies respiratoires inflammatoires chroniques [2, 3]. Ainsi, les connaissances sur le microbiome pulmonaire et le rôle qu’il joue dans la pathogenèse des maladies pulmonaires chroniques devraient nous permettre, à moyen terme, d’améliorer la prise en charge des malades et, à plus long terme, de découvrir de nouvelles possibilités thérapeutiques.
Tableau I.
| Pathologies |
Patients |
Principaux résultats |
Année [Référence] |
| BPCO |
Patients en exacerbation, comparaison en fonction de la prise en charge |
Patients avec antibiothérapie : diminution des protéobactéries ; patients avec corticoïdes : augmentation des protéobactéries et d’autres phyllies |
2014 [24] |
|
| BPCO |
BPCO et fumeurs non BPCO |
Grande variété dans le microbiome avec principalement des bactéries des genres : Prevotella, Sphingomonas, Pseudomonas, Acinetobacter, Fusobacterium, Megasphaera, Veillonella, Staphylococcus et Streptococcus
|
2013 [22] |
|
| BPCO |
BPCO et individus sains : microbiote post-infection à rhinovirus |
Augmentation du nombre de protéobactéries à J15 chez le sujet avec BPCO avec expansion d’un clone présent avant l’infection virale |
2013 [34] |
|
| BPCO |
BPCO |
30 % de patients avec des bactéries pathogènes |
2008 [25] |
|
| BPCO et mucoviscidose |
8 non-fumeurs ; 8 fumeurs
8 BPCO GOLD4 ; 8 mucoviscidoses |
Microbiome du patient mucoviscidosique plus abondant et moins divers ; moins d’acinétobactéries dans la mucoviscidose, plus de Firmicutes dans la BPCO |
2012 [33] |
|
| Mucoviscidose |
Patients à différents stades de mucoviscidose |
P. aeruginosa, H. influenzae, S. pneumoniae S. aureus sont des marqueurs du probable déclin du VEMS |
2014 [26] |
|
| Mucoviscidose |
23 patients |
Stabilité du microbiote pendant les périodes d’exacerbation ; lien entre faible diversité et diminution de la fonction ventilatoire |
2012 [19] |
|
| Mucoviscidose |
Patients avec mucoviscidose stable |
Lien entre faible diversité et diminution de la fonction ventilatoire, le S-K score et l’indice de masse corporelle |
2012 [11] |
|
| Mucoviscidose |
Enfants avec mucoviscidose suivis entre la naissance et 21 mois |
Lien entre facteurs nutritionnels, microbiote digestif et évolution du microbiote pulmonaire du patient avec une mucoviscidose |
2012 [13] |
|
| Mucoviscidose |
Mucoviscidose : prélèvements à 6 ans versus 8-9 ans |
Évolution du microbiote avec l’âge : diminution de la diversité bactérienne avec prédominance de la famille des Pseudomonadaceae
|
2012 [15] |
|
| Mucoviscidose |
Mucoviscidose de l’enfant à l’adulte de 72 ans |
Rôle de l’âge sur l’augmentation en richesse et la diminution de diversité du microbiome ; lien mutation CFTR et microbiome pulmonaire |
2010 [17] |
|
| Asthme |
Patients avec asthme cortico-résistant, ou cortico-sensible et patients non asthmatiques |
Plus d’acinétobactéries chez les asthmatiques cortico-résistants ; expansion de certaines protéobactéries chez certains asthmes cortico-résistants et cortico-sensibles |
2013 [23] |
|
| Asthme |
Enfants asthmatiques versus enfants sains |
Plus de protéobactéries chez les individus asthmatiques. Plus de Firmicutes et d’acinétobactéries chez les non asthmatiques |
2013 [27] |
|
| Asthme |
Enfants asthmatiques versus enfants sains |
Grande proportion de protéobactéries et staphylocoques alors que Bacteroides prédomine chez les enfants sains |
2010 [8] |
Principales études d’intérêt du microbiote pulmonaire des pathologies respiratoires chroniques. BPCO : broncho-pneumopathie chronique obstructive ; CFTR : cystic fibrosis transmembrane conductance regulator ; VEMS : volume expiratoire maximum seconde ; GOLD : global initiative for chronic obstructive lung disease, sont des recommandations pour le diagnostic et la prise en charge des BPCO ; S-K score : le score de Shwachman-Kulczycki détermine la sévérité de la mucoviscidose. |
|
Microbiome pulmonaire et mucoviscidose Dans la mucoviscidose, les voies respiratoires des patients représentent un environnement favorable pour la colonisation polymicrobienne. Cette colonisation et l’infection broncho-pulmonaire qui en résulte restent le problème majeur pour la surveillance et le traitement de cette maladie génétique touchant tous les continents [21]. L’infection est généralement chronique. Les exacerbations pulmonaires aiguës intercurrentes détériorent progressivement la fonction respiratoire conditionnant en grande partie le pronostic vital des patients [21]. Aujourd’hui encore, le suivi de ces colonisations/infections est réalisé essentiellement par les méthodes conventionnelles (examen direct et cultures microbiologiques). Cette approche permet le diagnostic des infections bactériennes, parfois fongiques, les plus courantes ; elle met en évidence un nombre restreint d’espèces bactériennes qui sont considérées comme ayant un rôle important dans l’évolution et/ou la physiopathologie de la mucoviscidose comme Pseudomonas aeruginosa. Cependant, cette approche ne permet pas de détecter les microorganismes non cultivables ; elle reflète peu les co-infections et la dynamique des populations microbiennes dans l’environnement pulmonaire. In fine, dans ce contexte de flore polymicrobienne, le concept développé dès les années 2000 de microbiome et microbiote pulmonaires prend toute sa signification [2, 3, 28]. Les techniques de séquençage à haut-débit ciblant essentiellement l’ADN ribosomique 16S des bactéries ont permis d’avoir une vue plus globale de la communauté bactérienne pulmonaire et une meilleure compréhension de la relation entre cette communauté, la pathogenèse de la maladie et l’évolution clinique et thérapeutique des patients atteints de mucoviscidose [9–11, 13–20, 26]. À ce jour, la majorité des données de séquençage à haut-débit concernant le bactériote et le bactériome qui a été publiée porte sur la mucoviscidose. Ainsi, d’après les résultats de Zhao et al. [15], le microbiote pulmonaire des patients évolue avec l’âge. La diversité de la communauté bactérienne est en effet diminuée chez les patients plus âgés chez lesquels la famille Pseudomonadaceae prédomine [15]. L’infection chronique par P. aeruginosa, qui est considéré comme le pathogène le plus fréquemment rencontré chez les patients atteints de mucoviscidose, est associée à la diminution de la fonction pulmonaire [7, 26]. À côté de P. aeruginosa, d’autres espèces ou genres (Streptococcus, Prevotella, Veillonella, Rothia, Actinomyces, Gemella, Granulicatella, Fusobacterium, Neisseria, Atopobium, etc.) ont été identifiés comme étant importants dans la constitution du microbiote pulmonaire au cours de la mucoviscidose [9, 11, 13–20]. L’ensemble de ces études tend à montrer qu’il existe une corrélation significative entre la diversité du microbiome pulmonaire et l’âge, le statut clinique et l’état de la fonction pulmonaire des patients [11, 15–17, 20, 26]. |
Microbiome pulmonaire et broncho-pneumopathie chronique obstructive La broncho-pneumopathie chronique obstructive (BPCO) est une maladie respiratoire chronique définie par une obstruction permanente et progressive des voies aériennes. Cette obstruction est causée par l’association en proportion variable des deux facteurs suivants : une diminution du calibre des bronchioles du fait de modifications anatomiques (remodelage) et une destruction des alvéoles pulmonaires (emphysème). Il s’y associe une réponse inflammatoire pulmonaire anormale à des toxiques inhalés via le tabagisme et/ou la pollution. La colonisation bactérienne est un facteur important contribuant à la progression de la BPCO [29] et à la survenue des exacerbations infectieuses [7]. De nombreuses pistes ont été décrites pour expliquer comment l’exposition tabagique détruit les défenses pulmonaires de l’hôte via la suppression de la production de peptides antimicrobiens, la destruction de la barrière épithéliale et la colonisation des voies aériennes inférieures par un certain nombre d’agents microbiologiques [25, 30, 31]. Jusqu’à récemment, les analyses microbiologiques réalisées chez les patients atteints de BPCO consistaient à détecter essentiellement P. aeruginosa, M. catarrhalis et H. influenzae. Ces techniques d’analyse sont toujours utilisées en routine. Néanmoins, depuis quelques années, les microbiotes et microbiomes pulmonaires des patients présentant une BPCO ont fait l’objet de nouvelles études. Ainsi, Zakharkina et al. ont comparé le microbiome de 9 individus sains et de 9 patients atteints de BPCO [22]. Ils ont mis en évidence une grande diversité bactérienne même si le noyau principal comprenait, dans les deux groupes d’individus, les genres Prevotella, Sphingomonas, Pseudomonas, Acinetobacter, Fusobacterium, Megaspheaera, Veillonella, Staphylococcus et Streptococcus, confirmant les premiers travaux publiés [1]. Cependant, les patients porteurs de Pseudomonas présentaient un microbiome avec une diversité réduite. Il semblerait donc que la sévérité de la maladie soit associée à une diminution de la diversité microbienne avec une surreprésentation de certaines espèces. Ainsi les Pseudomoanadaceae, Enterobacteriaceae et Helicobacteriaceae paraissent jouer un rôle crucial dans la survenue des exacerbations [32]. Sze et al. ont constaté que le microbiote différait surtout entre les patients les plus sévères (stade IV) et les patients non ou anciens fumeurs [33]. Le microbiote est également modifié par l’antibiothérapie et la corticothérapie [24]. Il est également différent chez les patients atteints de mucoviscidose. Pourtant, la vie des patients présentant une BPCO, comme celle des patients atteints de mucoviscidose, est émaillée de nombreuses exacerbations essentiellement d’origine infectieuse qui sont en grande partie responsables du déclin de la fonction respiratoire des patients. Les principaux agents responsables des exacerbations observées chez les patients atteints de BPCO sont les virus, en particulier le rhinovirus. Ces infections virales ont un impact négatif sur le microbiome de ces patients. Ainsi Molyneaux et al. ont comparé l’évolution du microbiome de volontaires sains, fumeurs ou non, et de patients après une infection à rhinovirus [34]. Contrairement à la stabilité du bactériome qui est observée chez les sujets sains après cette infection, le bactériome des patients infectés reste modifié, dysbiotique, avec une diminution de la diversité et une augmentation de certaines bactéries comme Haemophilius. Ces variations semblent persister au moins 42 jours après l’infection. Elles pourraient jouer un rôle dans la survenue secondaire d’autres infections bactériennes [34]. |
Microbiome pulmonaire et asthme La pathologie asthmatique est l’exemple même de l’interaction entre l’hôte, un pathogène et l’environnement (Figure 1). Un lien a été clairement démontré entre microbiote environnemental, sensibilisation/atopie et asthme [35–37]. Deux importantes études transversales et multidisciplinaires portant sur de larges cohortes d’enfants (PARISFAL et GABRIELA) ont en effet permis de démontrer le lien entre l’environnement dans lequel les enfants grandissent (milieu rural versus milieu urbain), sa contamination bactérienne et fongique, et le risque de développer une atopie ou un asthme [38, 39]. Par ailleurs, les infections virales, notamment les bronchiolites de l’enfance et en particulier celles à rhinovirus, semblent être impliquées dans la genèse de l’asthme, peut-être sur des terrains prédisposés [40–42]. Aujourd’hui, de nombreuses données tendent à montrer que plus le microbiote environnemental est diversifié, moins le risque de développer un asthme est important. À l’inverse, la colonisation par des bactéries sélectionnées serait un facteur de risque accru pour l’asthme. L’impact de la colonisation bactérienne sur l’asthme et sur la survenue d’exacerbations, a été observé dans de nombreuses études avec des taux de colonisation allant de 12 à 61 % selon les résultats [43–45]. Les enfants asthmatiques semblent également présenter un microbiote bactérien différent de celui des enfants sains avec une majorité de Proteobacteria (Haemophilius, Neisseria) et de Staphylococcus chez les jeunes asthmatiques alors que les Bacteroidetes, Firmicutes et/ou acinétobactéries sont plus souvent retrouvés chez les enfants sains [6, 27]. Le microbiote bactérien des enfants asthmatiques semble également influencer leur réponse au traitement par des corticoïdes. Ainsi, certaines bactéries à Gram négatif, notamment des bactéries appartenant au phylum des Actinobacteria, voient leur nombre augmenté chez les enfants ayant un asthme résistant aux corticoïdes [23], les bactéries participant à l’induction de la corticorésistance [23]. |
Conclusion : vers une identification et un suivi individuel du microbiome pulmonaire pour une prise en charge thérapeutique optimale ? Chaque individu, sain ou malade, comporte une flore polymicrobienne et multi-sites qui lui est propre et indispensable pour sa survie (Figure 2). Lors du développement d’une pathologie, on observe une modification du microbiome. Ainsi, dans les pathologies respiratoires chroniques, le microbiome pulmonaire est altéré, il est dysbiotique (Figure 3). Il évoluera dans le temps en fonction de la pathologie mais, quelle qu’elle soit (mucoviscidose, BPCO, ou asthme), la survenue d’une exacerbation sera associée à une modification du bactériote et du bactériome, voire du mycobiote et/ou du virome [8, 9, 11, 14, 31, 33, 34]. Cette dysbiose, largement étudiée en ce qui concerne la communauté bactérienne, moins documentée pour les micromycètes, les virus ou les phages, montre clairement son impact sur la fonction respiratoire, sur la réponse inflammatoire et immune locale, voire une association avec une réponse incomplète au traitement adapté à la pathologie pulmonaire [23]. Certaines associations, notamment de bactéries mais aussi de micromycètes ou de virus, pourraient représenter des signatures soit de la pathologie elle-même, soit de l’exacerbation, soit d’une réponse inadaptée au traitement de première intention. De telles associations sont considérées comme de potentiels biomarqueurs taxonomiques capables de prédire une évolution pour le patient vers une exacerbation, une détérioration de sa fonction respiratoire et/ou une réponse inappropriée au traitement. L’ensemble de ces résultats, bien que préliminaires, confirment la pertinence des approches de séquençage à haut-débit en pneumologie. Elles permettent en effet de déterminer, mieux que ne le font les méthodes de culture classiques, les constituants des flores bactériennes aérobie et anaérobie ainsi que des flores fongique et virale. Finalement, les approches intégrant une dimension écologique pour l’étude et la compréhension des données métagénomiques ont permis une analyse différente, novatrice et potentiellement informative sur l’évolution clinique et le devenir du patient [28, 46].
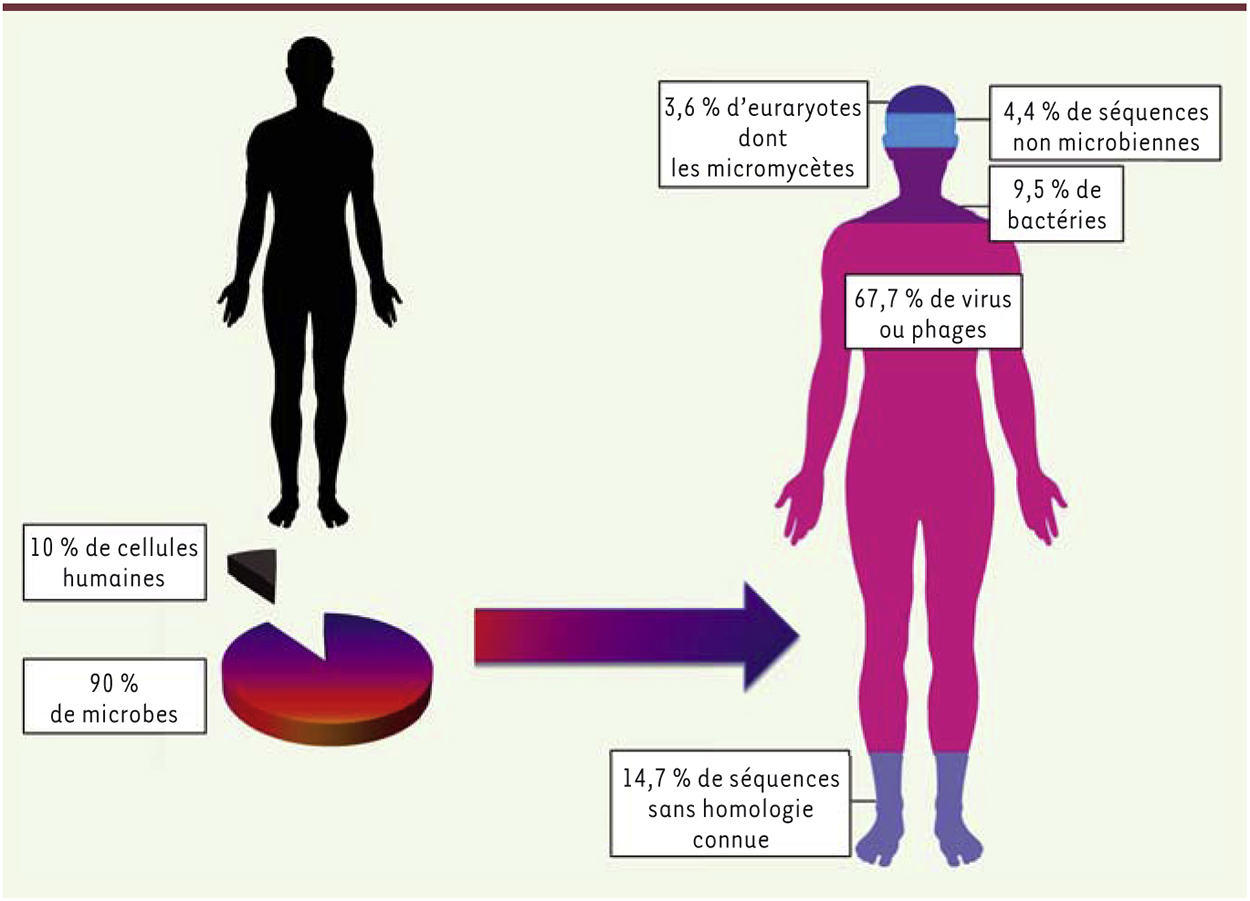
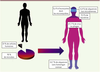 | Figure 2.
Proportions des différents microorganismes chez l’homme.
|
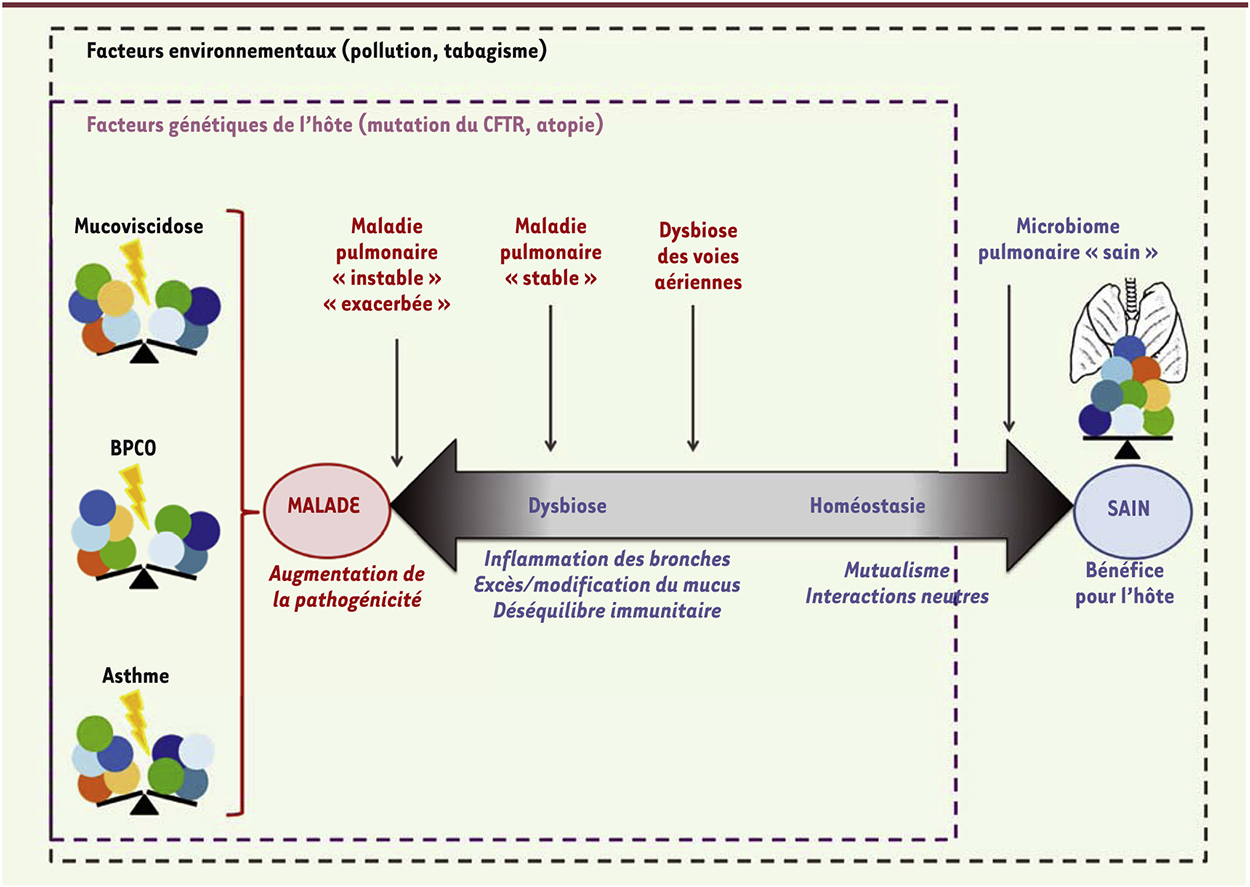
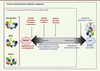 | Figure 3.
Hypothèses d’évolution du microbiome pulmonaire considéré comme entité pathogénique au cours des pathologies respiratoires chroniques. Les bactéries sont représentées par les ronds bleus, les virus par les ronds orange et les champignons par les ronds verts. BPCO : broncho-pneumopathie chronique obstructive ; CFTR : cystic fibrosis transmembrane conductance regulator. |
Le séquençage à haut-débit offre donc la possibilité d’étudier simultanément l’ensemble des microorganismes (bactéries, virus, phages et micromycètes) présents dans le tractus respiratoire. Cette technologie représente une stratégie méthodologique de choix qu’il sera nécessaire de standardiser et de valider [18, 20, 28, 46] avant de pouvoir l’appliquer en pratique quotidienne de pneumologie. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Par convention, sont désignés sous les termes « biote » – en particulier bactériote ou mycobiote – la communauté de bactéries ou de micromycètes ou de virus et phages présente dans un tissu ou organe défini (ici le poumon), tandis que les termes en « biome » – bactériome, mycobiome, virome – désignent l’ensemble des génomes composant cette communauté de bactéries, ou de micromycètes ou de virus et phages. Sur le même principe, le résistome fait référence à l’ensemble des gènes issus des génomes impliqués dans la résistance aux agents antimicrobiens présents dans une communauté microbienne donnée. |
1.
Erb-Downward JR, Thompson DL, Han MK, et al. Analysis of the lung microbiome in the healthy smoker and in COPD . PLoS One. 2011; ; 6 : :e16384.. 2.
Dickson RP, Erb-Downward JR, Huffnagle GB. The role of the bacterial microbiome in lung disease . Expert Rev Respir Med. 2013; ; 7 : :245.–257. 3.
Marsland BJ, Gollwitzer ES. Host-microorganism interactions in lung diseases . Nat Rev Immunol. 2014; ; 14 : :827.–835. 4.
Charlson ES, Bittinger K, Haas AR, et al. Topographical continuity of bacterial populations in the healthy human respiratory tract . Am J Respir Crit Care Med. 2011; ; 184 : :957.–963. 5.
Morris A, Beck JM, Schloss PD, et al. Comparison of the respiratory microbiome in healthy nonsmokers and smokers . Am J Respir Crit Care Med. 2013; ; 187 : :1067.–1075. 6.
Bouladoux N, Hand TW, Naik S, Belkaid Y. Microbiote et lymphocytes T : les meilleurs ennemis . Med Sci (Paris). 2013; ; 29 : :349.–352. 7.
Sze MA, Hogg JC, Sin DD. Bacterial microbiome of lungs in COPD . Int J Chron Obstruct Pulmon Dis. 2014; ; 9 : :229.–238. 8.
Hilty M, Burke C, Pedro H, et al. Disordered microbial communities in asthmatic airways . PLoS One. 2010; ; 5 : :e8578.. 9.
Zemanick ET, Harris JK, Wagner BD, et al. Inflammation and airway microbiota during cystic fibrosis pulmonary exacerbations . PLoS One. 2013; ; 8 : :e62917.. 10.
Tunney MM, Einarsson GG, Wei L, et al. Lung microbiota and bacterial abundance in patients with bronchiectasis when clinically stable and during exacerbation . Am J Respir Crit Care Med. 2013; ; 187 : :1118.–1126. 11.
Delhaes L, Monchy S, Fréalle E, et al. The airway microbiota in cystic fibrosis: a complex fungal and bacterial community . Implications for therapeutic management. PLoS One. 2012; ; 7 : :e36313.. 12.
Harrison MJ, Twomey KB, McCarthy Y, et al. The role of second-generation sequencing in describing the fungal microbiota in the adult cystic fibrosis (CF) airway and its correlation with clinical phenotype . J Cyst Fibros. 2013; ; 12 : :S16.. 13.
Madan JC, Koestler DC, Stanton BA, et al. Serial analysis of the gut and respiratory microbiome in cystic fibrosis in infancy: interaction between intestinal and respiratory tracts and impact of nutritional exposures . MBio. 2012; ; 3 : :e00251.–e00212. 14.
Lim YW, Evangelista JS 3rd, Schmieder R, et al. Clinical insights from metagenomic analysis of cystic fibrosis sputum . J Clin Microbiol. 2014; ; 52 : :425.–437. 15.
Zhao J, Schloss PD, Kalikin LM, et al. Decade-long bacterial community dynamics in cystic fibrosis airways . Proc Natl Acad Sci USA. 2012; ; 109 : :5809.–5814. 16.
Van der Gast CJ, Walker AW, Stressmann FA, et al. Partitioning core and satellite taxa from within cystic fibrosis lung bacterial communities . ISME J. 2011; ; 5 : :780.–791. 17.
Cox MJ, Allgaier M, Taylor B, et al. Airway microbiota and pathogen abundance in age-stratified cystic fibrosis patients . PLoS One. 2010; ; 5 : :e11044.. 18.
Goddard AF, Staudinger BJ, Dowd SE, et al. Direct sampling of cystic fibrosis lungs indicates that DNA-based analyses of upper-airway specimens can misrepresent lung microbiota . Proc Natl Acad Sci USA. 2012; ; 109 : :13769.–13774. 19.
Fodor AA, Klem ER, Gilpin DF, et al. The adult cystic fibrosis airway microbiota is stable over time and infection type, and highly resilient to antibiotic treatment of exacerbations . PLoS One. 2012; ; 7 : :e45001.. 20.
Maughan H, Wang PW, Diaz Caballero J, et al. Analysis of the cystic fibrosis lung microbiota via serial Illumina sequencing of bacterial 16S rRNA hypervariable regions . PLoS One. 2012; ; 7 : :e45791.. 21.
LiPuma JJ. The changing microbial epidemiology in cystic fibrosis . Clin Microbiol Rev. 2010; ; 23 : :299.–323. 22.
Zakharkina T, Heinzel E, Koczulla RA, et al. Analysis of the airway microbiota of healthy individuals and patients with chronic obstructive pulmonary disease by T-RFLP and clone sequencing . PLoS One. 2013; ; 8 : :E68302.. 23.
Goleva E, Jackson LP, Harris JK, et al. The effects of airway microbiome on corticosteroid responsiveness in asthma . Am J Respir Crit Care Med. 2013; ; 188 : :1193.–1201. 24.
Huang JY, Sethi S, Murphy T, et al. Airway microbiome dynamics in exacerbations of chronic obstructive pulmonary disease . J Clin Microbiol. 2014; ; 52 : :2813.–2823. 25.
Weinreich UM, Korsgaard J. Bacterial colonisation of lower airways in health and chronic lung disease . Clin Respir J. 2008; ; 2 : :116.–122. 26.
Ramsey KA, Ranganathan S, Park J, et al. Early respiratory infection is associated with reduced spirometry in children with cystic fibrosis . Am J Respir Crit Care Med. 2014; ; 190 : :1111.–1116. 27.
Marri PR, Stern DA, Wright AL, et al. Asthma-associated differences in microbial composition of induced sputum . J Allergy Clin Immunol. 2013; ; 131 : :346.–352. 28.
Rogers GB, Hoffman LR, Carroll MP, Bruce KD. Interpreting infective microbiota: the importance of an ecological perspective . Trends Microbiol. 2013; ; 21 : :271.–276. 29.
Sethi S. Infection as comorbidity of COPD . Eur Respir J. 2010; ; 35 : :1209.–1215. 30.
Heijink IH, Brandenurg SM, Postma DS, van Oosterhout AJ. Cigarette smoke impairs airway epithelial barrier function and cell-cell contact recovery . Eur Respir J. 2012; ; 39 : :419.–428. 31.
Pauly JL, Paszkiewicz G. Cigarette smoke, bacteria, mold, microbial toxins, and chronic lung inflammation . J Oncol. 2011; ; 2011 : :81929.. 32.
Huang YJ, Kim E, Cox MJ, et al. A persistent and diverse airway microbiota present during chronic obstructive pulmonary disease exacerbation . OMICS. 2010; ; 14 : :9.–59. 33.
Sze MA, Dimitriu PA, Hayashi S, et al. The lung tissue microbiome in chronic obstructive pulmonary disease . Am J Respir Crit Care Med. 2012; ; 185 : :1073.–1080. 34.
Molyneaux PL, Mallia P, Cox MJ, et al. Outgrowth of the bacterial airway microbiome after rhinovirus exacerbation of chronic obstuctrive pulmonary disease . Am J Respir Crit Care Med. 2013; ; 188 : :1224.–1231. 35.
Braun-Fahrlander C, Riedler J, Herz U, et al. Environmental exposure to endotoxin and its relation to asthma in school-age children . N Engl J Med. 2002; ; 347 : :869.–877. 36.
Fyhrquist N, Ruokolainen L, Suomalainen A, et al. Acinetobacter species in the skin microbiota protects against allergic sensitization and inflammation . J Allergy Clin Immunol. 2014; ; 134 : :1301.–1309. 37.
Trompette A, Gollwitzer ES, Yadava K, et al. Gut microbiota metabolism of dietary fiber influences allergic airway disease and hematopoiesis . Nat Med. 2014; ; 20 : :159.–166. 38.
Schram-Bijkerk D, Doekes G, Douwes J, et al. Bacterial and fungal agents in house dust and wheeze in children: the PARSIFAL study . Clin Exp Allergy. 2005; ; 35 : :1272.–1278. 39.
Ege MJ, Mayer M, Normand AC, et al. Exposure to environmental microorganisms and childhood asthma . N Engl J Med. 2011; ; 364 : :701.–709. 40.
Freymuth F, Vabret A, Dina J, et al. Les virus des bronchiolites aiguës . Arch Pediatr. 2010; ; 17 : :1192.–1201. 41.
Sigurs N, Aljassim F, Kjellman B, et al. Asthma and allergy patterns over 18 years after severe RSV bronchiolitis in the first year of life . Thorax. 2010; ; 65 : :1045.–1052. 42.
Bacharier LB, Cohen R, Schweiger T, et al. Determinants of asthma after severe respiratory syncytial virus bronchiolitis . J Allergy Clin Immunol. 2012; ; 130 : :91.–100. 43.
Nagayama Y, Tsubaki T, Nakayama S, et al. Bacterial colonization in respiratory secretions from acute and recurrent wheezing infants and children . Pediatr Allergy Immunol. 2007; ; 18 : :110.–117. 44.
Zhang Q, Illing R, Hui CK, et al. Bacteria in sputum of stable severe asthma and increased airway wall thickness . Respir Res. 2012; ; 13 : :35.. 45.
Bisgaard H, Hermansen MN, Buchvald F, et al. Childhood asthma after bacterial colonization of the airway in neonates . N Engl J Med. 2007; ; 357 : :1487.–1495. 46.
Whiteson KL, Bailey B, Bergkessel M, et al. The upper respiratory tract as a microbial source for pulmonary infections in cystic fibrosis. Parallels from island biogeography . Am J Respir Crit Care Med. 2014; ; 189 : :1309.–1315. 47.
Nguyen L, Delhaes L. Un nouveau concept : le mycobiome pulmonaire . Med Sci (Paris). 2015; ; 31 : :945.–947. 48.
Brégeon F, Rolain JM. Le résistome pulmonaire. Med Sci (Paris). 2015; ; 31 : :947.–950. |