| |
| Med Sci (Paris). 2015 December; 31(12): 1102–1108. Published online 2015 December 16. doi: 10.1051/medsci/20153112013.Les adénovirus non-humains Un risque zoonotique ? Fabien Loustalot,1,2 Sophie Creyssels,1,2 Sara Salinas,1,2 Mária Benkõ,3 Balázs Harrach,3 Franck J.D. Mennechet,1,2 and Eric J. Kremer1,2* 1Institut de Génétique Moléculaire de Montpellier, CNRS UMR 5535, 1919, route de Mende, 34293Montpellier, France 2Université de Montpellier, Montpellier, France 3Institute for Veterinary Medical Research, Center for Agricultural Research, Hungarian academy of sciences, H-1581Budapest, Hongrie |
Un grand nombre de facteurs virologiques, environnementaux et socioculturels interviennent dans l’émergence et la réémergence de maladies virales. Les virus présentent un temps de réplication très court et l’aptitude à produire des milliers de particules par cellule infectée. Les virus sont aussi des organismes qui évoluent rapidement grâce à une grande capacité d’adaptation et à des modifications génétiques. N’occupant pas une niche écologique de façon constante et définitive, les virus ont ainsi sélectionné au cours de l’évolution la possibilité de parasiter des espèces hôtes alternatives. Bien que restant un événement rare chez l’être humain, les zoonoses font l’objet d’une veille constante en raison de l’impact considérable que certains virus ont aujourd’hui sur la santé publique à l’échelle planétaire. |
Les animaux incubateurs de pathogènes humains Rendus célèbres par les médias, certains virus émergents ont surpris la communauté médicale et scientifique en raison de l’intensité de leur virulence, associée à un niveau élevé de contagion et de mortalité. Comme pour la plupart des virus émergents, ils sont à l’origine de zoonoses, c’est-à-dire de maladies et/ou d’infections qui se transmettent des animaux à l’homme. Les principales zoonoses virales connues à ce jour impliquent des virus à ARN (rage, chikungunya, grippes). Parmi les nombreuses espèces animales sauvages et/ou domestiques pouvant constituer le réservoir potentiel de ces virus, les chauves-souris sont particulièrement incriminées. Les chauves-souris forment l’ordre des chiroptères (Chiroptera) et représentent, avec presque un millier d’espèces répertoriées, le deuxième ordre des mammifères le plus important après celui des rongeurs (Rodentia). Réparties sur l’ensemble de la planète et parfois migratoires, les chauves-souris sont considérées comme le réservoir principal et naturel de nombreux pathogènes viraux à ARN comme les Rhabdoviruses, les Henipaviruses, les Coronoviruses et les Filoviruses [1, 2]. Au cours des vingt dernières années, les chauves-souris ont été suspectées d’être à l’origine de l’émergence de nombreux virus zoonotiques. Ainsi en 2003, plus de 8 000 individus à travers le monde furent contaminés par l’agent responsable du SARS-CoV (severe acute respiratory syndrome coronavirus) avec, dans près de 10 % des cas, une conséquence mortelle [3]. Plus récemment en 2010, le MERS-CoV (Middle East respiratory syndrome coronarivus) a emergé de la péninsule arabique et s’est étendu jusqu’en Europe, provoquant la mort d’environ 50 % des individus infectés [4]. Enfin, le virus Ebola souche Zaïre, appartenant à la famille des Filoviridae, a été à l’origine de plusieurs épidémies en Afrique de l’ouest dont le taux de létalité a parfois atteint 90 %. La dernière épidémie Ebola, amorcée en décembre 2013 en République démocratique du Congo, affichait un taux de mortalité de 40,9 % au 1er mars 2015 selon l’Organisation mondiale de la santé (OMS) (World health organisation, WHO). C’est donc dans ce contexte complexe, impliquant la virologie et les bouleversements socioculturels et environnementaux, que nous nous sommes demandé si des adénovirus (AdV) ayant pour réservoir naturel les chauves-souris (BtAdV, bat adenovirus) et/ou des primates non-humains (NHP-AdV, non-human primate adenovirus) pourraient un jour constituer un risque pour l’homme. |
Zoonoses adénovirales : des raisons de s’inquiéter ? Quelques observations tendent à montrer que la transmission inter-espèces des AdV serait un phénomène à ne pas négliger. Les virus à ADN comme les AdV sont généralement considérés comme étant plutôt spécifiques de leur espèce. Or, un AdV d’origine animale serait capable de changer d’espèce en conservant une partie de son pouvoir pathogène et contagieux chez son nouvel hôte [5]. Ainsi, dans un épisode zoonotique provoquant des atteintes respiratoires sévères, le virus baptisé TMAdV (adénovirus du singe Titi) avait préalablement causé la mort de 83 % des animaux qu’il avait infectés au sein d’une colonie de singes du Nouveau Monde (Callicebus cupreus), avant d’être clairement incriminé dans deux cas d’infections respiratoires chez l’homme. Parmi ces deux cas, le premier était un membre du personnel de recherche médicale en contact direct avec les animaux, alors que le second était un membre de sa famille n’ayant jamais été en contact direct avec les singes ou leur environnement [5]. La présence d’anticorps dirigés contre des AdV de babouins (BaAdV) et de chimpanzés (ChAdV) a été détectée dans le sérum de 2,5 % du personnel travaillant à proximité des animaux [6, 7]. L’analyse génétique des AdV a montré que les virus isolés de différentes espèces animales, comme les AdV canins (CAdV) types 1 et 2 et les BtAdV types 2 et 3, présentaient une similarité de séquence élevée pour les régions précoces 3 et 4 (early region 3 and 4 ; E3-E4) de leur génome [8], renforçant ainsi le regroupement phylogénétique qui avait déjà été établi entre ces deux virus [9]. Des isolats, réalisés chez des chimpanzés et des gorilles sauvages vivant dans leur milieu naturel, ont de même révélé la présence d’AdV dont l’analyse a mis en évidence un regroupement phylogénétique avec des AdV humains (HAdV) [10]. Ces différentes observations rendent ainsi compte de la possibilité d’une transmission inter-espèces des AdV de l’animal vers l’homme, mais aussi d’une probable propagation horizontale d’AdV non-humains d’homme à homme. |
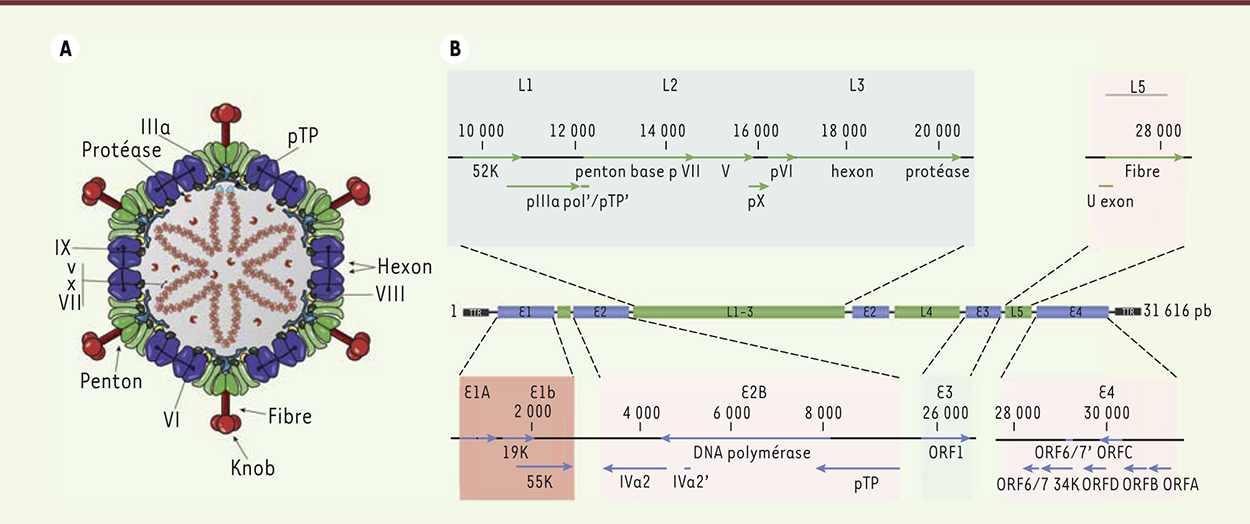
Les adénovirus : structure, génome et cycle infectieux La famille Adenoviridae comprend de nombreuses espèces classifiées en cinq genres [11]. Le genre Mastadenovirus englobe les AdV infectant les mammifères. Les genres Aviadenovirus, Siadenovirus, Atadenovirus, Ichtadenovirus infectent les principaux vertébrés (poissons, reptiles, oiseaux) [11]. Au sein du genre Mastadenovirus, sept espèces humaines (human Mastadenovirus A à G) avec plus de soixante types d’HAdV et deux espèces de chauves-souris (bat Mastadenovirus A et B) avec trois types de BtAdV ont été répertoriées. La dénomination des AdV humains suit une nomenclature récemment modifiée : la première lettre en majuscule identifie l’hôte (humain) suivi de AdV, puis de l’espèce (A à G) et enfin le type (chiffre), par exemple adénovirus humain de l’espèce C type 5, HAdV-C5. La famille Adenoviridae possède un tropisme varié infectant de nombreux types tissulaires. Les AdV peuvent provoquer des maladies très diverses causant principalement des troubles respiratoires, oculaires ou digestifs modérés. Plus rarement, certains HAdV peuvent provoquer la mort chez des individus à risque comme les personnes immunodéprimées, très jeunes ou âgées [12]. La transmission des AdV se fait principalement par contact direct ou féco-oral. Les AdV sont des particules virales non-enveloppées à ADN double brin linéaire non-segmenté [13]. D’un diamètre de 70 à 110 nm, la capside protéique à symétrie icosaédrique (20 faces triangulaires) est constituée de trois protéines majeures : l’hexon (II), la base du penton (III) et la fibre (IV), formant 252 capsomères1
(Figure 1A). Les 240 hexons occupent les faces alors que les 12 pentons (constitués de la base du penton et de la fibre) se répartissent sur les sommets. Chaque homopentamère de base du penton supporte une protéine homotrimérique appelée la fibre dont l’extrémité globulaire (ou knob) s’attache à la surface des cellules [14, 15]. Sur la face interne de la capside, quatre protéines mineures, IIIa, VI, VIII et IX, assurent une meilleure stabilité de la capside. Trois protéines basiques, V, VII et X, sont associées au génome viral. Ces protéines ont pour fonction de compacter l’ADN viral à la manière des histones. Enfin, chaque extrémité 5’ du génome viral est liée à une protéine TP (terminal protein) impliquée dans la réplication [16] (Figure 1A).
 | Figure 1.
Structure et organisation du génome viral. A. Représentation schématique d’un adénovirus. Vue d’ensemble de la capside montrant la localisation des protéines majeures (hexon, base du penton et fibre knob), des protéines mineures (IIIa, VI, VIII, IX et la protéase) ainsi que les protéines associées au génome (V, VII, X et pTP). B. Illustration de l’organisation génomique du BtAdV-2. Représentation des unités transcriptionnelles. La ligne horizontale noire représente l’ADN génomique avec, aux extrémités, les séquences répétées inverses (ITR). Les rectangles en bleu correspondent aux gènes précoces et ceux en vert représentent les gènes tardifs (partie centrale de l’illustration). Les régions codantes sont représentées par des flèches orientées dans le sens de la transcription (code couleur identique à ci-dessus). Le nom des gènes ainsi que leur position sur le génome sont indiqués. Les encadrés en vert, qui englobent les gènes, correspondent aux séquences génomiques qui présentent un fort taux de recombinaison dans les différentes espèces d’AdV. Graduellement en rouge sont représentées les régions qui pourraient se recombiner et permettre l’adaptation du virus à un changement d’hôte. |
Le cycle infectieux des AdV suit une chronologie bien établie avec (1) l’attachement, l’internalisation, et le trafic vers le noyau de la cellule ; (2) l’expression des gènes viraux et la réplication ; et enfin (3) la libération et la propagation des particules virales. L’émergence d’un virus au pouvoir zoonotique doit donc résulter d’une adaptation du virus à au moins l’une de ces étapes. L’attachement des virus à la surface des cellules est le résultat de l’interaction d’une ou plusieurs protéines virales avec un ou des récepteurs membranaires initiant l’internalisation de la particule. Lors d’un changement d’hôte, le virus va reconnaître un (des) récepteur(s) hétérologue(s). De nombreuses molécules de la membrane cellulaire peuvent interagir avec les HAdV [17]. Parmi celles-ci, CAR (coxsackievirus and adenovirus receptor), qui est bien caractérisé [18–20], est une protéine transmembranaire de type I2 qui appartient à la sous-famille CTX (cortical thymocyte marker in Xenopus), membre de la superfamille des immunoglobulines [21]. Exprimé par de nombreux tissus et types cellulaires, CAR présente un niveau élevé de conservation entre les espèces [20]. D’autres molécules réceptrices ont été également décrites dont CD46 (exprimé par toutes les cellules humaines nucléées et impliqué dans la protection contre la lyse cellulaire induite par le système du complément), l’acide sialique et la desmogléine-2 (molécule d’adhésion appartenant à la superfamille des cadhérines) [17]. Certains composants plasmatiques comme le facteur X (FX) de la coagulation peuvent également générer un « pont moléculaire » entre les HAdV et les cellules cibles, en particulier lors d’infection du foie et de la rate après injection par voie systémique de vecteurs adénoviraux [22, 23]. Les virus émergents SARS-CoV et MERS-CoV, récemment isolés de zoonoses à coronavirus (CoV), peuvent se fixer indépendamment sur deux récepteurs cellulaires spécifiques. Par son domaine S1, la protéine d’enveloppe Spike (S) du SARS-CoV interagit avec la molécule ACE-2 (angiotensin-converting enzyme 2) [24] et l’internalisation du MERS-CoV fait intervenir la protéine DPP4 (dipeptidyl peptidase 4) présente à la surface des cellules [25]. Notre expertise des AdV et de leur utilisation en tant que vecteurs nous portent à croire que l’étape initiale de reconnaissance cellulaire ne serait vraisemblablement pas l’étape déterminante pour l’émergence potentielle d’une zoonose adénovirale. Certains AdV non-humains comme le CAdV-2 [26] et le SAdV-5 (simian) peuvent en effet interagir avec CAR [19, 27] et donc être internalisés dans les cellules. De même, les vecteurs dérivés des CAdV et des HAdV peuvent pénétrer dans les cellules de plusieurs espèces incluant les rongeurs, les félins, les primates et même les poissons. Enfin, avec le développement de ces vecteurs, il a été montré qu’un même transgène pouvait être exprimé dans des cellules provenant d’espèces différentes, montrant que le transport intracellulaire des particules virales qui ont pénétré la cellule est efficace et permet la libération du génome viral dans le noyau cellulaire. En revanche, la réplication et la dissémination virale sont fortement réduites dans un système xénogénique. Ces dernières étapes du cycle viral limiteraient donc la transmission des AdV entre espèces différentes. Les génomes viraux présentent une densité de région codante inégalée dans le monde du vivant. Ceci est principalement dû au fait que la taille maximale de la molécule d’ADN qui peut être incorporée au sein d’une capside, dont la dimension est fixe, est limitée. Généralement, pour un virus à ADN, deux grand types de protéines sont synthétisés au cours d’un cycle : (1) les protéines régulatrices qui s’expriment lors de la phase précoce du cycle, une fois le génome libéré dans le noyau de la cellule hôte (gènes précoces) et (2) les protéines de structure s’exprimant lors de la phase tardive du cycle infectieux (gènes tardifs) [28]. Les protéines de structure vont permettre l’établissement de la capside protéique, des protéines associées à l’enveloppe du virus (pour les virus enveloppés), du tégument (Herpesviridae) ou des protéines se liant au génome viral. Les protéines régulatrices ont pour rôle de répliquer le génome viral, de contrôler la réponse immunitaire de l’hôte et d’interagir avec des protéines de la cellule afin d’induire un environnement intracellulaire propice à la réplication virale et à la libération des virions néo-synthétisés. La structure du génome des Mastadenovirus est similaire (Figure 1B). Il est constitué d’une molécule linéaire double brin d’ADN de 26 000 à 45 000 paires de bases dont les extrémités sont composées de séquences répétées inversées (ITR) qui forment les origines de réplication. Le génome des AdV peut être subdivisé en différentes unités de transcription (gènes précoces : E1, E2, E3 et E4 ; gènes tardifs : L1, L2, L3, L4 et L5) qui sont réparties indifféremment sur les deux brins de l’ADN et sont transcrites respectivement avant et après la réplication virale [28]. L’expression de ces différentes régions repose sur les ARN polymérases II/III de la cellule [29]. Lorsque le génome viral est libéré dans le noyau, la région précoce 1A (early region 1A ou E1A) est exprimée grâce à des facteurs de transcription cellulaires, indépendamment de facteurs viraux néo-synthétisés. Cette région capitale du génome est à l’origine de la synthèse de facteurs de trans-activation qui permettent l’expression de toutes les protéines codées par les régions précoces. Des protéines cellulaires interagissent avec les protéines virales E1A via des régions conservées (conserved region : CR) communes aux AdV d’une même espèce [28, 30]. La protéine majeure de la région E1A se lie ainsi à différents facteurs de transcription cellulaires comme la famille ATF (activating transcription factors) (tel c-Jun), la TBP (TATA binding protein), le coactivateur transcriptionnel p300, la pRb (retinoblastoma protein), ce qui conduit à l’expression des régions E2, E3 et E4 [31]. La réplication virale est alors initiée. La région E2 est nécessaire pour la synthèse de l’ADN du virus. Elle code une ADN polymérase, un précurseur de la protéine terminale (pTP) et une protéine de liaison à l’ADN (DNA-binding protein ou DBP) [32]. La région E3 code jusqu’à 9 protéines selon l’espèce d’AdV. La plupart d’entre elles ont un rôle dans le contrôle de la réponse immunitaire de l’hôte. En particulier, la protéine gp19K se lie aux molécules du complexe majeur d’histocompatibilité de classe I (CMH I) [33], bloquant la réponse immunitaire cytotoxique dépendant des lymphocytes T. Enfin, la région E4 produit plusieurs protéines qui ont toutes un rôle essentiel dans la réplication du virus [34]. La protéine E4 ORF3 (E4 gene open reading frame 3 protein) forme un complexe avec E1B-55K et recrute des composants associés aux corps nucléaires PML (promyelocytic leukemia) [35] qui interviennent dans les processus de réparation de l’ADN, de réplication, de défense antivirale et au niveau de la transcription [36]. Alors que l’expression des régions précoces repose sur des promoteurs indépendants, les gènes tardifs constituent un unique opéron placé sous le contrôle du promoteur tardif majeur (MLP : major late promoter). L’activation du MLP semble dépendre de la réplication virale. En effet, si la réplication n’est pas réalisée, l’activation du MLP n’est pas optimale malgré la présence des différents facteurs viraux (protéines IVa2 et IX) [37]. Ainsi, la production des particules via l’expression des protéines de structure ne s’effectue qu’à partir de génomes viraux néo-synthétisés (soit 105-106 molécules/cellule), ce qui permet de produire une quantité massive de virions (jusqu’à 100 000 virions/cellule). La limitation de la transmission inter-espèce des AdV serait donc plutôt associée à l’expression des gènes précoces sans lesquels le cycle viral ne peut être poursuivi et aboutir [38]. Au-delà des interactions protéines-protéines, faisant intervenir les produits de E1 et les protéines cellulaires qui permettent l’expression des régions précoces du virus, différents motifs régulateurs en cis ont été identifiés au sein du génome des AdV. Ainsi, quatre éléments de régulation ont été localisés dans la région promotrice de l’unité de transcription E2 des HAdV : un site d’ancrage pour la TBP, deux sites pour le facteur d’élongation E2F, et un site d’accroche pour des facteurs de transcription de la famille ATF. Les produits de l’unité de transcription E2 sont responsables de la réplication de l’ADN viral [32]. Or, les protéines issues de l’expression d’E2 ne permettent pas à elles-seules de répliquer le génome et différents facteurs cellulaires sont requis. C’est le cas du facteur nucléaire I (NF-I) qui interagit avec le complexe formé entre l’ADN polymérase virale (AdPol) et la pTP, et stabilise le complexe aux ITR (inverted terminal repeat) [39], ou du complexe NF-III/Oct-1 qui favorise les interactions du complexe d’initiation [28, 32]. L’homologie des protéines d’hôtes phylogénétiquement proches favoriserait ainsi la transmission des AdV. |
Mécanismes moléculaires pour l’émergence de zoonoses La réplication est une étape clé favorisant l’apparition de mutations pouvant mener à la création de nouveau(x) caractère(s) viraux « à risque(s) ». Les risques pourraient être d’autant plus importants si les mutations se situent au niveau de séquences régulatrices permettant le recrutement des facteurs cellulaires et/ou des protéines régulatrices virales impliquées dans les interactions avec ces facteurs cellulaires. En raison du grand nombre de copies d’ADN viral néo-synthétisées présentes par cellule infectée, l’apparition de mutations induites par l’AdPol pourrait être à l’origine de l’acquisition par le virus de nouvelles fonctions. Cependant, l’AdPol, tout comme ses homologues cellulaires, ne commet seulement qu’une erreur pour 109 nucléotides (soit un taux d’erreur d’environ 1 × 10-9) et possède une activité intrinsèque correctrice d’édition [32]. Le scénario le plus probable pour générer l’apparition d’AdV au pouvoir zoonotique ferait donc intervenir des remaniements génomiques par recombinaison. L’émergence du SARS-CoV et du MERS-CoV semble être la conséquence de tels événements de recombinaison apparus lors d’infection de chauves-souris de deux sous-ordres différents [40]. Un exemple d’événements de recombinaison peut être observé avec HAdV-D53, l’agent étiologique d’une kératoconjonctivite déclarée en 2006 chez douze patients d’une même clinique européenne [41]. Le séquençage de HAdV-D53 a révélé qu’il proviendrait de recombinaisons génétiques entre les souches HAdV-D22, HAdV-D8 et HAdV-D37 [42]. Ces dernières années, la littérature révèle un grand nombre de réarrangement génomique entre les différents HAdV expliquant en partie la voie évolutive des HAdV [43–46]. En revanche, ce processus de recombinaison génétique n’est pas restreint aux HAdV. Ces évènements peuvent en effet se réaliser entre AdV d’espèces différentes donnant naissance à de nouveaux pathogènes potentiellement dangereux pour l’homme et/ou pour les autres espèces. Prenons l’exemple du HAdV-E4 dont le génome contient de nombreuses similarités avec celui de certains adénovirus de chimpanzé (ChAdV) [47]. HAdV-E4 est un recombinant contenant les boucles 1 et 2 de l’hexon de HAdV-B16 au sein d’un génome de SAdV-26. Il a été proposée que l’adaptation de l’HAdV-E4 à son nouvel hôte résultait d’une part de la capture d’une partie du gène de l’hexon d’un HAdV, et d’autre part de l’intégration d’un site d’ancrage pour un facteur de transcription humain ayant pour origine un autre HAdV [48]. Les gènes de l’hexon, la base du penton et la fibre semblent se recombiner entre virus de l’espèce HAdV-D, au même titre que les gènes de la région E3 [49] (Figure 1B), unité de transcription impliquée dans la pathogénicité virale et l’échappement immunitaire. Comme pour HAdV-E4, l’analyse bio-informatique du génome de SAdV-35 a révélé que ce nouveau virus était issu d’événements de recombinaison entre des SAdV et des HAdV. Ainsi, il a été retrouvé au sein du génome de SAdV-35, au niveau des ITR, une séquence provenant d’un HAdV permettant le recrutement du facteur nucléaire cellulaire NF-I [50]. Ce virus pourrait donc être un agent zoonotique contenant des séquences impliquées dans l’adaptation à son nouvel hôte. |
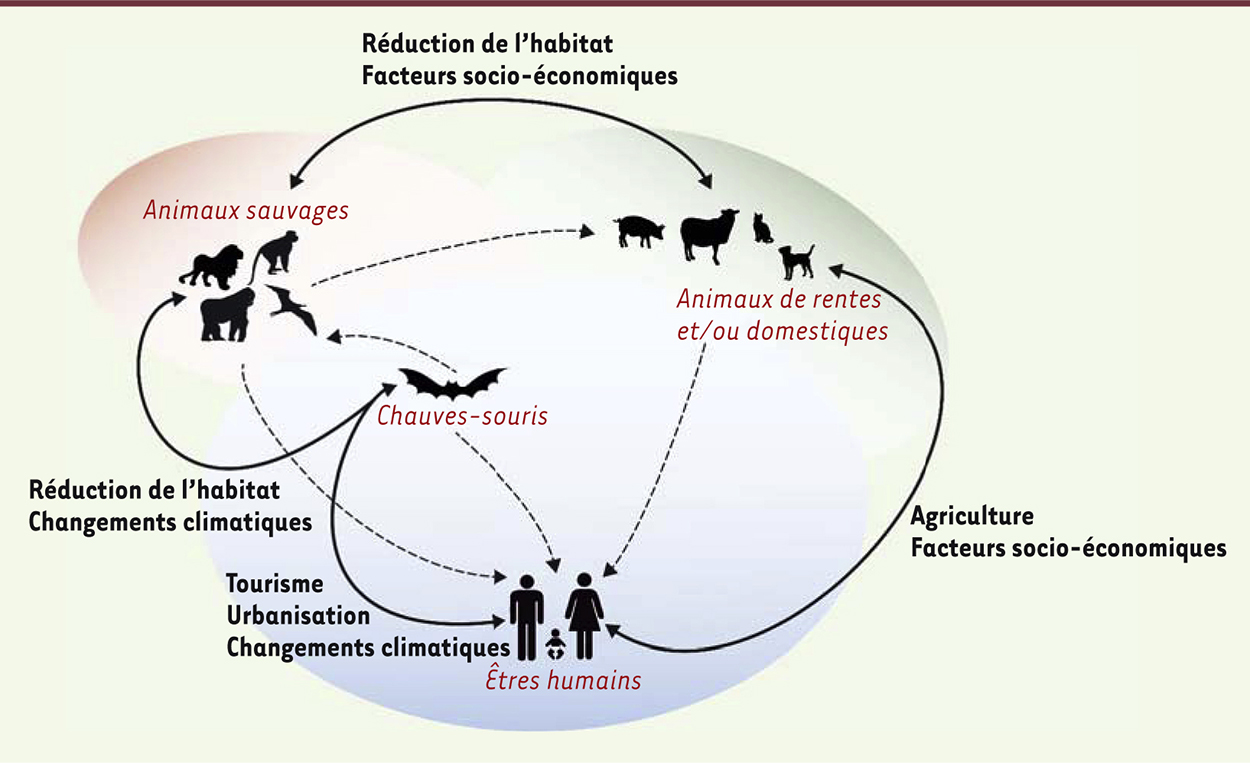
Facteurs extrinsèques intervenant dans l’apparition de zoonoses Parmi les facteurs favorisant les réarrangements génomiques et déterminants pour l’émergence de zoonoses, les phénomènes de rapprochement entre les nouveaux hôtes potentiels et les agents infectieux sont à prendre en considération. Ils favorisent les co-infections (par des AdV de différentes espèces ou des AdV de même espèce mais de types différents) et/ou les infections d’hôtes alternatifs (Figure 2). En effet, le développement des sociétés humaines à travers la planète réduit de manière considérable l’habitat animal augmentant ainsi les risques pour l’homme de se trouver en contact avec de nouveaux réservoirs animaux. De plus, les modifications de l’écosystème dues aux changements de modes de vie (migration, urbanisation, alimentation), ainsi que l’adaptation de l’environnement, sont sources de perturbation pour l’équilibre entre hôtes et agents infectieux (Figure 2). Cependant, malgré une colonisation presque planétaire des chauves-souris et la réduction de leur habitat naturel, les données récemment recueillies suggèrent que l’homme n’a pas encore été infecté par des adénovirus de chauve-souris, bien que certains BtAdV puissent se propager dans des cellules humaines [54] et que, inversement, certains HAdV puissent in vitro, se propager dans des cellules de chauves-souris (Zhengli Shi, Wuhan Institute of Virology, Hubei, China, communication personnelle).
 | Figure 2.
Représentation des interactions entre les différents réservoirs. Illustration des facteurs favorisant l’apparition de zoonoses. Les bulles correspondent à l’environnement dans lequel évoluent les animaux sauvages y compris les chauves-souris (rouge), les animaux d’élevages et/ou domestiques (vert) et les êtres humains (bleu). Les chevauchements de ces environnements représentent les interactions entre ces groupes. L’homme, ainsi que les animaux domestiques et/ou d’élevage, présentent une forte interaction entre eux. Les interactions sont moins importantes entre ces deux environnements et celui des animaux sauvages mais le chevauchement est de plus en plus grand à la suite de l’urbanisation et de la réduction de l’habitat. Les flèches (tirets) correspondent aux différentes voies de transmission, soit directement de la chauve-souris à l’homme, soit faisant intervenir un réservoir intermédiaire. Les flèches (continues) quant à elles, mettent en valeur les différents facteurs pouvant favoriser l’apparition de zoonoses. |
|
Cibles immunologiques en cas de zoonoses avérées Lors d’une infection à AdV, la réponse immunitaire de l’hôte est activée avec l’induction de la production de cytokines et de chimiokines antivirales [55]. Les PRR (pattern recognition receptors) mis en jeu lors d’une infection par les AdV sont partiellement caractérisés. Les réponses dépendent, ou non, des récepteurs de type Toll-like receptors (TLR) et d’autres récepteurs reconnaissant les acides nucléiques [56]. Les cellules dendritiques et les macrophages, outre leur rôle dans la production de molécules inflammatoires et immunomodulatrices, ont une fonction déterminante dans l’élimination du virus et dans les processus de présentation antigénique qui sont essentiels à la sélection et à l’activation des lymphocytes T. Le développement de la réponse immunitaire adaptative conduit à l’induction d’une réponse lymphocytaire T CD8 cytotoxique et à la production d’anticorps spécifiques. Si la présence d’anticorps semble importante pour la prévention des infections et de la propagation du virus, ce sont principalement les lymphocytes T CD4+
helper et CD8+ cytotoxiques qui protègent l’homme des pathologies induites par les AdV. Ainsi, alors que des patients atteints d’un déficit immunitaire touchant les lymphocytes B peuvent contrôler une infection par les AdV, des individus déficients pour les lymphocytes T en sont incapables et développent une pathologie sévère et/ou létale. Chez l’adulte, la réponse immunitaire dépendant des lymphocytes T est fortement ubiquitaire et majoritairement dirigée contre des épitopes conservés de la capside du virus (par exemple, la région interne de l’hexon). En raison de leur taux de conversion élevé, ces épitopes adénoviraux seraient donc des cibles immunitaires privilégiées lors de l’émergence de zoonoses adénovirales. |
Zoonoses adénovirales : à garder dans le viseur Les zoonoses font partie du risque sanitaire global. La veille épidémiologique permet de mieux surveiller, de s’adapter et de répondre à l’émergence des zoonoses. Le développement de la biologie moléculaire a permis de mieux comprendre l’origine des pathogènes. L’émergence d’AdV au pouvoir zoonotique résultera d’événements complexes faisant intervenir les phénomènes de réarrangements inter-génomes, de réplication du génome viral, le recrutement de protéines cellulaires impliquées dans le contrôle de la réponse immunitaire et dans l’expression des gènes viraux ainsi que dans le transport des ARN messagers viraux permettant l’adaptation du virus à son nouvel hôte. Malgré un risque évidemment plus faible que pour les virus à ARN, pour lesquels le mécanisme de réplication et la structure du génome favorisent les phénomènes de recombinaison génétique, l’émergence de variants pathogènes d’AdV transmissibles entre les espèces n’est pas à exclure. Il ne faut donc pas négliger le caractère pathogène des AdV qui, chez les individus immunodéprimés, peuvent provoquer des maladies sévères et/ou létales dont le risque épidémique est réel. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Smith I, Wang LF. Bats and their virome: an important source of emerging viruses capable of infecting humans . Curr Opin Virol. 2013; ; 3 : :84.–91. 2.
Leroy E, Pourrut X, Gonzalez J. Les chauves-souris, réservoirs du virus : Ebola, le mystère se dissipe . Med Sci (Paris). 2006; ; 22 : :78.–79. 3.
Peiris JSM, Guan Y, Yuen KY. Severe acute respiratory syndrome . Nat Med. 2004; ; 10 : :S88.–S97. 4.
Zaki AM, van Boheemen S, Bestebroer TM, et al. Isolation of a novel coronavirus from a man with pneumonia in Saudi Arabia . N Engl J Med. 2012; ; 367 : :1814.–1820. 5.
Chen EC, Yagi S, Kelly KR, et al. Cross-species transmission of a novel adenovirus associated with a fulminant pneumonia outbreak in a new world monkey colony . PLoS Pathog. 2011; ; 7 : :e1002155.. 6.
Xiang Z, Li Y, Cun A, et al. Chimpanzee adenovirus antibodies in humans, sub-Saharan Africa . Emerg Infect Dis. 2006; ; 12 : :10.–13. 7.
Chiu CY, Yagi S, Lu X, et al. A novel adenovirus species associated with an acute respiratory outbreak in a baboon colony and evidence of coincident human infection . MBio. 2013; ; 4 : :e00084.. 8.
Kohl C, Vidovszky MZ, Mühldorfer K, et al. Genome analysis of bat adenovirus 2: indications of interspecies transmission . J Virol. 2012; ; 86 : :1888.–1892. 9.
Jánoska M, Vidovszky M, Molnár V, et al. Novel adenoviruses and herpesviruses detected in bats . Vet J. 2011; ; 189 : :118.–121. 10.
Wevers D, Metzger S, Babweteera F, et al. Novel adenoviruses in wild primates: a high level of genetic diversity and evidence of zoonotic transmissions . J Virol. 2011; ; 85 : :10774.–10784. 11.
Harrach B, Benko M, Both G. Family Adenoviridae . In: Virus taxonomy . Oxford: : Elsevier; , 2011 : :125.–141. 12.
Leen AM, Rooney CM. Adenovirus as an emerging pathogen in immunocompromised patients . Br J Haematol. 2005; ; 128 : :135.–144. 13.
Green M, Piña M, Kimes R. Adenovirus DNA. I. Molecular weight and conformation . Proc Natl Acad Sci. USA 1967;; 57 : :1302.–1309. 14.
Seiradake E, Henaff D, Wodrich H, et al. The cell adhesion molecule CAR and sialic acid on human erythrocytes influence adenovirus in vivo biodistribution . PLoS Pathog. 2009; ; 5 : :e1000277.. 15.
Roelvink P, Lizonova A, Lee J. The coxsackievirus-adenovirus receptor protein can function as a cellular attachment protein for adenovirus serotypes from subgroups A, C, D, E, and F . J Virol. 1998; ; 72 : :7909.–7915. 16.
Rekosh D, Russell W, Bellet A, et al. Identification of a protein linked to the ends of adenovirus DNA . Cell. 1977; ; 11 : :293.–295. 17.
Arnberg N. Adenovirus receptors: implications for targeting of viral vectors . Trends Pharmacol Sci. 2012; ; 33 : :442.–448. 18.
Bergelson JM, Cunningham JA, Droguett G, et al. Isolation of a common receptor for coxsackie B viruses and adenoviruses 2 and 5 . Science. 1997; ; 275 : :1320.–1323. 19.
Salinas S, Bilsland LG, Henaff D, et al. CAR-associated vesicular transport of an adenovirus in motor neuron axons . PLoS Pathog. 2009; ; 5 : :e1000442.. 20.
Freimuth P, Philipson L, Carson SD. The coxsackievirus and adenovirus receptor . Curr Top Microbiol Immunol. 2008; ; 323 : :67.–87. 21.
Chrétien I, Marcuz A. CTX, a Xenopus thymocyte receptor, defines a molecular family conserved throughout vertebrates . Eur J Immunol. 1998; ; 28 : :4094.–4104. 22.
Waddington SN, McVey JH, Bhella D, et al. Adenovirus serotype 5 hexon mediates liver gene transfer . Cell. 2008; ; 132 : :397.–409. 23.
Kremer EJ. Mutagenesis of hexon FX hepatic tropism . Blood. 2009; ; 114 : :929.–930. 24.
Kuhn JH, Li W, Choe H, et al. Angiotensin-converting enzyme 2: a functional receptor for SARS coronavirus . Cell Mol Life Sci. 2004; ; 61 : :2738.–2743. 25.
Raj VS, Mou H, Smits SL, et al. Dipeptidyl peptidase 4 is a functional receptor for the emerging human coronavirus-EMC . Nature. 2013; ; 495 : :251.–254. 26.
Soudais C, Boutin S, Hong S. Canine adenovirus type 2 attachment and internalization: coxsackievirus-adenovirus receptors, alternative receptors, and an RGD-independent pathway . J Virol. 2000; ; 74 : :10639.–10649. 27.
Bangari DS, Mittal SK. Development of nonhuman adenoviruses as vaccine vectors . Vaccine. 2006; ; 24 : :849.–862. 28.
Seth P. Adenoviruses: basic biology to gene therapy . Austin, TX: : R.G. Landes Company; , 1999 : :314 p.. 29.
Weinmann R, Raskas HJ, Roeder RG. Role of DNA-dependent RNA polymerases II and III in transcription of the adenovirus genome late in productive infection . Proc Natl Acad Sci USA. 1974; ; 71 : :3426.–3439. 30.
Nevins J. Regulation of early adenovirus gene expression . Microbiol Rev. 1987; ; 51 : :419.–430. 31.
Jones N, Shenk T. An adenovirus type 5 early gene function regulates expression of other early viral genes . Proc Natl Acad Sci USA. 1979; ; 76 : :3665.–3669. 32.
Hoeben RC, Uil TG. Adenovirus DNA replication . Cold Spring Harb Perspect Biol. 2013; ; 5 : :a013003.. 33.
Ginsberg HS, Lundholm-Beauchamp U, Horswood RL, et al. Role of early region 3 (E3) in pathogenesis of adenovirus disease . Proc Natl Acad Sci USA. 1989; ; 86 : :3823.–3827. 34.
Bridge E, Medghalchi S, Ubol S, et al. Adenovirus early region 4 and DNA synthesis . Virology. 1993; ; 193 : :794.–801. 35.
Carvalho T, Seeler JS, Öhman K, et al. Targeting of adenovirus E1A and E4-ORF3 proteins to nuclear matrix-associated PML bodies . J Cell Biol. 1995; ; 131 : :45.–56. 36.
Evans JD, Hearing P. Distinct roles of the adenovirus E4 ORF3 protein in viral DNA replication and inhibition of genome concatenation . J Virol. 2003; ; 77 : :5295.–5304. 37.
Toth M, Doerfler W, Shenk T. Adenovirus DNA replication facilitates binding of the MLTF/USF transcription factor to the viral major late promoter within infected cells . Nucleic Acids Res. 1992; ; 20 : :5143.–5148. 38.
Hösel M, Schröer J, Webb D, et al. Cellular and early viral factors in the interaction of adenovirus type 12 with hamster cells: the abortive response . Virus Res. 2001; ; 81 : :1.–16. 39.
Armentero M, Horwitz M, Mermod N. Targeting of DNA polymerase to the adenovirus origin of DNA replication by interaction with nuclear factor I . Proc Natl Acad Sci USA. 1994; ; 91 : :11537.–11541. 40.
Lau SKP, Li KSM, Tsang AKL, et al. Recent transmission of a novel alphacoronavirus, bat coronavirus HKU10, from Leschenault’s rousettes to pomona leaf-nosed bats: first evidence of interspecies transmission of coronavirus between bats of different suborders . J Virol. 2012; ; 86 : :11906.–11918. 41.
Engelmann I, Madisch I, Pommer H, et al. An outbreak of epidemic keratoconjunctivitis caused by a new intermediate adenovirus 22/H8 identified by molecular typing . Clin Infect Dis. 2006; ; 43 : :e64.–e66. 42.
Walsh MP, Chintakuntlawar A, Robinson CM, et al. Evidence of molecular evolution driven by recombination events influencing tropism in a novel human adenovirus that causes epidemic keratoconjunctivitis . PLoS One. 2009; ; 4 : :e5635.. 43.
Gonzalez G, Koyanagi KO, Aoki K, et al. Intertypic modular exchanges of genomic segments by homologous recombination at universally conserved segments in human adenovirus species D . Gene. 2014; ; 547 : :10.–17. 44.
Lukashev AN, Ivanova OE, Eremeeva TP, et al. Evidence of frequent recombination among human adenoviruses . J Gen Virol. 2008; ; 89 : :380.–388. 45.
Walsh MP, Seto J, Jones MS, et al. Computational analysis identifies human adenovirus type 55 as a re-emergent acute respiratory disease pathogen . J Clin Microbiol. 2010; ; 48 : :991.–993. 46.
Robinson C, Singh G, Henquell C. Computational analysis and identification of an emergent human adenovirus pathogen implicated in a respiratory fatality . Virology. 2011; ; 409 : :141.–147. 47.
Purkayastha A, Ditty SE, Su J, et al. Genomic and bioinformatics analysis of HAdV-4, a human adenovirus causing acute respiratory disease: implications for gene therapy and vaccine vector development . J Virol. 2005; ; 79 : :2559.–2572. 48.
Dehghan S, Seto J, Liu EB, et al. Computational analysis of four human adenovirus type 4 genomes reveals molecular evolution through two interspecies recombination events . Virology. 2013; ; 443 : :197.–207. 49.
Singh G, Robinson CM, Dehghan S, et al. Homologous recombination in E3 genes of human adenovirus species D . J Virol. 2013; ; 87 : :12481.–12488. 50.
Dehghan S, Seto J, Jones MS, et al. Simian adenovirus type 35 has a recombinant genome comprising human and simian adenovirus sequences, which predicts its potential emergence as a human respiratory pathogen . Virology. 2013; ; 447 : :265.–273. 51.
Li Y, Ge X, Zhang H, et al. Host range, prevalence, and genetic diversity of adenoviruses in bats . J Virol. 2010; ; 84 : :3889.–3897. 52.
Nociari M, Ocheretina O, Schoggins JW, et al. Sensing infection by adenovirus : Toll-like receptor-independent viral DNA recognition signals activation of the interferon regulatory factor 3 master regulator . J Virol. 2007; ; 81 : :4145.–4157. 53.
Hendrickx R, Stichling N, Koelen J, et al. Innate immunity to adenovirus . Hum Gene Ther. 2014; ; 25 : :265.–284. |