| |
| Med Sci (Paris). 32(5): 506–508. doi: 10.1051/medsci/20163205019.Évaluer le risque épidémique à partir de données lacunaires Mathieu Génois,1 Christian Vestergaard,1 and Alain Barrat1* 1Aix Marseille Université, Université de Toulon, CNRS, CPT, UMR 7332, Centre de physique théorique, Campus de Luminy, 13288Marseille Cedex 9, France |
Vignette (Photo © Inserm-Frédérique Koulikoff). La structure des interactions entre personnes est un facteur clé pour comprendre comment une maladie infectieuse se propage au sein d’une population. La modélisation de cette propagation [1] (→) permet ainsi d’évaluer les risques épidémiques. Parmi les interactions entre personnes, les contacts face-à-face, en particulier, déterminent les chemins de propagation des maladies respiratoires telles que la grippe. Depuis quelques années, grâce à de récents progrès techniques, il est possible de mesurer in situ ces interactions. Par exemple, l’infrastructure mise au point par la collaboration SocioPatterns1 utilise des petits capteurs pouvant se détecter mutuellement ; ces capteurs, portés par les personnes participant aux mesures, sont réglés de manière à n’enregistrer que les contacts proches et face-à-face. On obtient ainsi un enregistrement direct et objectif des contacts au sein du contexte considéré (école, lycée, hôpital, immeuble de bureaux, conférence, etc. [2, 3]). La simulation de la propagation d’épidémies à partir de ces données permet ensuite d’améliorer la prédiction du risque épidémique dans ces populations.
(→) Voir la Nouvelle de L. Temime, m/s n° 2, février 2010, page 205
|
Les contacts entre personnes ainsi mesurés sont représentés sous la forme d’un réseau temporel qui rend compte de l’ordre chronologique des contacts [4] : à chaque participant, on associe un nœud du réseau, et à chaque contact enregistré entre deux personnes, on associe un lien entre les deux nœuds qui représentent ces personnes, lien qui n’existe qu’à l’instant de ce contact. On obtient donc un réseau de contacts qui évolue au cours du temps, et qu’on peut utiliser pour simuler la propagation d’une épidémie dans la population concernée. On considère par simplicité des modèles schématiques de maladies infectieuses, tels que le modèle SIR [5]. Dans ce modèle, les individus sains (S) deviennent infectieux (I) avec une certaine probabilité β à chaque contact avec un individu infectieux. Les individus infectieux (I) quant à eux passent à l’état guéri (R, recovered en anglais) avec une certaine probabilité μ à chaque instant, ces deux paramètres dépendant de la maladie considérée. L’épidémie se propage de façon stochastique au gré des contacts. La simulation s’arrête quand l’épidémie est terminée, c’est-à-dire quand il ne reste plus d’individus infectieux dans le réseau. On mesure alors la taille de l’épidémie, correspondant au nombre de personnes ayant été infectées. La simulation est répétée un grand nombre de fois, afin d’obtenir la statistique des tailles d’épidémies et de séparer les cas pour lesquels le risque épidémique est grand (probabilité non négligeable d’avoir une épidémie de grande taille) de ceux où le risque épidémique est faible. |
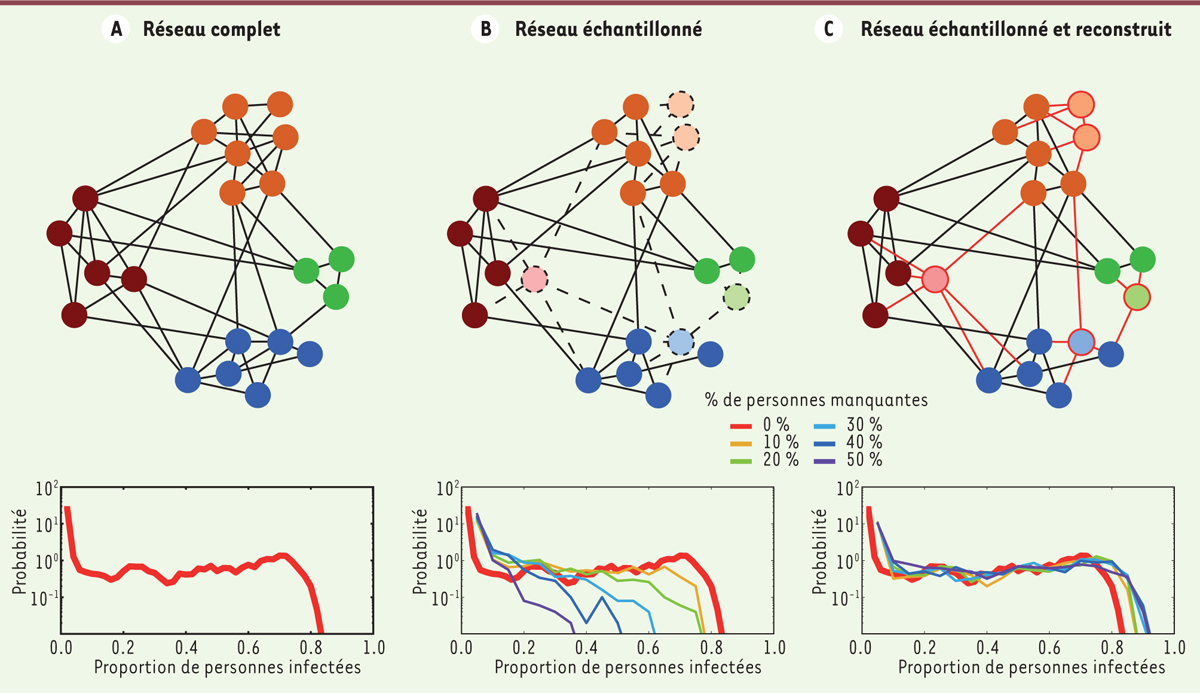
Le problème des données manquantes Les données décrivant les contacts entre personnes sont cruciales pour de telles simulations. Or ces données ne concernent jamais l’intégralité de la population étudiée. En effet, généralement, seule une partie de la population participe à l’étude et les contacts ne sont donc connus que pour une fraction de la population. Ceci a un impact sur les résultats des simulations car, du point de vue de la propagation simulée, les individus absents ont le même rôle que ceux qui seraient vaccinés contre la maladie et par lesquels elle ne peut donc se propager. À cause de la disparition des chemins de propagation correspondants, les simulations effectuées, en considérant seulement une partie du réseau de contacts, sous-estiment systématiquement la fraction de la population atteinte par l’épidémie et donc sous-estiment également le risque épidémique, parfois jusqu’à perdre complètement l’information sur la présence même d’épidémies (Figure 1).
 | Figure 1.
Schémas décrivant la procédure et résultats des simulations numériques. A. On part d’un réseau de contacts entre personnes qu’on considère comme complet. Les différentes couleurs des nœuds correspondent aux différents groupes présents dans la population étudiée (classes/départements/etc.). En réalisant des simulations de propagation de maladies infectieuses sur ce réseau, on obtient la probabilité pour une épidémie d’infecter une certaine proportion de la population. B. Si on retire une certaine fraction des personnes du réseau, et donc les contacts qu’elles ont avec le reste de la population (en pointillés), et qu’on réalise les simulations sur le réseau restant, on observe que, plus la fraction « inconnue » est grande, plus on sous-estime la probabilité d’avoir de grandes épidémies. C. En utilisant la méthode présentée dans [ 7] pour réintroduire cette fraction « inconnue », on peut construire une version alternative mais plausible des contacts manquants (en rouge) à partir des données partielles. On retrouve alors une très bonne évaluation du risque épidémique, même pour des fractions inconnues grandes (jusqu’à 50 %). |
|
Des données partielles mais riches en informations Le fait de n’avoir accès qu’à une partie des contacts modifie certaines caractéristiques du réseau comme la statistique des degrés des individus, le degré d’un individu étant le nombre de personnes distinctes avec qui il/elle a été en contact au moins une fois. Le degré moyen d’un individu dépend en effet linéairement du nombre de personnes présentes dans le réseau [6]. En revanche, d’autres caractéristiques restent stables. C’est le cas de la densité2 du réseau, de la « matrice de contact », qui donne les densités respectives de liens entre les différents groupes qui forment la population (par exemple les classes dans un lycée, les catégories de personnel dans un hôpital, les départements dans des bureaux, etc.), ainsi que des distributions statistiques des caractéristiques temporelles des contacts (durées de contact, temps entre deux contacts successifs, nombres de contacts entre deux personnes). Les propriétés statistiques de ces quantités, même mesurées sur une partie seulement des contacts, restent valables pour le réseau entier. Ces informations jouant un rôle dans les processus de propagation dans une population, on peut donc se baser sur elles pour construire des versions alternatives de la partie manquante du réseau qui aient les mêmes propriétés que le réseau entier du point de vue de la propagation d’épidémies (Figure 1). |
Une méthode robuste pour construire des données de remplacement Dans notre étude [7], nous avons considéré trois contextes distincts : un lycée, dans lequel le réseau de contacts est fortement structuré par les classes et sa dynamique par les horaires des cours ; une conférence, pour laquelle le réseau n’est pas structuré et la dynamique des contacts peu contrainte par des horaires ; un immeuble de bureau, structuré en départements, mais sans emploi du temps fixé. Pour chacune de ces situations, nous avons effectué une procédure de ré-échantillonnage. Pour cela, nous avons éliminé aléatoirement une partie des nœuds afin de générer un réseau partiel et ainsi simuler la non-participation d’une fraction contrôlée de la population. Nous avons ensuite construit une version alternative des contacts manquants en nous basant sur les caractéristiques mesurées sur les réseaux partiels : connaissant le nombre de nœuds manquants, la densité du réseau partiel nous permet de calculer le nombre de liens qu’il convient de rajouter pour lier ces nœuds au réseau partiel. À partir de la matrice de contact mesurée sur le réseau partiel, il est alors possible de répartir ces liens entre les différents groupes de la population. Nous avons ensuite construit, sur chacun de ces liens, une histoire (fictive mais statistiquement correcte) des contacts entre les deux personnes considérées. Pour cela, la technique du bootstrapping
3, a été utilisée. Elle se base sur les listes mesurées des nombres de contacts par lien, des durées de contact (séparées entre liens internes et externes4) et des intervalles entre contacts successifs, constituées à partir des données du réseau partiel. Plus précisément, à partir de ces trois listes respectives, sont tirés au hasard pour chaque lien : un nombre de contacts n, puis n durées de contact et n-1 intervalles entre contacts. L’instant du premier contact est tiré aléatoirement, puis contacts et intervalles entre contacts sont alternés. Nous avons ainsi obtenu une collection de versions échantillonnées puis reconstruites du réseau originel. Chacune de ces versions diffère des contacts ayant réellement eu lieu, mais est plausible et a les mêmes caractéristiques statistiques. Pour chacune de ces versions alternatives, nous avons effectué une simulation de la propagation d’une maladie infectieuse de type SIR et nous avons mesuré la distribution des tailles d’épidémies résultante. Comme montré en
Figure 1C
, nous avons retrouvé une distribution comparable au cas réel (bien que légèrement surestimée), et donnant une bien meilleure estimation du risque épidémique par rapport aux simulations utilisant seulement le réseau partiel. |
Cette étude montre donc, d’une part, que l’utilisation de données incomplètes dans des simulations peut mener à une forte sous-estimation d’un risque, mais, d’autre part, que ces données incomplètes contiennent suffisamment d’information pour construire des données de remplacement réalistes et obtenir une bonne évaluation du risque épidémique. Cette étude s’étant concentrée sur le cas d’un échantillonnage uniforme de la population, c’est-à-dire un cas en quelque sorte « idéal », de futurs travaux seront nécessaires pour considérer d’autres types d’échantillonnage ou l’utilisation d’autres types de données incomplètes comme celles obtenues par des questionnaires. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Temime L. Modélisation de la transmission des infections en service hospitalier : identification de super-propagateurs . Med Sci (Paris). 2010; ; 26 : :205.–208. 2.
Barrat A, Cattuto C, Tozzi AE, et al. Measuring contact patterns with wearable sensors: methods, data characteristics and applications to data-driven simulations of infectious diseases . Clin Microbiol Infect. 2014; ; 20 : :10.–16. 3.
Fournet J, Barrat A. Contact patterns among high school students . PLoS One. 2014; ; 9 : :e107878.. 4.
Holme P, Saramäki J. Temporal networks . Phys Rep. 2012; ; 519 : :97.–125. 5.
Anderson RM, May RM. Infectious diseases of humans: dynamics and control . Oxford: : Oxford University Press; , 1992 : :768. p. 6.
Cohen R, Erez K, ben Avraham D, Havlin S. Resilience of the Internet to random breakdowns . Phys Rev Lett. 2000; ; 85 : :4626.–4628. 7.
Génois M, Vestergaard CL, Cattuto C, Barrat A. Compensating for population sampling in simulations of epidemic spread on temporal contact networks . Nat Commun. 2015; ; 6 : :8860.. |