L’oxygène, sous sa forme dioxygène (O2), représente 20 % des composants de l’atmosphère. Tous les organismes aérobies l’utilisent pour respirer. Bien que l’O2 soit très stable et possède une faible réactivité, des dérivés (appelés reactive oxygen species [ROS] en anglais) peuvent survenir comme produits dérivés du métabolisme. Parmi ces dérivés on trouve notamment l’anion superoxyde (O2°-), le peroxyde d’hydrogène (H2O2) et le radical hydroxyle (HO°). Les ROS peuvent être produits par des rayonnements, via la radiolyse de l’eau par exemple, ou par des composés chimiques tels que l’eau de javel (HOCl). De par leur réactivité, les ROS peuvent altérer les macromolécules de la vie (ADN, protéines, lipides) et, par conséquent, le fonctionnement des systèmes cellulaires [1] (→).
(→) Voir la Synthèse de C. Migdal et M. Serres, m/s n° 4, avril 2011, page 405
C’est pourquoi l’ensemble des organismes vivants se sont dotés de systèmes anti-oxydants, incluant des composés chimiques simples (glutathion, ascorbate, etc.) mais aussi des enzymes (catalases, superoxyde dismutases, peroxydases, etc.) dont l’action vise à piéger ou éliminer les ROS. Un autre type de protection consiste en la réparation des dommages causés par les ROS [2].
La méthionine (Met), soit en tant qu’acide aminé à l’état libre, soit comme résidu d’une chaîne polypeptidique, est une cible privilégiée des ROS qui la convertissent en méthionine sulfoxyde (Met-SO). Les conséquences d’une telle oxydation sont évidemment variables et dépendent du rôle joué par le(s) résidu(s) Met dans la structure ou l’activité de la protéine ciblée. Plusieurs cas d’inactivation de protéines, soit par déstructuration soit par inhibition de leur activité, ont été rapportés. C’est le cas par exemple de la chaperonne1, bactérienne GroEL2 (growth), de la protéine appelée Ffh (fifty-four homolog) impliquée dans la translocation des protéines chez les bactéries, ou encore de la calmoduline, protéine centrale dans la régulation du calcium chez les eucaryotes [3]. Au contraire, on peut observer des cas dans lesquels l’action des ROS est bénéfique : par exemple, pour le régulateur HypT (hypochlorous transcription), l’oxydation de résidus Met permet de déclencher une voie de régulation permettant de lutter contre ce stress toxique [4]. Il existe un troisième cas de figure correspondant à l’exploitation de l’oxydation des résidus pour absorber les ROS et protéger ainsi l’environnement proche de leurs dégâts. La théorie d’une action des Met comme « éponge » contre le stress oxydant a été proposée par Earl Stadtman dans le cas des protéines, postulant que les résidus Met, à l’intérieur d’un polypeptide, protègeraient les acides aminés voisins [5]. Cette théorie peut également être invoquée pour la Met à l’état libre puisque plusieurs rapports ont fait état du rôle bénéfique de l’ajout de l’acide aminé Met pour l’homéostasie cellulaire [6]. En résumé, l’oxydation des Met peut être neutre, délétère dans le cas de résidus Met fonctionnellement importants, ou bénéfique dans leur rôle d’éponge anti-ROS.
Un niveau de sophistication supplémentaire a émergé au cours de l’évolution : il existe, dans la quasi-totalité des organismes vivants, un système enzymatique qui rend l’oxydation des Met en Met-SO réversible. Ainsi, les dommages potentiels, dus à la présence de Met-SO délétère, peuvent être prévenus par réduction des Met-SO en Met. Dans le cadre de l’hypothèse de Stadtman citée ci-dessus, la réduction des Met-SO permettrait de réinitialiser le processus, en d’autres termes d’essorer l’éponge.
Une première enzyme permettant la réduction des Met-SO, appelée méthionine sulfoxyde réductase (MsrA), a été découverte au début des années 1980 [7]. Ce système ne conduit cependant qu’à une réparation partielle des protéines oxydées. C’est au début des années 2000 que nous avons identifié une deuxième enzyme, MsrB. Nous avons en effet montré que l’action conjuguée de MsrA et MsrB était nécessaire pour une réparation totale des protéines oxydées [8]. Le fondement de la coopération entre MsrA et MsrB se trouve dans la chimie de l’oxydation de leurs substrats, Met. Le souffre de la chaîne latérale de Met est un atome asymétrique pouvant s’oxyder sous deux formes (R et S)3, donnant ainsi deux formes distinctes : la Met-S-SO et la Met-R-SO. Or, MsrA et MsrB sont chacune dia-stéréo-spécifiques, c’est-à-dire qu’elles ne réduisent efficacement, respectivement, que les formes -S et -R. L’oxydation des résidus Met en -S-SO ou en -R-SO étant a priori stochastique, chaque chaîne polypeptidique possèdera autant de chacune des deux formes. Ces études ont conduit au concept de système MsrAB. Il s’agit d’un système ubiquitaire retrouvé des bactéries à l’homme, dont l’importance fonctionnelle a été soulignée tant dans le cadre de l’adaptation des bactéries au stress, de la virulence des bactéries pathogènes, de certaines maladies neuro-dégénératives que dans le vieillissement cellulaire. Le système MsrAB est cytoplasmique chez les bactéries, excepté chez Neisseria (bactérie qui regroupe gonocoque et méningocoque). Chez les eucaryotes, plusieurs paralogues4 existent et des représentants sont trouvés dans différents compartiments : cytoplasme, chloroplaste, mitochondrie ou lumière du réticulum endoplasmique [9].
Les bactéries sont entourées d’une enveloppe cellulaire dans laquelle, chez les Gram-négatifs, existe un espace inter-membranaire appelé périplasme. L’enveloppe a évidemment un rôle de protection mais elle est aussi un lieu d’échange et de structuration de la cellule. C’est aussi un des sites les plus exposés aux toxiques environnementaux, y compris les ROS produits par les autres habitants du même biotope, hôte infecté ou autres bactéries. Dans ce contexte, il était tout à fait surprenant qu’aucun système MsrAB n’ait été identifié dans ce compartiment dont les protéines sont particulièrement exposées à l’oxydation. C’est en utilisant le modèle Escherichia coli et une approche génétique dite de recherche de mutations suppressives « gain de fonction », que nous avons identifié un nouveau système de type Msr ayant des propriétés génétiques et enzymologiques totalement nouvelles et localisé dans le périplasme des bactéries à Gram-négatif [10].
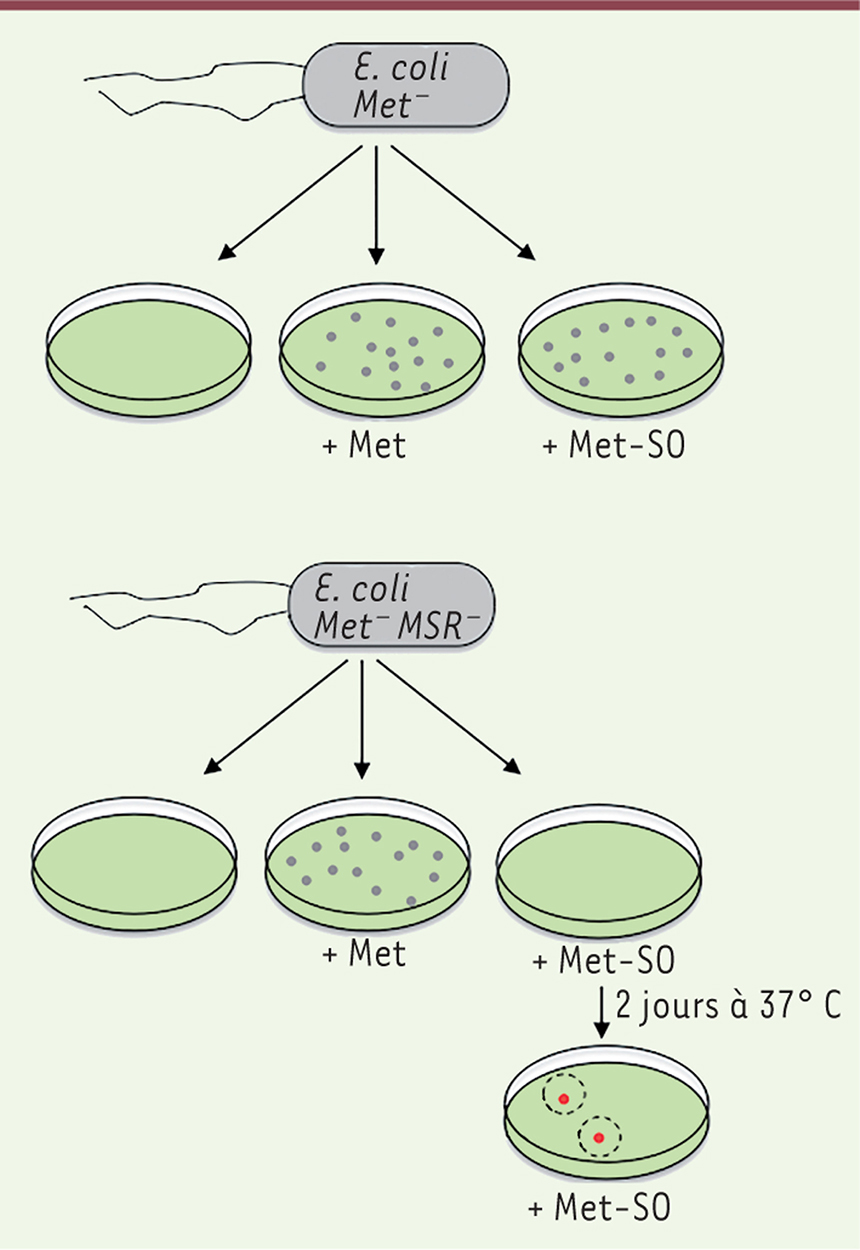
La réduction des Met-SO libres en Met par le système MsrAB permet de compenser une éventuelle carence en Met. C’est sur cette propriété que nous avons basé notre sélection génétique. Une souche auxotrophe5, de E. coli ne pouvant pas synthétiser la Met, et ne possédant plus les activités MsrAB, a été ensemencée sur un milieu de croissance contenant un mélange des 2 diastéréoisomères Met-S-SO et Met-R-SO. Quelques jours après incubation à 37°C, quelques rares colonies sont apparues, semblant donc avoir regagné la capacité à utiliser le Met-SO (Figure 1). Par cartographie, nous avons alors localisé la mutation conférant cet avantage sélectif. Il s’agit de l’insertion d’un élément mobile dans un gène régulateur (yedV), dont l’inactivation conduit à l’expression d’un opéron6 (yedYZ) situé immédiatement à côté. Bien qu’ils aient été l’objet de plusieurs études biochimiques et structurales, YedY et YedZ n’avaient, jusqu’alors, aucune fonction physiologique attribuée. Nous avons renommé YedY et YedZ, respectivement MsrP et MsrQ.
| Figure 1.
Isolement de révertants phénotypiques Met-SO+ par mutations suppressives. Des souches d’E. coli (met msrA msrB msrC bisC) sont ensemencées sur des boîtes de Petri contenant un milieu de culture en présence de Met ou Met-SO. La souche Met- (auxotrophe pour la méthionine) pousse dans les deux conditions. La souche Met- et MSR- (incapable de transformer la Met-SO en Met) ne pousse qu’en présence de Met. Deux jours après incubation à 37°C, des colonies révertantes capables d’utiliser la Met-SO comme source de Met apparaissent : leur analyse génétique montre qu’elles contiennent une insertion dans un gène régulateur qui permet de réverser le phénotype Met-SO- vers le phénotype sauvage. Met : méthionine ; Met-SO : méthionine sulfoxyde ; MSR : méthionine sulfoxyde réductase. |
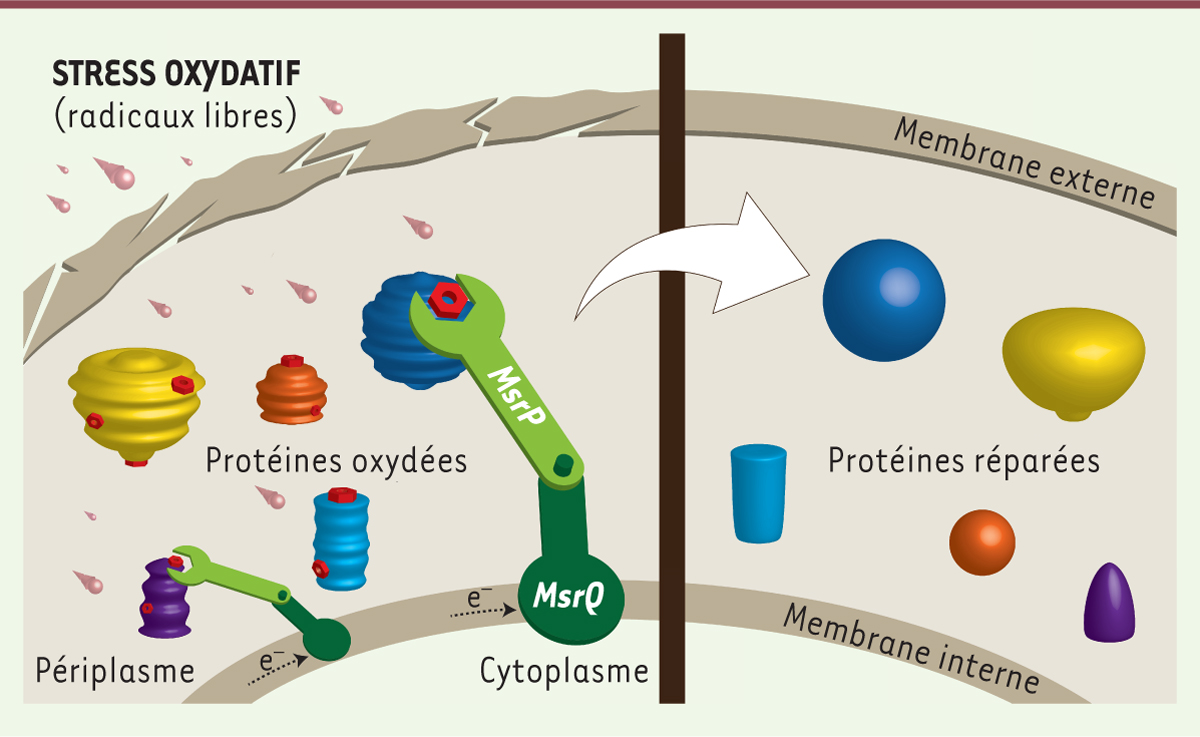
Le système MsrPQ présente plusieurs différences avec le système MsrAB. Ce système (1) est constitué de protéines à cofacteurs, molybdène7, dans le cas de MsrP, et hème8 dans le cas de MsrQ ; (2) est connecté à la chaîne respiratoire dont il détourne les électrons pour accomplir son cycle catalytique ; (3) a la capacité de réduire efficacement les 2 formes diastéréoisomériques –R-SO et -S-SO ; et enfin (4) est localisé dans le périplasme (Figure 2). Par une approche protéomique, nous avons identifié plus de 20 protéines candidates qui pourraient être sous la surveillance de MsrPQ. En prenant comme modèle SurA (survival), une protéine chaperonne dont le rôle est crucial dans le maintien de l’intégrité de l’enveloppe cellulaire bactérienne, nous avons montré que cette protéine peut être inactivée par oxydation de ses résidus Met, que cela conduit à une hyper-susceptibilité des bactéries à des ROS mais aussi aux radicaux chlorines contenus dans l’eau de javel, et que l’intervention de MsrPQ était nécessaire pour réparer les dommages.
| Figure 2.
Modèle d’action du système MsrPQ. L’enveloppe cellulaire d’une bactérie à Gram négatif, comprenant la membrane externe, l’espace intermembranaire (périplasme) et la membrane interne, est représentée. À gauche, la présence de radicaux libres induit un stress oxydatif qui endommage les protéines de l’enveloppe et, en particulier, les résidus méthionine qui sont transformés en méthionine sulfoxyde (représentés sous forme d’écrou rouge). Le système MsrPQ, ancré à la membrane interne et alimenté par les électrons de la chaîne respiratoire, réduit les méthionines sulfoxyde et répare ainsi les protéines (à droite). |
MsrPQ est ainsi un nouveau système de réparation des dommages causés par divers stress radicalaires qui est situé dans l’enveloppe cellulaire bactérienne et qui présente des propriétés totalement différentes du système orthodoxe MsrAB. De fait, une précédente étude réalisée chez la bactérie pathogène Campylobacter avait identifié le gène codant le système MsrPQ comme un facteur de virulence important mais de fonction inconnue [11]. Notre étude a donc clarifié cette fonction inconnue et ce système étant spécifique aux bactéries, fait émerger MsrPQ comme une potentielle nouvelle cible anti-bactérienne.