| |
| Med Sci (Paris). 32(11): 952–960. doi: 10.1051/medsci/20163211010.Microbiotes et maladies métaboliques De nouveaux concepts pour de nouvelles stratégies thérapeutiques Rémy Burcelin,1* Simon Nicolas,1 and Vincent Blasco-Baque1 1Institut national de la santé et de la recherche médicale (Inserm), Toulouse, France. Université Paul Sabatier (UPS), unité mixte de recherche (UMR) 1048, Institut des maladies métaboliques et cardiovasculaires (I2MC), équipe 2 (facteurs intestinaux de risque : diabète, insuffisance cardiaque et dyslipidémie), hôpital Rangueil, F-31432Toulouse Cedex 4, France |

Il aura fallu environ un siècle et demi à la communauté scientifique, depuis Louis Pasteur, pour décrypter par séquençage et répertorier dans un catalogue l’ensemble des gènes des bactéries qui sont présentes à la surface de nos épithéliums, regroupé sous le terme « métagénome » [69] (→). En 2014, près de 10 millions de gènes bactériens ont été identifiés à partir d’échantillons fécaux provenant de 1 268 patients originaires de trois continents différents [1]. Les bactéries qui nous colonisent seraient donc aussi, ou plus, nombreuses que nos cellules [2]. Parallèlement, la comparaison de cohortes issues de différentes sources (chinoise et danoise) a mis en évidence une répartition de ces gènes dépendant de l’origine des individus examinés. Ces gènes entrent dans la composition du génome de milliers d’espèces bactériennes dont la plupart ne sont actuellement pas encore identifiées. Aujourd’hui, les données concernant ces bactéries et ces gènes, recueillies au niveau international, sont collectées par un consortium (international microbiome project) afin de pouvoir classer ce métagénome en fonction des groupes d’individus étudiés et des maladies qu’ils présentent [66] (→).
(→) Voir la Synthèse de J. Weissenbach et A. Sghir, page 937 de ce numéro
(→) Voir la Synthèse de H.M. Blottière et J. Doré, page 944 de ce numéro
|
Les maladies métaboliques revisitées grâce à la métagénomique Une des avancées majeures de cette dernière décennie concerne les maladies métaboliques, notamment le diabète de type 2, l’obésité, et la stéatose hépatique non-alcoolique. S’il était intuitif jusqu’ici de concevoir que ces pathologies, toutes fortement liées à un déséquilibre nutritionnel, pouvaient être associées à un changement de l’écologie microbienne intestinale et buccale (les bactéries se nourrissant de ce que nous mangeons), ce n’est que récemment, grâce à l’avènement du séquençage à ultra-haut débit, que des associations entre pathologies métaboliques et modifications du microbiote ont pu être établies (Figure 1). Ainsi, les travaux du groupe d’O. Pedersen au Danemark [3, 4] ont montré sur une cohorte de plus de 300 patients, qu’il existait une signature caractéristique du microbiote intestinal du patient obèse et, dans une certaine mesure, du diabétique de type 2. En effet, on retrouve dans l’intestin d’une majorité de patients fortement obèses un nombre réduit de gènes de bactéries (moins de 500 000) [4], ce qui a introduit le concept de dysbiose intestinale. Parallèlement, la notion d’entérotypes a été avancée, notamment dans le contexte de la mise en place du microbiote qui a lieu au moment de la naissance [5], ou dans le cas de certaines maladies métaboliques [6] ou cardiovasculaires [7]. Il est également possible de définir une signature bactérienne, ou un entérotype bactérien, spécifique d’un sous-groupe de patients tous atteints de la même maladie. Ainsi, un entérotype donné, défini à partir des observations réalisées sur des individus ayant des microbiotes et une pathologie similaires, pourrait permettre de regrouper des patients non plus uniquement en fonction de leur phénotype, mais également sur la base de leur métagénome bactérien, identifié par l’analyse de leurs selles. À ce jour, l’identification de groupes bactériens (appelés taxons) qui constitueraient une signature du diabète de type 2 et de l’obésité n’est cependant pas sans ambiguïté. De façon générale, par rapport à des sujets apparemment sains, une restriction de la diversité microbienne1, est observée chez les sujets obèses. En effet, chez les sujets présentant un indice de masse corporelle (IMC) supérieur à 45, on observe une réduction de 30 à 70 % du nombre des gènes bactériens [4], réduction qui peut être modulée par un régime riche en fibres alimentaires. Il permet en effet d’augmenter la diversité en espèces microbiennes et, en parallèle, de diminuer le surpoids et, globalement, d’améliorer la santé [8], comme observé dans une étude restreinte de 50 patients obèses. Cette notion de réduction de diversité génétique bactérienne est donc à opposer à celle de l’implication d’une seule, ou de plusieurs, entités bactériennes particulières dans le développement de la maladie. Cependant, la réduction de diversité du microbiote intestinal n’est pas spécifique des maladies métaboliques. Elle est en effet également retrouvée dans d’autres pathologies dont les pathologies inflammatoires comme la maladie de Crohn, une maladie inflammatoire chronique du tube digestif (MICI) [9–11]. Ces spécificités de microbiote intestinal ne peuvent donc être utilisées de manière univoque pour le diagnostic d’une maladie métabolique. De plus, dans une proportion importante de patients obèses (environ 30 à 40 %), une telle altération du microbiote intestinal n’est pas observée, ce qui pourrait être lié au caractère multigénique et multi-étiologique de la maladie métabolique.
 | Figure 1.
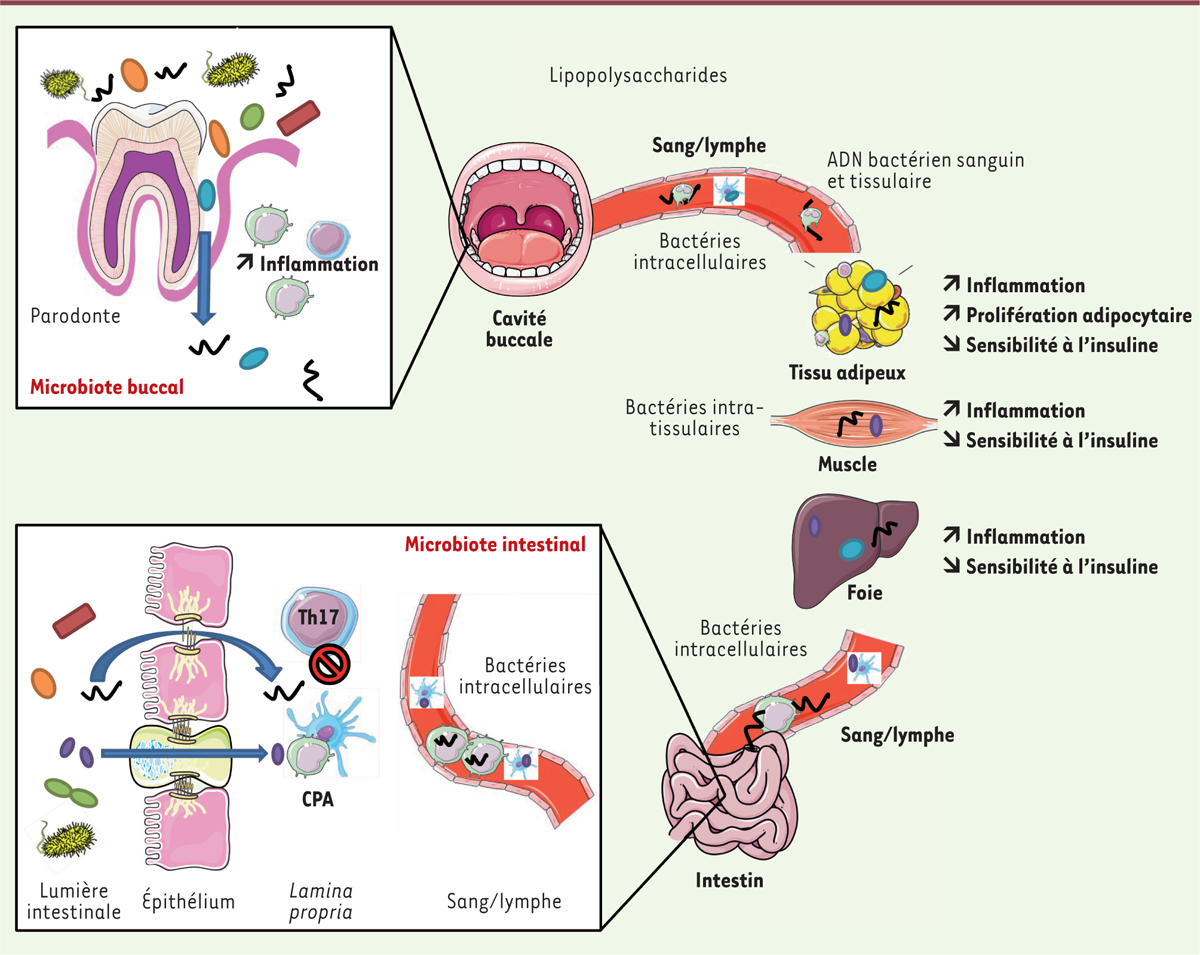
Les microbiotes épithéliaux, intracellulaires et intra-tissulaires. Le microbiote tapisse l’ensemble de nos épithéliums avec une diversité et une quantité qui dépend de la région concernée. Le système immunitaire, les cellules présentatrices d’antigènes (CPA), les lymphocytes, les polynucléaires, et les cellules lymphoïdes innées protègent notre organisme contre l’invasion par ces bactéries. Cependant, il existe une translocation physiologique de bactéries des épithéliums vers le sang, la lymphe et les tissus. Ce microbiote intracellulaire, localisé dans les cellules immunitaires circulantes est conduit jusqu’aux tissus. Il contribuerait non seulement à l’homéostasie des tissus mais également, en cas de dysbiose tissulaire, au développement de certaines pathologies. |
|
Un microbiote ou… des microbiotes ? Actuellement, une restriction importante de la stratégie de classification provient de ce que le microbiote étudié chez l’homme concerne essentiellement les fèces. Or, le microbiote fécal n’est pas représentatif de l’ensemble du microbiote intestinal. En effet, les proportions relatives de chaque bactérie le long du tractus digestif, de la bouche à l’anus, varient en fonction du pH, du gradient d’oxygène [12], de la teneur en acides biliaires et des aliments ingérés au cours des jours qui précèdent les analyses. Dans l’intestin grêle, le microbiote est composé de nombreuses espèces aérobies/anaérobies facultatives, telles les Protéobactéries, qui libèrent des lipopolysaccharides (LPS). Ces constituants de la paroi des bactéries à Gram négatif stimulent la production de cytokines inflammatoires par les cellules immunitaires, endothéliales [13] et pré-adipocytaires [14] via le TLR4 (toll-like receptor 4), l’un des récepteurs majeurs de l’immunité innée [15]. De plus, l’intestin grêle est davantage perméable aux fragments bactériens et aux bactéries que ne l’est le côlon [16]. Le mucus qui recouvre les épithéliums est en effet moins épais que dans le côlon [17–19]. Dans ce dernier résident essentiellement des familles de bactéries strictement anaérobies, telles que les Bifidobacteriaceae et les Clostridiaceae. Celles-ci ne possèdent pas de LPS mais un autre composant pariétal, le peptidoglycane, qui induit également une inflammation mais en activant des récepteurs différents, le TLR2 ou les NLR (Nod-like receptor) de type 1 ou 2 [20, 21]. La cavité buccale est également un réservoir bactérien. Bien que ne représentant que 1 % du contenu bactérien que l’on peut retrouver dans le côlon, elle est constituée de multiples niches écologiques avec une diversité de plus de 350 espèces de bactéries [22]. De nombreuses études épidémiologiques montrent une corrélation entre des maladies systémiques et les infections orales. C’est le cas de la maladie parodontale [23] qui touche plus de 65 % de la population âgée de plus de 50 ans [24]. Cette pathologie est définie comme une infection chronique d’origine bactérienne reposant sur la formation, à l’âge adulte, d’un biofilm composé principalement de bactéries à Gram négatif. Ce biofilm dysbiotique est constitué d’exopolysaccharides2. Il adhère à la dent et entraîne une réaction inflammatoire des tissus de soutien que sont les os et les gencives [25]. La composante microbiologique indispensable à sa formation n’est cependant pas suffisante au développement de la maladie parodontale. En effet, l’inflammation in situ, le stress, les désordres endocriniens ou le tabac constituent autant de facteurs qui sont également impliqués [26]. Il n’existe pas en fait de bactéries pathogènes spécifiques associées à cette pathologie. Porphyromonas gingivalis, Prevotella intermedia, Tannarella forsythia, etc. [27], appelées également parodontopathogènes ou « complexe rouge », sont en effet présentes sous la forme commensale (elles sont en équilibre avec l’hôte, n’induisant pas de pathologie). C’est une rupture de l’équilibre entre la proportion de ces bactéries et les capacités de défense du système immunitaire qui signe le développement de la maladie parodontale [28]. En l’absence de traitement anti-bactérien, le biofilm (ou plaque dentaire ou tartre) s’accumule donc, engendrant une réaction inflammatoire qui va détruire progressivement les tissus parodontaux jusqu’à causer la perte de l’enracinement des dents. La dysbiose microbienne engendrée qui induit la maladie parodontale représente en fait un nouveau facteur de risque et d’aggravation des maladies métaboliques [29], voire une des complications du diabète. Le traitement de la maladie parodontale permet ainsi de réduire le taux d’hémoglobine glyquée3 de 0,4 % chez les patients atteints de diabète de type 2 et, également, l’incidence des maladies cardiovasculaires associées [30]. Inversement, un état inflammatoire latent dû à la libération de composés comme les LPS résultant de ce microbiote parodontal chronique pourrait favoriser l’aggravation, ou l’émergence, d’une insulino-résistance. Il existe en fait une relation bidirectionnelle entre dysbiose de la cavité buccale et maladies métaboliques. En effet, l’hyperglycémie, la dyslipidémie ou l’inflammation systémique chronique, sont susceptibles de modifier le pH et l’humidité au niveau de la cavité buccale et donc la composition bactérienne de la plaque dentaire, entraînant ainsi une pression de sélection sur les microbiotes et le développement de la dysbiose. La mauvaise hygiène buccale ou les conditions environnementales facilitent ainsi le développement d’une dysbiose accompagnant possiblement les maladies métaboliques [31]. |
Microbiote et thérapie de l’obésité et du diabète de type 2 Un des moyens les plus efficaces pour contrôler l’obésité morbide est la chirurgie bariatrique4
(Figure 2). Parmi les différentes stratégies, le by-pass gastrique consiste à diminuer de 90 % la taille de l’estomac et à dévier le bol alimentaire directement vers le jéjunum, un segment intestinal distal de 1,5 mètre. À la suite de cette chirurgie, le duodénum et le jéjunum proximal ne reçoivent plus de nutriments. Ces derniers entrent donc directement au contact des microbiotes présents dans le jéjunum distal et l’iléon avec pour conséquence une modification drastique du métabolisme des bactéries qui y sont présentes [32, 33]. Ces changements infligés au microbiote, révélés par l’analyse du microbiote fécal, pourraient, au moins en partie, expliquer l’efficacité thérapeutique exceptionnelle de cette procédure chirurgicale sur l’obésité et sur les perturbations trouvant leur origine dans les maladies métaboliques qui lui sont associées. Un lien causal a pu être démontré dans le modèle murin. En colonisant des souris axéniques (dépourvues de bactéries et vivant en isolateur) avec un microbiote provenant de patients diabétiques et obèses et ayant subi ou non une chirurgie bariatrique, il a pu être montré que la prise de poids des souris, colonisées par le microbiote de patients opérés, était en effet inférieure à celle que l’on observe chez les souris traitées avec un microbiote provenant de patients non opérés [33]. Mais d’autres fonctions de l’intestin, telles que la sécrétion d’incrétines [34, 67] (→), la production intestinale de glucose [35] et la production d’acides biliaires [36, 37], pourraient aussi expliquer l’impact majeur de la chirurgie bariatrique sur le contrôle des maladies métaboliques. Ces fonctions étant également sous le contrôle du microbiote intestinal, elles pourraient participer au contrôle de la glycémie, du poids ou de la stéatose hépatique.
 | Figure 2.
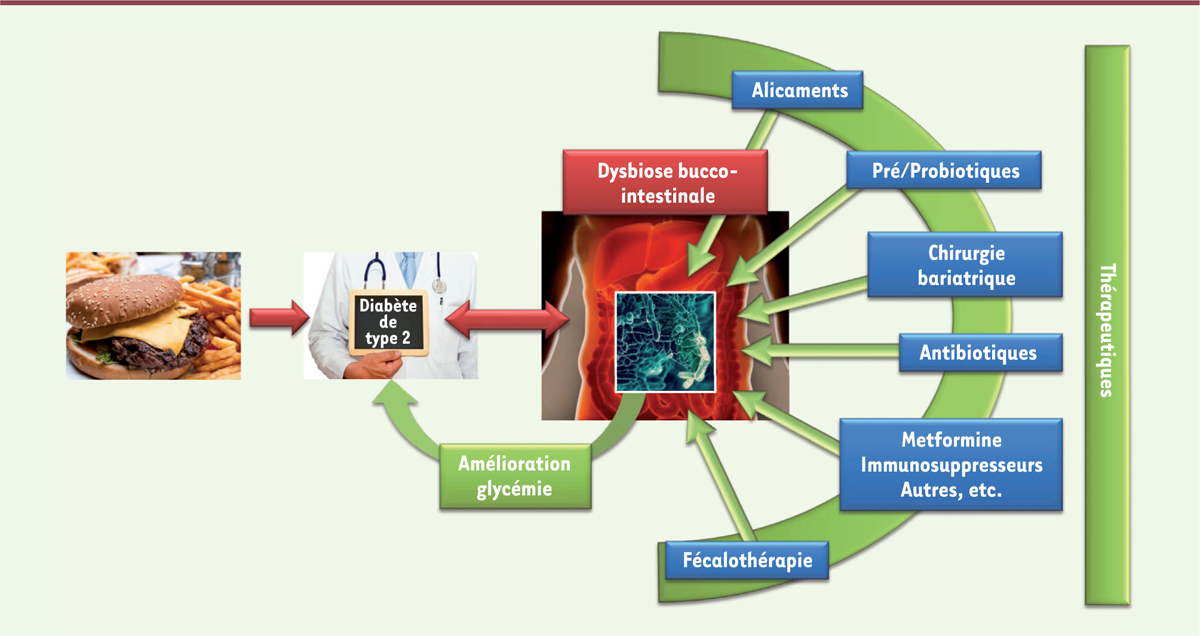
Diabète de type 2 et dysbiose du microbiote bucco-intestinal : une nouvelle cible thérapeutique dans la prise en charge personnalisée des patients. L’alimentation riche est un facteur de risque du développement du diabète de type 2. Les patients diabétiques présentent une dysbiose du microbiote intestinal et buccal avec une modification du rapport entre Bacteroides et Firmicutes. Cette dysbiose peut être ciblée et modifiée par différents agents thérapeutiques comme les alicaments, les pré- et probiotiques, les antibiotiques, la metformine ou la fécalothérapie. Ces thérapies permettent de moduler la dysbiose et d’améliorer de façon significative les paramètres glycémiques des patients diabétiques. |
(→) Voir la Synthèse de B. Thorens, m/s n° 8-9, août-septembre 2003, page 860
Une autre approche, nutritionnelle et donc moins drastique, consiste à administrer en prévention ou en accompagnement thérapeutique, des prébiotiques, des probiotiques ou des combinaisons des deux appelées symbiotiques. Les prébiotiques sont des extraits de fibres alimentaires. Ils sont de type inuline (polyfructose), polydextrose (polyglucose), polygalactose ou encore polysaccharose. Ils ne sont pas absorbés par l’hôte mais sont digérés et fermentés par certaines bactéries constitutives du microbiote intestinal. Les produits de cette fermentation qui sont, par exemple, les acides gras à chaîne courte (acétate, propionate, butyrate et leurs dérivés) interagissent avec les cellules intestinales, notamment les cellules épithéliales, immunitaires et nerveuses. Ils stimulent leurs fonctions endocrines en induisant la sécrétion de défensines, des peptides naturels ayant un rôle antimicrobien, la sécrétion d’hormones, comme le GLP1 (glucagon like peptide one) ou le NPY (neuropeptide Y) qui activent le système nerveux entérique et induisent la maturation de certains lymphocytes (T régulateurs, Th[T helper]17 et Th22) et participent à la défense immunitaire intestinale. Les produits de leur métabolisme interagissent non seulement avec l’hôte mais également avec l’ensemble de l’écologie microbienne, pouvant influer sur la dysbiose intestinale [38–40]. Les probiotiques, quant à eux, sont constitués de micro-organismes vivants (levures, bactéries) normalement inoffensifs. Bien que pouvant changer l’écologie du milieu dans lequel ils se trouvent, ils modifient peu le métagénome. Ils interagissent directement avec les cellules de l’hôte et contribuent ainsi à renforcer ses défenses et à moduler les fonctions des cellules intestinales. Certains traitements antidiabétiques, comme la metformine utilisée en première ligne thérapeutique pharmacologique dans le diabète de type 2, agissent sur le microbiote intestinal [41]. En effet, la metformine est métabolisée au niveau de l’intestin par des bactéries comme Escherichia coli qui produisent des dérivés foliques capables d’interagir avec les cellules de l’hôte [42]. Les fonctions biologiques engendrées par ces dérivés métaboliques bactériens ne sont pas clairement établies mais pourraient impliquer le stress oxydant et les défenses intestinales. Ainsi, les effets thérapeutiques avérés de la metformine sur la glycémie pourraient dépendre de l’impact induit sur le microbiote. À noter que deux tiers des patients diabétiques traités par des doses importantes de metformine présentent des diarrhées. La croissance de certaines bactéries que l’on observe pourrait ainsi participer à l’effet thérapeutique que l’on obtient avec la metformine. Il est donc d’ores et déjà concevable que les patients présentant une dysbiose puissent être identifiés sur la base de la description de leur microbiote respectif (intestinal et buccal) et de la dysbiose qu’ils présentent, et donc être traités par des approches qui prennent en compte la modification de ce microbiote ainsi que leur sensibilité à cette dysbiose. Il est néanmoins difficile d’envisager de séquencer à bas coût le microbiote intestinal et buccal de tout patient présentant une maladie métabolique. Il est donc nécessaire d’identifier des biomarqueurs simples, et de coût réduit, qui soient révélateurs d’une telle dysbiose. Une signature précoce du diabète de type 2, notamment une diminution de la fréquence d’Akkermansia muciniphila, de nombreuses espèces de Bactériodes et Eubactérium, pour n’en citer que quelques unes, a, par exemple, pu être mise en évidence à partir du microbiote fécal provenant de jumeaux homozygotes d’origine coréenne [43]. Depuis quelques années, une approche permettant de normaliser le microbiote au niveau intestinal a émergé. Il s’agit de la thérapie fécale. Cette méthode consiste en l’introduction d’un microbiote fécal d’un individu sain dans l’intestin d’un individu malade afin de remédier aux dysbioses consécutives à la pathologie. Cette thérapie semble très efficace pour le traitement d’infections intestinales ayant pour origine la bactérie Clostridium difficile [68] (→).
(→) Voir la Synthèse de J.C. Lagier et D. Raoult, page 991 de ce numéro
Néanmoins, la notion de donneur sain reste encore mal définie – il est en effet essentiel d’établir à partir de larges banques de données, une écologie microbienne qui puisse être associée à un « bon état de santé ». L’Agence nationale de sécurité du médicament et des produits de santé (ANSM) a publié des recommandations et un essai clinique de thérapie fécale, conduit par une équipe hollandaise, a été réalisé chez des patients diabétiques de type 2. Le transfert du microbiote intestinal de personnes non diabétiques, provenant de l’entourage des patients, a ainsi permis d’augmenter leur sensibilité à l’insuline et de diminuer significativement leur hémoglobine glyquée. Cet effet observé, a pu être associé à une profonde modification du microbiote intestinal des patients traités. Il se caractérise, entre autres, par une augmentation de bactéries qui sont productrices d’acides gras à chaîne courte [44]. Néanmoins, l’efficacité de ce type de traitement est courte (3-6 mois). L’implication d’une réaction immunitaire associée à ce type de transplantation fécale n’est pas prouvée à ce jour. Il pourrait cependant être envisagé d’effectuer des greffes successives afin d’« éduquer » le système immunitaire de l’hôte et ainsi favoriser la greffe d’un microbiote qui serait non diabétogène. L’ensemble de ces questions est en cours d’étude. Dans cette dualité fonctionnelle qui existe entre microbiote et hôte, les deux protagonistes doivent être considérés afin de pouvoir établir une stratégie thérapeutique efficace et adaptée à chaque individu. Les fonctions de fermentation et la production d’acides gras à chaîne courte, ainsi que d’autres fonctions bactériennes potentiellement impliquées dans le métabolisme des acides biliaires, la production d’acide folique, de vitamines, le métabolisme du fer, les nitrosylations et autres sulfatations, sont étudiées actuellement afin d’identifier un lien causal possible et donc d’en dériver des stratégies thérapeutiques. Les fonctions de l’hôte affectées par la dysbiose et qui pourraient être contrôlées par ces stratégies, doivent également être étudiées afin d’agir efficacement en prévention et pour le traitement des maladies. Il est également envisageable de cibler des groupes de patients qui présentent des dysbioses s’avérant similaires afin d’adapter plus précisément les stratégies thérapeutiques à chacun des déséquilibres que l’on observe pour le microbiote. Cette notion de « biomarqueur compagnon » et de médecine personnalisée s’adapte particulièrement aux maladies métaboliques dont la diversité est grande tant par la dysbiose intestinale que par les mécanismes de l’hôte qui sont affectés. |
Le versant bactérien Le diabète de type 2 et l’obésité sont caractérisés par une inflammation métabolique chronique à bas bruit. Les tissus impliqués dans le contrôle du poids et de la glycémie, comme le foie, le tissu adipeux, le muscle, le pancréas endocrine et le cerveau, sont infiltrés par des cellules immunitaires inflammatoires. La transmission du signal insulinique est alors altérée à la suite de la stimulation, dans ces cellules, de différentes voies de signalisation (impliquant des phosphatases, des kinases, etc.) à l’origine de la production de cytokines qui induisent une résistance à l’insuline au niveau du muscle et du foie. Ces cytokines peuvent également favoriser une désensibilisation au glucose des cellules beta-pancréatiques pour la sécrétion d’insuline ou le contrôle de la prise alimentaire par le cerveau. Les mécanismes à l’origine de cette activation du système immunitaire tissulaire restent une question majeure. La dysbiose, intestinale et buccale, pourrait être à l’origine de cette inflammation, mais par quels mécanismes ? Nous avons montré qu’un régime riche en graisses, notamment saturées, qui induit un diabète de type 2 et une obésité, génère une dysbiose du microbiote intestinal et buccal. Ceci favoriserait l’absorption de molécules d’origine bactérienne, comme les LPS [ 45, 46]. Les LPS sont empaquetés dans les chylomicrons, des complexes lipides-protéines, puis sont échangés avec les autres lipoprotéines et véhiculés jusqu’aux tissus qui les métabolisent. Une anomalie du transfert et de l’élimination de ces LPS, comme on les observe au cours des maladies métaboliques [ 47], serait ainsi à l’origine d’une sur-activation du système immunitaire au niveau tissulaire. Ils seraient libérés au voisinage de ces tissus où ils induiraient une réaction inflammatoire. Ainsi, l’infiltration des cellules immunitaires et leur activation seraient la conséquence d’une dysbiose intestinale avec pour effet l’absorption de fragments bactériens inflammatoires, comme les LPS et les peptidoglycanes. Dans le sang, des conglomérats des LPS sont pris en charge, en plus des lipoprotéines, par des protéines de transport comme la LPS-binding protein (LBP) et le CD14 soluble (la forme soluble du récepteur des LPS). Alors que les lipoprotéines protègent contre l’inflammation en stockant les LPS et en les éliminant, la LBP les linéarise, les rendant ainsi aptes à se fixer sur le TLR4 et le CD14 membranaire présents à la surface de nombreuses cellules, dont les cellules immunitaires, déclenchant ainsi une réaction inflammatoire dite métabolique. Ceci est aussi le cas des autres composants de paroi bactérienne de type peptidoglycane qui peuvent également induire une telle réaction inflammatoire [ 16, 48]. Le versant hôte Du côté de l’hôte, une augmentation de la perméabilité intestinale permettrait le passage des antigènes et des molécules pro-inflammatoires ayant pour origine les bactéries. Si cette hypothèse reste à démontrer chez l’homme [ 65] ( →), chez la souris, un régime enrichi en graisses saturées modifie la perméabilité intestinale [ 46]. Cela reposerait sur une modification de la localisation des protéines de jonctions serrées, et donc une augmentation de la perméabilité para-cellulaire (entre cellules épithéliales) [ 46, 49]. Une perméabilité trans-cellulaire (au travers des cellules) permettrait également aux fragments bactériens de traverser l’épithélium sans qu’il y ait intervention des jonctions serrées, via les cellules caliciformes, les cellules qui produisent le mucus [ 50]. Dans différentes affections, il existe, en plus du passage de fragments bactériens, une translocation de bactéries commensales vivantes, du tractus gastro-intestinal vers des sites extra-intestinaux comme les ganglions lymphatiques, le foie, la rate, le rein ou la circulation sanguine [ 51– 53]. Plusieurs mécanismes ont été décrits pour expliquer ce passage transépithélial de bactéries. Des phagocytes présents dans la lumière intestinale peuvent en effet capturer les bactéries qui adhèrent à l’épithélium, normalement protégé par une couche de mucus. Les bactéries peuvent également traverser les cellules M de l’épithélium présentes au niveau des plaques de Peyer, un organe lymphoïde secondaire 5. Les bactéries entrent alors directement en contact avec le système immunitaire du côté baso-latéral. Elles sont prises en charge par le système immunitaire inné et dégradées en peptides antigéniques qui seront présentés aux lymphocytes, déclenchant ainsi une réaction immunitaire adaptative spécifique. Au cours du diabète de type 2, la couche de mucus est réduite [ 54]. L’adhérence des bactéries luminales (présentes dans la lumière) sur l’épithélium et leur translocation dans les tissus sont donc favorisées [ 16]. Ce processus, qui est décrit dans de nombreuses maladies inflammatoires [ 55], a été caractérisé pour la première fois chez la souris diabétique [ 56]. Certaines bactéries sont retrouvées sous forme de fragments dans les tissus, d’autres sont transportées vivantes par les phagocytes dans les organes. Ceci a pu être montré dans un modèle murin où des souris diabétiques ont été colonisées par gavage avec des Escherichia coli productrices de beta-lactamase qui leur confère une résistance à l’ampicilline : les rétro-cultures en présence d’ampicilline des tissus de ces souris mettent en effet en évidence la présence de colonies résistantes à l’antibiotique, preuve du passage de la bactérie vivante [ 16]. Le temps de survie des bactéries dans les tissus n’est pas connu. Des fragments d’ADN bactériens sont également retrouvés dans les organes et le sang chez la souris comme chez l’homme [ 57– 59]. Certaines de ces séquences d’ADN sont prédictives de l’apparition du diabète de type 2 [ 59] tandis que d’autres sont associées à l’apparition d’événements cardiovasculaires [ 58]. Dans les tissus, les bactéries, ou leurs fragments, induisent une stimulation de la prolifération de précurseurs pré-adipocytaires et de macrophages, contribuant ainsi au développement de l’obésité et de l’insulino-résistance. Les LPS des bactéries ainsi transloqués, circulent et se fixent aux complexes récepteurs CD14/TLR4-MD2 présentés par les cellules des tissus comme le foie et le tissu adipeux [ 60] générant la production de cytokines qui interfèrent avec la signalisation de l’insuline et induisent l’insulino-résistance et le diabète.
(→) Voir la Synthèse de L. Gemser et al., m/s n° 5, mai 2016, page 461
Nous avons montré que la dysbiose affectant le microbiote intestinal et buccal s’installe dans les jours qui suivent un changement de régime pour un régime gras [61, 62]. Au niveau buccal, certaines bactéries qui traversent l’épithélium parodontal via des micro-ouvertures induites par l’inflammation ainsi provoquée sont retrouvées dans les tissus métaboliques [63]. L’ADN correspondant à ces bactéries est présent dans le sang et dans les organes clés du métabolisme glucidique que sont le foie, le tissu adipeux et le muscle [64]. Dans l’intestin, la dysbiose induite par le régime, en inhibant la co-activation par les cellules présentatrices d’antigène de l’intestin, altère la stimulation de lymphocytes T intestinaux particuliers, les lymphocytes Th17, qui sont impliqués précisément dans la défense intestinale. De façon intéressante, le transfert du microbiote dysbiotique ainsi obtenu à des souris axéniques induit un diabète chez ces souris receveuses. Il altère également la co-activation des lymphocytes Th17 par les phagocytes intestinaux et ceci indépendamment du régime gras. L’administration d’un prébiotique (le polydextrose) permet de restaurer l’intégrité de la défense intestinale. Il diminue la translocation bactérienne et corrige la dysbiose intestinale et tissulaire [16]. La prévention de l’effet délétère du microbiote dysbiotique sur le nombre de lymphocytes Th17 représente ainsi une voie thérapeutique originale. L’identification des bactéries qui transloquent vers les tissus permettrait également d’envisager la constitution d’un vaccin spécifique afin d’éduquer le système immunitaire contre la dysbiose intestinale. Ces nouvelles voies thérapeutiques ouvrent de multiples opportunités qui représenteront les médicaments de demain. Afin d’augmenter leur efficacité thérapeutique, l’identification de biomarqueurs spécifiques, comme l’ADN circulant des bactéries, permettrait de cibler facilement, et à moindre coût, les patients présentant une dysbiose intestinale et tissulaire. L’obésité et le diabète de type 2 sont typiquement des maladies qui pourront bénéficier de ces nouvelles approches thérapeutiques. |
Le décryptage du microbiote tissulaire et sanguin personnalisé nécessitera de mettre en œuvre les moyens informatiques et mathématiques adaptés aux méga-données qui seront obtenues à partir de larges cohortes de patients. Les bactéries qui nous entourent ont été sélectionnées au cours de 3 milliards d’années d’évolution. Nous commençons seulement notre plongée dans les profondeurs, à la découverte de la face immergée de l’iceberg, mais les premières informations recueillies sont déjà riches en promesses thérapeutiques. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Li J, Jia H, Cai X, et al. An integrated catalog of reference genes in the human gut microbiome . Nature Biotechnol. 2014; ; 32 : :834.–841. 2.
Sender R, Fuchs S, Milo R. Are we really vastly outnumbered? Revisiting the ratio of bacterial to host cells in humans . Cell. 2016; ; 164 : :337.–340. 3.
Qin J, Li Y, Cai Z, et al. A metagenome-wide association study of gut microbiota in type 2 diabetes . Nature. 2012; ; 490 : :55.–60. 4.
Le Chatelier E, Nielsen T, Qin J, et al. Richness of human gut microbiome correlates with metabolic markers . Nature. 2013; ; 500 : :541.–546. 5.
Bergstrom A, Skov TH, Bahl MI, et al. Establishment of intestinal microbiota during early life: a longitudinal, explorative study of a large cohort of Danish infants . Appl Environ Microbiol. 2014; ; 80 : :2889.–2900. 6.
Qin J, Li R, Raes J, et al. A human gut microbial gene catalogue established by metagenomic sequencing . Nature. 2010; ; 464 : :59.–65. 7.
Emoto T, Yamashita T, Sasaki N, et al. Analysis of gut microbiota in coronary artery disease patients: a possible link between gut microbiota and coronary artery disease . J Atheroscler Thromb. 2016; ; 23 : :908.–921. 8.
Cotillard A, Kennedy SP, Kong LC, et al. Dietary intervention impact on gut microbial gene richness . Nature. 2013; ; 500 : :585.–588. 9.
Duboc H, Rajca S, Rainteau D, et al. Connecting dysbiosis, bile-acid dysmetabolism and gut inflammation in inflammatory bowel diseases . Gut. 2013; ; 62 : :531.–539. 10.
Rajca S, Grondin V, Louis E, et al. Alterations in the intestinal microbiome (dysbiosis) as a predictor of relapse after infliximab withdrawal in Crohn’s disease . Inflamm Bowel Dis. 2014; ; 20 : :978.–986. 11.
Lamas B, Richard ML, Leducq V, et al. CARD9 impacts colitis by altering gut microbiota metabolism of tryptophan into aryl hydrocarbon receptor ligands . Nat Med. 2016; ; 22 : :598.–605. 12.
Albenberg L, Esipova TV, Judge CP, et al. Correlation between intraluminal oxygen gradient and radial partitioning of intestinal microbiota . Gastroenterology. 2014; ; 147 : :1055.–1063e8. 13.
Kim F, Pham M, Luttrell I, et al. Toll-like receptor-4 mediates vascular inflammation and insulin resistance in diet-induced obesity . Circ Res. 2007; ; 100 : :1589.–1596. 14.
Song MJ, Kim KH, Yoon JM, Kim JB. Activation of Toll-like receptor 4 is associated with insulin resistance in adipocytes . Biochem Biophys Res Commun. 2006; ; 346 : :739.–745. 15.
Shi H, Kokoeva MV, Inouye K, et al. TLR4 links innate immunity and fatty acid-induced insulin resistance . J Clin Invest. 2006; ; 116 : :3015.–3025. 16.
Amar J, Chabo C, Waget A, et al. Intestinal mucosal adherence and translocation of commensal bacteria at the early onset of type 2 diabetes: molecular mechanisms and probiotic treatment . EMBO Mol Med. 2011; ; 3 : :559.–572. 17.
Hansson GC, Johansson ME. The inner of the two Muc2 mucin-dependent mucus layers in colon is devoid of bacteria . Gut Microbes. 2010; ; 1 : :51.–54. 18.
Atuma C, Strugala V, Allen A, Holm L. The adherent gastrointestinal mucus gel layer: thickness and physical state in vivo . Am J Physiol Gastrointest Liver Physiol. 2001; ; 280 : :G922.–G929. 19.
Swidsinski A, Loening-Baucke V, Theissig F, et al. Comparative study of the intestinal mucus barrier in normal and inflamed colon . Gut. 2007; ; 56 : :343.–350. 20.
Carneiro LA, Magalhaes JG, Tattoli I, et al. Nod-like proteins in inflammation and disease . J Pathol. 2008; ; 214 : :136.–148. 21.
Kufer TA, Sansonetti PJ. Sensing of bacteria: NOD a lonely job . Curr Opin Microbiol. 2007; ; 10 : :62.–69. 22.
Koren O, Spor A, Felin J, et al. Human oral, gut, and plaque microbiota in patients with atherosclerosis . Proc Natl Acad Sci USA. 2011; ; 108 ((suppl 1)) : :4592.–4598. 23.
D’Aiuto F, Sabbah W, Netuveli G, et al. Association of the metabolic syndrome with severe periodontitis in a large U.S. population-based survey . J Clin Endocrinol Metab. 2008; ; 93 : :3989.–3994. 24.
Preshaw PM, Alba AL, Herrera D, et al. Periodontitis and diabetes: a two-way relationship . Diabetologia. 2012; ; 55 : :21.–31. 25.
Blasco-Baque V, Serino M, Vergnes JN, et al. High-fat diet induces periodontitis in mice through lipopolysaccharides (LPS) receptor signaling: protective action of estrogens . PLoS One. 2012; ; 7 : :e48220.. 26.
Preshaw PM, Lauffart B, Zak E, et al. Progression and treatment of chronic adult periodontitis . J Periodontol. 1999; ; 70 : :1209.–1220. 27.
Borrell LN, Papapanou PN. Analytical epidemiology of periodontitis . J Clin Periodontol. 2005; ; 32 ((suppl 6)) : :132.–158. 28.
Lakhssassi N, Elhajoui N, Lodter JP, et al. Antimicrobial susceptibility variation of 50 anaerobic periopathogens in aggressive periodontitis: an interindividual variability study . Oral Microbiol Immunol. 2005; ; 20 : :244.–252. 29.
Holm NR, Belstrom D, Ostergaard JA, et al. Identification of individuals with undiagnosed diabetes and pre-diabetes in a Danish cohort attending dental treatment . J Periodontol. 2016; ; 87 : :395.–402. 30.
Field CA, Gidley MD, Preshaw PM, Jakubovics N. Investigation and quantification of key periodontal pathogens in patients with type 2 diabetes . J Periodontal Res. 2012; ; 47 : :470.–478. 31.
Demmer RT, Jacobs DR, Jr, Singh R, et al. Periodontal bacteria and prediabetes prevalence in ORIGINS: the oral infections, glucose intolerance, and insulin resistance study . J Dent Res. 2015; ; 94 : :S201.–S211. 32.
Zhang H, DiBaise JK, Zuccolo A, et al. Human gut microbiota in obesity and after gastric bypass . Proc Natl Acad Sci USA. 2009; ; 106 : :2365.–2370. 33.
Tremaroli V, Karlsson F, Werling M, et al. Roux-en-Y gastric bypass and Vertical banded gastroplasty induce long-term changes on the human gut microbiome contributing to fat mass regulation . Cell Metab. 2015; ; 22 : :228.–238. 34.
Bueter M, Abegg K, Seyfried F, et al. Roux-en-Y gastric bypass operation in rats . J Vis Exp. 2012; ; 64 : :e3940.. 35.
Troy S, Soty M, Ribeiro L, et al. Intestinal gluconeogenesis is a key factor for early metabolic changes after gastric bypass but not after gastric lap-band in mice . Cell Metab. 2008; ; 8 : :201.–211. 36.
Parker HE, Wallis K, le Roux CW, et al. Molecular mechanisms underlying bile acid-stimulated glucagon-like peptide-1 secretion . Br J Pharmacol. 2012; ; 165 : :414.–423. 37.
Cani PD, Everard A, Duparc T. Gut microbiota, enteroendocrine functions and metabolism . Curr Opin Pharmacol. 2013; ; 13 : :935.–940. 38.
Davis LM, Martinez I, Walter J, et al. Barcoded pyrosequencing reveals that consumption of galactooligosaccharides results in a highly specific bifidogenic response in humans . PLoS One. 2011; ; 6 : :e25200.. 39.
Baer DJ, Stote KS, Henderson T, et al. The metabolizable energy of dietary resistant maltodextrin is variable and alters fecal microbiota composition in adult men . J Nutr. 2014; ; 144 : :1023.–1029. 40.
Delzenne NM, Neyrinck AM, Cani PD. Gut microbiota and metabolic disorders: How prebiotic can work? . Br J Nutr. 2013; ; 109 ((suppl 2)) : :S81.–S85. 41.
Forslund K, Hildebrand F, Nielsen T, et al. Disentangling type 2 diabetes and metformin treatment signatures in the human gut microbiota . Nature. 2015; ; 528 : :262.–266. 42.
Cabreiro F, Au C, Leung KY, et al. Metformin retards aging in C. elegans by altering microbial folate and methionine metabolism . Cell. 2013; ; 153 : :228.–239. 43.
Yassour M, Lim MY, Yun HS, et al. Sub-clinical detection of gut microbial biomarkers of obesity and type 2 diabetes . Genome Med. 2016; ; 8 : :17.. 44.
Vrieze A, Van Nood E, Holleman F, et al. Transfer of intestinal microbiota from lean donors increases insulin sensitivity in individuals with metabolic syndrome . Gastroenterology. 2012; ; 143 : :913.–916e7. 45.
Cani PD, Amar J, Iglesias MA, et al. Metabolic endotoxemia initiates obesity and insulin resistance . Diabetes. 2007; ; 56 : :1761.–1772. 46.
Cani PD, Bibiloni R, Knauf C, et al. Changes in gut microbiota control metabolic endotoxemia-induced inflammation in high-fat diet-induced obesity and diabetes in mice . Diabetes. 2008; ; 57 : :1470.–1481. 47.
Verges B, Duvillard L, Lagrost L, et al. Changes in lipoprotein kinetics associated with type 2 diabetes affect the distribution of lipopolysaccharides among lipoproteins . J Clin Endocrinol Metab. 2014; ; 99 : :E1245.–E1253. 48.
Schertzer JD, Tamrakar AK, Magalhaes JG, et al. NOD1 activators link innate immunity to insulin resistance . Diabetes. 2011; ; 60 : :2206.–2215. 49.
Cani PD, Possemiers S, Van de Wiele T, et al. Changes in gut microbiota control inflammation in obese mice through a mechanism involving GLP-2-driven improvement of gut permeability . Gut. 2009; ; 58 : :1091.–1103. 50.
Hirose S, Ono HK, Omoe K, et al. Goblet cells are involved in translocation of staphylococcal enterotoxin A in the intestinal tissue of house musk shrew (Suncus murinus) . J Appl Microbiol. 2016; ; 120 : :781.–789. 51.
Koh IH, Liberatore AM, Menchaca-Diaz JL, et al. Bacterial translocation, microcirculation injury and sepsis . Endocr Metab Immune Dis Drug Targets. 2006; ; 6 : :143.–150. 52.
Sandek A, Bauditz J, Swidsinski A, et al. Altered intestinal function in patients with chronic heart failure . J Am Coll Cardiol. 2007; ; 50 : :1561.–1569. 53.
Gutierrez A, Holler E, Zapater P, et al. Antimicrobial peptide response to blood translocation of bacterial DNA in Crohn’s disease is affected by NOD2/CARD15 genotype . Inflamm Bowel Dis. 2011; ; 17 : :1641.–1650. 54.
Everard A, Belzer C, Geurts L, et al. Cross-talk between Akkermansia muciniphila and intestinal epithelium controls diet-induced obesity . Proc Natl Acad Sci USA. 2013; ; 110 : :9066.–9071. 55.
Berg RD. Bacterial translocation from the gastrointestinal tract . Trends Microbiol. 1995; ; 3 : :149.–154. 56.
Amar J, Burcelin R, Ruidavets JB, et al. Energy intake is associated with endotoxemia in apparently healthy men . Am J Clin Nutr. 2008; ; 87 : :1219.–1223. 57.
Burcelin R, Serino M, Chabo C, et al. Metagenome and metabolism: the tissue microbiota hypothesis . Diabetes Obes Metab. 2013; ; 15 ((suppl 3)) : :61.–70. 58.
Amar J, Lange C, Payros G, et al. Blood microbiota dysbiosis is associated with the onset of cardiovascular events in a large general population: the D.E.S.I.R. study . PLoS One. 2013; ; 8 : :e54461.. 59.
Amar J, Serino M, Lange C, et al. Involvement of tissue bacteria in the onset of diabetes in humans: evidence for a concept . Diabetologia. 2011; ; 54 : :3055.–3061. 60.
Luche E, Cousin B, Garidou L, et al. Metabolic endotoxemia directly increases the proliferation of adipocyte precursors at the onset of metabolic diseases through a CD14-dependent mechanism . Mol Metab. 2013; ; 2 : :281.–291. 61.
Garidou L, Pomie C, Klopp P, et al. The gut microbiota regulates intestinal CD4 T cells expressing RORgammat and controls metabolic disease . Cell Metab. 2015; ; 22 : :100.–112. 62.
Blasco-Baque V, Garidou L, Pomié C, et al. E. Periodontitis induced by Porphyromonas gingivalis drives periodontal microbiota dysbiosis and insulin-resistance via an impaired adaptive immune response . Gut. 2016 ; doi : 10.1136/gutjnl-2015-309897. 63.
Ishikawa M, Yoshida K, Okamura H, et al. Oral Porphyromonas gingivalis translocates to the liver and regulates hepatic glycogen synthesis through the Akt/GSK-3beta signaling pathway . Biochim Biophys Acta. 2013; ; 1832 : :2035.–2043. 64.
Reichert S, Altermann W, Stein JM, et al. Individual composition of human leukocyte antigens and periodontopathogens in the background of periodontitis . J Periodontol. 2013; ; 84 : :100.–109. 65.
Genser L, Poitou C, Brot-Laroche E, et al. L’altération de la perméabilité intestinale : chaînon manquant entre dysbiose et inflammation au cours de l’obésité ? . Med Sci (Paris). 2016; ; 32 : :461.–469. 66.
Blottière HM, Doré J. Impact des nouveaux outils de métagénomique sur notre connaissance du microbiote intestinal et de son rôle en santé humaine : enjeux diagnostiques et thérapeutiques . Med Sci (Paris). 2016; ; 32 : :944.–951. 67.
Thorens B. Incrétines, sécrétion d’insuline et diabète . Med Sci (Paris). 2003; ; 19 : :860.–863. 68.
Lagier JC, Raoult D. Greffe de microbiote fécal et infections : mise au point, perspectives . Med Sci (Paris). 2016; ; 32 : :991.–997. 69.
Weissenbach J, Sghir A. Microbiotes et métagénomique . Med Sci (Paris). 2016; ; 32 : :937.–943. |