| |
| Med Sci (Paris). 32(12): 1071–1075. doi: 10.1051/medsci/20163212009.Le rôle des protéines BET dans l’intégration des γ-rétrovirus Olivier Albagli1* and Hélène Pelczar2** 1Inserm U1016 – CNRS UMR 8104 – Institut Cochin, groupe hospitalier Cochin-Port-Royal, bâtiment Cassini, 123, boulevard de Port-Royal, 75014Paris, France 2ER6, Université Pierre et Marie Curie, 4, place Jussieu, 75005Paris, France MeSH keywords: Animaux, Transformation cellulaire virale, Humains, Retroviridae, Facteurs de transcription, Intégration virale, physiologie, génétique |
La réplication des rétrovirus1 implique l’intégration de leur matériel génétique dans celui de la cellule hôte. L’ARN génomique viral simple brin est rétrotranscrit en un ADN double brin qui s’intègre dans le génome de l’hôte pour former le provirus. Ces deux étapes sont respectivement catalysées par la rétrotranscriptase (RT) et l’intégrase (IN), deux enzymes codées par le gène viral pol [1–4] (→).
(→) Voir la Synthèse de M. Marin et al., m/s n° 3, mars 1994, page 318
Cette propriété d’intégration présentée par les rétrovirus a suscité le développement d’outils de transfert de gènes, les rétrovecteurs1. Parmi ceux-ci, les γ-rétrovecteurs (γRV1) dérivés du γ-rétrovirus prototypique Moloney murine leukemia virus (MLV) sont utilisés en recherche fondamentale et dans plusieurs essais de thérapie génique [5] (Figure 1A).
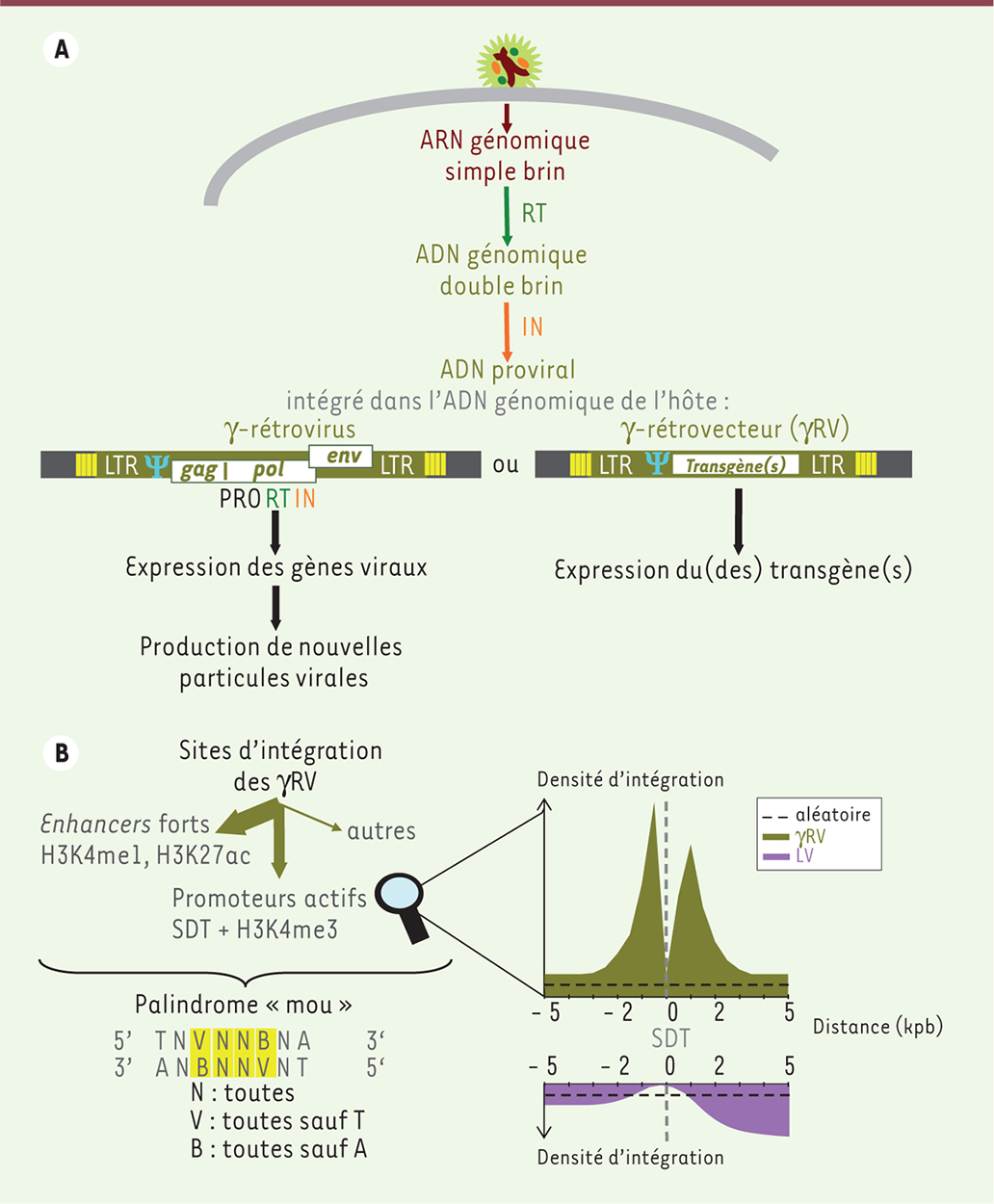
 | Figure 1.
Intégration des γ-rétrovirus et des γRV. A. De l’ARN génomique rétroviral au provirus. L’ARN génomique simple brin positif du γ-rétrovirus ou du γRV (γ-rétrovecteur) dérivé (deux exemplaires par particule virale) est rétrotranscrit par la rétrotranscriptase (RT) en ADN double brin qui s’intègre dans le génome de l’hôte (en noir) grâce à l’intégrase (IN). RT et IN sont codées par le gène viral pol et présentes dans la particule virale (en vert et orange). Ces étapes sont identiques pour le γ-rétrovirus et le γRV. Seule différence : les protéines virales impliquées ne sont pas codées par le γRV lui-même (les régions codantes du γ-rétrovirus sont remplacées par le(s) transgène(s)), mais sont apportées par transcomplémentation lors de la production du γRV. Les provirus schématisés sont ceux du γ-rétrovirus « modèle » MLV ( Moloney murine leukemia virus) et d’un γRV dérivé. L’intégration provoque la duplication de 4 pb (paires de bases) du génome de l’hôte (barres jaunes) de part et d’autre du provirus. Le codon stop (barre verticale) qui sépare gag et pol est traduit en glutamine avec une faible efficacité, générant le précurseur Gag-Pol. La partie 3’ de pol chevauche la partie 5’ de env, traduit dans une autre phase de lecture. PRO, codée aussi par pol, est une protéase responsable des clivages des précurseurs Gag et Gag-Pol, et de l’un des clivages affectant Env (voir [ 1– 4] pour des références de revues plus détaillées). B. Les γRV s’intègrent préférentiellement dans les enhancers forts et les promoteurs actifs. Les enhancers forts sont identifiés par leur enrichissement en histone H3 monométhylée sur la lysine 4 (H3K4me1) et en histone H3 acétylée sur la lysine 27 (H3K27ac). Le profil d’intégration des γRV dans les promoteurs actifs, identifiés par un SDT (site de démarrage de la transcription) et un enrichissement en histone H3 triméthylée sur la lysine 4 (H3K4me3), est présenté (profil vert) ; il diffère de celui des LV (lentivecteurs, profil violet). Les lignes horizontales pointillées indiquent le profil correspondant à des intégrations aléatoires (adapté de [ 8]). Un léger biais en faveur d’une séquence palindromique dégénérée est observé au niveau des sites d’intégration ; les 4 pb dupliquées à l’issue de l’intégration sont surlignées [ 1, 2, 9]. LTR : long terminal repeat ; Psi (Ψ) : séquence d’encapsidation. |
|
Les γRV s’intègrent préférentiellement dans les promoteurs actifs et les enhancers forts L’intégration permet un transfert stable de gènes mais elle présente deux inconvénients : elle soumet l’expression du(des) gène(s) transféré(s) à l’influence de son (leur) environnement génomique et elle expose le génome de l’hôte à l’inactivation ou la dérégulation de gènes. L’efficacité et la dangerosité des rétrovecteurs dépendent donc en partie de leurs sites d’intégration [6]. Les progrès du séquençage ont permis l’analyse d’un nombre croissant de sites d’intégration. Ces études ont révélé que deux types de région concentrent les intégrations des γRV : les promoteurs2 actifs et les enhancers forts [7–9]. Par exemple, dans des cellules souches hématopoïétiques humaines, 15 % des intégrations d’un γRV ont lieu dans les promoteurs actifs, soit 14 fois ce que prédit une intégration aléatoire [8] (Figure 1B). L’intégration est d’autant plus fréquente que le promoteur est actif ; les promoteurs inactifs sont même sous-représentés [8]. Les enhancers forts concentrent la majorité des autres intégrations [8] (Figure 1B). Une catégorie d’enhancer fort est jusqu’à 40 fois plus représentée que le prédit une intégration aléatoire [9]. Notons que ces biais sont observés dans des conditions permettant d’exclure qu’ils ne procèdent que d’une sélection des cellules portant des intégrations favorables à leur survie ou leur prolifération [8, 9]. |
Profil d’intégration et oncogénicité Les régions privilégiées par les γRV pour l’intégration sont donc des éléments cis-régulateurs3, actifs de la transcription. Ce tropisme contribue probablement à l’expression des γRV [6–9] mais il explique aussi, en partie [10], leur oncogénicité marquée car il accroît le risque de dérégulation de gènes de l’hôte [2–12]. Les essais de thérapie génique témoignent de ces deux aspects. Dans l’ensemble, les γRV corrigent la déficience génétique mais ils déclenchent parfois des leucémies en s’intégrant dans des régions cis-régulatrices de proto-oncogènes [5, 6]. Les rétrovirus des autres genres présentent un profil d’intégration généralement distinct de celui des γRV. Ainsi, les lentivirus et les lentivecteurs (LV1) évitent les promoteurs et s’intègrent préférentiellement dans le corps des gènes transcrits (Figure 1B) [2–4, 6–12]. Les LV sont moins oncogéniques que les γRV [10]. D’autres rétrovirus s’intègrent de façon peu ou pas biaisée ; les vecteurs dérivés pourraient donc s’avérer encore moins oncogéniques que les LV [2, 4, 6]. |
Les protéines BET : partenaires spécifiques de IN des γ-rétrovirus Le tropisme distinct des γRV et des LV a permis d’identifier les protéines virales impliquées dans la sélection des sites d’intégration. En effet, un LV hybride codant IN de MLV tend à reproduire la prédilection des γRV pour l’intégration au sein des promoteurs [4]. Pour comprendre comment IN de MLV sélectionne les sites d’intégration, trois équipes ont cherché les protéines cellulaires capables d’interagir avec elle. Les partenaires identifiés sont trois protéines apparentées, BRD2 (bromodomain-containing protein 2), BRD3 et BRD4, qui présentent deux bromodomaines (motifs conservés de reconnaissance des lysines acétylées) et un domaine caractéristique baptisé ET (extraterminal) [2, 3, 11, 12]. Chez les mammifères, ces trois protéines d’expression ubiquitaire constituent avec BRDT (bromodomain testis-specific protein), qui est uniquement détectée dans les testicules, la famille des protéines BET (bromodomain and extraterminal) [13]. Le domaine ET et un court peptide situé à l’extrémité C-terminale de IN de MLV baptisé EBM (ET binding motif), sont nécessaires et suffisants pour l’interaction [14] (Figure 2A). Cette interaction est directe [2, 3 11, 12] et spécifique : EBM n’existe que dans les IN des γ-rétrovirus et seules ces IN, mais pas celles des autres rétrovirus, interagissent avec les protéines BET [2, 3]. EBM, dont le rôle était énigmatique jusqu’ici, paraît avoir évolué pour reproduire, sous une forme compacte et très affine, l’interface d’interaction présentée par plusieurs partenaires cellulaires du domaine ET [14].
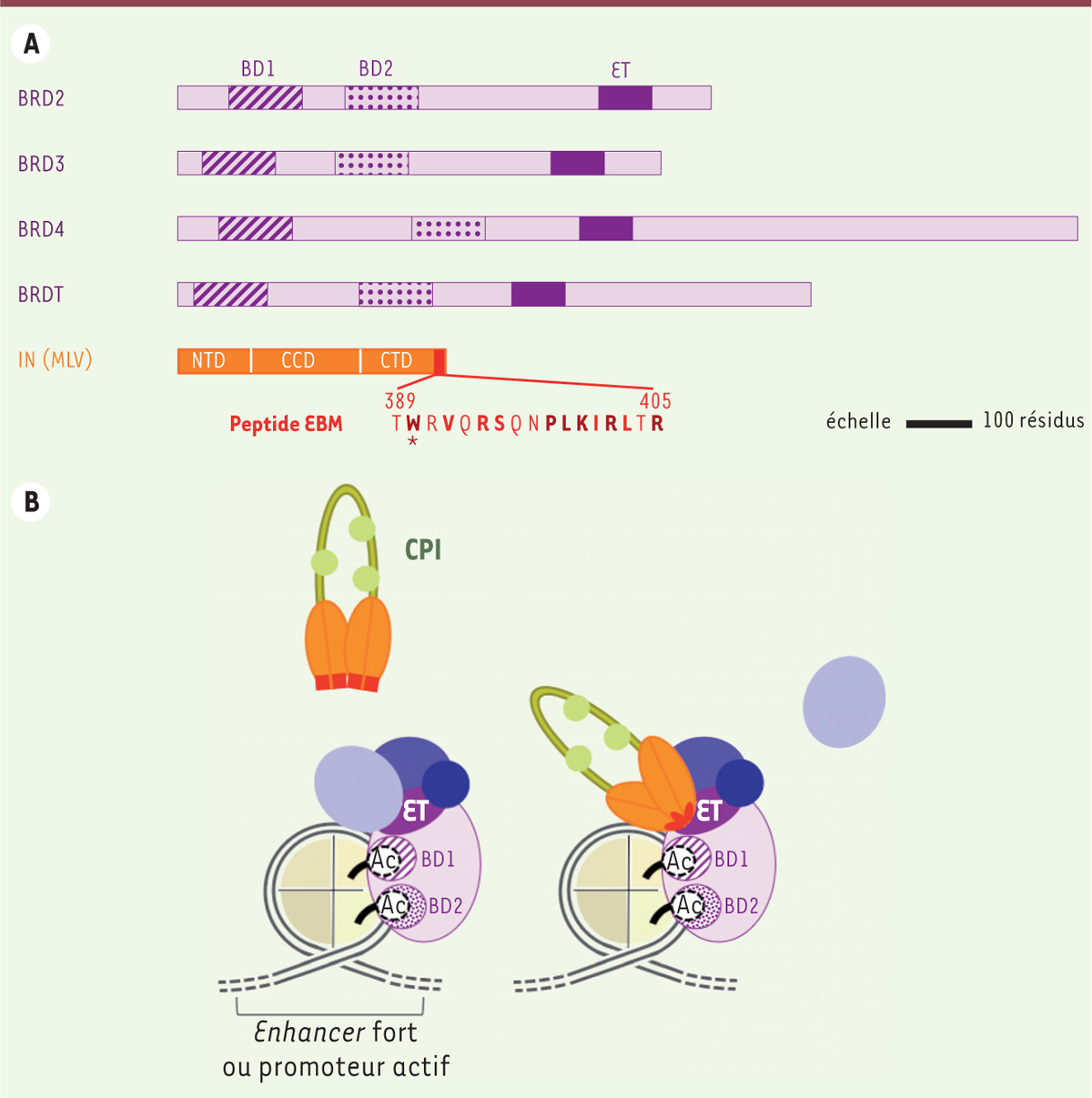
 | Figure 2.
Interaction entre l’intégrase IN des γ-rétrovirus et les protéines BET. A. Structure des protéines BET ( bromodomain and extraterminal) et de IN (intégrase) de MLV ( Moloney murine leukemia virus). Les protéines BET se caractérisent par deux bromodomaines (BD1 et BD2) et un domaine extraterminal (ET). D’autres domaines conservés ont été identifiés (non représentés) [ 2, 12, 13]. Plusieurs isoformes existent pour BRD2 ( bromodomain-containing protein 2), BRD4 et BRDT ( bromodomain testis-specific protein) [ 2, 13], une seule est schématisée. Le domaine ET des protéines BRD2, BRD3 et BRD4 (celui de BRDT n’a pas été testé) interagit directement avec le peptide EBM ( ET-binding motif) situé à l’extrémité C-terminale de IN de MLV. EBM est très conservé parmi les IN des γ-rétrovirus : sont indiqués les résidus conservés (identiques en grenat gras ; similaires en rouge gras) avec les IN de sept autres γ-rétrovirus, dont celle de FeLV ( feline leukemia virus) qui interagit aussi avec les protéines BET. Par contre, EBM n’existe pas dans les IN des rétrovirus des autres genres et aucune interaction n’est détectée entre elles et les protéines BET [ 2, 3]. EBM est codé précisément par la partie du gène pol chevauchant le gène env [ 2] (Figure 1A). B. Les protéines BET servent d’amarres pour le complexe de préintégration des γ-rétrovirus. À gauche, une protéine BET (en rose) liée à un nucléosome grâce à ses deux bromodomaines, fixant certaines lysines acétylés (Ac) des histones (en beige), et par interaction directe avec l’ADN (en gris) [ 2]. Les protéines BET recrutent de nombreux partenaires (ici trois, en bleu) grâce, entre autres, au domaine ET [ 14]. Le complexe de préintégration (CPI) du γ-rétrovirus (ou du γRV), composé (entre autres) de l’ADN double brin génomique (vert amande), de protéines virales codées par gag (vert clair) et de IN (en orange, un dimère à chaque extrémité de l’ADN double brin, en rouge le peptide EBM), fixe la protéine BET grâce à l’interaction directe entre le peptide EBM de IN et le domaine ET. Cette interaction guide l’intégration au niveau des sites génomiques riches en protéines BET, c’est-à-dire principalement les promoteurs actifs et les enhancers forts. L’interaction EBM-ET structure le peptide EBM [ 2] et peut déplacer in vitro au moins un partenaire du domaine ET [ 14], ce qui reste à confirmer in vivo. L’étape suivante, intégration de l’ADN viral et relargage des autres composants du CPI, n’est pas représentée. D’autres virus appartenant à des familles variées utilisent les protéines BET en exploitant au moins deux de leurs propriétés : (1) leurs fonctions transrégulatrices, pour moduler l’expression virale, et/ou (2) leur capacité à rester fixées aux chromosomes condensés durant la mitose, pour assurer la persistance du génome viral sous forme épisomique à travers les divisions cellulaires [ 2, 3, 13]. NTD : zinc-binding N-terminal domain ; CCD : core catalytic domain ; CTD : C-terminal domain. |
|
Les protéines BET guident et stimulent l’intégration des γRV Les protéines BET lisent l’information épigénétique : elles fixent la chromatine riche en histones acétylées grâce à leurs bromodomaines et recrutent des régulateurs transcriptionnels et des complexes de remodelage de la chromatine via, notamment, leur domaine ET [13] (Figure 2B). Leur distribution sur le génome est remarquablement similaire à celle des intégrations γ-rétrovirales : elles sont préférentiellement associées aux promoteurs et aux enhancers de gènes activement transcrits [15]. Plusieurs études confirment directement la fréquente présence des protéines BET, particulièrement BRD2, au voisinage des sites d’intégration des γRV [2, 11, 12]. Cette co-distribution suggère que les protéines BET guident l’intégration des γRV en arrimant IN aux promoteurs et enhancers actifs du génome de l’hôte (Figure 2B). Cette hypothèse est étayée par l’expérience : l’inhibition transitoire des protéines BET par un composé chimique (JQ14,) ou leur déplétion par knock-down diminue la tendance des γRV à s’intégrer dans les promoteurs (Tableau I) [2, 11].
Tableau I.
| Buts |
Moyens |
Résultats |
|
Neutralisation Minimiser l’activation transcriptionnelle de gènes de l’hôte suite à l’intégration |
γRV SIN : élimination des séquences cis-régulatrices de la transcription dans les LTR |
Oncogénicité réduite [5, 6, 10] |
|
|
Désorientation Réduire la fréquence des intégrations dans les enhancers et les promoteurs des gènes de l’hôte |
inhibition transitoire (pharmacologique ou knock-down) des protéines BET
γRV BIN : inactivation du peptide EBM de IN (délétion ou mutation ponctuelle)
|
Intégration plus aléatoire [2, 11, 16] |
|
|
Réorientation Rediriger l’intégration des γRV |
Expression dans les cellules d’une protéine artificielle fusionnant un domaine ET et un domaine responsable d’un adressage « épigénomique » choisi |
Intégration dans le corps des gènes transcrits avec une chimère entre le domaine ET* de BRD4 et le domaine de liaison à la chromatine de LEDGF/p75 [12] |
Stratégies pour diminuer l’oncogénicité des γRV. La stratégie courante pour diminuer le risque de dérégulation de gènes cellulaires par des γRV (γ-rétrovecteur) intégratifs consiste à éliminer les éléments cis-régulateurs de la transcription ( enhancer et promoteur) des LTR ( long terminal repeat ; γRV SIN, self-inactivating). Cette stratégie (« neutralisation ») nécessite d’ajouter au γRV un promoteur interne. Elle diminue nettement l’oncogénicité des γRV, surtout si le promoteur interne est faible [ 5, 6, 10] ; elle réduit en outre les risques d’apparition de dérivés réplicatifs et peut permettre d’obtenir une expression du/des transgène(s) spécifique(s) d’un tissu [ 6]. Le rôle clé des protéines BET ( bromodomain and extraterminal) dans le ciblage de l’intégration suggère d’autres pistes. La première est d’empêcher le γRV d’utiliser les protéines BET (« désorientation »), ce qui rend l’intégration plus aléatoire. Cette désorientation est obtenue de deux façons [ 2, 11, 16] : (1) déplétion ( knock-down) ou inhibition (pharmacologique) transitoires des protéines BET au moment de l’infection. De nombreux inhibiteurs (JQ1, I-BET 151, etc.) fixent les bromodomaines des protéines BET et bloquent ainsi leur interaction avec les lysines acétylées ; (2) invalidation du peptide EBM ( ET[extraterminal]-binding motif) de IN (intégrase) (Figure 2A) (γRV BIN, BET-independent). Différentes versions de γRV BIN existent : celle portant une IN mutée sur le résidu tryptophane (W) conservé (astérisque sur la Figure 2A) garde un titre et une capacité intégrative quasi-normaux alors que celle portant une IN complètement amputée de EBM est plus affectée. Enfin, une autre stratégie consiste à « réorienter » l’intégration des γRV par l’expression d’un domaine ET fusionné à un domaine d’adressage « épigénomique » hétérologue (différent de leurs bromodomaines). LEDGF( lens epithelium derived growth factor)/p75 joue un rôle clé pour le ciblage de l’intégration des lentivirus (et des lentivecteurs LV) dans le corps des gènes transcrits. LEDGF/p75 peut être divisée en deux parties : le domaine N-terminal (résidus 1-324) reconnaît des marques épigénétiques spécifiques du corps des gènes transcrits, la partie C-terminale reconnaît IN des lentivirus [ 2]. L’expression d’une chimère fusionnant le domaine ET de BRD4 ( bromodomain-containing protein 4) au domaine 1-324 de LEDGF conduit le γRV à adopter un profil d’intégration lentiviral, biaisé en faveur du corps des gènes transcrits et moins concentré dans les promoteurs [ 12]. Pour d’autres stratégies, voir [ 6, 16]. *La chimère LEDGF-ET contient, en plus du domaine ET, un court domaine de BRD4 (domaine SEED, Ser-Glu-Glu-Asp) adjacent au domaine ET [ 12]. |
L’inhibition des protéines BET lors de l’infection diminue aussi le nombre total de génomes γ-rétroviraux intégrés sans affecter la rétrotranscription. Réciproquement, la surexpression d’une portion de BRD2 contenant le domaine ET augmente ce nombre et, in vitro, BRD2, BRD4 ou (moins efficacement) un domaine ET seul, favorise l’intégration dans un système constitué d’ADN et de IN de MLV purifiée [2, 3, 11, 12]. Les protéines BET stimulent donc l’intégration des γRV, probablement par deux mécanismes : l’activation allostérique de IN et l’arrimage de IN à l’ADN cible [2, 3]. |
La stratégie usuelle pour diminuer l’oncogénicité des γRV consiste à utiliser des γRV SIN (self-inactivating) dans lesquels ont été éliminées les séquences enhancers et promoteurs de leurs LTR (long terminal repeat) afin de limiter les conséquences dérégulatrices des intégrations [5, 6, 10] (Tableau I). Les travaux présentés ici suggèrent une autre possibilité : « désorienter » les γRV pour prévenir les intégrations dangereuses. L’inhibition transitoire des protéines BET réduit effectivement la fréquence des intégrations à proximité des proto-oncogènes [2]. De même, un γRV portant une IN dépourvue d’un peptide EBM fonctionnel (γRV BIN, BET-independent) montre un profil d’intégration qui n’est plus corrélé à la distribution des protéines BET et une moindre prédilection pour les promoteurs, notamment ceux des proto-oncogènes [2, 16] (Tableau I). On peut aussi réorienter l’intégration des γRV en modifiant l’adressage « épigénomique » du domaine ET. En effet, l’expression d’une chimère comportant le domaine ET de BRD4 et le domaine d’interaction avec la chromatine de LEDGF (lens epithelium derived growth factor)/p75, une protéine cellulaire jouant un rôle clé dans le ciblage de l’intégration des LV, redirige l’intégration d’un γRV dans le corps des gènes transcrits [12]. D’autres chimères avec le domaine ET sont envisageables mais l’efficacité de cette stratégie implique d’obtenir leur expression à un niveau plus élevé que celui des protéines BET endogènes [16]. |
Les résultats résumés ici apportent un éclairage sur un phénomène resté longtemps mystérieux : le caractère non-aléatoire de l’intégration des γRV. Ils montrent que la préférence pour certains sites n’est pas simplement liée à « l’accessibilité » de la chromatine, mais qu’elle résulte d’une interaction directe et spécifique entre IN des γ-rétrovirus et une famille de régulateurs transcriptionnels cellulaires. Ces travaux ouvrent une voie possible pour rendre plus sûrs les γRV en perturbant les mécanismes de leur intégration. Ils suggèrent une même cause pour la « propension » des γRV à s’intégrer à proximité de proto-oncogènes [7, 10] et pour l’effet sélectif de JQ1 sur la croissance tumorale [15] : le rôle central des protéines BET dans la régulation de gènes déterminant l’identité et la prolifération cellulaires. L’évolution pourrait avoir sélectionné cette « propension » en raison de son effet favorable à la propagation des γ-rétrovirus [7, 9]. Ces résultats laissent néanmoins plusieurs points en suspens. Comment certaines protéines codées par le gène gag de MLV potentialisent la sélection exercée par IN [4] ? Existe-t-il des facteurs de ciblage alternatifs lorsque IN et les protéines BET ne peuvent plus interagir ? Quelles sont précisément les marques épigénétiques reconnues par les protéines BET dans les régions privilégiées d’intégration ? La fraction des protéines BET associée à des régions transcriptionnellement inactives [13] joue-t-elle un rôle pour l’intégration ? Les conséquences de l’interaction sur la fonction des protéines BET restent aussi à éclaircir. La capacité du peptide EBM à empêcher l’association entre le domaine ET et au moins l’un de ses partenaires cellulaires [14] (Figure 2B) suggère que IN bloque certaines fonctions des protéines BET. Si tel est le cas, EBM pourrait inspirer de nouveaux inhibiteurs spécifiques des protéines BET, ciblant, non pas les bromodomaines comme les composés actuels, mais le domaine ET [14]. Les travaux présentés ici pourraient permettre alors d’améliorer le traitement des nombreuses pathologies, notamment tumorales, impliquant les protéines BET [13].
|
Note Ajoutée Aux Épreuves À la suite de l’écriture de cet article, une étude dans des lymphocytes humains a montré l’intégration préférentielle du transposon piggyBac dans les promoteurs et les enhancers actifs, et l’interaction entre la transposase de piggyBac et le domaine ET des protéines BET. Cette interaction et celle décrite dans cette revue représentent probablement une convergence évolutive entre piggyBac et les γ-rétrovirus et non deux phénomènes phylogénétiquement reliés [17]. |
|
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Marin M, Etienne-Julan M, Piechaczyk M, Noël D. L’intégration des rétrovirus : faits et croyances . Med Sci (Paris). 1994; ; 10 : :318.–324. 2.
Kvaratskhelia M, Sharma A, Larue RC, et al. Molecular mechanisms of retroviral integration site selection . Nucleic Acids Res. 2014; ; 42 : :10209.–10225. 3.
Gupta SS, Maetzig T, Maertens GN, et al. Bromo- and extraterminal domain chromatin regulators serve as cofactors for murine leukemia virus integration . J Virol. 2013; ; 87 : :12721.–12736. 4.
Santoni FA, Hartley O, Luban J. Deciphering the code for retroviral integration target site selection . PLoS Comput Biol. 2010; ; 6 : :e1001008.. 5.
Hacein-Bey-Abina S, Pai SY, Gaspar HB, et al. A modified γ-retrovirus vector for X-linked severe combined immunodeficiency . N Engl J Med. 2014; ; 371 : :1407.–1417. 6.
Suerth JD, Labenski V, Schambach A. Alpharetroviral vectors: from a cancer-causing agent to a useful tool for human gene therapy . Viruses. 2014; ; 6 : :4811.–4838. 7.
Cattoglio C, Pellin D, Rizzi E, et al. High-definition mapping of retroviral integration sites identifies active regulatory elements in human multipotent hematopoietic progenitors . Blood. 2010; ; 116 : :5507.–5517. 8.
De Ravin SS, Su L, Theobald N, et al. Enhancers are major targets for murine leukemia virus vector integration . J Virol. 2014; ; 88 : :4504.–4513. 9.
LaFave MC, Varshney GK, Gildea DE, et al. MLV integration site selection is driven by strong enhancers and active promoters . Nucleic Acids Res. 2014; ; 42 : :4257.–4269. 10.
Montini E, Cesana D, Schmidt M, et al. The genotoxic potential of retroviral vectors is strongly modulated by vector design and integration site selection in a mouse model of HSC gene therapy . J Clin Invest. 2009; ; 119 : :964.–975. 11.
Sharma A, Larue RC, Plumb MR, et al. BET proteins promote efficient murine leukemia virus integration at transcription start sites . Proc Natl Acad Sci USA. 2013; ; 110 : :12036.–12041. 12.
De Rijck J, de Kogel C, Demeulemeester J, et al. The BET family of proteins targets Moloney murine leukemia virus integration near transcription start sites . Cell Rep. 2013; ; 5 : :886.–894. 13.
Belkina AC, Denis GV. BET domain co-regulators in obesity, inflammation and cancer . Nat Rev Cancer. 2012; ; 12 : :465.–477. 14.
Crowe BL, Larue RC, Yuan C, et al. Structure of the Brd4 ET domain bound to a C-terminal motif from γ-retroviral integrases reveals a conserved mechanism of interaction . Proc Natl Acad Sci USA. 2016; ; 113 : :2086.–2091. 15.
Loven J, Hoke HA, Lin CY, et al. Selective inhibition of tumor oncogenes by disruption of super-enhancers . Cell. 2013; ; 153 : :320.–334. 16.
Schambach A. Toward a safer integration profile of MLV-based retroviral vectors . Mol Ther. 2014; ; 22 : :1405.–1406. 17.
Gogol-Döring A, Ammar I, Gupta S, et al. Genome-wide profiling reveals remarkable parallels between insertion site selection properties of the MLV retrovirus and the piggyBac transposon in primary human CD4+ T cells . Mol Ther. 2016; ; 24 : :592.–606. |