| |
| Med Sci (Paris). 33(2): 126–130. doi: 10.1051/medsci/20173302004.Sans lamine A, la chromatine s’emballe Eldad Kepten1 and Judith Miné-Hattab2* 1Institut Curie, PSL Research University, CNRS, Génétique et biologie du développement, UMR 3215, 26, rue d’Ulm, F-75005, Paris, France 2Institut Curie, PSL Research University, CNRS, Dynamique du noyau, UMR 3664, 26, rue d’Ulm, F-75005, Paris, France |
À l’intérieur du noyau de nos cellules, la chromatine est organisée à différentes échelles, allant du nucléosome jusqu’à l’assemblage de larges territoires chromosomiques, en passant par des structures intermédiaires dont la nature reste mal comprise. L’organisation de la chromatine ainsi que sa dynamique ont un rôle essentiel pour le bon fonctionnement de la cellule. Il est donc fondamental de mieux comprendre ces mécanismes. Différents modèles d’organisation ont été proposés : certains s’inspirent de la physique des polymères [1, 2], d’autres sont structuraux. Les modèles structuraux décrits dans la littérature s’appuient sur l’observation de protéines de la membrane nucléaire maintenant la chromatine [3] ou bien sur l’existence d’une « matrice nucléaire » faisant office de structure à la chromatine [4, 5]. Cependant, aucun modèle ne se suffit à lui-même et ne fait actuellement consensus. Pour mieux comprendre les mécanismes régissant l’organisation nucléaire, l’équipe de Yuval Garini a choisi d’étudier l’effet de la lamine A sur la dynamique de différentes régions génomiques dans des cellules mammifères. Dans un article récemment publié dans le journal Nature Communications, Bronshtein et al. ont ainsi montré que l’absence de lamine A entraîne une augmentation spectaculaire de la mobilité du génome [6]. Les lamines A, B et C forment la lamina nucléaire, une structure marquant la membrane intérieure du noyau et participant aux liaisons chromatine/membrane nucléaire (Figure 1A). Des mutations de ces protéines laminaires ont un impact sévère sur la plasticité du noyau [7] et sur l’organisation de la chromatine [8] et sont associées à de nombreuses maladies telles que la progéria1, ou les laminopathies2 [9–11] (→).
(→) Voir la Synthèse de C.L. Navarro et al., m/s n° 10, octobre 2008, page 833
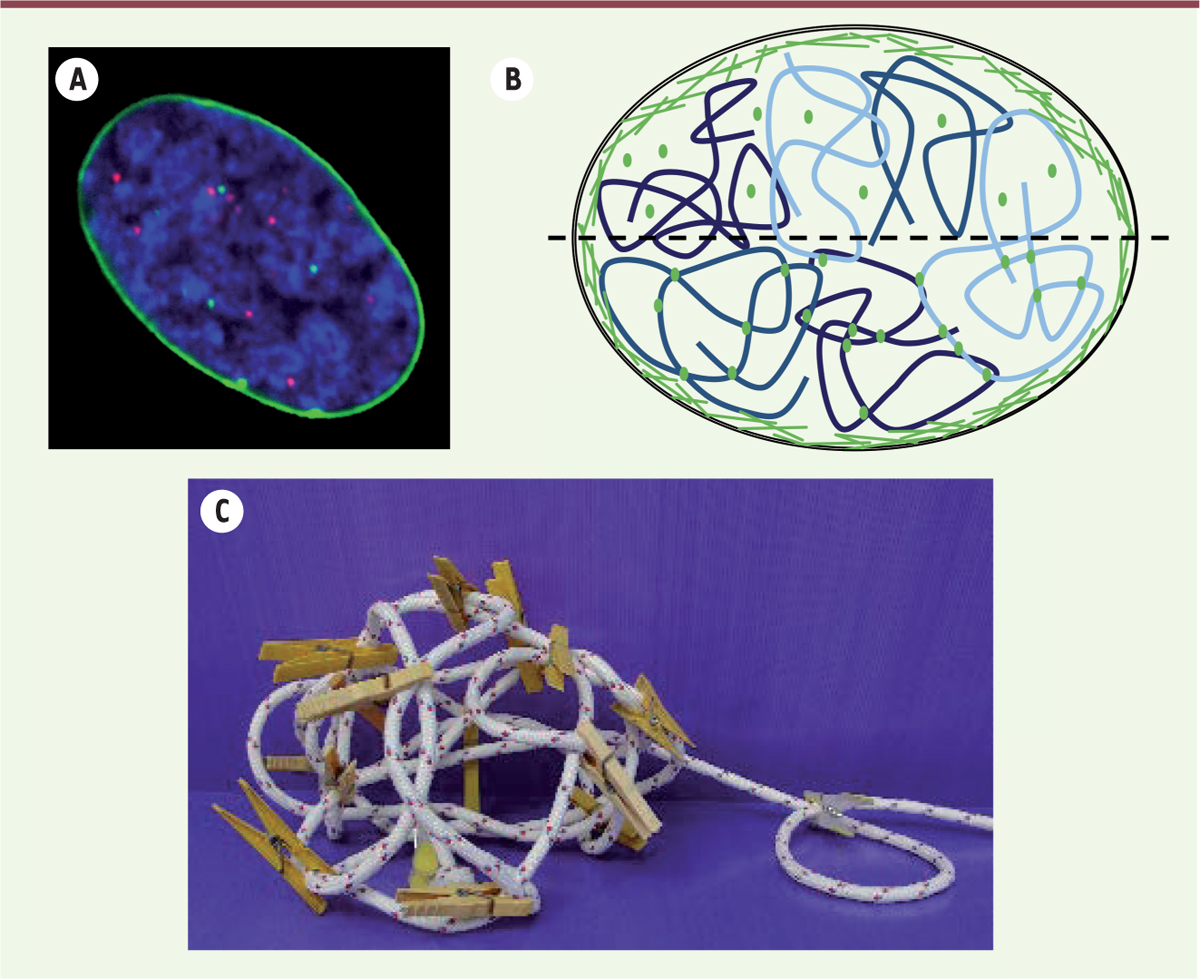
 | Figure 1.
La lamine A dans le noyau : observation et modèles. A. Image en microscopie de florescence d’un noyau de fibroblaste embryonnaire de souris (MEF) contenant de la pré-lamine A 4 marquée avec de la eGFP ( enhanced green fluorescent protein ; vert). Les télomères sont localisés grâce au marquage du TRF1 ( telomeric repeat factor 1) marqué par la DsRed ( Discosoma sp. red fluorescent protein ; rouge), et l’ADN est coloré par du Hoechst 33258 (bleu). La lamine A est principalement localisée à la périphérie nucléaire tandis que les télomères sont dispersés dans le noyau. L’image montre un seul plan z et seulement quelques télomères sont visibles (image © Yuval Garini). B. Illustration du modèle de « lamine A libre » (hémisphère supérieur) et du modèle de « lamine A liée » (hémisphère inférieur). Dans les 2 modèles, la majorité de la lamine A est située à la périphérie nucléaire sous forme de filaments (lignes vertes). Cependant, comme Bronshtein et al. le montrent dans leur étude [ 6], il existe une portion de lamine A figée à l’intérieur du noyau, jouant le rôle de cross-linkers6 et affectant de manière très importante la dynamique de l’ADN (représenté en bleu). Ces résultats renforcent ainsi le modèle de la « lamine A liée ». C. Illustration du modèle de la « lamine A liée » : quelques cross-linkers ou nœuds de réticulation (pinces à linge) suffisent à créer une structure rigide à partir d’un polymère flexible représenté par une corde (image adaptée de Bronshtein et al. [ 6]). |
À la périphérie nucléaire, la lamine A se trouve sous forme de filaments (Figure 1B). Cependant, une quantité importante de lamine A est également localisée à l’intérieur du noyau [12] ; son rôle reste inconnu et, même s’il est possible d’observer la lamine A par microscopie sous forme de fond diffus dans le noyau, sa structure et ses interactions potentielles avec la chromatine n’ont jamais été élucidées [8]. Deux modèles non exclusifs ont été proposés : (1) la lamine A diffuserait à l’intérieur du noyau sans interagir avec la chromatine (
Figure 1B
, hémisphère supérieur) ; (2) la lamine A serait attachée à la chromatine et lui servirait de matrice structurante (
Figure 1B
, hémisphère inférieur). Comme nous allons le détailler dans le reste de cette Nouvelle, l’étude de Bronshtein et al. montre qu’en l’absence de lamine A, la chromatine passe d’une diffusion lente et très localisée à une diffusion libre, soulignant le rôle clé de la lamine A dans l’organisation dynamique du génome [6]. Les résultats de cette équipe montrent également que 40 % de la lamine A se trouvant à l’intérieur du noyau serait en fait immobile. Cette lamine pourrait ainsi être liée à la chromatine de façon à freiner sa dynamique, comme proposé dans le 2e modèle (
Figure 1B, hémisphère inférieur ;
Figure 1C
). |
Comprendre la diffusion de la chromatine dans le noyau de cellules L’attachement des télomères à la lamina nucléaire contraint leur diffusion. Il est cependant peu probable que cet attachement à lui seul puisse contraindre la chromatine à l’intérieur du noyau. Un ou plusieurs autres mécanismes permettent donc de contrôler la dynamique du génome dans le noyau. Un de ces mécanismes pourrait impliquer la lamine A, dont la fonction à l’intérieur du noyau était jusqu’à présent inconnue. Bronshtein et al. ont ainsi testé son rôle potentiel dans le maintien de la dynamique chromatinienne au sein du noyau [6]. Afin d’élucider le rôle de la lamine A, les auteurs de cette étude ont caractérisé la diffusion de différents locus marqués par fluorescence dans des cellules mammifères vivantes. Grâce à l’évolution récente des techniques de microscopie et d’analyse d’images, il est en effet possible de suivre en temps réel le mouvement d’une région génomique spécifique, appelée locus, à l’intérieur du noyau de cellules vivantes. Le mouvement d’un locus peut être déterminé à partir de ses positions au cours du temps en calculant une fonction mathématique appelée déplacement quadratique moyen3 (mean square displacement, MSD). La forme de la MSD révèle alors le type de diffusion du locus observé. Lorsqu’un locus évolue librement dans le noyau, son mouvement est une « diffusion brownienne3 libre » aussi appelée « diffusion normale ». À l’inverse, lorsqu’un locus est ralenti par l’environnement qui l’entoure, on observe une sous-diffusion. Un mouvement sous-diffusif indique par exemple qu’il existe des interactions entre les constituants du nucléoplasme et le locus étudié, empêchant ainsi la chromatine de bouger librement en freinant son mouvement. Lorsqu’un mouvement est caractérisé par l’équation MSD = A tα + b, la diffusion est qualifiée d’anormale. Le coefficient anormal α permet de quantifier la contrainte exercée par le milieu entourant le locus observé : pour un mouvement sous-diffusif, α est toujours compris entre 0 et 1, 1 indiquant une diffusion libre. Une faible valeur de α indique que le locus, même s’il se déplace, a tendance à revenir proche de sa position précédente, restant ainsi longtemps dans le même environnement et interagissant souvent avec les molécules voisines ; au contraire, une valeur de α élevée indique qu’un locus est capable d’explorer de nouvelles régions. Dans les cellules mammifères, la diffusion de la chromatine est anormale, avec des coefficients variant de 0,4 à 0,7 selon la position des locus observés [6, 13] (Figures 2A et 2B). Ce type de diffusion reflète un mouvement très lent et localisé : typiquement, cela prendrait plusieurs jours à un locus pour diffuser à travers un territoire chromosomique faisant plus de 1 µm.
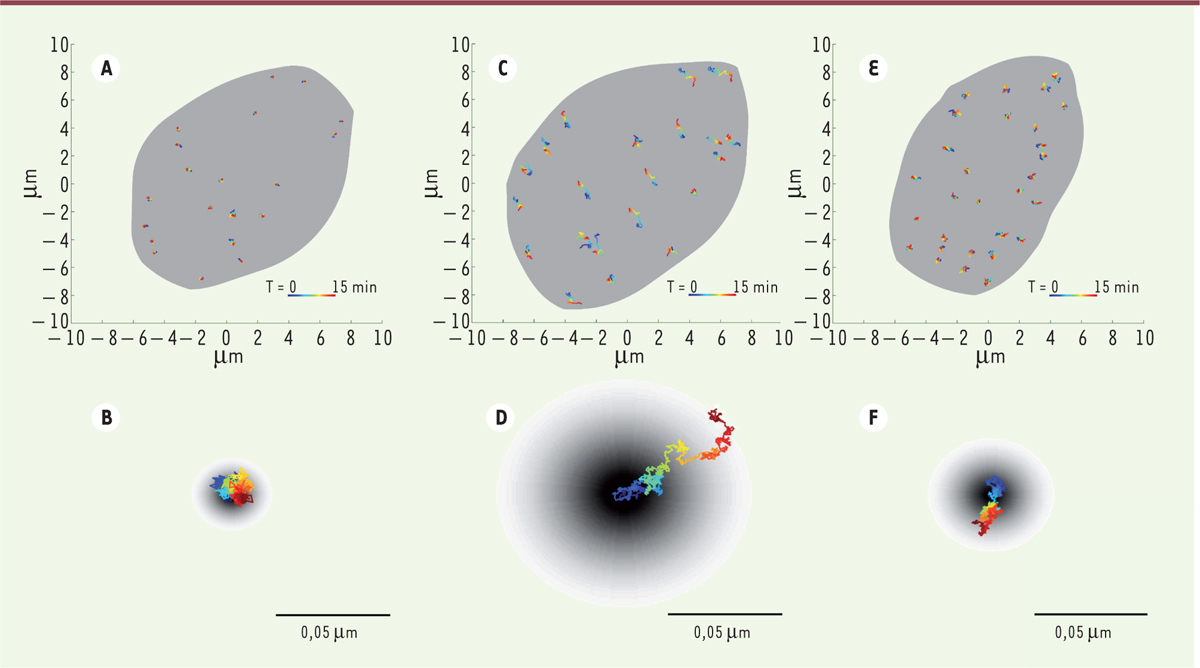
 | Figure 2.
Exemples de trajectoires de télomères dans 3 types de lignées cellulaires. A. Trajectoires de télomères dans des fibroblastes embryonnaires de souris (MEF) Lmna
+/+ ayant un niveau normal de lamine A. Ces télomères présentent une diffusion lente et localisée : leur mouvement est sous-diffusif. B. Agrandissement des trajectoires de télomères dans des cellules Lmna
+/+. C-D. Trajectoires de télomères dans des cellules de fibroblastes embryonnaires de souris (MEF) Lmna
-/- (sans lamine A). Ces télomères présentent une diffusion rapide normale sur de grandes distances. E-F. Trajectoires de télomères dans des fibroblastes embryonnaires de souris (MEF) Lmna
-/- transitoirement transfectés avec un plasmide codant pour le mutant de lamine A N195K. Un retour partiel à la dynamique relevée dans les cellules des lignées Lmna
+/+ est observé, indiquant que ce mutant ne compense que partiellement l’absence de lamine A. Des images de toutes les cellules ont été observées pendant 15 minutes ; les trajectoires sont colorées de bleu à rouge en fonction du temps. Le fond gris dans les parties A, C et E indique l’ensemble du noyau (images ©Yuval Garini). |
|
En l’absence de lamine A, la chromatine diffuse librement dans le noyau Afin de tester la fonction de la lamine A dans l’organisation de la chromatine, Bronshtein et al. ont mesuré la diffusion de plusieurs locus dans différents types cellulaires en l’absence de lamine A. L’effet obtenu est spectaculaire : dans des cellules de fibroblastes embryonnaires de souris déplétées en lamine A (MEF Lmna-/-), la diffusion des télomères devient libre pour des temps supérieurs à 7 secondes, avec un coefficient α atteignant 1 ± 0,2 (Figures 2C et 2D). Le même effet est visible dans des cellules U2OS dans lesquelles l’expression de la lamine A a été réduite par siARN (small interfering RNA). Ce changement de mode de diffusion de la chromatine est réversible puisqu’après transfection de pré-lamine A4 dans des cellules MEF Lmna-/-, le mouvement de la chromatine redevient anormal (α = 0,6 ± 0,1). Il faut souligner que l’absence de lamine A affecte aussi bien la dynamique des télomères que celle des centromères [6]. Les télomères interagissant avec la lamine A au sein de la lamina nucléaire, un changement de mobilité de ces derniers en l’absence de lamine A était attendu. Cependant, le détachement de ces télomères à la membrane nucléaire ne permet pas, à lui seul, d’expliquer la mobilité des autres sites du génome testés, en particulier les centromères. Bronshtein et al. ont donc proposé une nouvelle fonction de la lamine A présente au sein du noyau : celle-ci pourrait interagir directement ou indirectement avec la chromatine en créant des liaisons structurantes entre fibres de chromatine, comme illustré dans la
Figure 1C
. |
Une nouvelle fonction pour la lamine A : le contrôle de la dynamique chromatinienne à l’intérieur du noyau Afin de tester les interactions possibles entre la lamine A et la chromatine au sein du noyau, Bronshtein et al. ont utilisé une technique de microscopie appelée photo-blanchiment en continu5,. Ils ont montré que 40 % de la lamine A se trouvant à l’intérieur du noyau est immobile [6]. Cette fraction de lamine A pourrait alors interagir directement ou indirectement avec la chromatine en freinant sa diffusion de manière à passer d’une diffusion libre à une diffusion anormale (Figure 1B). Afin de mieux comprendre la nature des interactions entre la lamine A et la chromatine, ces chercheurs ont ensuite testé la mobilité de différents locus en présence de lamine A mutante (lignées cellulaires Lmna-/- transfectées avec un mutant de la lamine). En présence de lamine A mutée, on observe une diffusion anormale assez rapide (0,61 < α < 0,85) et non une diffusion libre, comme c’était le cas en l’absence totale de lamine A (Figure 2E et F). Les mutants de lamine A sont ainsi capables de contraindre l’organisation dynamique de la chromatine, mais seulement de manière partielle. Les mutations testées se situent dans le domaine « tête » (mutants L85R et N195K) ou « queue » (mutants R482W et L530) de la lamine A, indiquant que ces domaines ont un rôle essentiel dans le maintien de l’organisation du génome. Le domaine « tête » intervient dans la dimérisation de la lamine A, alors que le domaine « queue » interagit directement avec les histones et l’ADN. Ces résultats suggèrent donc l’existence d’un mécanisme de liaison entre la chromatine et les oligomères de lamine A [6]. |
L’équipe de Yuval Garini a mis à jour une nouvelle fonction de la lamine A dans la régulation de la dynamique de la chromatine, avec un rôle essentiel des domaines « tête » et « queue » de la lamine A [6]. Leur modèle présente une vision nouvelle de la chromatine, proposant qu’une structure rigide puisse émerger à partir d’un polymère flexible dont la diffusion est contrainte par des liaisons transverses6
(Figure 2C). Leurs observations ont été validées dans les différents types cellulaires testés. De nouvelles questions quant à la fonction de la lamine A se posent désormais. Il sera intéressant d’étudier son rôle au cours du cycle cellulaire, lorsque les chromosomes se condensent puis se décondensent, ainsi qu’à différents stades de différenciation. L’étude des relations entre la lamine A et la cohésine ou la condensine, qui jouent aussi un rôle fondamental dans l’organisation du génome, est également un défi excitant pour de futures recherches [14]. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Ce travail a été financé par l’ANR-12-PDOC-0035-01 (J.M.H.) et par une bourse EMBO Long-Term Fellowhip (E.K). Nous remercions Antoine Coulon pour ses commentaires et discussions sur ce travail.
|
Footnotes |
1.
Mirny LA. The fractal globule as a model of chromatin architecture in the cell . Chromosome Res. 2011; ; 19 : :37.–51. 2.
Nicodemi M, Pombo A. Models of chromosome structure . Curr Opin Cell Biol. 2014; ; 28 : :90.–95. 3.
Bickmore WA, van Steensel B. Genome architecture: domain organization of interphase chromosomes . Cell. 2013; ; 152 : :1270.–1284. 4.
Nickerson J. Experimental observations of a nuclear matrix . J Cell Sci. 2001; ; 114 : :463.–474. 5.
Pienta KJ, Coffey DS. A structural analysis of the role of the nuclear matrix and DNA loops in the organization of the nucleus and chromosome . J Cell Sci. 1984; ; 1 ((suppl)) : :123.–135. 6.
Bronshtein I, Kepten E, Kanter I, et al. Loss of lamin A function increases chromatin dynamics in the nuclear interior . Nat Commun. 2015; ; 6 : :8044.. 7.
Pajerowski JD, Dahl KN, Zhong FL, et al. Physical plasticity of the nucleus in stem cell differentiation . Proc Natl Acad Sci USA. 2007; ; 104 : :15619.–15624. 8.
Dechat T, Adam SA, Taimen P, et al. Nuclear lamins . Cold Spring Harb Perspect Biol. 2010; ; 2 : :a000547.. 9.
Mounkes L, Kozlov S, Burke B, Stewart CL. The laminopathies: nuclear structure meets disease . Curr Opin Genet Dev. 2003; ; 13 : :223.–230. 10.
Gilchrist S, Gilbert N, Perry P, et al. Altered protein dynamics of disease-associated lamin A mutants . BMC Cell Biol. 2004; ; 5 : :46.. 11.
Navarro CL, Poitelon Y, Lévy N. Lamines A et syndromes progéroïdes : une farnésylation persistante aux conséquences dramatiques . Med Sci (Paris). 2008; ; 24 : :833.–840. 12.
Moir RD, Yoon M, Khuon S, Goldman RD. Nuclear lamins A and B1: different pathways of assembly during nuclear envelope formation in living cells . J Cell Biol. 2000; ; 151 : :1155.–1168. 13.
Barkai E, Garini Y, Metzler R. Strange kinetics of single molecules in living cells . Phys Today. 2012; ; 65 : :29.–35. 14.
Fudenberg G, Imakaev M, Lu C, et al. Formation of chromosomal domains by loop extrusion . Cell Rep. 2016; ; 15 : :2038.–2049. |