| |
| Med Sci (Paris). 33(5): 484–485. doi: 10.1051/medsci/20173305008.Le dixième gène du VIH Élodie Cassan,1,2 Anne-Muriel Arigon-Chifolleau,1 Jean-Michel Mesnard,2 Antoine Gros,2 and Olivier Gascuel1,3* 1IBC-LIRMM, CNRS et Université de Montpellier, France 2CPBS, CNRS et Université de Montpellier, France 3C3BI, CNRS et Institut Pasteur, Paris, France |
Les manuels de biologie ont longtemps écrit que le génome du virus de l’immunodéficience humaine (VIH) était composé de neuf gènes : les trois gènes rétroviraux structuraux (gag, pol et env) et un ensemble de six gènes régulateurs ou accessoires. Suggérée à la fin des années 1980 [1], l’existence d’un dixième gène, asp, restait débattue. Nous venons de confirmer sa présence dans le groupe pandémique du VIH, en analysant près de 23 000 séquences du VIH-1 et du SIV (virus d’immunodéficience simien) [2, 3]. (→).
(→) Voir la Synthèse de M. Peeters et al., m/s n°6-7, juin-juillet 2008, page 621
|
Brin sens et brin anti-sens Le VIH est un rétrovirus. Son matériel génétique est contenu dans un ARN qui est rétrotranscrit en ADN puis intégré dans l’ADN génomique de la cellule infectée. Ce génome proviral a deux brins : le brin sens et le brin anti-sens, l’ensemble formant la double hélice de l’ADN. On a longtemps pensé que, chez les rétrovirus, la production de la totalité des protéines virales dépendait de l’expression du transcrit du brin sens initié par un promoteur de la transcription contenu dans le LTR (long terminal repeat) 5’. L’existence d’une transcription anti-sens chez les rétrovirus a été mise en évidence au tournant des années 2000, notamment grâce à l’étude d’une protéine virale chez le virus HTLV-1 (human T cell leukemia/lymphoma virus type 1). Cette protéine nommée HBZ (basic leucine zipper) est codée par un transcrit anti-sens initié depuis le LTR 3’ [4]. Cette découverte a relancé l’intérêt pour les gènes situés sur le brin anti-sens, en particulier pour le gène asp, situé en chevauchement du gène env sur le brin anti-sens du génome proviral du VIH, d’où son nom : antisense protein gene. Nous avons réalisé la première étude évolutive et bioinformatique à grande échelle du gène asp [2]. |
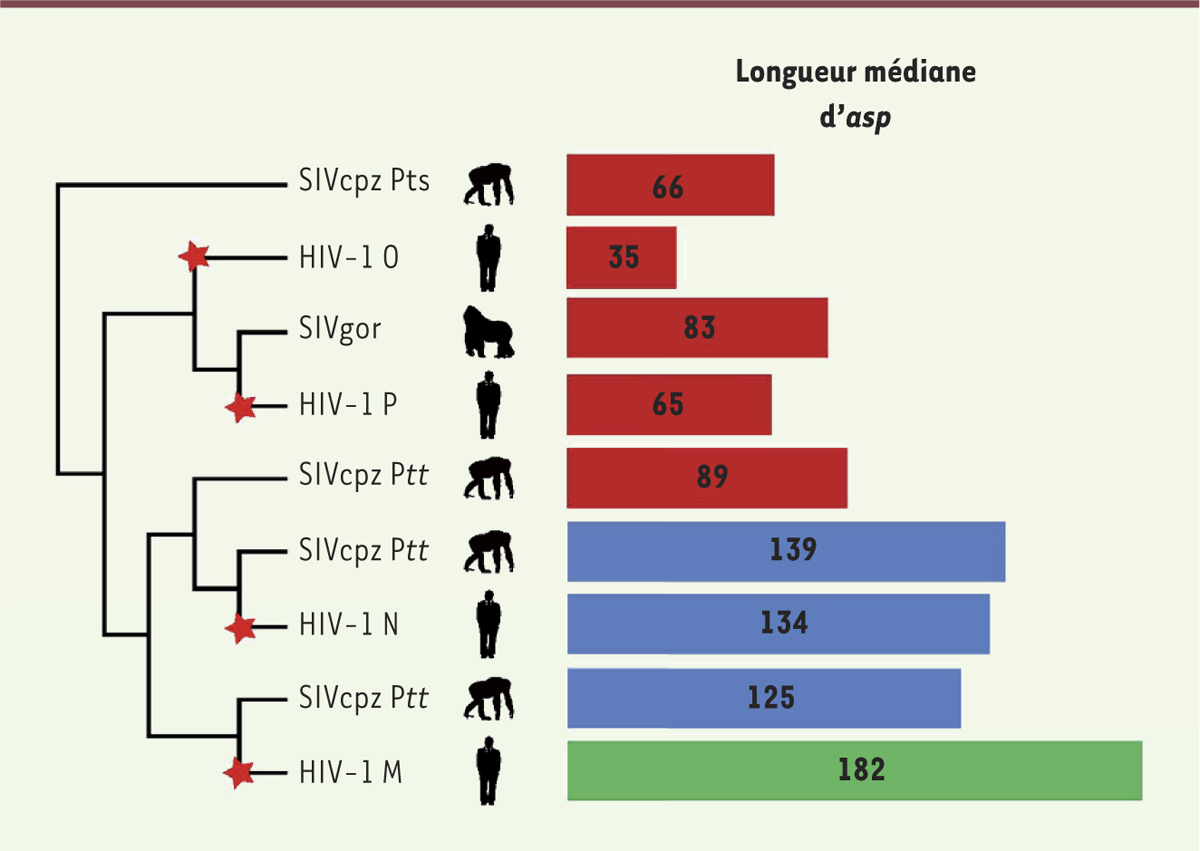
Conservation et évolution du gène asp L’objectif de notre étude était d’analyser la conservation et l’évolution d’asp depuis l’origine de l’introduction du VIH-1 chez l’homme. Nous avons étudié près de 23 000 séquences du virus, représentant l’ensemble des séquences disponibles dans les banques de données publiques, afin de déterminer si le gène asp était conservé dans les différents groupes du VIH-1 et de son équivalent chez le singe, le SIV. Nous avons ainsi montré que le gène asp n’existe que dans le groupe de virus responsable de la pandémie humaine, le groupe M (pour majeur) du VIH-1. Le gène asp pourrait donc jouer un rôle dans la transmission du virus. Une analyse phylogénétique montre également (Figure 1) que son apparition est concomitante à l’émergence du VIH-1 chez l’homme, datée vers le début du xx
e siècle [5].
 | Figure 1.
Arbre phylogénétique montrant la propagation du VIH-1 (virus de l’immunodéficience humaine) à l’homme. Les introductions des groupes O, P, N (très peu prévalents) et M (à l’origine de la pandémie) sont indiquées par une étoile rouge. En regard de chaque groupe, pour l’ensemble des séquences disponibles, la longueur médiane d’asp est indiquée en nombre de codons compris entre le début (start) et le premier stop. Le gène asp s’est allongé en se rapprochant du groupe M, pour dépasser 180 codons dans ce groupe, ce qui ne peut pas être observé par hasard. Le gène est alors devenu fonctionnel et utile au virus, la pression de sélection agissant pour maintenir son intégrité. SIV : virus d’immunodéficience simien ; cpz : chimpanzé ; Ptt : Pan troglodytes troglodytes ; Pts : Pan troglodytes schweinfurthii ; gor : gorille. |
|
La difficulté concernant l’étude d’asp venait principalement du fait que ce gène est chevauchant. Il est en chevauchement avec le gène env qui code les protéines de l’enveloppe virale. Le code génétique est redondant : dans les gènes non-chevauchants, chaque codon (séquence de trois nucléotides) code un unique acide aminé, et la plupart des acides aminés peuvent être codés par plusieurs codons différents. La redondance du code génétique est à la base des approches classiques pour étudier la pression de sélection. Avec les gènes chevauchants, au contraire, une même portion de séquence code deux (voire trois) protéines. Comme si un mot avait plusieurs sens possibles lorsqu’on décale le début de la lecture d’une à deux lettres, ou qu’on le lit à l’envers. Le codage de l’information génétique est alors très contraint : les deux protéines chevauchantes sont fortement corrélées et de nombreuses combinaisons sont impossibles. De ce fait, les approches bioinformatiques classiques sont largement inopérantes. |
Afin de mettre en évidence la présence d’une pression de sélection, il a été nécessaire de développer une nouvelle méthode originale fondée sur l’étude fine des codons d’arrêt (stop). Les codons stop correspondent à des signaux d’arrêt dans le mécanisme de synthèse des protéines et ne sont donc pas présents à l’intérieur des gènes. Nous avons montré que dans le cas du gène asp, certains stop étaient « obligatoires » et imposés par la protéine chevauchante ENV, alors que d’autres étaient « potentiels » et pouvaient apparaître ou disparaître sans que ENV ne soit modifiée. En l’absence de pression de sélection, on s’attendait à ce que la présence effective de ces stop potentiels soit aléatoire dans la région d’asp. À l’inverse, si le gène et la protéine ASP sont fonctionnels et utiles au virus, on s’attend à ce que les stop potentiels soient systématiquement évités. En analysant les 23 000 séquences, nous avons montré que les stop potentiels sont effectivement évités dans le groupe M du VIH-1 à l’origine de la pandémie humaine, alors que ces codons stop sont présents aléatoirement dans les autres groupes du VIH-1 ainsi que chez les SIV des singes. Ce résultat indique donc une forte pression de sélection. D’autres résultats sont venus conforter cette analyse. En utilisant des simulations informatiques, nous avons montré que le taux de présence très élevé d’asp au sein du groupe pandémique M, a une probabilité quasi-nulle d’être obtenu par hasard. Nous avons également observé que dans certains sous-types viraux, une mutation ayant fait disparaître asp avait été compensée par une nouvelle mutation permettant quand même la synthèse de la protéine ASP. |
La particularité du positionnement du gène asp, en anti-sens et en chevauchement du gène de l’enveloppe virale, ne permettait pas jusqu’à présent de caractériser une pression évolutive avec les méthodes de bioinformatique conventionnelles. Les avancées en bioinformatique évolutive ont permis de mettre en évidence la pression de sélection en faveur du maintien du gène asp dans le groupe M. Nous avons aussi montré qu’il existe une forte corrélation entre la présence d’asp et la prévalence des groupes et sous-types du VIH-1. La fonction d’ASP dans la multiplication virale ainsi que son rôle exact dans la pandémie de SIDA (syndrome d’immunodéficience acquise) restent pour autant largement inconnus. À la suite de cette étude, de nouvelles recherches devraient émerger, visant à comprendre le rôle d’ASP dans l’infection par le VIH-1. Ce gène supplémentaire représente une nouvelle cible thérapeutique potentielle, particulièrement sensible en raison de son chevauchement avec le gène env. Les méthodes bioinformatiques qui ont été développées devraient permettre également l’analyse systématique d’autres virus pour lesquels des gènes chevauchants seraient passés inaperçus ou nécessiteraient des confirmations évolutives. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Miller R H. Human immunodeficiency virus may encode a novel protein on the genomic DNA plus strand . Science. 1988; ; 239 : :1420.–1422. 2.
Cassan E, Arigon-Chifolleau AM, Mesnard JM, et al. Concomitant emergence of the antisense protein gene of HIV-1 and of the pandemic . Proc Natl Acad Sci U S A. 2016; ; 113 : :11537.–11542. 3.
Peeters M, Chaix ML, Delaporte E Phylogénie des SIV et des VIH . Med Sci (Paris). 2008; ; 24 : :621.–628. 4.
Gaudray G, Gachon F, Basbous J, et al. The complementary strand of the human t-cell leukemia virus type 1 rna genome encodes a bzip transcription factor that down-regulates viral transcription . J Virol. 2002; ; 76 : :12813.–12822. 5.
Faria NR, Rambaut A, Suchard MA, et al. The early spread and epidemic ignition of HIV-1 in human populations . Science. 2014; ; 346 : :56.–61. |