| |
| Med Sci (Paris). 33(6–7): 591–593. doi: 10.1051/medsci/20173306011.Des gènes synthétiques à l’assaut du cancer Vers des traitements plus efficaces grâce à des programmes génétiques autonomes Mathieu Morel1* 1Laboratoire PASTEUR, département de chimie, École normale supérieure, PSL Research University, Sorbonne Universités, UPMC-Université Paris 6, CNRS, 75005Paris, France |
Les promesses d’une édition simple du génome, récemment apportées par la technologie CRISPR-Cas9 [1], a replacé la thérapie génique au centre des débats scientifiques et éthiques [2] (→). Si ces ciseaux moléculaires s’avèrent très puissants et simples d’utilisation, ils ne sont que des outils. Les questions restent ouvertes quant à notre capacité à reprogrammer le génome ou quant à la fonctionnalité des séquences que l’on peut désormais insérer. Depuis plus de quinze ans, des laboratoires travaillent au développement de réseaux de gènes synthétiques [3], (→) c’est-à-dire à l’établissement des « programmes » génétiques permettant la lecture en temps réel de l’état d’une cellule, au traitement de ces informations et au déclenchement d’une réponse adaptée [4, 5]. L’intégration d’activités moléculaires spécifiques permet une prise de décision autonome de ces « programmes » en fonction des niveaux d’activités cibles détectés, haut ou bas. Insérés dans le génome d’une cellule, ces circuits génétiques peuvent déclencher une réponse faisant suite à ce diagnostic en cas de dérèglement, ou même provoquer la mort cellulaire lorsque plusieurs marqueurs pathologiques sont activés. Bien que ces approches soulèvent encore de nombreuses questions éthiques, l’introduction de ces circuits est susceptible de permettre, néanmoins, le développement d’une thérapie génique de deuxième génération avec des « programmes » ciblant plus spécifiquement les cellules pathologiques et s’adaptant à l’évolution d’une maladie. Ce domaine est donc très prometteur pour le traitement de cancers, de maladies auto-immunes, ou de dérèglements métaboliques [6]. Dans le cas d’une tumeur cancéreuse, les augmentations d’activités liées à la malignité peuvent être détectées, intégrées et analysées par le « programme » afin de décider de la mort de ces cellules de manière autonome. Cependant, l’efficacité de ces réseaux de gènes synthétiques reste encore à optimiser, surtout pour des applications de ciblage thérapeutique qui demande une grande spécificité de la réponse. Les chercheurs se sont inspirés de l’électronique numérique afin de concevoir des circuits génétiques sur un principe binaire, c’est-à-dire détectant des signaux ou donnant une réponse du type 0 ou 1. Or les signaux biologiques ne peuvent pas être limités à cette description binaire : ils sont soumis à une variabilité due aux fluctuations moléculaires. Nos récents travaux montrent ainsi que la variabilité intrinsèque du circuit et celle des niveaux d’entrées, due à l’hétérogénéité cellulaire, vont induire des limitations sur l’efficacité du ciblage thérapeutique, à la fois en terme de sensibilité ou de spécificité optimale [7].
(→) Voir la Nouvelle de H. Gilgenkrantz, m/s n° 12, décembre 2014, p. 1066
(→) Voir la Chronique génomique de B. Jordan, m/s n° 6–7, juin-juillet 2016, page 651
|
Des circuits génétiques inspirés de l’électronique Sous la direction du Pr Bar-Ziv de l’Institut Weizmann, nous avons mis au point des circuits génétiques synthétiques fonctionnant sur le principe simple de la porte logique « ET » utilisée en électronique numérique : ceux-ci renvoient une réponse positive uniquement si les deux signaux d’entrées sont détectés comme positifs [11] (→). La qualité de détection de tels circuits est améliorée du fait de la validation simultanée de la lecture de deux activités cibles, limitant ainsi le risque de faux positifs. Ces circuits génétiques, appelés dual promoter integrator (DPI, intégrateurs à doubles promoteurs), lisent deux activités transcriptionnelles grâce à des copies de promoteurs endogènes. Ils utilisent ensuite le principe du double-hybride afin de former un facteur de transcription synthétique lorsque la cellule présente les deux activités cibles simultanément [8, 9] (Figure 1). Le circuit est ainsi constitué de trois éléments : deux séquences « entrées » contenant chacune une copie d’un des promoteurs cibles et une partie du dimère de transcription synthétique ; une séquence « sortie » contenant les gènes effecteurs, rapporteur fluorescent et/ou gène tueur, sous le contrôle du facteur de transcription synthétique formé. Il est également possible de modifier les paramètres internes de ce circuit afin d’amplifier le signal de sortie ou de changer les seuils de déclenchement d’une réponse positive. Une étude sur un grand nombre de cellules uniques, faisant appel à des rapporteurs fluorescents et à des analyses en cytométrie en flux, a permis de mesurer la fonction de réponse de ces circuits (le niveau de sortie en fonction des niveaux d’entrées). Nous avons ainsi obtenu une cartographie précise du fonctionnement de ces portes « ET » biologiques, grâce à l’obtention d’une réponse si les deux entrées atteignaient un niveau suffisant, mais montrant également une variabilité du seuil de déclenchement de la porte ainsi qu’un signal de sortie bruité autour d’un niveau moyen. L’ajout d’un module amplificateur aide à séparer proprement les niveaux bas et hauts de la sortie mais amplifie également le bruit interne : on obtient alors des circuits très sensibles pour détecter des cellules cancéreuses, mais qui augmentent les erreurs commises sur les niveaux bas, et donc peu spécifiques. Au contraire, en utilisant un module de faible intégration des entrées, une plus grande activité des entrées est nécessaire pour activer la sortie : on améliore la spécificité du circuit, mais au détriment de sa capacité à détecter toutes les cellules cancéreuses. De manière générale, l’hétérogénéité cellulaire impose de faire un compromis entre une grande sensibilité (la capacité à détecter toutes les cellules cancéreuses, mais également des cellules saines) et une meilleure spécificité du circuit (la capacité à cibler uniquement les cellules cancéreuses, mais au risque d’en manquer une partie).
 | Figure 1.
Principe du circuit génétique synthétique DPI (intégrateurs à doubles promoteurs). Des facteurs de transcription endogènes surexprimés dans une cellule cancéreuse déclenchent une réponse positive du circuit : l’expression de protéines fluorescentes ou d’enzymes de conversion d’une pro-drogue. DPI : dual promoter integrator. |
(→) Voir la Synthèse de M. Madec et al., m/s n° 2, février 2017, page 159
|
Vers des traitements plus spécifiques ? Nous avons développé un modèle in vitro de ciblage thérapeutique d’une tumeur du poumon afin d’optimiser la sensibilité et la spécificité des circuits dans un environnement plus représentatif de l’hétérogénéité cellulaire. L’étude a été réalisée à partir de co-cultures de cellules de deux lignées de même origine (cellules fœtales du poumon WI-38) mais présentant des niveaux de malignité différents [10] : des cellules à la malignité agressive, représentant une « tumeur », dans un environnement « sain » de cellules pré-malignes. Dans ces cellules cancéreuses, les circuits détectent une forte activité des facteurs de transcription responsables de la production d’histone H2A.1 et de la protéine SSX1 (synovial sarcoma X) impliquée dans le sarcome synovial, et déclenchent alors la réponse : l’expression d’une protéine fluorescente et de la protéine TK1-HSV (thymidine kinase type 1 de l’herpès simplex virus), qui peut induire la mort cellulaire en cas d’ajout d’une pro-drogue. Au contraire, dans les cellules pré-malignes, la plus faible activité des entrées ne permet pas de déclencher la réponse du circuit. Il en résulte une forte différence du niveau d’expression du gène tueur TK1-HSV entre la tumeur et son environnement sain, avec pour conséquence un traitement plus spécifique lors de l’ajout de la pro-drogue (Figure 2). Les paramètres internes du circuit, amplification et seuil, peuvent alors être optimisés afin d’obtenir un ciblage plus sensible ou au contraire plus spécifique en fonction du traitement utilisé, du type de cancer ou de son environnement.
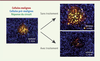 | Figure 2.
Ciblage thérapeutique de cellules de tumeur du poumon. L’évolution des « tumeurs » (en rouge, sur l’image de gauche à 5 jours) est suivie par microscopie de fluorescence pendant environ un mois (images de droite à 15 jours). Sans ajout de la pro-drogue, les tumeurs se développent au détriment du « tissu sain » (en bleu), alors qu’avec un traitement conditionné à la réponse du circuit (en vert/jaune), elles régressent sans perturber le tissu environnant. |
Notre étude reste limitée à des systèmes biologiques simples : elle devra être confirmée par des modèles plus complexes intégrant des cultures en 3-dimensions ou des biopsies. Ces circuits devront in fine être confrontés au ciblage de tumeurs in vivo. L’efficacité de ces circuits pourrait également être améliorée grâce à l’utilisation de signaux d’entrées moins bruités, par exemple des promoteurs endogènes ou synthétiques plus spécifiques des cellules cibles. Ce travail souligne néanmoins l’importance de l’analyse de la variabilité cellulaire et de la reconstruction de microenvironnements pour le développement et l’optimisation de circuits de ciblage à de véritables fins thérapeutiques. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Doudna JA, Charpentier E. The new frontier of genome engineering with CRISPR-Cas9 . Science. 2014; ; 346 : :1258096.. 2.
Gilgenkrantz H. La révolution des CRISPR est en marche . Med Sci (Paris). 2014; ; 30 : :1066.–1069. 3.
Jordan B. Synthétique, vous avez dit « Synthétique » ? . Med Sci (Paris). 2016; ; 32 : :651.–653. 4.
Rinaudo K, Bleris L, Maddamsetti R, et al. A universal RNAi-based logic evaluator that operates in mammalian cells . Nat Biotechno. 2007; ; 25 : :795.–801. 5.
Ausländer S, Ausländer D, Müller M, et al. Programmable single-cell mammalian biocomputers . Nature. 2012; ; 487 : :1.–6. 6.
Weber W, Fussenegger M. Emerging biomedical applications of synthetic biology . Nat Re Genet. 2011; ; 13 : :21.–35. 7.
Morel M, Shtrahman R, Rotter V, et al. Cellular heterogeneity mediates inherent sensitivity-specificity tradeoff in cancer targeting by synthetic circuits . Proc Natl Acad Sci U S A. 2016; ; 113 : :8133.–8138. 8.
Nissim L, Beatus T, Bar-Ziv R. An autonomous system for identifying and governing a cell’s state in yeast . Phys Biol. 2007; ; 4 : :154.–163. 9.
Nissim L, Bar-Ziv RH. A tunable dual-promoter integrator for targeting of cancer cells . Mol Syst Biol. 2010; ; 6 : :444.. 10.
Milyavsky M, Tabach Y, Shats I, et al. Transcriptional programs following genetic alterations in p53, INK4A, and H-Ras genes along defined stages of malignant transformation . Cancer Res. 2005; ; 65 : :4530.–4543. 11.
Madec M, Haiech J, Rosati E, et al. Application à la biologie synthétique des méthodes et outils de CAO de la microélectronique . Med Sci (Paris). 2017; ; 33 : :159.–168. |



