| |
| Med Sci (Paris). 33(6–7): 613–619. doi: 10.1051/medsci/20173306018.La synthèse des ribosomes, au cœur du contrôle de la prolifération cellulaire Clément Madru,1 Nicolas Leulliot,1 and Simon Lebaron1,2* 1Laboratoire de Cristallographie et RMN Biologiques, UMR, CNRS 8015, Université Paris Descartes, Faculté de Pharmacie, Sorbonne Paris Cité, 75006Paris, France 2Institut national de la santé et de la recherche médicale, Paris, France |

Vignette (Photo © Inserm - Patrick Delapierre). Les ribosomes assurent la traduction des ARN messagers (ARNm) en protéines. Ils sont ainsi au cœur de l’expression génique. La stabilité structurale et la fidélité du système de traduction expliquent certainement la conservation du ribosome au cours de l’évolution. Son utilisation est commune à l’ensemble des cellules vivantes. Ces macro-particules sont formées de deux sous-unités dénommées chez les eucaryotes, petite ou 40S et grande ou 60S. Chez l’homme, la petite sous-unité 40S est composée d’un ARN ribosomique (ARNr) appelé 18S, associé à 33 protéines ribosomiques (RPS pour ribosomal protein of the small subunit). La grande sous-unité est constituée de 3 ARNr (5S, 5,8S et 28S) et de 47 protéines (RPL pour ribosomal protein of the large subunit). Les ARNr sont au centre de l’activité du ribosome, ils définissent sa structure globale et supportent l’activité peptidyl-transférase qui catalyse la formation des liaisons peptidiques. Dans les cellules eucaryotes, la production de ribosomes matures suit un processus complexe et hautement régulé. En effet, la voie de synthèse dédiée à la biogenèse des ribosomes est très élaborée, marquée par une forte production et par conséquent excessivement coûteuse en énergie [1]. Ce processus fait donc l’objet de nombreux contrôles et régulations. La synthèse des ribosomes commence dans le nucléole, un compartiment nucléaire formé autour des unités répétées d’ADN ribosomique (NOR, nucleolar organisation regions) sur la chromatine. C’est dans ce nucléole que les ARNr 18S, 28S et 5,8S sont co-transcrits par l’ARN polymérase I (Pol I) sous la forme d’un long transcrit primaire (47S) qui contient, outre les séquences des ARNr matures, des séquences additionnelles dites espaceurs internes (ITS) et externes (ETS). Ces pré-ARNr sont immédiatement reconnus par deux classes de protéines pour former des particules pré-ribosomiques 90S : les protéines ribosomiques et les facteurs d’assemblage requis pour la maturation des pré-ribosomes mais absents des sous-unités matures. C’est au sein de ces particules ribonucléoprotéiques (RNP) que les pré-ARNr subissent de nombreuses étapes de maturation telles que des modifications chimiques ou l’élimination des séquences additionnelles par digestions endo- et exo-nucléolytiques [2, 3]. Ces modifications du composant ARN s’accompagnent de réorganisations structurales et de changements dans la composition protéique des différentes particules pré-ribosomiques. À la suite d’une coupure spécifique dans l’espaceur interne 1 (ITS1), ces pré-ribosomes précoces vont être scindés en deux, formant les particules pré-60S et pré-40S. Les particules pré-40S et pré-60S suivent ensuite des voies de maturation distinctes, transitant par le nucléoplasme avant d’être exportées dans le cytoplasme (Figure 1). Il a été montré récemment chez la levure Saccharomyces cerevisiae que, durant les dernières étapes de maturation de la petite sous-unité ribosomique dans le cytoplasme, la particule pré-40S pouvait s’associer à une grande sous-unité ribosomique mature. Cette étape servirait de contrôle qualité, testant la capacité des sous-unités pré-40S à initier la traduction avant qu’elles ne soient dirigées vers les ultimes étapes de maturation [4, 5].
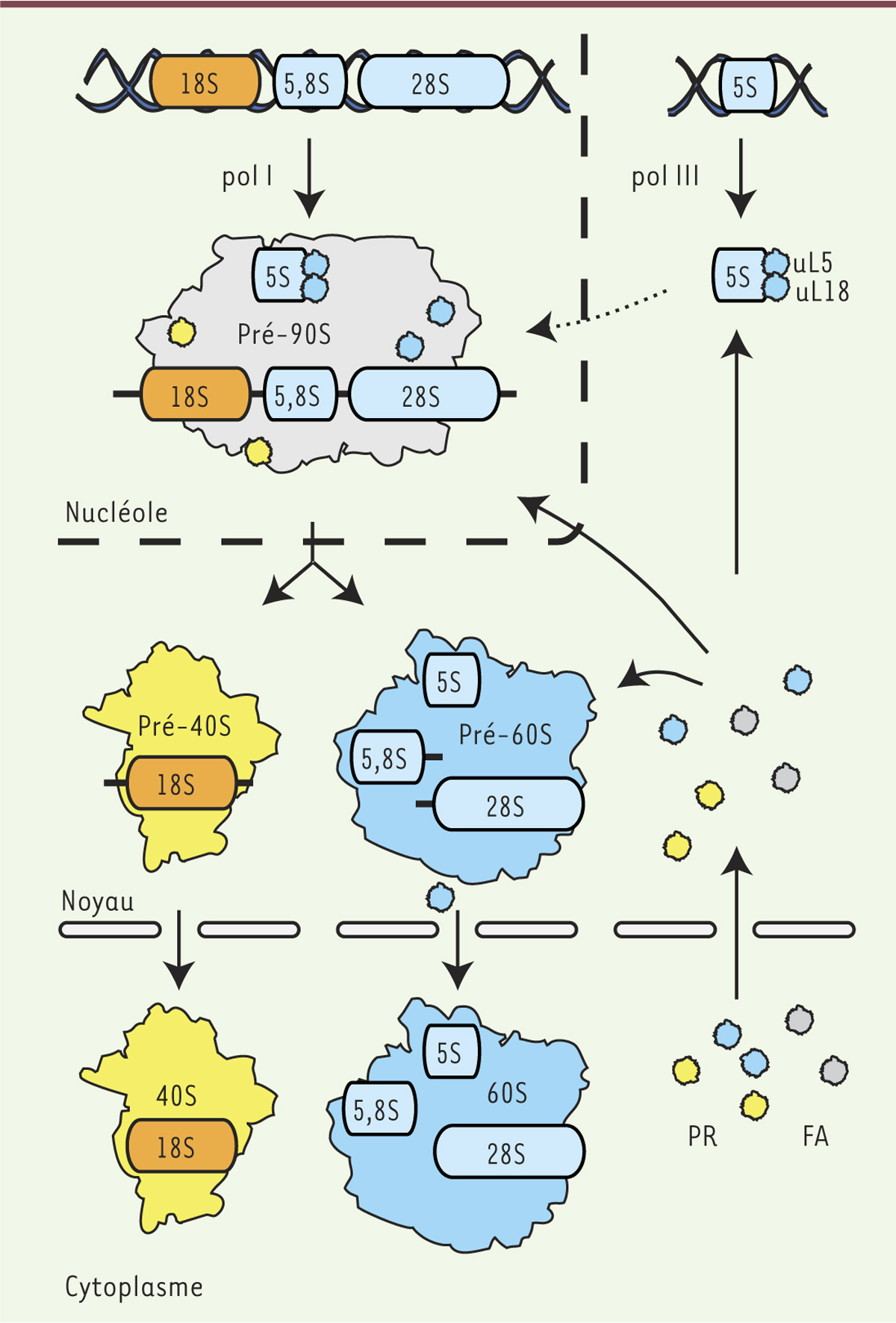
 | Figure 1.
La synthèse des ribosomes chez l’homme. La biogenèse du ribosome débute dans le nucléole avec la transcription du pré-ARN 47S par l’ARN polymérase I. Ce long transcrit primaire qui contient les séquences des ARNr 5,8S, 18S et 28S s’associe de manière co-transcriptionnelle avec plusieurs protéines ribosomiques (PR) et différents facteurs d’assemblage (FA) pour former le préribosome 90S. L’ARNr 5S suit une voie de synthèse et d’assemblage totalement différente, puisqu’il est d’abord transcrit par l’ARN polymérase III en périphérie du nucléole, reconnu par deux protéines ribosomiques, uL18 et uL5, et s’associe ensuite aux particules pré-ribosomiques en cours d’assemblage. Ces particules vont subir différentes étapes de clivages et de maturations dans le nucléoplasme, puis le cytoplasme, qui aboutiront à la formation de sous-unités 40S et 60S matures, capables d’assurer la traduction des ARNm. |
L’ARNr 5S est transcrit indépendamment par l’ARN polymérase III. Il est rapidement reconnu par deux protéines ribosomiques (RPL5/uL18 and RPL11/uL5), formant ce que l’on appelle la particule 5S. Celle-ci est ensuite intégrée aux particules pré-ribosomiques pré-60S précoces. De nombreuses études menées durant les vingt dernières années ont permis d’identifier près de 200 facteurs intervenant dans la biogenèse des ribosomes [2, 3]. Cependant, les fonctions moléculaires précises de la majorité d’entre eux ne sont toujours pas connues. Outre la caractérisation structurale et fonctionnelle de ces différents facteurs d’assemblage dans la biogénèse du ribosome, beaucoup d’études récentes ont été menées pour étudier les liens entre la synthèse des ribosomes et le contrôle de la prolifération cellulaire. |
Liens entre synthèse des ribosomes et cancers De par son coût énergétique et sa complexité, le processus de synthèse des ribosomes doit être finement régulé et doit rapidement s’adapter à l’environnement cellulaire. Ainsi, les cellules qui se multiplient ont un besoin accru en protéines dont la production ne peut être assurée que par un grand nombre de ribosomes. La régulation de la production des ribosomes a été observée pour la première fois chez la levure par la mise en évidence du régulon Ribi
1, constitué de plus de 200 gènes contrôlant la synthèse des ribosomes en réponse à des signaux environnementaux ou génétiques [6-8]. La synthèse des ribosomes est ainsi directement contrôlée par la voie métabolique de réponse aux nutriments mTOR (target of rapamycin) mais également par le facteur de transcription Runx1 (runt-related transcription factor 1) qui contrôle, entre autre, l’hématopoïèse [9, 10]. Le facteur de transcription c-Myc, un proto-oncogène qui stimule la prolifération cellulaire et dont l’expression est altérée dans de nombreux cancers, a également été impliqué comme régulateur de la biogénèse des ribosomes. Il contrôle l’expression de plusieurs gènes, notamment ceux du régulon Ribi [11]. Ainsi, la surexpression de c-Myc stimule la synthèse des ribosomes en agissant directement sur la transcription des pré-ARNr (via Pol I et Pol III) et sur l’expression de facteurs d’assemblage et de protéines ribosomiques. La prolifération cellulaire et la synthèse des ribosomes sont donc deux processus étroitement liés. La dérégulation de la synthèse des ribosomes peut être à l’origine d’altérations du cycle cellulaire et de la croissance cellulaire, aboutissant à une prolifération incontrôlée propice au développement de cancers. La dérégulation de la synthèse des ribosomes provoque également un phénomène appelé stress nucléolaire qui conduit le plus souvent à une modification de la taille et du nombre de nucléoles. L’observation des nucléoles est d’ailleurs utilisée comme indicateur de sévérité clinique ou de réponse à la chimiothérapie [12]. Différentes équipes ont observé une surexpression de plusieurs facteurs impliqués dans la synthèse des ribosomes en corrélation avec la sévérité de certains cancers. Parmi ces facteurs, on retrouve la protéine GPATCH2 (G-patch domain- containing protein 2) qui régule l’activité de l’hélicase Prp43 (pre-mRNA-splicing factor ATP-dependent RNA helicase), le facteur d’assemblage hCINAP (human coilin-interacting nuclear ATPase protein), l’endonucléase Nob1 (NIN1/PSMD8 binding protein 1 homolog) et la kinase RIOK2 (RIO kinase 2) [13-15]. L’ensemble de ces observations ont engendré différentes hypothèses sur l’existence de liens directs entre synthèse des ribosomes et progression tumorale [16]. Mais ce n’est que depuis une dizaine d’année que les composants impliqués dans ces connections ont pu être mis en évidence. Contrôle de la stabilité de p53 par les protéines ribosomiques La protéine p53 est un facteur de transcription qui promeut l’arrêt du cycle cellulaire, pouvant engager la cellule dans un programme apoptotique en réponse à des lésions de l’ADN ou à des stress oncogéniques. Cette protéine est un acteur majeur de la réponse anti-tumorale [ 57] ( →). Ce suppresseur de tumeur, aussi appelé « gardien du génome », est le facteur pro-apoptotique le plus fréquemment muté dans les lignées cancéreuses : 50 % des cancers présentent une ou plusieurs mutation(s)/délétion(s) au sein de ce gène. Dans la majorité des autres cancers, c’est sa régulation qui est affectée. Les premiers liens entre la synthèse des ribosomes et la régulation de p53 ont été établis en 2001 dans une étude montrant que la délétion de la protéine Bop1 ( block of proliferation 1), requise pour les premières étapes de maturation des ribosomes, provoquait un arrêt de croissance induit par p53 [ 17]. En 2004, une autre étude réalisée chez le poisson zèbre montrait que plusieurs mutations de gènes codant des protéines ribosomiques provoquaient l’apparition de cancers [ 18].
(→) Voir la Synthèse de O. Albagli, m/s n° 10, octobre 2015, page 869
Pourtant, aucune interaction directe n’avait jusqu’alors été montrée entre des protéines ribosomiques et p53. C’est à partir de l’étude de son principal régulateur, Mdm2 (murine double minute 2), que les premières bases moléculaires de la régulation de p53 par des protéines ribosomiques ont été découvertes. Mdm2 est une ubiquitine-ligase qui cible spécifiquement p53 et permet sa dégradation après adressage au protéasome. Lors d’un stress oncogénique, plusieurs signaux inhibent cette interaction, ce qui a pour effet de stabiliser p53. De nombreuses études ont mis en évidence l’existence d’interactions entre Mdm2 et des protéines ribosomiques en réponse à un stress nucléolaire [19]. Parmi celles-ci, on retrouve uS3 et eS25, appartenant à la petite sous-unité, et uL5 et uL18, composants de la grande sous-unité [20-23]. D’autres protéines ont également été impliquées mais leur interaction avec Mdm2 reste controversée [24]. Les protéines ribosomiques interagissent avec Mdm2 au niveau de son domaine acide et/ou de son domaine à doigt de zinc, dans lesquels des mutations sont retrouvées dans plusieurs cancers [25]. La formation des deux sous-unités ribosomiques étant indépendantes, il n’est pas surprenant de retrouver des protéines appartenant à chacune des sous-unités impliquées dans le processus de régulation de p53. Ce mécanisme pourrait ainsi permettre à la cellule de signaler rapidement des défauts d’assemblage des deux sous-unités, mais cette hypothèse fait actuellement débat. En effet, il semblerait que seule la déplétion de certaines protéines de la grande sous-unité soit capable d’induire significativement la stabilisation de p53 (dont le taux est alors 5 fois supérieur au taux normal) [26]. Néanmoins, il est clair qu’en cas de stress nucléolaire, qui peut conduire à la production de ribosomes déficients, certaines protéines ribosomiques, non encore assemblées, sont capables d’induire la stabilisation de p53 et de promouvoir un arrêt du cycle cellulaire, voire une apoptose de la cellule. Le rôle particulier de la particule 5S Parmi les différentes protéines ribosomiques interagissant avec Mdm2, les protéines uL5 et uL18 semblent être au cœur de la régulation de p53. Ces deux protéines s’associent en dehors du ribosome avec l’ARN 5S pour former la particule 5S. L’intégration de cette particule au sein des ribosomes pré-60S est une étape cruciale qui intervient très précocement dans l’assemblage de la grande sous-unité. En son absence, la maturation de la grande sous-unité est stoppée dans le noyau, ce qui conduit rapidement à un déficit en sous-unités 60S fonctionnelles [ 27]. Toute altération de la synthèse des ribosomes a en fait pour effet de réduire la production de particules pré-60S, augmentant ainsi la quantité de particules 5S libres dans le nucléoplasme. La découverte, en 1994, d’un complexe extra-ribosomique formé de l’assemblage de Mdm2, uL18 et de l’ARNr 5S, a suggéré un rôle de la particule 5S dans la régulation de p53 [22]. uL18, uL5, et l’ARNr 5S ont depuis été identifiés comme des partenaires de Mdm2, capables d’inhiber son activité ubiquitine-ligase et ainsi de stabiliser p53. La particule 5S joue donc un rôle particulier dans le contrôle de p53. En l’absence d’un de ces composants, l’inhibition de synthèse des ribosomes ne conduit plus à la stabilisation de p53 [28, 29]. L’examen de l’impact de la déplétion des différentes protéines ribosomiques sur l’intégrité du nucléole et la stabilisation de p53 a montré que l’effet le plus important est observé en l’absence de la particule 5S [26]. Récemment, la structure de la protéine uL5 associée aux domaines acide et doigt de zinc de Mdm2 a pu être résolue par cristallographie aux rayons X [23]. Cette analyse structurale révèle que Mdm2 interagit avec uL5 au niveau du site de liaison de uL5 à l’ARNr 28S, montrant ainsi que l’interaction entre les deux protéines ne peut se produire qu’en absence de l’ARNr 28S et donc vraisemblablement en dehors du ribosome, ou via un changement de conformation de la particule 5S au sein du ribosome. La particule 5S est au cœur de la coordination entre la synthèse des ribosomes et la prolifération cellulaire. Il est intéressant de noter que l’ARN 5S est transcrit par l’ARN polymérase III, indépendamment des autres ARNr qui sont transcrits par l’ARN polymérase I. Ainsi, l’inhibition de la transcription dépendant de la polymérase I par différents signaux, ne s’accompagne pas inévitablement de l’inhibition de la transcription par la polymérase III, créant ainsi un déséquilibre entre la quantité de particules 5S libres et celle associée aux ribosomes. La particule 5S pourrait ainsi véritablement jouer le rôle de détecteur fonctionnel de la biogenèse du ribosome, indiquant à la cellule quel état adopter pour la prolifération. La double régulation de la synthèse des ribosomes et de la stabilité de p53 en réponse à des stress oncogéniques La synthèse des ribosomes est régulée par de nombreux signaux cellulaires. Parmi les différents acteurs de cette régulation, on retrouve certains facteurs pro-oncogéniques. La protéine p14 ARF est un suppresseur de tumeur provenant de l’épissage alternatif du locus Ink4a/Arf ( ADP-ribosylation factor ou Cdkn2A [ cyclin dependent kinase inhibitor 2A]), deuxième locus le plus fréquemment muté après celui de P53 dans les cancers humains [ 30]. La protéine p14 ARF s’accumule dans les cellules à la suite de signaux oncogéniques comme ceux produits par c-Myc et Ras [ 30]. Dans les cellules sauvages, p14 ARF, en s’associant directement avec Mdm2, est à l’origine de la stabilisation de p53 [ 31]. Outre ce rôle dans le contrôle de la prolifération cellulaire par l’intermédiaire de p53, p14 ARF régule aussi directement la synthèse des ribosomes [ 32, 33] ( →). Deux équipes ont en effet montré que la particule 5S était nécessaire à la stabilisation de p53 par p14 ARF [ 28, 34], p14 ARF pouvant s’associer avec Mdm2 et avec la particule 5S. Il est donc possible qu’à la suite de son activation par des signaux oncogéniques, p14 ARF altère la prolifération cellulaire à deux niveaux : en modifiant la synthèse des ribosomes afin de réduire la production des protéines et en formant un complexe avec la particule 5S et Mdm2 pour activer p53. Le facteur d’épissage Srsf1 ( serine and arginine-rich splicing factor 1) est un proto-oncogène impliqué dans l’épissage alternatif de différents oncogènes. En réponse à sa surexpression induite par les signaux oncogéniques, la cellule inhibe la dégradation de p53 évitant la mise en place d’un programme oncogénique [ 35]. Cette stabilisation de p53 nécessite l’association de Srsf1 avec la protéine ribosomique uL18 [ 35] démontrant que Srsf1 est également un facteur qui relie p53 et la synthèse des ribosomes (Figure 2).
(→) Voir la Synthèse de O. Ayrault et al., m/s n° 5, mai 2006, page 519
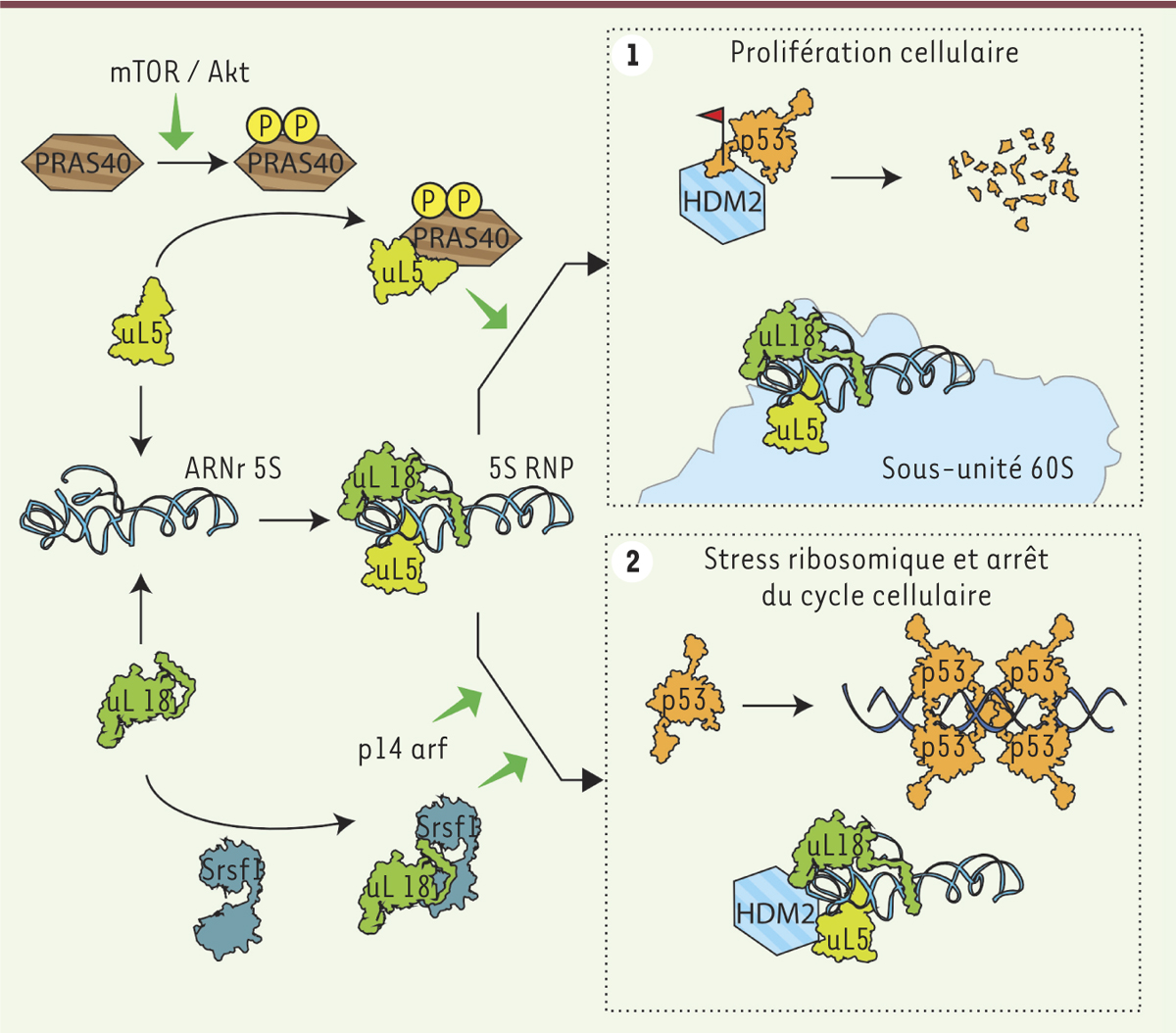
 | Figure 2.
La particule 5S régule la prolifération cellulaire. Dans le nucléoplasme, l’ARNr 5S s’associe avec les deux protéines ribosomiques uL18 et uL5 pour former la particule 5S, composant essentiel de la grande sous-unité ribosomique. (1) La phase de prolifération cellulaire est caractérisée par un très grand nombre de ribosomes, nécessaires à la production des protéines. Dans ce contexte, le taux de synthèse des ribosomes est élevé et la particule 5S s’intègre aux nombreuses particules pré-ribosomiques produites. D’autre part, HDM2 (homologue chez l’homme de mdm2 [murine double minute 2]) catalyse l’ubiquitination de p53, conduisant à sa dégradation, et assure ainsi le maintien de la phase de prolifération cellulaire. (2) En cas de stress de la biogenèse des ribosomes, la particule 5S s’accumule sous forme libre, en dehors du ribosome. Cette particule 5S peut s’associer avec HDM2, et inhiber son activité ubiquitine ligase. p53 est alors stabilisée, et peut s’associer sous forme de tétramères pour remplir son rôle de facteur de transcription promouvant l’expression de différents gènes impliqués dans l’arrêt du cycle cellulaire et l’apoptose. (3) L’équilibre entre ces deux voies est assuré par des protéines anti-apoptotiques comme la protéine PRAS40 (proline-rich Akt substrate of 40 kDa), qui, sous le contrôle de la voie mTOR (mammalian target of rapamycin)/Akt (protéine kinase B), peut interagir avec uL5 et ainsi inhiber son association avec HDM2. Par ailleurs, certaines protéines pro-apoptotiques, comme le facteur d’épissage Srsf1 en s’associant avec uL18, ou la protéine p14ARF, favorisent la stabilisation de p53. Les protéines dont les structures ne sont pas entièrement connues sont représentées en hachuré. |
À l’inverse, la protéine PRAS40 (proline-rich Akt substrate of 40 kDa) est un inhibiteur de la voie mTOR qui, à la différence des autres inhibiteurs de cette voie, participe à la prolifération cellulaire et la tumorigenèse. Il a récemment été démontré qu’une fois phosphorylée par mTOR et Akt, la protéine PRAS40 interagit avec uL5 dans le noyau, ce qui a pour effet d’inhiber son interaction avec Mdm2 et de promouvoir ainsi la dégradation de p53 [36]. Le cas particulier des ribosomopathies Les ribosomopathies regroupent différentes pathologies associées à des défauts d’assemblage des ribosomes [ 37]. Ces pathologies, bien que présentant toutes des défauts de synthèse des ribosomes, varient dans leurs symptômes [ 38]. L’une de leurs particularités est le rôle joué par p53. Dans les différents modèles cellulaires et animaux de ces maladies, p53 s’accumule dans les cellules en réponse aux défauts de synthèse des ribosomes. Réciproquement, l’inactivation de p53 peut compenser certains symptômes. Ainsi, dans les modèles de cellules de moelle osseuse CD34 + mimant l’anémie de Diamond-Blackfan 2 (DBA), l’inactivation de p53 compense les défauts d’érythropoïèse induits par la délétion de la protéine ribosomale eS19 [ 39]. De même, on observe une absence d’anomalies cranio-faciales associées au syndrome de Treacher-Collins 3 (TCS, une autre ribosomapthie) dans des modèles murins dans lesquels p53 est inactivée [ 40]. Bien qu’associées à une stabilisation de p53, ces pathologies prédisposent les patients au développement de cancers. Il est possible que l’accumulation de p53 en réponse à ces défauts de synthèse des ribosomes exerce une pression sélective sur les cellules de ces patients. Les clones dans lesquels la régulation, l’activité ou les effecteurs de p53 sont altérés seraient ainsi favorisés. |
Liens entre prolifération cellulaire et synthèse des ribosomes p53 indépendants Le processus impliquant Mdm2, les protéines ribosomiques et p53 est fondamental pour les liens existant entre prolifération cellulaire et synthèse des ribosomes. Cependant, de tels liens ont pu être observés au sein d’organismes n’ayant pas d’équivalent de p53, ou au sein de modèles cellulaires dans lesquelles l’expression ou la fonction de p53 sont altérées. Chez l’homme, la déplétion de Pescadillo (PES), une protéine impliquée dans les premières étapes de maturation des ribosomes, conduit à une perte de ribosomes et perturbe le cycle cellulaire [41]. Cet effet est néanmoins maintenu dans des cellules exprimant une protéine p53 mutée. Il semble donc que l’action de Pescadillo sur la croissance cellulaire ne repose pas exclusivement sur p53 [42]. En fait, l’effet de Pescadillo dépendrait de la protéine du rétinoblastome, pRB [42]. pRB est un suppresseur de tumeur dont l’expression est affectée dans plusieurs cancers. Il agit en séquestrant le facteur de transcription E2F-1 (E2 factor transcription factor 1), bloquant ainsi le cycle cellulaire [43]. Phosphorylée, pRB ne peut plus interagir avec E2F-1, ce qui permet la progression dans le cycle cellulaire. PPAN (Peter Pan homolog), une protéine partenaire de la protéine Pescadillo, est impliquée dans l’assemblage des ribosomes. Lors de perturbations de la synthèse des ribosomes, PPAN se relocalise dans la mitochondrie et induit une apoptose de la cellule indépendamment de p53 [44]. L’une des premières observations pointant un mécanisme reliant défauts de synthèse des ribosomes et prolifération cellulaire, indépendant de p53, a été obtenue chez la levure S. cerevisiae. Dans cet organisme modèle, il n’existe pas d’homologue identifié de la protéine p53. Des défauts de synthèse des ribosomes affectent pourtant la prolifération cellulaire avant même que le niveau de traduction des protéines ne soit affecté [45]. Whi5, l’équivalent fonctionnel du suppresseur de tumeur pRB chez la levure est impliqué dans cette régulation [45]. Certaines protéines ribosomiques peuvent également directement influencer le cycle cellulaire, indépendamment de la voie Mdm2-RP-P53. Ainsi, l’interaction de uL5 avec Mdm2 inhibe la protection du facteur E2F-1 par Mdm2, E2F-1 étant lui-même contrôlé par pRB [46]. uL5 est capable aussi d’interagir avec le régulateur c-Myc. Il inhibe son activité et peut se lier à son ARN messager, ce qui conduit à sa dégradation, ceci même en l’absence de p53 [47]. L’importance des protéines ribosomiques uL3 et eL27 dans la régulation du cycle cellulaire en absence de p53 a également été montrée [48, 49]. Les liens entre la synthèse des ribosomes et la prolifération cellulaire indépendants de p53, ont été étudiés par une équipe qui a examiné l’effet de la déplétion des protéines uL5 et uL18 sur la progression du cycle cellulaire. Dans ces modèles, p53 ne s’accumule pas dans les cellules en réponse aux défauts de synthèse des ribosomes. La déplétion de uL5 et de uL18 conduit dans ce cas à une baisse du taux de traduction ne provoquant pas d’arrêt du cycle mais une progression plus lente vers celui-ci, limitant ainsi la prolifération cellulaire [50]. |
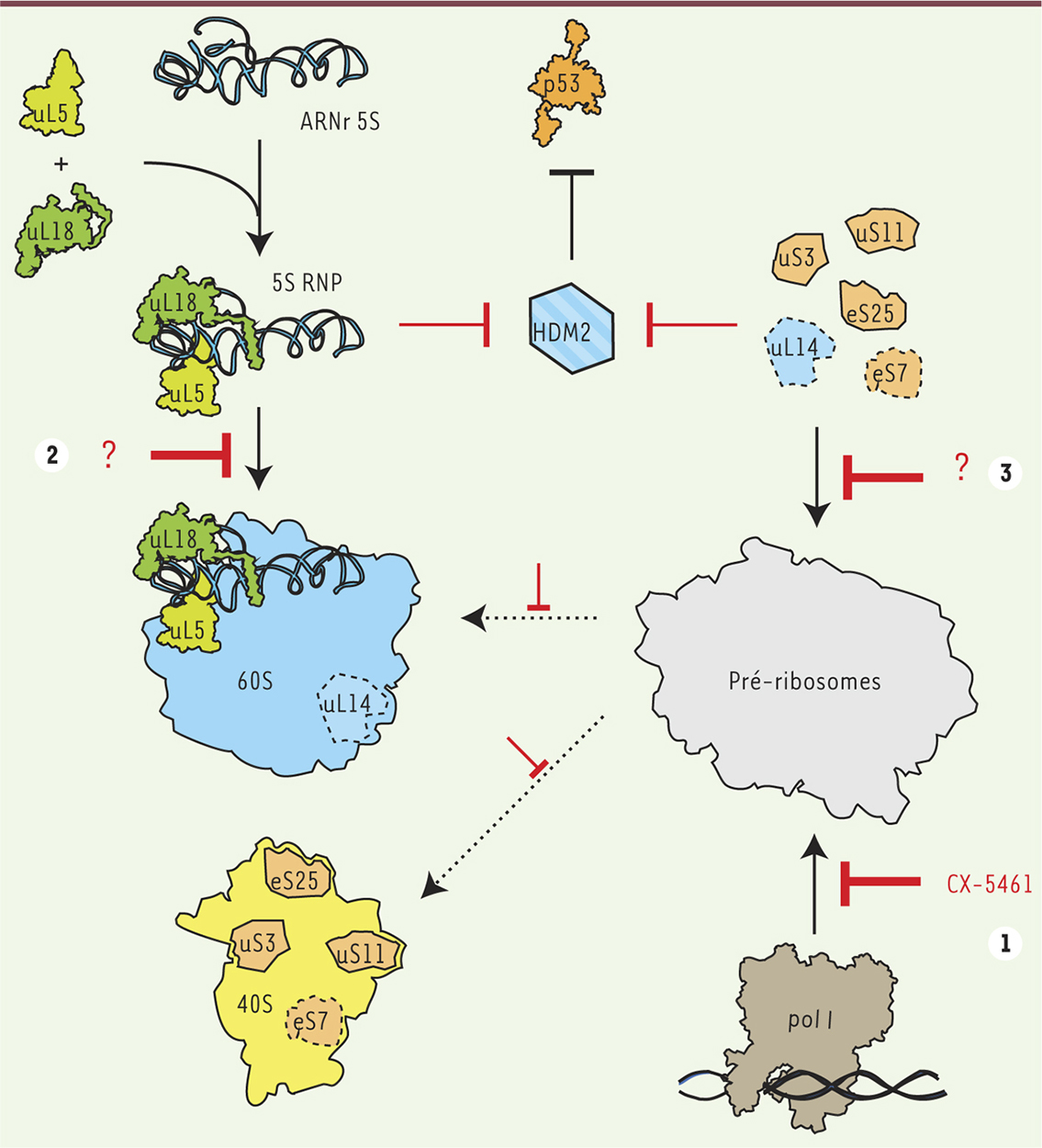
Perspectives sur le développement de nouvelles drogues Les liens entre la synthèse des ribosomes et la prolifération cellulaire font de la biogenèse des ribosomes une nouvelle cible thérapeutique pour le traitement des cancers. L’inhibition de la synthèse des ribosomes perturbe en effet la prolifération cellulaire à deux niveaux différents : en affectant directement la capacité de la cellule à synthétiser des protéines, et en stabilisant indirectement le suppresseur de tumeur p53 (Figure 3). Une première drogue, le CX-5461, qui inhibe l’activité de l’ARN polymérase I, est en cours d’essais cliniques [51]. Des résultats obtenus dans des modèles murins indiquent que cette drogue pourrait être utilisée en combinaison avec d’autres molécules qui ciblent la traduction afin d’optimiser la réponse [51]. Le CX-5461 semble particulièrement indiqué pour les cancers présentant une dérégulation de l’expression du facteur oncogénique c-Myc [52].  | Figure 3.
Inhibition de la biogenèse du ribosome et réponse pro-apoptotique. L’étude de la biogenèse des ribosomes permet aujourd’hui l’identification de nouvelles stratégies moléculaires en thérapeutique anti-cancéreuse. En effet, l’inhibition de certaines étapes clés de la synthèse des sous-unités ribosomiques pourrait conduire à une stabilisation de p53. (1) L’inhibition de l’ARN polymérase I (pol I) par le CX-5461 conduit à un arrêt de la synthèse des sous-unités ribosomiques et provoque l’accumulation de particule 5S (5S RNP [particule ribonucléique]) dans le noyau, capable d’inhiber HDM2. (2) L’autre moyen de favoriser cette interaction serait alors d’inhiber l’assemblage de la particule 5S au sein de la grande sous-unité du ribosome. (3) Certaines protéines ribosomiques comme uS3, uS11 et eS25 peuvent lier directement HDM2. Certaines études, qui restent controversées, ont montré que les protéines uL14 et eS7 (en pointillés) seraient également impliquées dans cette régulation. La surexpression de ces protéines mène d’ailleurs à la stabilisation de p53. Ces données font de ces protéines ribosomiques de nouvelles cibles thérapeutiques anti-cancéreuses potentielles. |
Les récentes avancées sur la compréhension des mécanismes moléculaires impliqués dans la synthèse des ribosomes ainsi que la résolution de nombreuses structures de facteurs d’assemblages apportent les fondements nécessaires au développement de nouvelles drogues anti-cancéreuses [53-56]. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Footnotes |
1.
Warner JR. The economics of ribosome biosynthesis in yeast . Trends Biochem Sci. 1999; ; 24 : :437.–440. 2.
Henras AK, Soudet J, Gérus M, et al. The post-transcriptional steps of eukaryotic ribosome biogenesis . Cell Mol Life Sci. 2008; ; 65 : :2334.–2359. 3.
Henras AK, Plisson-Chastang C, O’Donohue M-F, et al. An overview of pre-ribosomal RNA processing in eukaryotes . Wiley Interdiscip Rev RNA. 2015; ; 6 : :225.–242. 4.
Lebaron S, Schneider C, van Nues RW, et al. Proofreading of pre-40S ribosome maturation by a translation initiation factor and 60S subunits . Nat Struct Mol Biol. 2012; ; 19 : :744.–753. 5.
Strunk BS, Novak MN, Young CL, et al. A translation-like cycle is a quality control checkpoint for maturing 40S ribosome subunits . Cell. 2012; ; 150 : :111.–121. 6.
Wade C, Shea KA, Jensen RV, et al. EBP2 is a member of the yeast RRB regulon, a transcriptionally coregulated set of genes that are required for ribosome and rRNA biosynthesis . Mol Cell Biol. 2001; ; 21 : :8638.–8650. 7.
Wade CH, Umbarger MA, McAlear MA. The budding yeast rRNA and ribosome biosynthesis (RRB) regulon contains over 200 genes . Yeast Chichester Engl. 2006; ; 23 : :293.–306. 8.
Jorgensen P, Rupes I, Sharom JR, et al. A dynamic transcriptional network communicates growth potential to ribosome synthesis and critical cell size . Genes Dev. 2004; ; 18 : :2491.–2505. 9.
Cai X, Gao L, Teng L, et al. Runx1 Deficiency Decreases Ribosome Biogenesis and Confers Stress Resistance to Hematopoietic Stem and Progenitor Cells . Cell Stem Cell. 2015; ; 17 : :165.–177. 10.
Iadevaia V, Liu R, Proud CG. mTORC1 signaling controls multiple steps in ribosome biogenesis . Semin Cell Dev Biol. 2014; ; 36 : :113.–120. 11.
van Riggelen J, Yetil A, Felsher DW. MYC as a regulator of ribosome biogenesis and protein synthesis . Nat Rev Cancer. 2010; ; 10 : :301.–309. 12.
Montanaro L, Treré D, Derenzini M. Nucleolus, Ribosomes, and Cancer . Am J Pathol. 2008; ; 173 : :301.–310. 13.
Lin M-L, Fukukawa C, Park J-H, et al. Involvement of G-patch domain containing 2 overexpression in breast carcinogenesis . Cancer Sci. 2009; ; 100 : :1443.–1450. 14.
Bai D, Zhang J, Li T, et al. The ATPase hCINAP regulates 18S rRNA processing and is essential for embryogenesis and tumour growth . Nat Commun. 2016; ; 7 : :12310.. 15.
Liu K, Chen H-L, Wang S, et al. High Expression of RIOK2 and NOB1 Predict Human Non-small Cell Lung Cancer Outcomes . Sci Rep. 2016; ; 6 : :28666.. 16.
Ruggero D, Grisendi S, Piazza F, et al. Dyskeratosis congenita and cancer in mice deficient in ribosomal RNA modification . Science. 2003; ; 299 : :259.–262. 17.
Pestov DG, Strezoska Z, Lau LF. Evidence of p53-dependent cross-talk between ribosome biogenesis and the cell cycle: effects of nucleolar protein Bop1 on G(1)/S transition . Mol Cell Biol. 2001; ; 21 : :4246.–4255. 18.
Amsterdam A, Sadler KC, Lai K, et al. Many ribosomal protein genes are cancer genes in zebrafish . PLoS Biol. 2004; ; 2 : :E139.. 19.
Zhou X, Liao WJ, Liao JM, et al. Ribosomal proteins: functions beyond the ribosome . J Mol Cell Biol. 2015; ; 7 : :92.–104. 20.
Yadavilli S, Mayo LD, Higgins M, et al. Ribosomal protein S3: A multi-functional protein that interacts with both p53 and MDM2 through its KH domain . DNA Repair. 2009; ; 8 : :1215.–1224. 21.
Zhang X, Wang W, Wang H, et al. Identification of ribosomal protein S25 (RPS25)-MDM2-p53 regulatory feedback loop . Oncogene. 2013; ; 32 : :2782.–2791. 22.
Marechal V, Elenbaas B, Piette J, et al. The ribosomal L5 protein is associated with mdm-2 and mdm-2-p53 complexes . Mol Cell Biol. 1994; ; 14 : :7414.–7420. 23.
Zheng J, Lang Y, Zhang Q, et al. Structure of human MDM2 complexed with RPL11 reveals the molecular basis of p53 activation . Genes Dev. 2015; ; 29 : :1524.–1534. 24.
Fumagalli S, Ivanenkov VV, Teng T, et al. Suprainduction of p53 by disruption of 40S and 60S ribosome biogenesis leads to the activation of a novel G2/M checkpoint . Genes Dev. 2012; ; 26 : :1028.–1040. 25.
Lindström MS, Jin A, Deisenroth C, et al. Cancer-associated mutations in the MDM2 zinc finger domain disrupt ribosomal protein interaction and attenuate MDM2-induced p53 degradation . Mol Cell Biol. 2007; ; 27 : :1056.–1068. 26.
Nicolas E, Parisot P, Pinto-Monteiro C, et al. Involvement of human ribosomal proteins in nucleolar structure and p53-dependent nucleolar stress . Nat Commun. 2016; ; 7 : :11390.. 27.
Zhang J, Harnpicharnchai P, Jakovljevic J, et al. Assembly factors Rpf2 and Rrs1 recruit 5S rRNA and ribosomal proteins rpL5 and rpL11 into nascent ribosomes . Genes Dev. 2007; ; 21 : :2580.–2592. 28.
Sloan KE, Bohnsack MT, Watkins NJ. The 5S RNP couples p53 homeostasis to ribosome biogenesis and nucleolar stress . Cell Rep. 2013; ; 5 : :237.–247. 29.
Donati G, Peddigari S, Mercer CA, et al. 5S ribosomal RNA is an essential component of a nascent ribosomal precursor complex that regulates the Hdm2-p53 checkpoint . Cell Rep. 2013; ; 4 : :87.–98. 30.
Sherr CJ. The INK4a/ARF network in tumour suppression . Nat Rev Mol Cell Biol. 2001; ; 2 : :731.–737. 31.
Weber JD, Kuo ML, Bothner B, et al. Cooperative signals governing ARF-mdm2 interaction and nucleolar localization of the complex . Mol Cell Biol. 2000; ; 20 : :2517.–2528. 32.
Sugimoto M, Kuo M-L, Roussel MF, et al. Nucleolar Arf tumor suppressor inhibits ribosomal RNA processing . Mol Cell. 2003; ; 11 : :415.–424. 33.
Ayrault O, Andrique L, Larsen CJ, et al. La régulation négative de la biogenèse des ribosomes . Med Sci (Paris). 2006; ; 22 : :519.–524. 34.
Dai MS, Challagundla KB, Sun XX, et al. Physical and functional interaction between ribosomal protein L11 and the tumor suppressor ARF . J Biol Chem. 2012; ; 287 : :17120.–17129. 35.
Fregoso OI, Das S, Akerman M, et al. Splicing-factor oncoprotein SRSF1 stabilizes p53 via RPL5 and induces cellular senescence . Mol Cell. 2013; ; 50 : :56.–66. 36.
Havel JJ, Li Z, Cheng D, et al. Nuclear PRAS40 couples the Akt/mTORC1 signaling axis to the RPL11-HDM2-p53 nucleolar stress response pathway . Oncogene. 2015; ; 34 : :1487.–1498. 37.
Narla A, Ebert BL. Ribosomopathies: human disorders of ribosome dysfunction . Blood. 2010; ; 115 : :3196.–3205. 38.
Danilova N, Gazda HT. Ribosomopathies: how a common root can cause a tree of pathologies . Dis Model Mech. 2015; ; 8 : :1013.–1026. 39.
Dutt S, Narla A, Lin K, et al. Haploinsufficiency for ribosomal protein genes causes selective activation of p53 in human erythroid progenitor cells . Blood. 2011; ; 117 : :2567.–2576. 40.
Jones NC, Lynn ML, Gaudenz K, et al. Prevention of the neurocristopathy Treacher Collins syndrome through inhibition of p53 function . Nat Med. 2008; ; 14 : :125.–133. 41.
Wu CL, Zukerberg LR, Ngwu C, et al. In vivo association of E2F and DP family proteins . Mol Cell Biol. 1995; ; 15 : :2536.–2546. 42.
Grimm T, Hölzel M, Rohrmoser M, et al. Dominant-negative Pes1 mutants inhibit ribosomal RNA processing and cell proliferation via incorporation into the PeBoW-complex . Nucleic Acids Res. 2006; ; 34 : :3030.–3043. 43.
Li J, Yu L, Zhang H, et al. Down-regulation of pescadillo inhibits proliferation and tumorigenicity of breast cancer cells . Cancer Sci. 2009; ; 100 : :2255.–2260. 44.
Pfister AS, Keil M, Kühl M. The Wnt target protein Peter Pan defines a novel p53-independent nucleolar stress-response pathway . J Biol Chem. 2015; ; 290 : :10905.–10918. 45.
Bernstein KA, Bleichert F, Bean JM, et al. Ribosome biogenesis is sensed at the Start cell cycle checkpoint . Mol. Biol. Cell. 2007; ; 18 : :953.–964. 46.
Donati G, Brighenti E, Vici M, et al. Selective inhibition of rRNA transcription downregulates E2F–1: a new p53-independent mechanism linking cell growth to cell proliferation . J Cell Sci. 2011; ; 124 : :3017.–3028. 47.
Dai M-S, Arnold H, Sun X-X, et al. Inhibition of c-Myc activity by ribosomal protein L11 . EMBO J. 2007; ; 26 : :3332.–3345. 48.
Pagliara V, Saide A, Mitidieri E, et al. 5-FU targets rpL3 to induce mitochondrial apoptosis via cystathionine-β-synthase in colon cancer cells lacking p53 . Oncotarget. Aug 2; ; 7((31))::50333.–50348. 49.
Alkhatabi HA, McLornan DP, Kulasekararaj AG, et al. RPL27A is a target of miR-595 and may contribute to the myelodysplastic phenotype through ribosomal dysgenesis . Oncotarget. 2016; ; 7 : :47875.–47890. 50.
Teng T, Mercer CA, Hexley P, et al. Loss of tumor suppressor RPL5/RPL11 does not induce cell cycle arrest but impedes proliferation due to reduced ribosome content and translation capacity . Mol Cell Biol. 2013; ; 33 : :4660.–4671. 51.
Devlin JR, Hannan KM, Hein N, et al. Combination therapy targeting ribosome biogenesis and mRNA translation synergistically extends survival in MYC-driven lymphoma . Cancer Discov. 2016; ; 6 : :59.–70. 52.
Poortinga G, Quinn LM, Hannan RD. Targeting RNA polymerase I to treat MYC-driven cancer . Oncogene. 2015; ; 34 : :403.–412. 53.
Leidig C, Thoms M, Holdermann I, et al. 60S ribosome biogenesis requires rotation of the 5S ribonucleoprotein particle . Nat Commun. 2014; ; 5 : :3491.. 54.
Kornprobst M, Turk M, Kellner N, et al. Architecture of the 90S pre-ribosome: A structural view on the birth of the eukaryotic ribosome . Cell. 2016; ; 166 : :380.–393. 55.
Loc’h J, Blaud M, Réty S, et al. RNA Mimicry by the Fap7 adenylate kinase in ribosome biogenesis . PLoS Biol. 2014;; 12 : :e1001860.. 56.
Madru C, Lebaron S, Blaud M, et al. Chaperoning 5S RNA assembly . Genes Dev. 2015; ; 29 : :1432.–1446. 57.
Albagli O. Protéger et sévir : p53, métabolisme et suppression tumorale . Med Sci (Paris). 2015; ; 31 : :869.–880. |