| |
| Med Sci (Paris). 33(10): 843–845. doi: 10.1051/medsci/20173310010.Comment la ré-initiation de la transcription est-elle gouvernée chez le virus de la rougeole ? Louis-Marie Bloyet,1 Philippe Roche,2,3,4 Denis Gerlier,1 and Sonia Longhi5,6* 1CIRI, Centre international de recherche en infectiologie, Inserm U1111, université Claude Bernard Lyon 1, CNRS, UMR5308, École Normale Supérieure de Lyon, université Lyon, 21, avenue Tony Garnier, F-69007, Lyon, France 2Aix Marseille Univ, Institut Paoli-Calmettes, centre de recherche en cancérologie de Marseille (CRCM), 27, boulevard Lei Roure, 13273Marseille Cedex 9, France 3CNRS, centre de recherche en cancérologie de Marseille (CRCM) UMR 7258, Marseille, France 4Inserm, centre de recherche en cancérologie de Marseille (CRCM) U1068, Marseille, France 5Aix Marseille Univ, Architecture et Fonction des Macromolécules Biologiques (AFMB) UMR 7257, Marseille, France 6CNRS, Architecture et Fonction des Macromolécules Biologiques AFMB, UMR 7257, 163, avenue de Luminy, 13288Marseille, France MeSH keywords: Génome viral, Humains, Rougeole, Virus de la rougeole, Transcription génétique, Réplication virale, physiologie, virologie, génétique |
Le virus de la rougeole appartient au genre Morbillivirus, au sein de la famille des Paramyxoviridae, ordre des Mononegavirales. Cet ordre viral se caractérise par un génome d’ARN simple brin, non segmenté et de polarité négative. Pour être traduits, les gènes de ce type de génome doivent préalablement être transcrits en ARN messagers (ARNm) [1]. Les Mononegavirales regroupent de très nombreux virus pathogènes pour l’homme et/ou pour les animaux avec un impact socioéconomique et/ou en santé publique majeur. Les virus Ebola, Nipah, de la rage, et le virus respiratoire syncytial appartiennent également à l’ordre des Mononegavirales. La rougeole est une maladie virale grave, extrêmement contagieuse. En dépit de la disponibilité et de l’efficacité d’un vaccin, qui a conduit à une réduction par quatre de la mortalité entre 2000 et 2007, ce virus reste un problème de santé publique majeur à travers le monde. L’Organisation mondiale de la santé (OMS) recense chaque année plus de 100 000 décès et aucune diminution n’a été observée ces dix dernières années. L’extrême contagiosité du virus et les refus de vaccination exprimés par une fraction malheureusement grandissante de la population, expliquent les flambées épidémiques récurrentes de rougeole en Europe avec, chaque année, plusieurs milliers de cas et des dizaines de morts (voir les revues sur l’épidémie ayant sévi en France entre 2008 et 2012 [2, 3]). L’éradication de la rougeole nécessiterait de maintenir une couverture vaccinale supérieure à 95 %, ce qui n’est plus le cas en Europe depuis quelques années à cause d’une défiance individuelle prenant le pas sur la protection collective [4] (→). (→) Voir l’éditorial d’A. Fisher, m/s n° 2, février 2017, page 119 Outre cette difficulté sociétale, aucun traitement thérapeutique n’est disponible à ce jour contre le virus de la rougeole, comme d’ailleurs contre aucun autre Mononegavirales. Il est donc impératif de découvrir des alternatives thérapeutiques ou préventives puissantes et spécifiques afin de contenir les ravages épidémiques causés par ces virus. Les protéines du complexe réplicatif de ces virus constituent une cible biologique de choix pour le développement d’agents antiviraux. Elles n’ont, en effet, pas d’homologues connus dans les tissus humains ou animaux. Mais, nos connaissances sur les mécanismes moléculaires de la transcription et de la réplication de ces virus restent fragmentaires. La compréhension des mécanismes moléculaires gouvernant les interactions entre protéines nécessaires pour la transcription et la réplication du virus reste donc un des prérequis pour l’identification et la conception d’agents antiviraux et/ou pour comprendre le mécanisme d’action d’inhibiteurs qui seraient identifiés par un criblage « aveugle ». |
Le complexe réplicatif du virus de la rougeole Comme tous les Mononegavirales, le virus de la rougeole possède un mécanisme de transcription et de réplication très singulier reposant sur trois protéines virales : la polymérase (L), la nucléoprotéine (N) et la phosphoprotéine (P). Chez ces virus, l’ARN génomique n’est pas infectieux car il n’est pas reconnu comme matrice par la polymérase virale. Le génome doit être encapsidé dans un homopolymère hélicoïdal de protéines N pour pouvoir servir de matrice pour la transcription et pour la réplication du génome viral. Lors de la transcription, sont produits les ARN messagers complémentaires, de polarité positive, à partir desquels seront traduites les protéines virales. Lors de la réplication, une copie intégrale et complémentaire du génome viral de polarité positive (appelée antigénome), est synthétisée. Cet antigénome sera lui-même encapsidé pour être copié, à son tour, en ARN génomique négatif qui sera la base génétique de la progéniture virale. L’assemblage entre ARN et protéine N, appelé nucléocapside, est reconnu par la polymérase L via son cofacteur, la phosphoprotéine P. Celle-ci se lie à la nucléocapside par son domaine C-terminal (appelé PXD) [5] qui reconnaît spécifiquement le domaine C-terminal de la protéine N (ou NTAIL
). NTAIL
constitue un appendice flexible qui est exposé à la surface de la nucléocapside [6, 7]. L’organisation de la nucléocapside est ainsi semblable à celle d’une chenille dont les « poils » hérissés seraient les appendices NTAIL
de chaque monomère de protéine N (Figure 1).
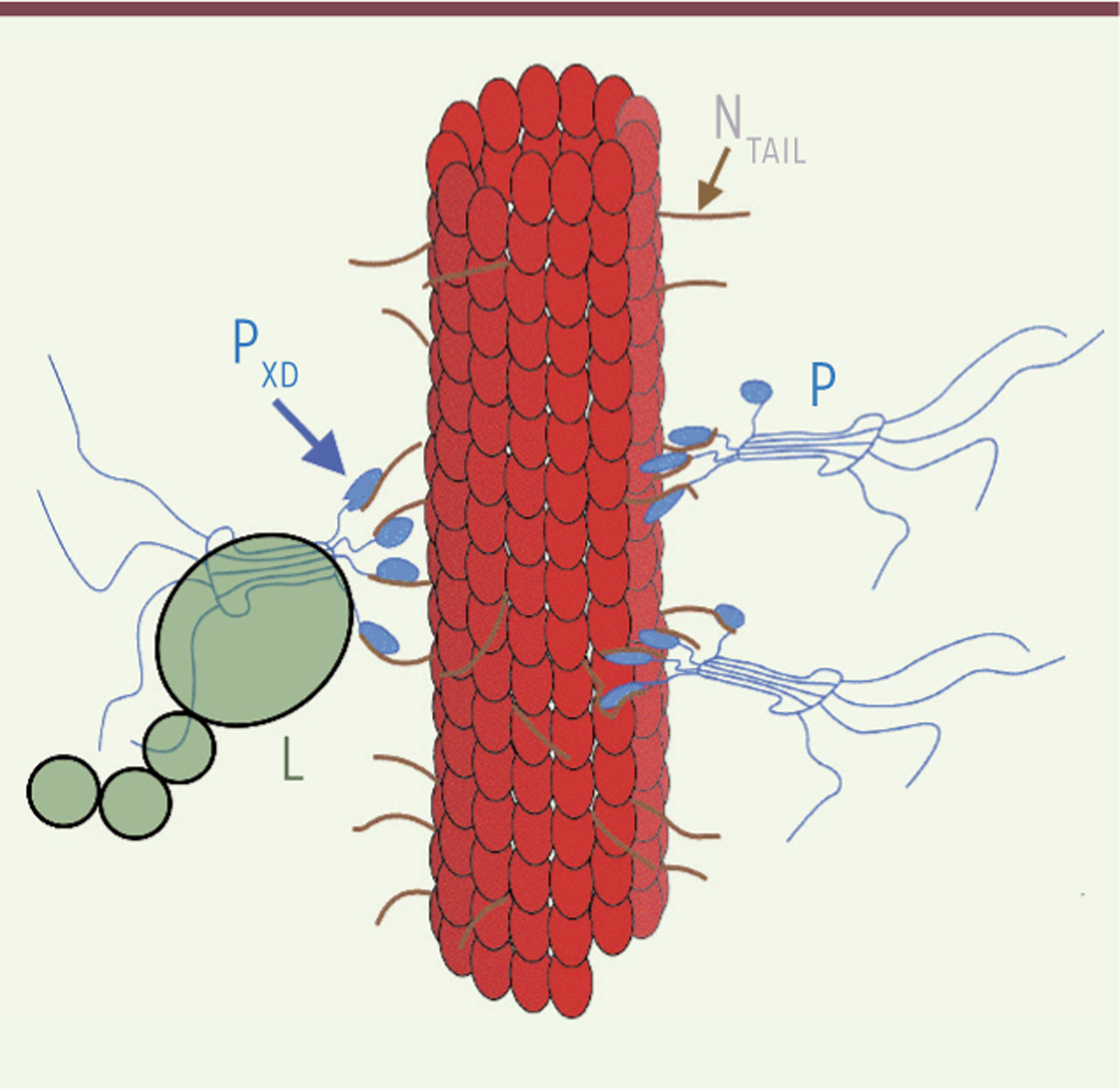
 | Figure 1.
Modèle de nucléocapside. La nucléocapside est constituée d’un homopolymère de nucléoprotéines N (en rouge) entourant l’ARN génomique (non représenté) et hérissé de sous-unités NTAIL (lignes ondulées en marron, il faut imaginer autant de NTAIL
que d’ovales rouges) sur lesquels viennent se fixer le complexe formé de la protéine L (polymérase en vert) et de la phosphoprotéine P (en bleu) via les extrémités PXD (ovales bleus). |
L’interaction entre NTAIL
et PXD est donc fondamentale : elle permet la transcription et la réplication du virus. Elle constitue, en conséquence, une cible attractive pour des stratégies antivirales : son inhibition permettrait de bloquer le recrutement de la polymérase et donc la transcription et la réplication du virus. Mais par quel mécanisme l’interaction NTAIL
-PXD affecte-t-elle la transcription et la réplication virales ? |
La transcription des six gènes viraux selon un gradient À la suite de l’infection d’une cellule par le virus de la rougeole, les nucléocapsides sont libérées dans le cytoplasme de la cellule hôte qui sera le siège de la transcription et de la réplication du génome viral. La transcription de chacun des six gènes viraux débute immédiatement et s’effectue séquentiellement, l’un après l’autre. La polymérase virale utilise, pour la synthèse des transcrits, les ribonucléosides triphosphates produits par la cellule hôte. Le point de départ de la transcription est la première base complémentaire située à l’extrémité 3’ du génome viral. Les six gènes, présents dans le génome du virus de la rougeole, sont séparés par des régions dites inter-géniques (IGR) (Figure 2). Quand la polymérase virale a terminé de transcrire le premier gène (ou gène amont), elle parcourt la région IGR à la recherche du signal d’initiation du gène aval suivant pour débuter sa transcription. L’efficacité de cette ré-initiation de la transcription au niveau du gène aval, n’est cependant pas systématique. L’abondance relative de chaque ARNm décroît donc au fur et à mesure que l’on s’éloigne de l’extrémité 3’ du génome, selon un gradient décroissant caractéristique [8]. Ce gradient induit un ajustement de la quantité d’ARNm transcrits à partir de chacun des gènes et donc, de la quantité de chaque protéine virale. Ainsi, la protéine N, codée par le premier gène, et la protéine L (ou polymérase virale), codée par le dernier gène, sont donc respectivement produites en grande et faible quantités.
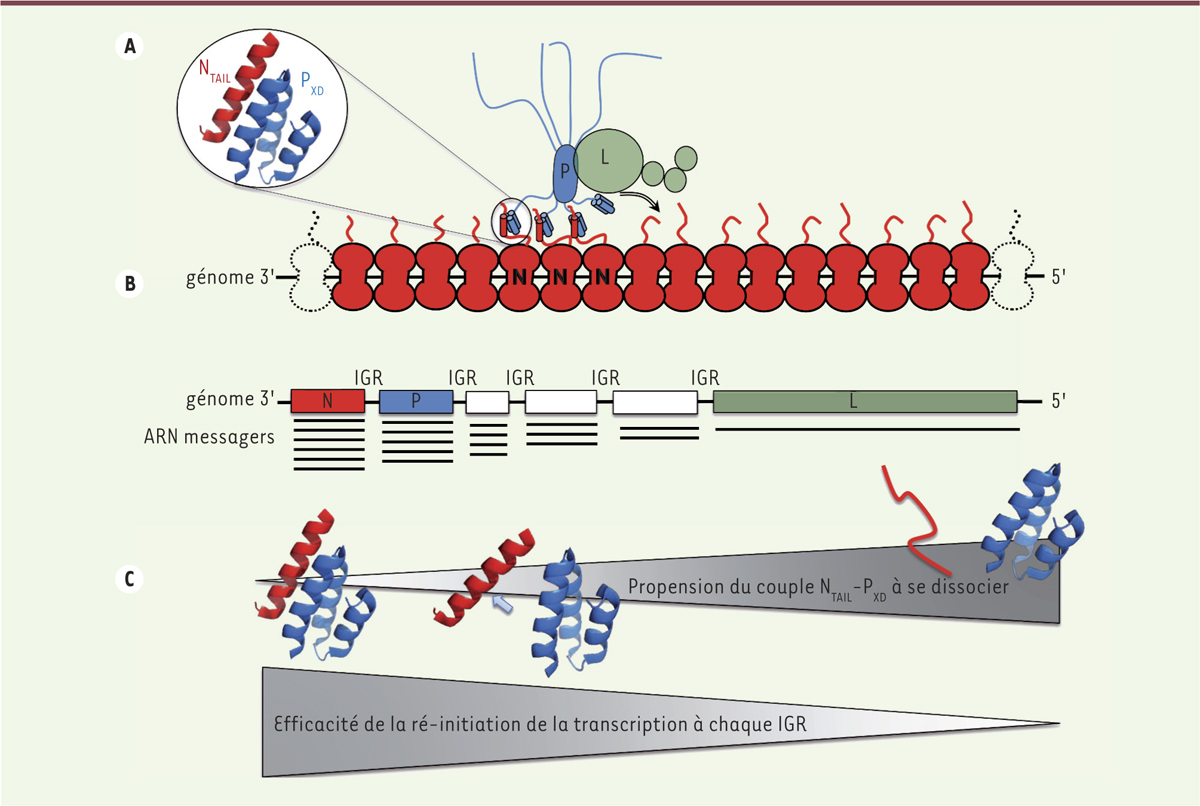
 | Figure 2.
Illustration schématique de l’activité de transcription de la polymérase virale. A. représentation schématique du déplacement de la polymérase virale (L, en vert) en complexe avec la phosphoprotéine (P, en bleu) le long de la nucléocapside, faite du génome viral encapsidé par la nucléoprotéine N (en rouge). La protéine P est représentée sous forme de tétramère, avec sa région centrale responsable de l’oligomérisation montrée en ovale allongé. Les lignes ondulées représentent la région N-terminale de P ainsi que la région flexible reliant la partie centrale de P à son domaine X (PXD). Les hélices au sein de PXD et de NTAIL
sont représentées sous forme de cylindre. La liaison du complexe L-P à la nucléocapside repose sur l’interaction entre le domaine X de P (PXD) et le domaine NTAIL
de N. B.organisation schématique du génome du virus de la rougeole avec ses six gènes, séparés par des régions inter-géniques (IGR). En dessous de chaque gène sont représentés, dans leur abondance relative, les ARN messagers respectivement transcrits. C. l’efficacité avec laquelle la polymérase virale ré-initie la transcription à chaque région intergénique (IGR) dépend de l’affinité du couple NTAIL
-PXD. Une propension plus prononcée du complexe NTAIL
-PXD à la dissociation (de la gauche vers la droite) est ainsi corrélée à une plus faible efficacité de ré-initiation de la transcription. |
|
Le rôle de l’interaction NTAIL-P XD dans le contrôle de l’efficacité de ré-initiation de la transcription Trois équipes françaises ont réalisé des expériences de biochimie, de biophysique, de dynamique moléculaire et de biologie cellulaire afin d’identifier les acides aminés de NTAIL
impliqués dans son interaction avec PXD [9]. La première avancée a été l’obtention d’une gamme de variants de NTAIL
ayant une affinité variable pour PXD. La seconde a été la possibilité de comprendre, par modélisation, comment la substitution de certains résidus pouvait altérer l’affinité entre NTAIL
et PXD, explication qui ne pouvait pas être déduite de la structure cristallographique de ce complexe publiée antérieurement [5]. La troisième avancée a été d’observer que l’affinité des variants de NTAIL
pour PXD, mesurée en solution saline avec des protéines recombinantes purifiées, est remarquablement similaire à celle déterminée dans des milieux biologiques très complexes comme le cytoplasme de la bactérie Escherichia coli et celui d’une cellule humaine en culture. La quatrième a été la conception d’un minigénome du virus de la rougeole constitué de deux gènes rapporteurs modifiés de manière à pouvoir mesurer l’expression du second gène (et ses variations) avec une sensibilité élevée. La conclusion remarquable à laquelle ces études ont permis de parvenir, grâce à ces quatre avancées, et, « cerise sur le gâteau », avec des virus recombinants, est la suivante : pour chaque IGR, l’efficacité avec laquelle la polymérase ré-initie la transcription du gène aval dépend de l’affinité entre NTAIL
et PXD [9]. Une faible affinité entre les deux réduit ainsi l’efficacité de la polymérase à ré-initier la transcription (Figure 2). L’interaction entre NTAIL
et PXD est donc naturellement optimale pour permettre à la polymérase virale de synthétiser chacun des ARNm viraux dans des proportions assurant une production équilibrée de chacune des protéines du virus. Cette interaction apparaît comme l’une des contraintes majeures auxquelles ce couple de protéines a été soumis au cours de l’évolution [9, 10]. Ces résultats apportent un éclairage sur une interaction protéine-protéine essentielle pour la transcription virale. Ils constituent ainsi un socle pour la conception rationnelle d’antiviraux. Un autre point remarquable qui est à noter, est que ce mécanisme est très probablement conservé chez les Paramyxoviridae, et, dans son principe, pourrait également l’être pour les autres Mononegavirales. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Blocquel D, Bourhis JM, Eléouët JF, et al. Transcription et réplication des Mononégavirales : une machine moléculaire originale . Virologie. 2012; ; 16 : :225.–257. 2.
Antona D, Levy-Bruhl D, Baudon C, et al. Measles elimination efforts and 2008–2011 outbreak, France . Emerg Infect Dis. 2013; ; 19 : :357.–364. 3.
Lupo J, Bernard S, Wintenberger C, et al. Fatal measles without rash in immunocompetent adult, France . Emerg Infect Dis. 2012; ; 18 : :521.–523. 4.
Fisher A. La vaccination en péril ? . Med Sci (Paris). 2017; ; 33 : :119.–120. 5.
Kingston RL, Hamel DJ, Gay LS, et al. Structural basis for the attachment of a paramyxoviral polymerase to its template . Proc Natl Acad Sci USA. 2004; ; 101 : :8301.–8306. 6.
Longhi S, Receveur-Brechot V, Karlin D, et al. The C-terminal domain of the measles virus nucleoprotein is intrinsically disordered and folds upon binding to the C-terminal moiety of the phosphoprotein . J Biol Chem. 2003; ; 278 : :18638.–18648. 7.
Ringkjøbing Jensen M, Communie G, Ribeiro ED Jr., et al. Intrinsic disorder in measles virus nucleocapsids . Proc Natl Acad Sci USA. 2011;; 108 : :9839.–9844. 8.
Ray J, Fujinami RS. Characterization of in vitro transcription and transcriptional products of measles virus . J Virol. 1987; ; 61 : :3381.–3387. 9.
Bloyet L, Brunel J, Dosnon M, et al. Modulation of re-initiation of measles virus transcription at intergenic regions by PXD to NTAIL binding strength . PLoS Pathog. 2016; ; 12 : :e1006058.. 10.
Brunel J, Chopy D, Dosnon M, et al. Sequence of events in measles virus replication: role of phosphoprotein-nucleocapsid interactions . J Virol. 2014; ; 88 : :10851.–10863. |